猪流行性腹泻病毒云南省流行毒株S1 基因序列分析
2022-09-08王鑫文吴静楠郭红瑞张松梅邓成松鲁金强邹丰才张以芳
王鑫文,吴静楠,郭红瑞,张松梅,邓成松,鲁金强,邹丰才,张以芳,柴 俊
(1.云南农业大学动物医学院,云南昆明 650201;2.德宏州尚善品味农业有限公司,云南梁河 679200)
猪流行性腹泻病毒(porcine epidemic diarrhea virus,PEDV)引起的猪流行性腹泻(PED)是养猪业中最常见、危害较严重的病毒性腹泻病,具有高传染性、高致病性和季节性暴发特征[1]。仔猪由于生理机能尚未发育完善,体内抗体水平低,通常最易感染PEDV[2]。疫苗免疫在PED 防控中起重要作用,但近年来免疫猪只也频繁发生PED。相关报道和多项研究已证实,PEDV 目前已发生基因突变出现新的变种,并且这种基因突变主要发生在S1基因,这或是造成免疫失败的原因[3]。
PEDV 为尼多病毒目冠状病毒科冠状病毒属的,单股正链有囊膜结构的RNA 病毒,基因组长约28 kb,包括2 个非编码区和7 个开放阅读框(ORF)。这7 个开放阅读框编码4 个结构蛋白(S、E、M、N)与3 个非结构蛋白(ORF1a、ORF1b 和ORF3)[4-5]。其中,S基因全长4 152 bp,其编码的S 蛋白是位于病毒粒子表面的纤突蛋白[4],在PEDV与宿主细胞表面受体结合和入侵宿主细胞过程中起着巨大作用,同时还与介导动物机体产生抗体有着重要联系,被认为是能够抵抗PEDV 的主要抗原。根据其他冠状病毒S 蛋白序列可以将PEDV 人为划分为S1(1~735 aa,抗原区)和S2(736~1 383 aa,膜融合区)两个结构域。研究[5-7]表明,S1 结构域易发生变异,能够更好地适合多种宿主细胞,更能体现病毒变异情况。因此研究S1基因有利于了解、分析毒株特点与变异趋向[5-6]。本研究对2017 年以来采自云南省疑似PED 发病猪只的病料样品进行PEDV 检测,然后对检出的阳性样品进行S1基因测序分析,以期为PED 防制提供依据。
1 材料与方法
1.1 样品来源
采自云南省12 个地区部分猪场腹泻仔猪的粪便样品120份、小肠组织样品62份,共计182份(表1)。
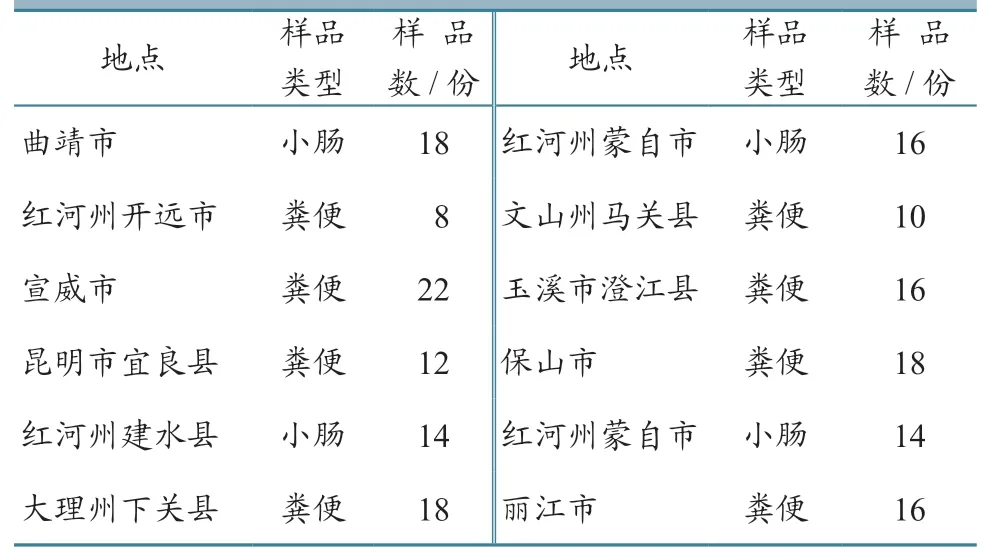
表1 腹泻仔猪样品信息
1.2 主要试剂和仪器
RNA 提取试剂盒、胶回收试剂盒、质粒提取试剂盒、DL 2 000 和DL 5 000 DNA Marker,购自宝生物工程有限公司;2×EasyTaqSuper Mix、感受态细胞DH5α、PMD19-T 载体、AMP、IPTG、X-gal,购自北京全式金生物技术有限公司;LB 肉汤、LB 琼脂,购自广东环凯微生物科技有限公司。试验所用仪器,均由云南农业大学动物医学院实验室提供。
1.3 引物设计与合成
参考GenBank 中PEDV 毒株N基因保守区和CV777 株全基因序列(登录号AF353511),分别设计PEDV 检测引物(F、R)和S1基因扩增测序引物(SF1/SR1、SF2/SR2),由昆明擎科生物科技有限公司合成。检测引物序列见表2。

表2 引物序列及扩增长度
1.4 总RNA 提取,目的基因扩增、克隆及基因测序
根据试剂盒说明书提取RNA,配制cDNA 反转录体系(20.0 μL):5×TransScript All-in-One SuperMix For PCR 4.0 μL、RNase-free water 8.0 μL、Total-RNA 8.0 μL。反转录程序:42 ℃30 min,85 ℃ 5 s。产物于-20 ℃保存。PCR 检测反应体系(25.0 μL):2×EasyTaqSuper Mix 12.5 μL,ddH2O 9.5 μL,F、R 引物各1.0 μL,模板1.0 μL。PCR 反应程序:94 ℃预变性3 min;94 ℃变性30 s,57 ℃退火30 s,72 ℃延伸1 min,扩增32 个循环;72 ℃延伸5 min。PCR 产物用1%琼脂糖凝胶电泳鉴定。S1基因前段(SF1/SR1)PCR 扩增反应程序:94 ℃预变性3 min;94 ℃变性30 s,54.6 ℃ 30 s,72 ℃ 100 s,34 个循环。后段(SF2/SR2)PCR 反应程序:94 ℃预变性3 min;94 ℃ 30 s,54 ℃ 30 s,72 ℃ 80 s,扩增34 个循环。DNA 产物用胶回收试剂纯化,连接至pMD19-T 载体,转化至感受态细胞DH5α,提取重组克隆质粒进行PCR 鉴定,并送昆明擎科生物科技有限公司测序。
1.5 S1 基因序列分析
利用DNAstar 软件对测序获得的两段S1基因进行拼接,获得2 376 bp 序列;利用MEGA7.0 及itol 在线软件,进行同源性、氨基酸变异位点分析,以及遗传进化树构建、修饰和标定。利用DNAstar中的Jameson-Wolf 法进行抗原性变化分析。参考序列与疫苗株信息见表3。
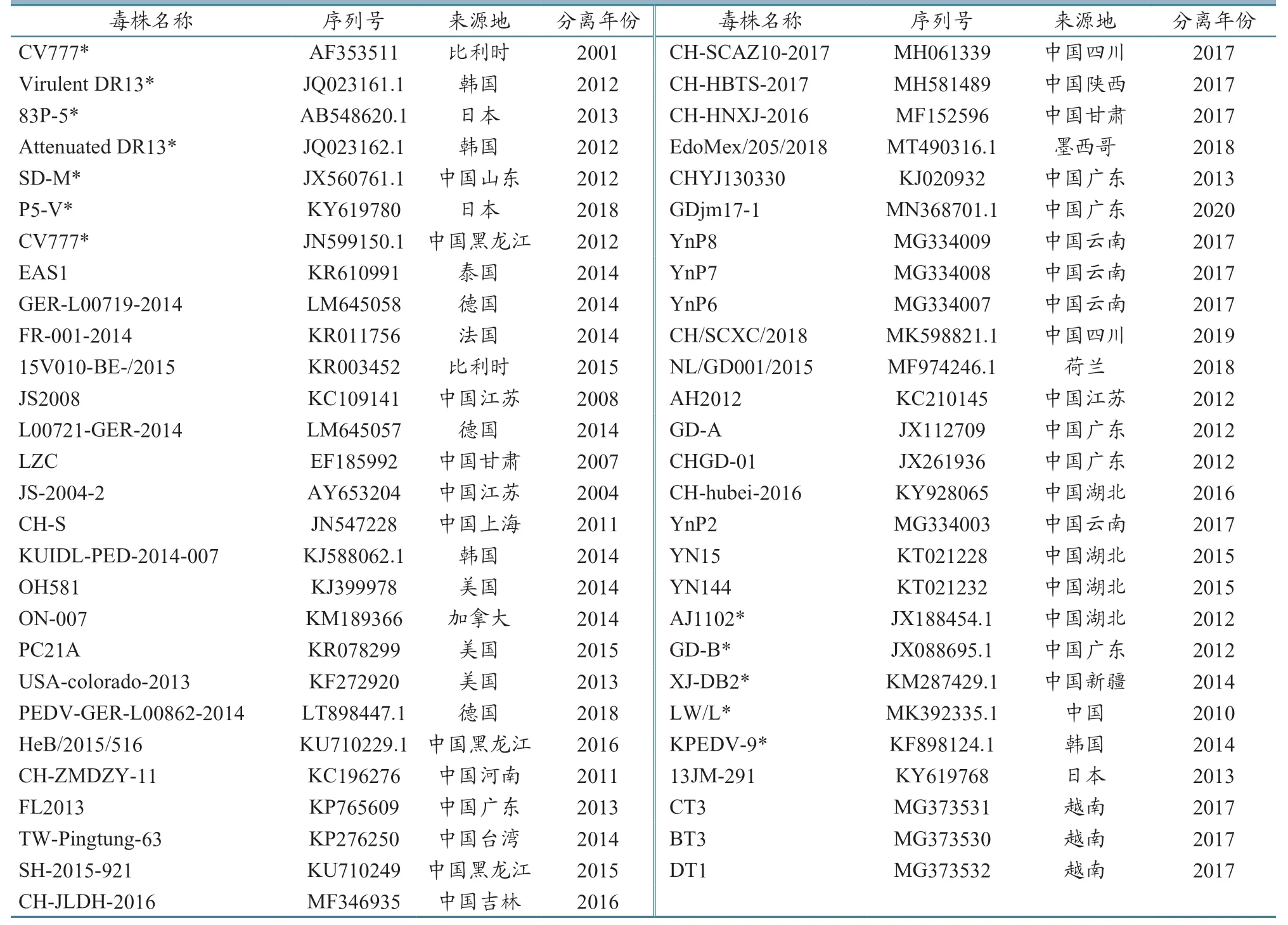
表3 PEDV S1 基因序列参考毒株信息
2 结果与分析
对2017—2022 年云南省12 个地区的182 份腹泻仔猪样品进行RT-PCR 检测,结果在9 个地区的110 份样品中检出PEDV 阳性,样品阳性率为61.20%。选取代表性样品进行基因测序及拼接,共获得9 个样品的S1序列,分别命名为YN-QJ-2017(曲靖)、YN-MZ-2018(蒙自)、YN-XW-2018(宣威)、YN-JS-2019(建水)、YN-DLXG-2020(大理)、YN-LJ-2020(丽江)、YN-BS-202(保山)、YN-CJ-2021(澄江)、YN-XW-2022(宣威)。
2.1 样品RT-PCR 检测
样品RT-PCR 扩增产物的1%琼脂糖凝胶电泳检测结果(图1)显示,在778 bp 左右位置出现清晰的特异性条带,与预期相符。
2.2 S1 基因扩增纯化、克隆及鉴定
对检测阳性的样品,用引物S1F/S1R、S2F/S2R,对S1全基因序列分两段进行RT-PCR 扩增,扩增产物用1%琼脂糖凝胶电泳分析,结果在约1 516、1 187 bp 位置可见清晰的特异性扩增条带(图2),与预期目的片段大小一致。对纯化的PCR 产物进行克隆、鉴定及测序,得到与预期结果一致的大小约1 516 bp 和1 187 bp(图3)的目的片段,从而确定重组质粒pMD-19-S1-1 和pMD-19-S1-2构建成功。
2.3 S1 基因核苷酸遗传进化分析
S1基因遗传进化分析结果(图4)显示,所构建的遗传进化树主要分为两个大分支,即G1 和G2基因群,其中G1基因群包括G1a亚群(经典株)、G1b 亚群(变异株),G2 基因群包括G2a 和G2b亚群。本研究获得的9 份样品S1基因可分为1 份G1a 亚群(YN-QJ-2017)、1 份G1b 亚群(YNXW-2022)和7 份G2b 亚群。其中,YN-QJ-2017与国内疫苗株SD-M、CV777-hlj 和P5-V 亲缘关系最近,7 份G2b 基因亚群样品与2011—2018 年的4 株北美洲毒株、1 株韩国毒株、18 株国内流行毒株(包括3 株云南株)和2 株国内疫苗株位于同一个分支,与国内市售G2 基因群疫苗株(AJ1102、L/W/L)亲缘关系较远,而该分支也包含了国内外最近几年的许多主要流行毒株。
2.4 S1 基因核苷酸及推导氨基酸序列同源性分析
对 9 份阳性样品与35 株代表性PEDV 参考毒株进行S1基因核苷酸和推导氨基酸序列同源性分析。结果显示:2 份G1 基因群样品与7 份G2 基因群样品的核苷酸和推导氨基酸同源性分别为93.1%~93.6% 和91.1%~91.9%。7 份G2b 基因亚群样品与CV777、CH-S 及CV777、P5-V、KPEDV-9、83P-5、SD-M 及DR13 等疫苗株的亲缘关系很远,它们间的核苷酸和推导氨基酸同源性分别为91.3%~93.1%和89.4%~92%;与AJ1102和L/W/L 疫苗株以及国内的高致病性变异强毒株CHYJ130330、变异减毒株FL2013、云南流行毒株YNP6 等参考毒株位于不同分支,它们间的核苷酸和推导氨基酸同源性分别为96.1%~98.7%和89.9%~97.9%;而与同一分支的美国变异强毒株USA-Colorador-2013 以及国内的TW-Pingtung-63、CH-hubei-2016、CH-JLDH-2016 等变异流行毒株亲缘关系最近,它们间的核苷酸和氨基酸同源性分别为97.5%~99.2%和96.5%~98.6%,说明存在共同祖先,具有密切关系;与3 株云南参考株的核苷酸和推导氨基酸同源性较高,分别为97.6%~98.8%和96.2%~98.2%;与同一分支的国内疫苗株GD-B和XJ-DB2 的核苷酸和推导氨基酸同源性较高,分别为97.0%~97.7%和95.6%~97.6%。
2.5 S1 基因与疫苗株氨基酸突变位点分析
结合系统发育分析结果可知;处于G1 基因群的阳性样品YN-XW-2022 与同群的疫苗株存在较多一致的突变位点,与G2 基因群疫苗株和阳性样品在第2、15、361、370、522、554、599、724、729 和771 位发生了一致的突变,分别为R→K、P→S、I→T、E→Q、A→S、T→S、G→S、N→S、N→S、Y→S,此外在第3、42、71、87、152、168、226、306、333 和568 位还发生了与G1 和G2 基因群疫苗株均不一致的突变位点,分别为I→N、Q→H、I→L、S→A、K→R、I→L、G→S、D→G、A→V、K→N;处于G2 基因群的7 份阳性样品与AJ1102、XJ-DB24等4 株疫苗株在第13、139、289 和363 位发生了不一致的突变位点,分别为V→L、N→D、I→M、A→T。这提示,现有的G2 基因群疫苗株对现有流行毒株不能提供完全保护甚至发生免疫失败现象。
2.6 S1 蛋白抗原位点变化分析
S1 蛋白主要抗原中和位点存在于COE 和SS2 区域。阳性样品与疫苗株(CV777、LW/L、AJ1102)的S1 抗原位点分析结果(图6)显示:处于G1 分支的2 份样品与同源经典疫苗株CV777相比抗原性差异较小,但YN-QJ-2017 株S1 蛋白主要抗原中和表位COE 区域在第605~613 位明显增加,第540~548 位明显减少。处于G2 分支的疫苗株与阳性样品YN-MZ-2018、YN-JS-2019、YNDLXG-2020 和YN-CJ-2021 在第240~254 位明显减少,与YN-XW-2018 在第320~326 位明显减少,与YN-JS-2019 在第389~404 位明显减少;在COE区域,YN-CJ-2021 的第502~510 位,YN-LJ-2020的 第529~537、605~613 位,YN-JS-2019 的第540~548 位明显减少。这可能导致云南省流行毒株与疫苗株在抗原性上存在差异。
3 讨论
2010 年以来,PED 的暴发流行给我国养猪业造成了巨大经济损失。Zuo 等[15]对2012—2017年从云南省采集的435 份猪腹泻样品检测发现,PEDV 样品阳性率逐年上升。本研究从2017—2022年阳性样品中扩增得到9 份样品PEDVS1基因,且样品来源猪群均已接种CV777、AJ1102 和L/W/L 株疫苗,说明这些疫苗株未能对PEDV 流行提供很好的预防。通过S1基因遗产进化和同源性分析得知,9 份PEDV 阳性样品位于G1 基因群和G2b基因亚群。这两种基因型毒株也导致了美国和韩国PED 疫情暴发。李晓成等[8]分析2017 年我国猪群腹泻疫病流行状况,发现PEDV 为主要病原,其中G2 基因群占90%,说明G2 基因群是我国的优势流行株;杨汉春等[9]对我国部分省份进行PEDVS1基因检测也得出相同的结论。可以看出,云南省和国内其他地区当前的PEDV 流行株基本一致。从疫苗株的S1 蛋白氨基酸突变位点分析来看,2份G1 基因群样品和7 份G2 基因群样品与各自同群的疫苗株均存在较多相同的突变位点,但也存在不一致的突变位点。值得注意的是,G1 基因群样品YN-XW-2022 与G2 基因群样品和疫苗株在第1~163 位存在10 处一致的突变位点,同时也存在9 处与G1 和G2 基因群毒株都不一致的突变位点。这提示PEDV 流行毒株在疫苗免疫和繁育过程中可能发生了G1 和G2 基因群的基因重组或变异现象。Cuo 等[10]对PEDV 流行毒株进行遗传进化和基因型分析,发现存在G1 和G2 基因群重组毒株。王若木等[11]研究G2 基因型PEDV 疫苗免疫后的毒株遗传进化特点发现,广西地区PEDV 毒株在免疫压力下S1 蛋白氨基酸序列发生了独特的突变。从这方面看,流行毒株S1 蛋白的这一系列变化可能会导致云南省PED 免疫失败。
PEDV S 蛋白与细胞表面受体结合、入侵宿主细胞以及介导动物机体产生抗体有着重要联系,是研发疫苗的首要目的蛋白;不同毒株S基因间存在不同程度的核苷酸插入、缺失和突变等现象,进而影响其抗原性[12-13]。Chen 等[14]通过PEDV 变异株基因序列分析,首次公布了PEDV 变异株的主要变异区为S1基因。朱海侠等[12]通过分析4 株分离株与疫苗株PEDVS1基因主要抗原表位区抗原位点变化,推测抗原表位变化可能是影响PEDV 感染的主要原因。因此,通过PEDVS1基因主要抗原表位区的位点变化,结合遗传进化和同源性分析,比较分离株与疫苗株S1基因的差异,可推测现有疫苗的保护情况。本研究发现,阳性样品与不同基因群的3 株疫苗株相比,在S1 蛋白主要抗原中和表位区COE 区域和以外区域均出现了不同程度变化,提示现有疫苗对云南省PEDV 流行毒株不具有较好的免疫效果,迫切需要研发能保护猪只免受新型PEDV 攻击,并且安全、有效以及低价的疫苗[15-16]。
4 结论
综上所述,云南省部分地区同时流行G1 基因群和G2b 基因亚群两种PEDV 基因型毒株,但G2b 为优势流行基因亚型,其与同群的北美洲毒株有密切亲缘关系;云南流行株和疫苗株间存在较远的遗传亲缘关系,已发生了部分氨基酸突变,尤其在抗原表位区,推测现有疫苗株对云南省部分PEDV 流行毒株不具有较好的保护效果,这很可能是导致现在PED 控制不理想的原因。
