花生疮痂病菌全基因组PKS基因簇挖掘及毒素生物合成基因ESCB1生物信息学分析
2022-09-03张晓天付祎玮焦文莉朴静子李洋李自博周如军
张晓天,付祎玮,焦文莉,朴静子,李洋,李自博,周如军
(沈阳农业大学植物保护学院,辽宁 沈阳,110866)
花生疮痂病是花生产业中重要病害之一,在我国各主要花生产区均有发生危害,植物发病叶片、叶柄和茎秆等产生大量褐色、圆形或卵圆形病斑,病斑中部稍凹陷,呈“火山口”状,可导致发病部位扭曲畸形,植株矮化,一般田间发病率10%~30%,损失严重[1]。花生疮痂病菌(Elsinoë arachidis)可产生一种具有光敏活性的苝醌类真菌毒素—痂囊腔菌素(Elsinchrome,ESC),在光照条件下产生大量活性氧,导致细胞膜脂过氧化、电解质泄漏、膜通透性降低,最终细胞死亡,是花生疮痂病菌关键的致病因子,在病菌侵染和病斑扩展过程中发挥着重要作用[2~4]
植物病原真菌毒素致病作用、生物合成途径及调控网络研究对于揭示病菌的侵染机制、致病机理和病原寄主互作具有重要意义。前期研究表明,毒素的生物合成一般由编码聚酮合酶(polyketide synthase,PKS)的基因所在的基因簇进行调控[5]。如涉及尾孢菌素(Cercosporin)、黑粉菌素(Ustilaginoidins)和伏马菌素(Fumonisin)生物合成的CTB、Ugs 和FUM基因簇等,当PKS基因被靶向敲除或沉默时,毒素生物合成途径受到阻滞,毒素生物合成受到抑制[6~8]。目前ESC的生物合成途径及调控机制研究尚处于初始阶段,仅在柑橘疮痂病菌(Elsinoë fawcettii)中初步阐明了聚酮合酶EfPKS1基因是毒素生物合成的关键基因[9,10],锌指转录因子EfSTE12基因缺失,毒素积累减少,果胶酶和蛋白水解酶活性升高[11],而基因簇中其他基因功能尚不清晰,生物合成途径和调控网络尚未解析,需进一步研究揭示。
近年来,全基因组测序技术和生物信息学的快速发展,可以便捷地从基因组中挖掘特定功能基因或基因簇,研究其生物功能,在基因水平为植物病原真菌遗传、次生代谢、生物合成途径及病原寄主互作科学问题提供可靠的分子证据[12,13]。前期研究发现花生疮痂病菌ESC 的积累量与致病性呈显著性正相关,是病菌重要的毒力因子[14],项目组对病菌进行了全基因组测序,大小为33.18 Mb[15]。本文在此基础上,进一步开展了E.arachidis次生代谢相关PKS 基因簇挖掘、毒素生物合成相关基因ESCB1克隆和生物信息学分析,旨在为解析ESC 生物合成途径、构建调控网络和阐明病菌致病机制提供前期理论基础。
1 材料与方法
1.1 材料
供试菌株:花生疮痂病菌(E.arachidisLN-JHC01),分离自辽宁锦州花生种植基地。
1.2 PKS基因簇的挖掘
利用antiSMASH 在线网站(https://bitbucket.org/antismash/antiSMASH5.0/)预测和分析次级代谢产物基因簇,通过NRPS-PKS knowledgebase(http://www.nii.ac.in/~pksdb/sbspks/master.html)对预测结果进行验证后,筛选出PKS基因簇。
1.3 PKS基因簇的功能预测
根据antiSMASH 搜索已知功能的同源基因簇,并预测基因簇中合成基因对应产物的核心结构,初步判断基因簇代谢产物,利用NCBI BLAST(https://blast. ncbi. nlm. nih. gov/Blast. cgi)对基因簇所有同源序列进行同源比对,初步确定基因功能。
1.4 ESCB1基因的克隆
根据ESCB1基因组序列,利用Primer5.0 引物设计软件设计全长引物ESCB1-F(TCGATTGCTTTCCCTGGAA)/ ESCB1R (CCTTGATGCTCCTCTTGATA),以DNA 为模板PCR 扩增,扩增条件为:95℃预变性10 min;95℃变性30 s,63℃退火30 s,72℃延伸2 min,35个循环;72℃终延伸10 min。1%琼脂糖凝胶电泳,PCR产物由上海生工生物工程公司测序。
1.5 ESCB1基因生物信息学分析
利用ORF Finder(http://www. bioinformatics. org/sms2/orf_find.html)、ProtParam(https://web.expasy.org/protparam)、NetPhos 3.1 Server(http://www.cbs.dtu. dk/services/NetPhos/)、InterPro(http://www. ebi.ac. uk/interpro/scan. html)、蛋白二级结构预测网站(https://npsa-prabi. ibcp. fr/cgi-bin)、蛋白三级结构预测网站(https://www. swissmodel. expasy. org)等在线软件获得开放阅读框、外显子、内含子信息,预测编码蛋白的理化性质、保守结构域等。通过MEGA 7.0 软件构建系统发育树。为了可视化蛋白质结构域架构,使用软件DOG 2.0 绘制ESCB1基因结构域。
1.6 ESC毒素的提取及含量测定
ESC毒素的提取及含量测量参考刘璐方法[16]。
1.7 ESCB1的表达分析
菌悬液涂布于PDA 培养基中黑暗培养14 d 后光照培养2 d,刮取菌丝,利用Eastep Super 总RNA提取试剂盒(普洛麦格生物技术有限公司)提取总RNA,HiFiScript cDNA Synthesis Kit 反转录试剂盒(康为世纪公司)进行反转录得到cDNA,以Actin基因为内参,以黑暗培养16 d 为对照,qPCR 测定ESCB1表达量。
2 结果与分析
2.1 全基因组中PKS基因簇预测分析与筛选
通过antiSMASH5.0 预测和NRPS-PKS knowledgebase 验证,E.arachidis基因组中共发现次级代谢基因簇22 条,其中t1PKS 基因簇5 条,PKS-NRPS基因簇1条,结果如表1所示。
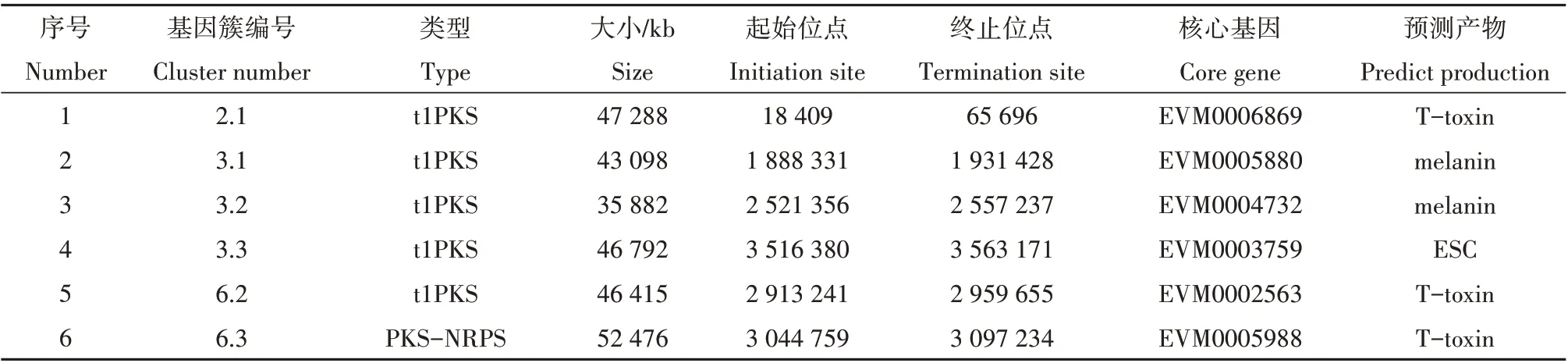
表1 PKS基因簇信息表Table 1 PKS sequence information
基因簇中所含有的编码基因数量及分布各不相同。绘制PKS基因结构域发现PKS的保守结构域包括酰基转移酶(acyltransferase,AT)、酮基合酶(ketosynthase,KS)、脱水酶(dehydratase,DH)、甲基转移酶(methyltransferase,MeT)、酰基载体蛋白(acylcarier protein,ACP)和具有还原活性的烯脂酰还原酶(enoylreductase,ER)、酮体还原酶(ketoreductase,KR)等结构域(图1)。根据其结构域类别不同划分为 非 还 原 型(EVM0004732、EVM0003759、EVM0005880) 和 还 原 型 (EVM0005988、EVM0002563、EVM0006869)两类。根据基因簇产物类型,大小和结构组成,编号为3.3 的基因簇为ESC 生物合成关键基因簇,命名为ESCB 基因簇,核心PKS基因EVM0003759命名为ESCB1。

图1 聚酮合酶结构域分析Fig.1 Analysis of Elsinoë arachidis polyketide synthases(PKS)proteins domains
2.2 ESCB基因簇功能分析
该基因簇位于基因组的3 516 380~3 563 171 bp位置,长度约为47 kb,含有12 个编码基因,分别命名为ESCB1~ESCB12(图2)。以PKS 编码基因为核心[17~19],包括超家族转运蛋白(major facilitator superfamily transporter,MFS)编码基因[6],锌指转录因子(zinc finger transcription factor)编码基因[11],细胞色素P450(cytochrome P450)编码基因[20],O-甲基转移酶(o-methyltransferase)编码基因[21]和单加氧酶编码(monooxygenase)基因[22]等生物合成相关基因。

图2 ESCB基因簇分布图Fig.2 Distribution of the ESCB gene cluster
2.3 ESCB1基因克隆
以DNA 为模板,使用全长引物ESCB1F/ ESCB1R 和长链高保真酶进行PCR 扩增,琼脂糖凝胶电泳(图3),获得的清晰明亮PCR 条带,测序分析后确定ESCB1基因全长为6636 bp。
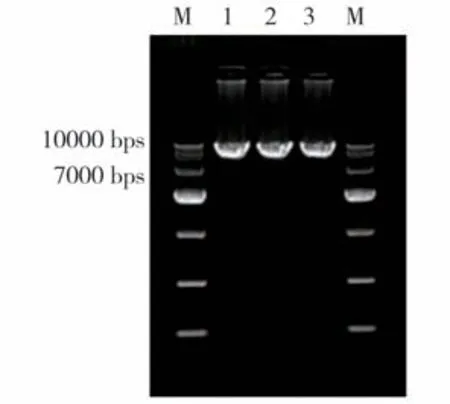
图3 ESCB1基因PCR电泳图Fig.3 PCR electrophoresis result of ESCB1 gene
2.4 ESCB1生物信息学分析
2.4.1 系统进化分析 将ESCB1基因编码蛋白与柑橘疮痂病菌(Elsinoë australis)、柳树疮痂病菌(Sphaceloma murrayae)、竹黄菌(Shiraiasp.)等真菌编码的聚酮合酶编码蛋白进行相似性分析。结果显示,ESCB1 蛋白与E.australis的Eapks 蛋白及S.murrayae的CQ-2017a 蛋白亲缘关系最近(图4),相似性为91%(图5)。Eapks 和CQ-2017a 均参与ESC生物合成,因此,初步认为ESCB1基因参与花生疮痂病菌ESC的生物合成。

图4 ESCB1系统进化树分析Fig.4 Phylogenetic tree of ESCB1

图5 ESCB1基因编码蛋白与其他蛋白序列对比分析Fig.5 Amino acid sequences of ESCB1 and other protein
2.4.2 开放阅读框、外显子和内含子分析 利用ORF Finder 对ESCB1基因进行分析,ESCB1基因具有一个完整的开放阅读框(图6)。该基因编码长度为2212 个氨基酸的蛋白序列。利用在线分析工具Softberry 对ESCB1基因全长的外显子和内含子进行预测,并与cDNA 进行序列对比。结果显示,ESCB1基因包含4个外显子区域及3个内含子区域。

图6 ESCB1基因ORF分析Fig.6 ORF analysis of ESCB1
2.4.3 编码蛋白结构域分析 利用IntroPro 在线分析网站对蛋白的结构域进行分析,ESCB1基因编码蛋白包括长度为240 个氨基酸的启动单元(SAT),434 个氨基酸的KS,301 个氨基酸的AT,316个氨基酸的DH,长度分别为77 个氨基酸和75 个氨基酸的ACP以及253个氨基酸的TE(图1)。
2.4.4 蛋白理化性质分析及亲疏水性 利用Prot-Param 对ESCB1基因编码蛋白的理化性质进行预测,编码蛋白包含2212 个氨基酸,分子量为238.84 kDa,理论等电点(pI)5.65。推测该蛋白分子式为C10594H16718N2890O3240S76,原子总数为33 518。该蛋白脂溶性系数为86.92,不稳定系数(Ⅱ)为41.44,亲水性系数(GRAVY)为-0.144。亲疏水性结果分析表明(图7),ESCB1编码蛋白在643 位的I 有最大值为2.333,在2015 位的E 有最小值为-2.789,亲水性最强,多肽链表现出亲水性。
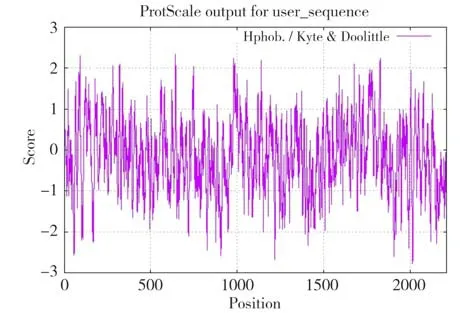
图7 ESCB1基因编码蛋白亲疏水性Fig.7 Hydrophilicity of ESCB1 protein
2.4.5 编码蛋白磷酸化位点分析及亚细胞定位利用NetPhos 3.1 Server 在线工具软件对ESCB1基因编码蛋白磷酸化位点进行分析,结果显示,该蛋白含有多个可被磷酸化氨基酸残基,说明该蛋白可被激酶磷酸化后发挥作用(图8)。利用亚细胞定位在线分析工具分析,ESCB1 蛋白定位在细胞叶绿体中。

图8 ESCB1蛋白磷酸化位点预测Fig.8 prediction of ESCB1 protein phosphorylation sites
2.4.6 编码蛋白二、三级结构 根据分析结果可知ESCB1基因编码蛋白含有的二级结构中,α-螺旋和无规卷曲是蛋白酶中的主要结构元件占总蛋白的73.72%,而β-转角和延伸链则散布于整个蛋白中(图9A)。利用在线Swiss-model 分析工具发现ESCB1基因编码蛋白的三级结构与Cercosporin合成相关基因CTB1基因编码的蛋白三级结构覆盖率为56.68%。以该蛋白晶体结构作为模板,构建了三维结构模型如图9B所示。

图9 ESCB1基因编码蛋白二级结构(A)和三级结构预测(B)Fig.9 Secondary structure(A)and tertiary structure(B)prediction of ESCB1 coding protein
2.4.7 蛋白信号肽及跨膜结构预测 利用在线工具分析ESCB1 蛋白是否存在信号肽(SPI)及信号肽的剪切位点(CS),结果显示编码蛋白不存在Sec/SPI结构与CS,说明蛋白可能是非分泌蛋白,主要分布于细胞内。使用在线工具预测ESCB1基因跨膜结构,分析结果可知,ESCB1基因编码蛋白不存在跨膜结构域,该蛋白质为非膜蛋白。
2.4.8ESCB1表达分析 前期光生物学研究证实E.arachidis中ESC的生物合成在光照和黑暗条件下具有显著差异[23],光照条件下菌落为红色,周围产生红色晕圈,产毒量为54 nmol/plug,黑暗条件下菌落为淡黄色,未检测到毒素的合成。为进一步明确ESCB1是否参与ESC 的生物合成,分别检测该基因在光照和黑暗条件下的mRNA 表达水平,结果表明,光照条件下ESCB1的表达量为黑暗对照的3.24倍,且表达模式与毒素在光暗条件下的变化趋势相同(图10)。
3 结论与讨论
基因组挖掘是以基因簇序列为指导预测次生代谢产物的一种新型生物信息学分析方法,同时还能直接将次生代谢产物结构与合成途径关联,方便进行生物合成和组合生物合成研究等[24,25]。AntiSMASH 是微生物次生代谢产物生物合成基因簇预测及组成结构分析网站,可以在整个基因组范围内进行PKS 及NRPS 基因簇筛选,对基因簇组成结构和产物进行初步分析和预测,为解析生物合成途径提供理论基础。真菌次生代谢产物生物合成基因一般在基因组中紧密成簇,称为次生代谢产物生物合成基因簇[5]。如C.nicotianae和Ustilaginoidea virens等真菌中Cercosporin 和Ustilaginoidins 生物合成相关基因均成簇存在,组成CTB 基因簇和Ugs基因簇,大量研究解析了基因簇的组成及相关基因功能,极大促进了病菌致病机制领域的研究进展[7,20]。本研究在全基因组测序的基础上,发现E.arachidis基因组中含有6 条与次生代谢密切相关的PKS 和PKSNRPS 基因簇,通过生物信息学分析,挖掘出ESC 生物合成ESCB 基因簇。该基因簇上包含PKS 编码基因,MFS 编码基因,锌指转录因子编码基因等12 个预测功能基因,但基因簇中各基因的生物学功能尚需要进一步验证。
目前研究表明,ESC 毒素在E.fawcettii中经由PKS 途径生物合成,但基因簇中各编码基因的功能尚未完全解析。本文通过基因克隆、同源聚类和生物信息学分析,E.arachidis中PKS 编码基因ESCB1与Cercosporin 和Hypocrellin 生物合成PKS编码基因CTB1和SbaPKS高度同源[18,19],初步认为ESCB1基因是花生疮痂病菌中ESC 生物合成的关键编码基因,并得到了ESCB1基因的序列全长、结构域和磷酸位点等大量分子信息,为该基因功能的深入研究提供了分子数据基础。为了进一步验证,本文进一步开展了毒素含量与表达量相关性研究,结果表明,ESCB1表达量与ESC 含量趋势基本相同,且受光照正向调控,调控模式与C.nicotianae相似[19],但与Aspergillus nidulans光调控模式不同[26],说明PKS 编码基因在不同真菌中虽然生物功能类似,但调控表达模式差异较大,因此,为了明确花生疮痂病菌毒素生物合成途径及致病机制,应进一步开展ESCB1基因功能、基因簇解析及调控网络研究工作。
