小麦组蛋白TaHis3.2 的克隆及盐胁迫下的表达分析
2022-08-12吴丹萌胡子全王志强刘一晓潘萍萍王从磊袁卉馥王建贺
吴丹萌 ,梁 丹 ,刘 丹 ,张 欣 ,胡子全 ,王志强 ,梁 晨 ,刘一晓 ,潘萍萍 ,王从磊 ,袁卉馥 ,王建贺
(1.河北北方学院,河北 张家口 075000;2.天津市农业科学院 农作物研究所/天津市农作物遗传育种重点实验室,天津 300384;3.界首市农业技术推广中心,安徽 界首 236500;4.天津市宝坻区农业发展服务中心,天津 301800;5.天津市农业发展服务中心,天津 300061)
小麦是我国乃至世界的重要粮食作物[1-2]。随着对小麦相关育种技术的研究不断深入和小麦新品种选育及其配套栽培技术不断应用,我国乃至世界小麦单产已经发展到较高的水平。但是,由于土壤条件及区域生态环境的影响,小麦生产水平存在地域之间及田块之间的差异,存在超高产田的同时,也存在中低产田,特别是土壤盐渍化较重的地区,小麦单产较低。近年随着地下水不断开采,土壤次生盐渍化程度也有不断上升趋势,据统计,目前有20%的耕地土壤盐渍化或正在逐步盐渍化,严重限制了小麦产量的进一步提高,形成了大量的小麦中低产田[3]。
作物育种的实践表明,育种上的突破性进展在于关键性基因、关键材料的挖掘、研究与利用。如小麦山融3 号通过体细胞融合技术,融入野生近缘种中的染色体片段,提高了其耐盐性,在0.3%~0.5%的盐渍化土壤种植,单产达到7 350 kg/hm2。目前,公认的小麦耐盐品种较少,耐盐机制研究进展缓慢。同时,由于缺乏与盐胁迫密切相关基因作用机制研究及分子标记,严重制约了耐盐小麦新品种的选育进程,从而制约着小麦耐盐性的进一步提高。因此,挖掘小麦耐盐基因,研究其在盐胁迫下的信号通路,探明其耐盐的分子机制,并挖掘优异单倍型,开发分子标记,辅助耐盐育种,对提升小麦耐盐育种水平、进一步培育耐盐性突出的小麦新品种具有重要意义。
研究表明,耐盐性是一个由多基因控制的农艺性状[4-7]。目前在拟南芥中明确SOS 信号转导途径与耐盐性密切相关[8-10]。研究发现,SOS 信号转导途径主要有2 个方面作用,一是维持细胞内的离子稳态;二是清除盐胁迫造成的自由基。目前在拟南芥SOS 信号转导途径上已经发现6 个重要成员,分别是SOS1、SOS2(AtCIPK24)、SOS3(AtCBL4)、SOS4、SOS5 和SOS6,这6 个成员形成了SOS 途径的主体网络[8-10]。同时,小麦中的蛋白磷酸酶信号转导通路、油菜素内酯信号通路、MAPK 信号通路上的部分基因均能受到盐胁迫的响应,能够提升转基因材料的耐盐性[3,11-14]。如异源三聚体蛋白磷酸酶的PP2A、PP2C 亚基均受到了盐胁迫的影响。但是在漫长的进化过程中,植物形成了多种应答盐胁迫的信号系统,仍然需要通过基因组、转录组、生物统计学进行耐盐基因的挖掘,为耐盐分子育种提供丰富的优异单倍型及分子标记。
表观遗传修饰参与了植物对非生物胁迫的抵御过程,如DNA 的甲基化修饰、染色质重塑以及长链非编码RNA 和小RNA 对下游靶基因的调控,从而影响植物对非生物胁迫的响应。目前,典型的表观修饰主要是DNA 的甲基化和组蛋白的乙酰化。细胞核中的染色体为高度浓缩状态,每146 bp 的双链DNA 围绕组蛋白形成核小体,再浓缩为染色质[15]。其中,核小体为组蛋白八聚体,这些组蛋白由H2A、H2B、H3、H4 构成[16]。组蛋白的编码基因在真核生物中高度保守,控制着染色质的结构,也影响基因的表达、修复等生物学功能。组蛋白通过表观修饰,如组蛋白的甲基化、乙酰化参与基因的表达。目前关于组蛋白家族成员的数目、在染色体上的位置及在非生物胁迫中的作用报道相对较少。前人研究表明,家禽的组蛋白编码基因约10 个拷贝,哺乳类动物约20 个拷贝,而有的生物则有约上百个拷贝,如海胆中包含600 个组蛋白基因。说明不同物种基因的拷贝数差异较大。而且组蛋白基因成簇存在,如人类的组蛋白编码位点主要位于6 号染色体,少部分位于1 号染色体;小鼠的组蛋白编码位点主要位于13 号染色体,少部分位于3 号染色体上。目前关于植物组蛋白编码成员基因的数量及染色体分布研究的较少。
本研究通过盐胁迫下转录组测序技术,挖掘差异表达基因,重点关注组蛋白成员表达模式变化,最终挖掘到了一个编码组蛋白的编码基因His3.2,并在小麦中分离该基因。通过生物信息学分析,明确其保守结构域,并分析小麦组蛋白全家族编码基因在染色体组上的分布,明确其序列特征,通过reads 数目明确该基因在盐胁迫的表达模式,初探其功能,并在小麦二倍体祖先种(AA/DD)、四倍体材料(BBAA)、六倍体栽培种(BBAADD)中分离序列,明确克隆序列的来源,通过重测序数据序列分析,初探该基因在不同材料中的变异情况,为探讨利用该基因分离单倍型,开发培育耐盐小麦新品系的分子标记奠定基础。
1 材料和方法
1.1 试验材料
所用小麦材料为:祖先种AA01(AA,二倍体,A基因组祖先种)、DD01(DD,二倍体,D 基因组祖先种)、DD02(DD,二倍体,D 基因组祖先种);四倍体小麦津硬8号(BBAA,四倍体)、kros(BBAA,四倍体);栽培小麦沧麦14(BBAADD,六倍体冬小麦)、沧6002(BBAADD,六倍体冬小麦)、以色列-1(BBAADD,六倍体冬小麦)、津农6 号(BBAADD,六倍体冬小麦)、辽春10 号(BBAADD,六倍体春小麦)、津强7 号(BBAADD,六倍体春小麦),均由天津市农业科学院农作物研究所保藏。试验材料具有不同的冬、春性,地理来源不同,亲缘关系较远且在耐盐、抗旱、抗冻水培上存在较大差异,用于序列分析。
1.2 DNA、RNA 提取及cDNA 第一链的合成
在12 h 光 照、12 h 黑暗、22oC 下培养小麦 幼苗至一心一叶期,一部分单株用于DNA 提取;另一部分进行50 mmol/L NaCl 处理1 h,迅速取叶片及根系于2.0 mL 的离心管中,贮存于-80oC 超低温冰箱中,用于提取RNA。
采用CTAB 法和多糖多酚植物总RNA 提取试剂盒(DP441)分别进行DNA 和RNA 提取,采用thermo 的RevertAid Strand cDNA Sythesis Kit 进 行cDNA 合成。DNA 提取后储存-20oC 冰箱中,RNA 及cDNA 提取后储存-80oC 冰箱中,待使用。
1.3 聚合酶链式反应
以50 mmol/L NaCl 转录组测序挖掘到的与盐胁迫潜在相关的转录本为母序列(query sequence),利用Blastp 对A、D 基因组进行序列比对,找到小麦中His3.2完整序列信息;通过其上下游序列采用Primer premier 5.0 本地软件设计克隆引物[17],引物如表1 所示。

表1 基因克隆引物Tab.1 Primer for gene cloning
编码区及基因分离分别以cDNA 和DNA 为模板进行,采用15 µL 体系进行PCR 扩增。所用高保真酶为TransStart®FastPfu DNA Polymerase(全式金,北京)。
1.4 基因克隆测序
将上述PCR 产物,采用1.2%的琼脂糖进行凝胶电泳,并对目的条带进行切胶回收,连接于Blunt载体中(全式金,北京),通过热击法转入大肠杆菌Top10 中,挑取12 个单克隆进行测序,获取基因编码区序列及基因组序列。
1.5 生物信息学分析
采用DNAman 软件进行核苷酸序列比对;通过HMMER 网站(https://www.ebi.ac.uk/Tools/hmmer/)预测氨基酸结构域,采用HMM 算法通过HMMER 软件进行全家族成员调取,并使用mapchart 进行染色体定位可视化[18],使用TBtools进行序列统计分析及可视化[19]。通过小麦基因组变异联合数据库(http://wheat-cau-edu-cn.vpn.cau.edu.cn:8118/WheatUnion/)进行重测序数据的基因序列分析。
2 结果与分析
2.1 TaHis3.2 基 因cDNA 克 隆
通过保守引物(Ta-4-F/Ta-3-R),在冬小麦津农6 号(BBAADD)的cDNA 中进行扩增,并且连接到克隆载体上,随机挑取12 个克隆进行测序,结果表明,共分离到2 种核苷酸序列,其长度均为411 bp,2 条序列存在9 个核苷酸的碱基差异(图1);2 种核苷酸序列均编码136 个氨基酸残基,且氨基酸序列一致率为100%,即2 种序列编码同样的氨基酸序列。
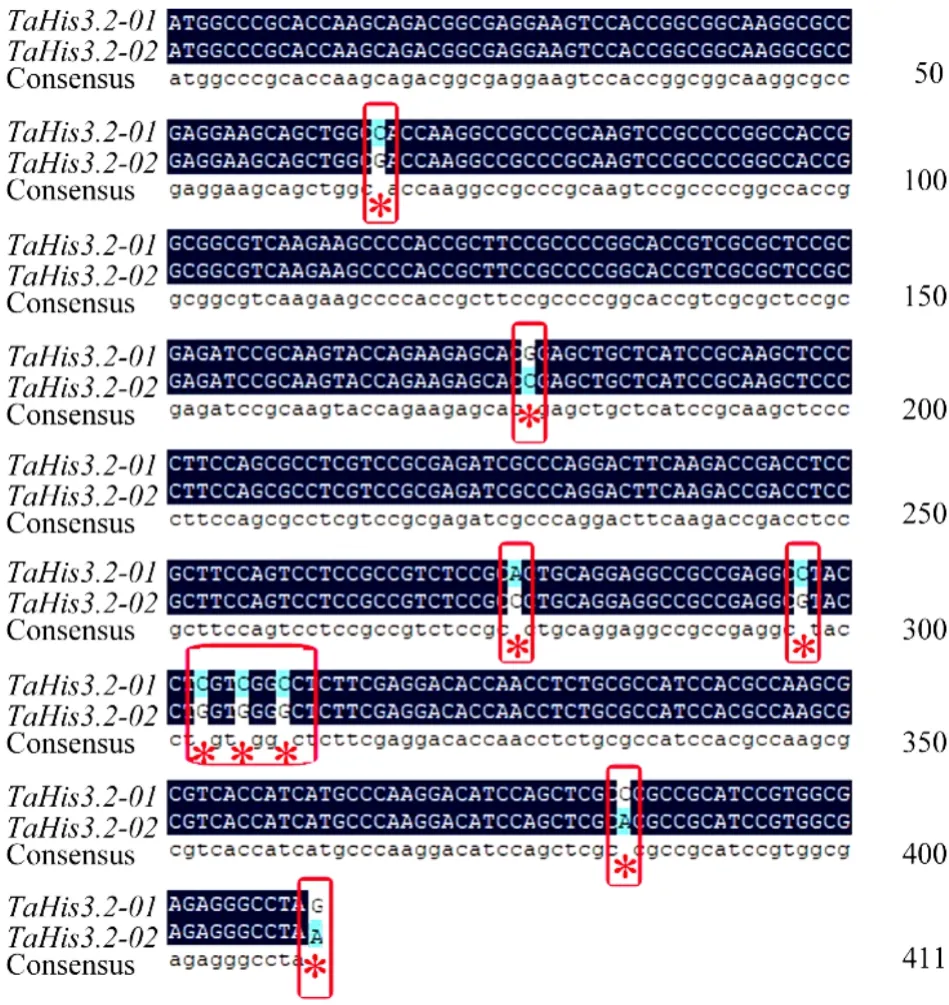
图1 小麦TaHis3.2 核苷酸序列比对Fig.1 Nucleotide sequence alignment of wheat TaHis3.2
经过氨基酸保守结构域分析发现,该基因编码的氨基酸仅包含一个结构域,为Histon3 结构域(Pfam:PF00125)(图2)。

图2 小麦TaHis3.2 结构域Fig.2 Domain of TaHis3.2
2.2 小麦组蛋白全基因组染色体分布
为了进一步研究该家族成员在小麦中的分布情况,明确家族成员数量及染色体分布,依据Histon 保守结构域PF00125 进行小麦组蛋白全家族成员调取,发现该家族存在398 个成员,在小麦的所有染色体上均有分布,且主要分布在染色体的两端(图3);研究表明,该家族成员在六倍体小麦的第1、6、7 同源群上分布较多,分别有70、90、77 个成员,在第2 同源群上分布最少,仅有17 个成员(图4);编码序列分析发现,该家族成员由染色体上的57 390 bp 核苷酸编码,最长的编码序列为482 bp,最短的编码序列仅66 bp(表2)。说明组蛋白家族成员众多,可能存在功能分化及功能冗余。

表2 小麦组蛋白DNA 编码序列特征Tab.2 Characteristics of the coding sequence of the wheat histone protein

图3 小麦组蛋白家族成员在染色体上的分布Fig.3 The wheat chromosomal distribution of histone family members
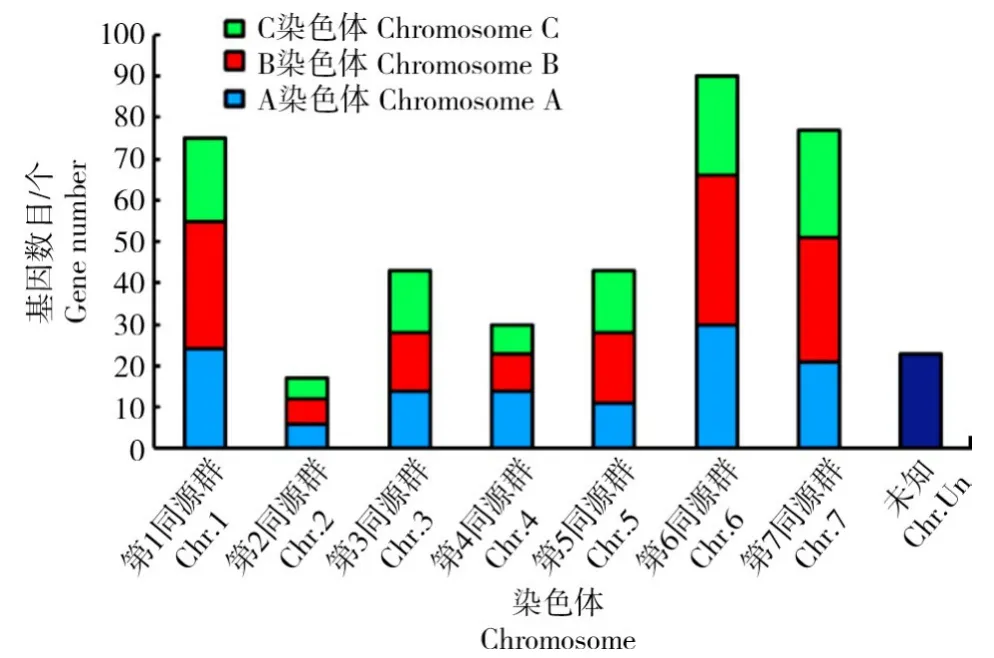
图4 小麦组蛋白家族成员在染色体上的分布数目Fig.4 The distribution number of histone family members of wheat on chromosomes
2.3 盐胁迫下TaHis3.2 表达模式分析
小麦TaHis3.2表达模式如图5 所示。
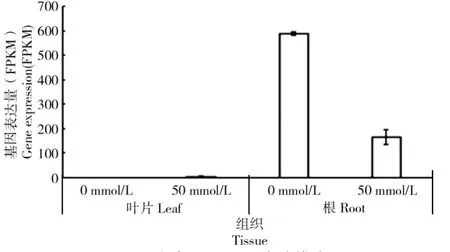
图5 小麦TaHis3.2 表达模式Fig.5 Expression pattern of TaHis3.2 in wheat
为了明确本研究克隆到的TaHis3.2基因表达是否具有组织特异性,是否受到盐胁迫的影响,本研究具体分析了其表达模式,结果表明,自然状态下,小麦TaHis3.2主要在根部表达,叶片几乎探测不到表达,暗示该基因可能主要在根部行使相关功能;盐胁迫处理下,根部表达受到抑制,叶片探测到的reads 较少(图5),说明该基因在小麦苗期主要在根部表达,且盐胁迫抑制该基因的表达。
2.4 TaHis3.2 不同品种间序列特异性分析
通过设计基因组特异引物(Ta-2-F/Ta-1-R),并在二倍体(AA 和DD)、四倍体(BBAA)、六倍体(BBAADD)小麦材料中进行扩增。D 基因组特异引物在没有D 基因组成分的材料中均无扩增,仅在有D 基因组供体的材料中有扩增(图6)。同时在多种材料中扩增该基因,基因区未发现核苷酸序列差异,说明D 基因组在不同来源的材料中序列高度保守。

图6 小麦TaHis3.2 基因D 基因组特异引物不同倍性材料扩增Fig.6 Amplification of TaHis3.2 in genome D by different ploidy materials of specific primers
2.5 TaHis3.2 单倍型及变异频率分析
为了进一步明确D 基因组上His3.2在自然界中的变异情况,探讨该基因的保守性,本研究对667 份小麦重测序数据进行分析,调取该基因的变异信息。发现在667 份来自不同地域的小麦材料中,该基因存在2 种单倍型,单倍型Ⅰ为主要的单倍型,单倍型Ⅱ包含的材料数目少于单倍型Ⅰ(图7-A)。2 种单倍型在编码区仅存在一个无义突变,并未导致氨基酸差异(图7-B)。说明His3.2 在进化上高度保守,也暗示His3.2 具有重要功能,一旦发生氨基酸突变,可能导致植株无法正常生存。

图7 小麦TaHis3.2 基因D 基因组单倍型Fig.7 Haplotype of TaHis3.2 in D genome
3 结论与讨论
小麦作为重要的农作物,耐盐性水平影响小麦的广适性,据统计,京津冀区域中的滨海新区、宁河、宝坻、静海、沧州、衡水、邢台、唐山等地区含有大量的盐渍化土壤,其面积达到京津冀总耕地面积的25%,提高这部分盐渍化区域小麦单产,成为京津冀区域小麦总产量进一步提高的重要途径。除栽培措施以外,培育筛选耐盐性突出小麦新种质也是小麦中低产田产量进一步提升关键所在。通过耐盐基因的克隆,挖掘优异耐盐单倍型,并开发成标记,能够加速小麦耐盐育种进程,提升小麦耐盐水平。因此,转录组测序技术、重测序技术被广泛用于耐盐基因的挖掘。
本研究通过盐胁迫下转录组测序,结合小麦AA、DD 基因组祖先种供体乌拉尔图小麦、粗山羊草的基因组测序数据,挖掘到了一个受到盐胁迫抑制的基因,经过生物信息学分析,该基因含有一个典型的组蛋白保守结构域,初步认定该基因为组蛋白编码基因TaHis3.2,在六倍体小麦中共分离到2 种编码序列,其中一条序列来自于D 基因组,通过序列比对发现,该基因在不同基因组上序列一致性较高,具有高度的保守性。前人研究表明,组蛋白为染色体上的重要基因,对维系染色体结构具有重要意义[20-21]。因此,该基因的变异可能会严重影响染色体结构,从而影响DNA 的复制,最终影响生物体的各项生命进程。
通过组蛋白保守结构域(PF00125)在中国春基因组中调取该家族全部成员,发现在六倍体小麦中共存在398 个成员,且长短不一。组蛋白在各条染色体上均有分布,在同一个同源群上分布接近。目前,发现组蛋白除了H2A、H2B、H3、H4 外,还有多 种 变 体[22-24],如H2AZ,当DNA 双 链 断 裂 时,H2AZ 可能通过改变核小体的稳定性,与核小体重塑复合体部分冗余,并参与转录控制,从而维系染色体结构的稳定性,说明组蛋白不同变体具有不同的作用,影响组蛋白修饰等[25-27]。同时,染色体上的多个组蛋白可能具有功能的协同作用,如果1 个组蛋白基因发生突变,功能缺失,其余组蛋白编码基因可继续行使相关功能,确保生命体新陈代谢不受过多影响。另外,组蛋白编码基因也可能是不同组蛋白在不同发育时期、不同组织中起到相关作用,如本研究中克隆的TaHis3.2主要在根部表达,而在叶片中几乎检测不到。
盐胁迫主要导致小麦发育迟缓,生长缓慢,抽穗期晚于对照,株高、根长都受到明显抑制[28]。本研究发现,组蛋白TaHis3.2的表达受到了盐胁迫的抑制,表明盐胁迫影响了组蛋白的表达,可能影响了DNA 的复制,与盐胁迫抑制植物生长的现象相吻合。本研究克隆的TaHis3.2主要在根部表达,在叶片几乎探测不到表达水平,根系的发育情况与植物抵御非生物胁迫密切相关,暗示该基因与逆境胁迫密切相关。
本研究设计了D 基因组特异性引物,在六倍体小麦中分离到D 基因组上的TaHis3.2-D基因序列,在亲缘关系较远的小麦材料序列分析中发现,TaHis3.2-D基因序列高度保守,并未发现序列差异。目前,由于D 基因的高度保守性,小麦大部分的单倍型主要发现于B、A 基因组,D 基因组由于变异较小,开发的标记也相对较少。同时,已经公布的基因组重测序数据中,也发现TaHis3.2-D在667 份来自于不同区域的材料中仅存在2 种单倍型,并且编码同一种氨基酸。由于组蛋白基因功能的重要性[15],一旦发生变异,可能导致生物体的致死突变,因此,基因的变异频率本身较少。
组蛋白虽然受到了盐胁迫的抑制,但是该基因的D 基因组上未发现序列差异,因此,不适合在基因区开发标记进行单倍型研究,用于小麦耐盐分子育种。在今后的研究中,可以尝试在该基因的D 基因组的启动子区域进行SNP 的挖掘,同时设计基因组特异引物区分B、A、D 的表达,明确哪条基因组上的基因与盐胁迫最相关。同时,利用B、A 基因组上该基因的编码区及启动区进行SNP 挖掘,开发相关标记。
本研究重点关注组蛋白编码基因,利用同源克隆法从小麦中获得组蛋白编码基因His3.2,明确其蛋白序列特征TaHis3.2 包含PF00125 保守结构域,通过HMMER 软件进行全家族调取,发现小麦中该家族共398 个成员,在各个染色体上均有分布,且主要分布在染色体的两端。表达模式分析显示,TaHis3.2 主要在根部表达,盐胁迫抑制该基因的表达。自然群体序列分析发现,在D 基因组该基因高度保守,仅存在2 种单倍型。本研究为挖掘小麦耐盐相关基因、开发盐胁迫下的分子标记提供了新的思路。
