‘横冲89’油茶转录组及光合油脂代谢途径基因表达分析
2022-07-20王保明颜士华王胜清陈永忠商建波
王保明,颜士华,王胜清,陈永忠,商建波
(1. 湖南应用技术学院 农林科技学院,湖南 常德 415000;2. 临沂科技职业学院 现代农业系,山东 临沂 276000;3. 湖南省林业科学院 国家油茶研究工程中心,湖南 长沙 410004)
高通量RNA 测序分析(RNA-Seq)包括测序、组装、基因注释、基因表达分析过程,是研究基因功能的有效途径,采用该技术能够快速、高效、全面地比较分析转录组,发掘功能基因,准确研究基因表达水平及表达调控[1-5]。如,通过对C4植物狗尾草Setaria viridis的叶、茎、节、冠、根、小穗和种子转录组分析,揭示了其营养生长和生殖生长的规律,并发掘出大量与光合效率有密切关系的光合基因,从而为优良种的筛选和品种改良奠定了理论基础[6]。
油茶是我国重要的食用油料树种,从茶籽中提取的茶油是优质食用植物油,含有82%~84%的不饱和脂肪酸,其中油酸含量为67.7%~76.7%,其品质可与橄榄油相媲美[7]。但是,低产油量目前已成为油茶产业发展的瓶颈。因此,开展油茶遗传改良,培育出优质高产油品种已经成为当务之急。研究油茶代谢途径及基因表达对改良品种具有重要的参考价值。目前,已有利用转录组研究小果油茶与普通油茶间的系统发育关系[8]、进行优良单株间的比较[9],以及有关转录因子[10]、油脂合成代谢[11]、基因表达与叶花可溶性糖含量的关系等研究的报道,如,在幼苗期、幼树期、初果期等生长期,基因表达量与可溶性糖含量、可溶性蛋白含量具有因果关系[12-13]。但是,目前对光合作用、CO2固定、脂肪酸以及油茶代谢机制的解析及基因代谢作用网络的研究仍然不足,这严重影响了功能基因研究以及品种改良进程。在前期研究中,本课题组已经鉴定了油茶光合作用关键酶Rubisco 亚基基因rbcL和rbcS,以及油脂代谢途径脂肪酸合成的关键酶异质型ACCase 基因Co-accC和Co-accD[14-15]。但是,对这些基因是否含有同源基因、结构和功能变化以及网络调控机制的认识仍然缺乏。在本研究中,笔者利用Illumina RNA-seq 高通量技术开发油茶叶花成熟种子转录组,并进行分析,旨在了解油茶的代谢途径及调控机制,为揭示光合作用、脂肪酸和油脂合成机制及挖掘功能基因提供参考。
1 材料与方法
1.1 RNA 提取和转录组文库构建
以湖南省林业科学院国家油茶工程技术研究中心试验地的优良油茶品种‘横冲89’为研究对象。采集叶、花芽、雄蕊、雌蕊、子房、成熟种子,立即液氮冷冻,-70 ℃贮藏。利用CTAB 裂解法分别提取上述组织的总RNA。以花芽、雄蕊、雌蕊、子房提取的总RNA 组成花混合样构建花的转录文库,其分别占混合样总量的3/6、1/6、1/6、1/6。用DNase Ⅰ(NEB 有限公司,北京)除去残留的基因组DNA。用Qubit 2.0 荧光定量仪(Life Technologies,美国)测定总RNA浓度,用Agilent 2100 生物分析仪系统(Agilent Technologies,美国)测定RNA 的浓度和完整性。用磁珠分离富集mRNA(Life technologies,美国)。将mRNA 短片化,以mRNA 为模板,用六碱基随机引物合成第1 链cDNA,然后加入缓冲液、dNTPs 和DNA 聚 合 酶Ⅰ合 成 第2 链cDNA,用AMPure XP beads 核酸纯化试剂盒(Beckman Coulter,美国),去除dNTP、引物、引物二聚体、盐离子和其他杂质,选择200 bp 文库片段两端连接头,用通用PCR 引物和Index primer(NEB)经PCR 扩增富集cDNA 文库。用Qubit 2.0 荧光定量仪初步定量,用Agilent 2100 生物分析仪系统精准检测文库质量,用Illumina HiSeq 2000 测序仪进行2×100 bp 测序。
1.2 测序数据质量评估和转录组装配
加工消除原始数据中的poly-N、低质量以及接头读长序列,计算测序数据中Q20、Q30、GC含量,将原始测序数据进行统计和评估,对其进行质控分析,得到高质量的净读长序列[16]。以FASTQ 文件格式储存,用Trinity 软件[17]进行De Novo 拼接。将测序得到的读长(reads)与前面所得的拼接结果进行比对。
1.3 基因功能注释、KEGG 通路的富集分析以及光合作用、脂肪酸合成基因的挖掘
根据NCBI 非冗长蛋白序列(non-redundant protein sequences,Nr)、非冗长核酸序列(nonredundant nucleotide sequences,Nt)及蛋白质家族Pfam、Swiss-Prot、KOG 进行基因功能注释。以GO(gene ontology)数据库和KOG 数据库对基因进行功能分类;采用BLAST 算法将基因与KEGG基因数据库(GENES)进行比对,再根据KOG 数据库的KO 编号查找生物学通路,提供所分析基因可能参与的生物学通路[18]。分析糖代谢、能量代谢、油脂代谢途径,挖掘参与光合作用CO2固定的关键酶Rubisco 基因、脂肪酸合成乙酰辅酶A羧化酶(acetyl-CoA carboxylase,ACCase)基因。
1.4 转录组光合作用、脂肪酸形成基因的差异表达分析
利用RSEM 估算基因的表达水平,用DEGseq(2010)R 包分析油茶叶花果转录组基因表达差异[4]。在此基础上,研究参与光合作用CO2固定的Rubisco 基因、脂肪酸合成关键酶ACCase 基因以及其他油脂合成基因,分析同源基因结构功能差异,比较其表达差异[4-5]。
2 结果与分析
2.1 油茶各组织器官RNA 转录组文库
利用CTAB 裂解法提取油茶幼叶、花芽、雄蕊、雌蕊、子房、成熟种子的总RNA,电泳结果如图1 所示。用DNase Ⅰ除去残留的基因组DNA。各样本RNA 提取液的A260/A280值分别为2.119、1.868、2.083、1.933、1.919 和2.093。总RNA 的浓度达到建库要求,用Agilent 2100 生物分析仪系统测定RNA 的完整性,结果表明RIN(RNA integrity number)值为8.2 ~8.9,符合建库标准。分离富集叶、种子的mRNA,合成叶和种子的cDNA 转录组。以花芽、雄蕊、雌蕊、子房的mRNA 为模板构建花转录组,其中,花芽的mRNA占1/2,雄蕊、雌蕊、子房的各占1/6。

图1 所提取油茶各组织器官的总RNA 的电泳结果Fig. 1 The total RNAs of different tissues of C. oleifera
2.2 油茶各组织器官转录组数据
去除含有接头的序列,过滤低质量序列,纯化原始序列,在叶、花、种子中分别有18 019 152、17 313 103、24 209 779 条原始读长序列。叶的净读长序列为17 338 672 条(占96.22%),含N 序列为150 009 条(占0.83%),低质量序列为506 630条(占2.81%),接头序列为23 841 条(占0.13%);花的净读长序列为16 647 147 条(占96.13%),含N 序列为150 589 条(占0.87%),低质量序列为491 526 条(占2.84%),接头序列为27 677 条(占0.16%);成熟种子的净读长序列为22 896 112 条(94.57%),含N 序列为171 751 条(0.71%),低质量序列为1 103 179 条(4.56%),接头序列为38 737 条(0.16%)。获得的叶转录组为4.50 Gb,获得的花转录组为4.24 Gb,获得的种子转录组为4.58 Gb,总计13.32 Gb。通过转录组质控分析发现,Q20、Q30 分别超过97%和90%,叶、花、种子转录组的GC 含量分别为44.69%~44.81%、44.77%~44.87%和47.29%~47.39%,质控数据优于大豆(41.5%)[19]、苜蓿(39.5%)[19]、鹰嘴豆(40.3%)[16]、拟南芥(42.5%)[5]。因此,这些转录组数据完全符合建库要求,可以进一步用于拼接和序列特征分析。
除去油茶转录组原始读长中的接头序列、低质量序列和含N 序列,得到高质量的净读长序列,然后进行De Novo 拼接,其转录本和Unigene 的长度分布如图2 所示。由图2 可以看出,拼接的油茶各组织器官转录组的Unigen 和转录本数量分别为53 382 和80 291,长度为200 ~500 bp。最小序列长度为201 bp,最大序列为14 543 bp,Unigen和转录本的平均长度分别为670 bp 和966 bp。油茶各组织器官转录组基因数量如图3 所示。由图3 可以看出,叶、花、种子中的Unigene 数量分别为66 379、64 328、57 613,获得的3 个转录组基因总数为82 964。其中,叶、花共有基因数为54 113,叶、种子共有基因数为49 110,花、种子共有基因数47 232。叶、花、种子中特异基因数分别为6 377、6 204 和4 492。
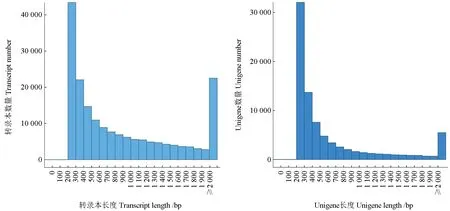
图2 油茶转录组的转录本和Unigene 的长度分布Fig. 2 The length distribution of the transcripts and Unigenes of C. oleifera transcriptomes

图3 油茶各组织器官转录组的基因数量Fig. 3 Gene numbers of C. oleiera transcriptomes of different tissues
通过比较Unigene 长度以及进行Nr、Nt、Swiss-Prot、Pfam、GO、KO、KOG 分 析, 发 现叶、花、种子转录组中分别有73.79%、76.30%和75.17%的测序序列匹配到拼接的转录组上。这些Unigenes 与NCBI、SwissProt 数据库基因相似性约为46.51%,与Nr 蛋白数据的相似性为53.49%。
2.3 油茶各组织器官转录组基因功能
将油茶各组织器官转录组中的Unigene 在GO分类功能数据库注释比对,结果如图4 所示。由图4 可以看出,油茶叶、花、种子3 个转录组的注释Unigene 数量分别为28 818、29 673、25 896,分属于生物过程、细胞组分和分子活性3 大类的57 个小类。生物过程包括应激反应、多细胞生物过程、细胞繁殖等24 小类。其中,多细胞生物过程中的注释Unigene 数量最多,叶、花、种子转录组的注释Unigene 数量分别为20 091、19 283、17 460;代谢过程次之,3 个转录组的注释Unigene 数量分别为18 574、17 759、16 070;细胞杀死中的注释Unigene 数量最少,3 个转录组的注释Unigene 数量分别为26、26 和21。细胞组分包括细胞、膜内腔等19 小类。其中:细胞中的注释Unigene 数量最多,叶、花、种子转录组的注释Unigene 数量分别为18 400、17 718、16 271;其次是细胞组分,3 个转录组的注释Unigene 数量分别为18 322、17 639、16 211;再次为细胞器,3 个转录组的注释Unigene 数量分别为14 356、13 855、12 831。分子活性包括金属伴侣、受体活性、通道调节、催化活性等14 类。其中:结合中注释Unigene 数量最多,叶、花、种子转录组的注释Unigene 数量分别为17 373、16 875、15 285;催化活性次之,3 个转录组的注释Unigene 数量分别为15 009、14 431、12 977。

图4 油茶各组织器官转录组注释Unigene 的GO 分类Fig. 4 Gene ontology (GO) classification of the annotated unigenes of C. oleifera different tissue transcriptomes
以KEGG 数据库为参考,将24 313 条Unigene(分别包括叶、花、种子转录组的8 030、8 868、7 415 条Unigene)分为细胞过程、环境信息过程、遗传信息过程、代谢、生物系统5 类,共31 小类,251 个代谢途径,如图5 所示。由图5 可以看出,Unigene 数量较多的为A 类的运输和分解代谢,B类的信号传导、折叠分类降解,C 类的翻译,D 类的糖代谢、能量代谢、脂质代谢,E 类的免疫系统。

图5 油茶各组织器官转录组注释Unigene 的KEGG 分类Fig. 5 KEGG classification of the annotated unigenes of C. oleifera different tissue transcriptomes
以KOG 数据库为参考,将油茶叶、花、种子转录组中的11 286、13 849、10 638 条Unigene 进行分类,结果如图6 所示。其中:分布最多的是普遍功能预测类,共有2 185 条,分别包括叶、花、种子转录组的2 023、2 070、1 879 条注释Unigene;其次是翻译后修饰、蛋白转运、伴侣蛋白类(O 类),有1 564 条,包括3 个转录组的1 368、1 518、1 302 条注释Unigene。
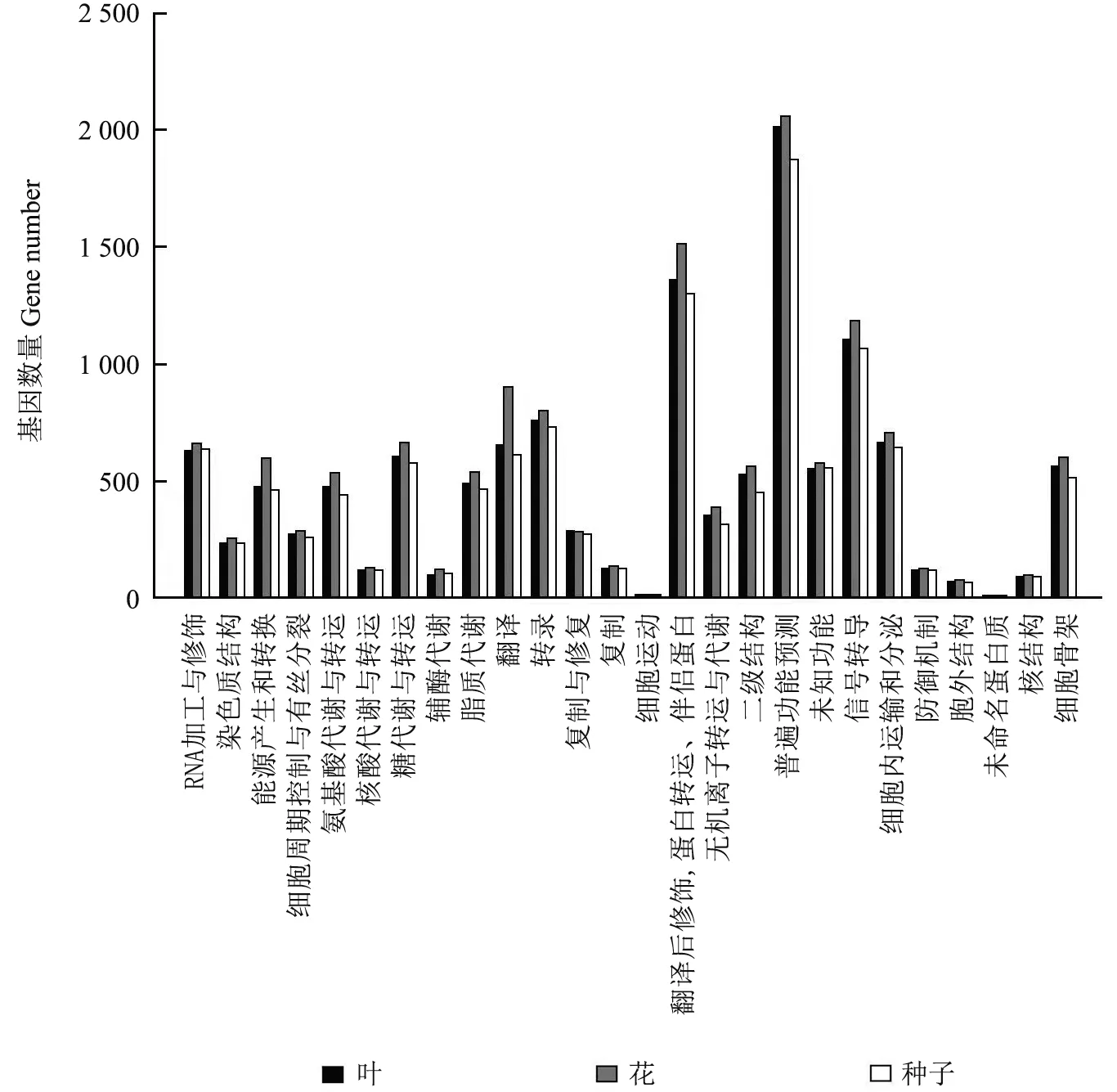
图6 油茶各组织器官转录组注释Unigene 的KOG 分类Fig. 6 KOG classification of the annotated Unigenes of different tissue transcriptomes of C. oleifera
2.4 油茶各组织器官转录组在部分代谢途径的参与情况
使用KEGG 数据库,对糖代谢、能量代谢以及油脂代谢途径进行分析,结果见表1。由表1 可知,1 439 条基因通过乙醛和二羧酸代谢、戊糖葡萄糖醛酸代谢等14 个途径参与了糖代谢,其中共有98 条基因参与乙醛和二羧酸代谢(ko00630)途径。共有864 条基因通过光合作用(ko00195)、光合生物固碳(ko00710)、光合作用天线蛋白(ko00196)等8 个途径参与能量代谢。共有666条基因通过脂肪酸合成、脂肪酸代谢等15 个代谢途径参与油脂代谢。
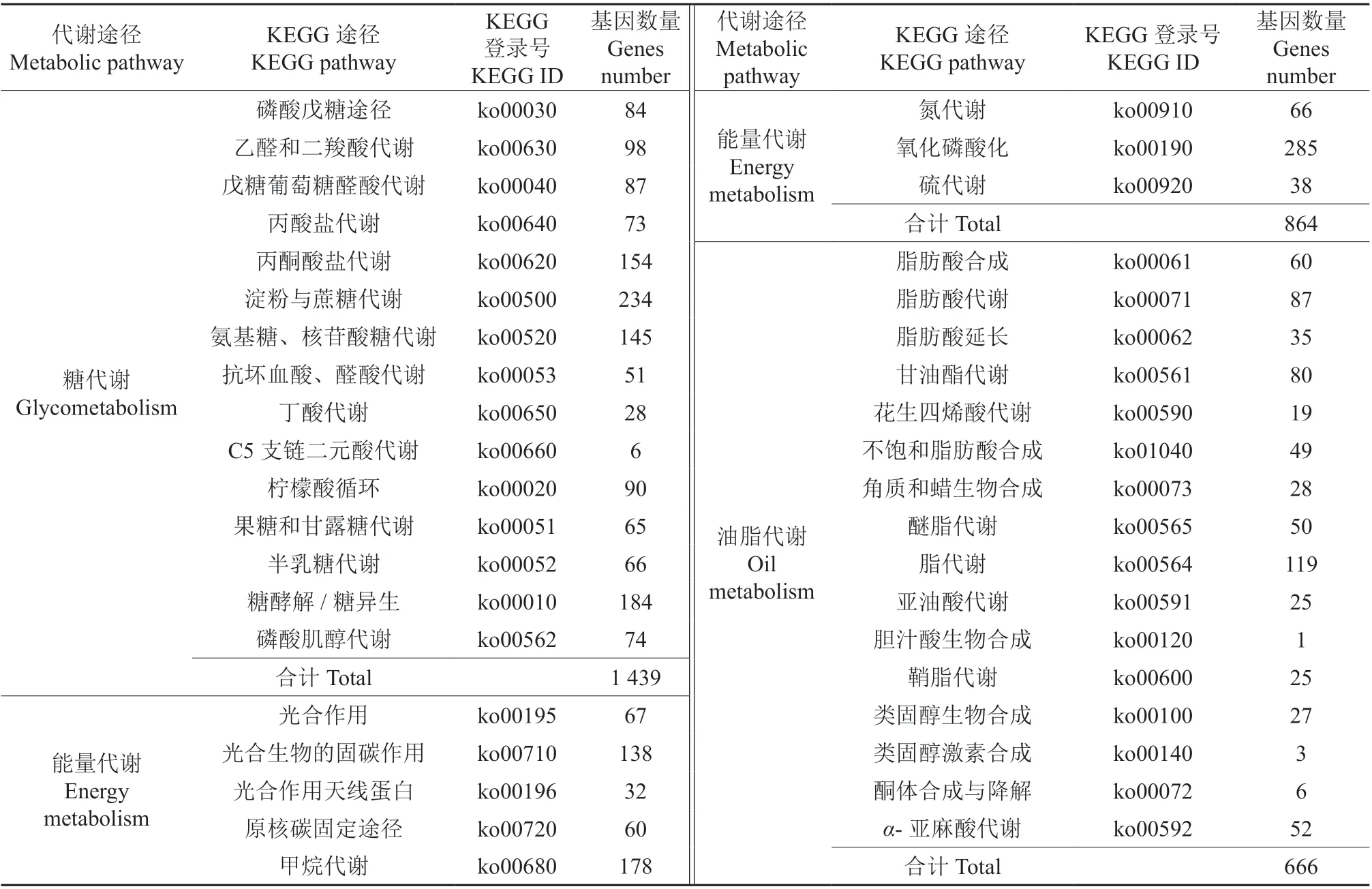
表1 油茶各组织器官转录组参与糖代谢、能量代谢、脂质代谢途径的KEGG 分析结果Table 1 KEGG analyses of different tissue transcriptomes of C. oleifera participated in the photosynthetic, carbohydrate and lipid metabolism pathways
油茶各组织器官转录组参与糖代谢、能量代谢、油脂代谢途径的KO 分析结果见表2。由表2可知,参与光合作用中CO2固定的关键酶Rubisco的亚基基因中,有1 条大亚基基因,有7 条小亚基基因。在光合作用途径(ko00195)中,有CP47 叶绿素载脂蛋白(K02704,comp87827_c1)、CP43叶绿素载脂蛋白(K02705,comp127116_c0)、细 胞 色 素b559 的α亚 基 蛋 白(K02707,comp80177_c1)、PsbI 蛋 白(K02710,comp56372_c1)、光系统Ⅱ氧增强子蛋白2(K02717,comp77787_c0)、PsbY 蛋白(K02723,comp73953_c0)、光系统Ⅱ Psb27 蛋白(K08902,comp73953_c0)、光系统Ⅰ亚基Ⅱ(K02692,comp76836_c0)、光系统Ⅰ亚基V(K08905,comp78080_c1)、光系统Ⅰ亚基X(K02698,comp76156_c0)的编码基因。在脂肪酸合成途径(ko00061)中,脂肪酸合成关键酶ACCaseα-羧基转移酶(α-CT)、生物素羧基载体蛋白(BCCP)、β-羧基转移酶(β-CT)、生物素羧化酶(BC)亚基的编码基因accA、accB、accD、accC存在同源基因,其中,α-CT 编码基因4 条、BCCP 编码基因4 条、β-CT 编码基因7 条、BC 编码基因20 条。
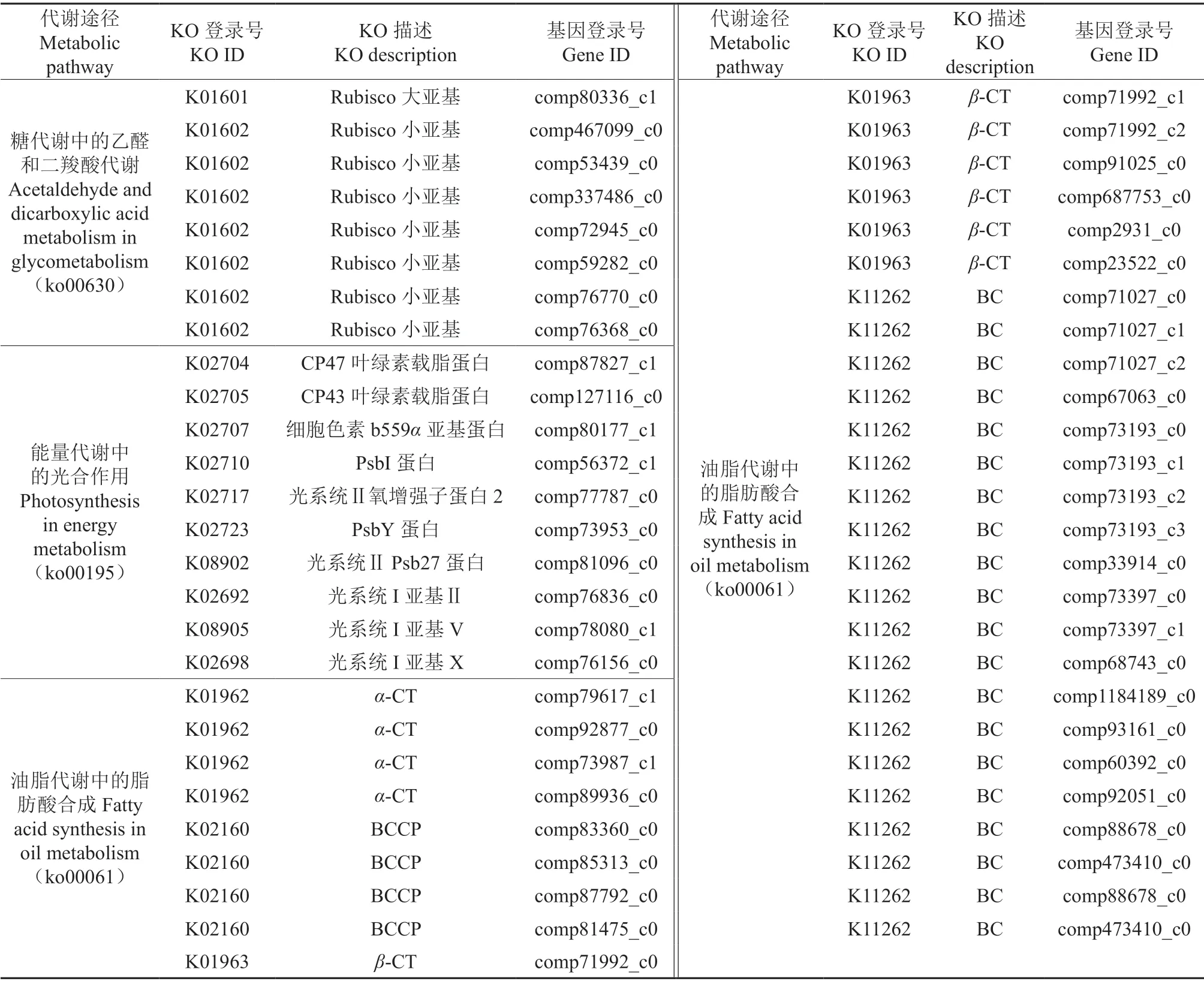
表2 油茶各组织器官转录组参与糖代谢、能量代谢、油脂代谢途径的KO 分析结果Table 2 The KO analyzed result of carbohydrate, energy metabolism and lipid metabolism pathways of different tissue transcriptomes of C. oleifera
2.5 油茶各组织器官转录组的差异表达
约有60 002 条(90.39%)Unigene 在油茶叶、花、种子中表达。其中,叶、花中共有54 113 条Unigene,叶、种子中共有49 110 条Unigene,花、种子中共有47 232 条Unigene,叶、花、种子共有43 221 条Unigene(图3)。在参与的代谢途径中存在叶、花、种子基因表达的差异,各组织器官转录组的差异表达基因数量如图7 所示。叶-花、叶-种子、花-种子差异表达基因分别为4 358、4 675 和4 942 条。如,光合作用代谢途径中,叶绿素a/b 结合蛋白4(comp79853_c0)在叶中的相对表达量为2 402.56,而在种子中为0.13。果糖1,6-二磷酸醛缩酶(comp46464_c0)和叶绿素a/b 结合蛋白(comp68492_c1)在叶中的表达量是种子中的500 倍。在脂肪酸合成途径(ko00061)中的脂肪酸合成酶(comp8554_c0)、类脂肪酸合成酶异构体2(comp5764_c0)等基因仅存在于花转录组中。而甘油脂代谢途径(ko00561)中乙醇脱氢酶、NADP+(comp548255_c0)等基因仅存在于种子中。

图7 油茶各组织器官转录组的差异表达基因数量Fig. 7 Gene number of differential expression different tissue transcriptomes of C. oleifera
3 结论与讨论
本研究结果揭示了参与糖代谢、能量代谢、油脂代谢途径的油茶叶、花、种子转录组Unigene,及其在光合作用及油脂合成过程中的差异表达。
RNA-Seq 技术包括测序、组装、基因注释、基因表达分析等过程,使用该技术能够快速高效、高通量比较转录组[20]。在本研究中,以66 520 000条油茶净读长序列组装185 190 个转录本,82 964条Unigene。通过将这些Unigene 与其他山茶属以及油料树种的基因比较,可发掘出新的功能基因,提高品种的遗传改良效率。通过对代谢途径分析,特别是对脂类代谢、RNA 降解糖代谢、脂类代谢等过程的分析,可发掘出参与光合作用和油脂代谢的关键基因。在本研究中共发现光合作用相关基因237 条,油脂代谢相关基因666 条,其中与脂肪酸合成相关Unigene 60 条。
通过对KEGG 代谢途径的研究,可以从细胞过程、结合、代谢过程、细胞组分等方面解释细胞分子相互作用网络、生物体中的变化。在油茶光合代谢途径(ko00195)中鉴定出许多参与光合作用的基因。Zhong 等[21]的研究结果表明,在糖代谢中,叶、花、种子转录组的Unigene 分别为741、868、661 条,三者共有Unigene 902 条。将叶、花、种子转录组的Unigene 进行KOG 基因功能分类,找寻直系同源基因,可以提高基因功能注释的准确性。已有研究结果证明Rubisco 在油茶光合作用中发挥重要的作用[14]。高产、高含油量油茶种质的连续和长期性选择能够增加Rubisco 基因的丰度,进而提高光合性能、光合效率。这种选择行为会提高脂肪酸合成的量,从而提高油茶产油量。在本研究中鉴定出参与光合作用的Rubisco 编码基因的1 条大亚基、7 条小亚基,这与在C4 植物狗尾草中发现的5 个拷贝rbcS同源基因存在一定的结构差异[6]。深入了解Rubisco 大小亚基的功能有助于利用Rubisco 大小亚基基因分子标记识别高产油品种,缩短油茶育种时间,提高育种效率[14]。
油茶异质型ACCase 亚基α-CT、BC、β-CT的编码基因在脂肪酸与油脂合成中发挥重要作用[11,15]。本研究结果表明,参与脂肪酸合成的异质型ACCaseα-CT、BCCP、β-CT、BC 亚基的编码基因存在多个同源基因,分别为4、4、7、20条, 并 且 发 现comp71027_c0、comp71027_c1、comp71027_c2、comp67063_c0、comp60392_c0 可能为编码同质型ACCase 的同源基因[15]。
本研究仅聚焦于油茶叶、花、果基因的表达,油茶基因表达、代谢网络与果实主要性状指标及产量的关系有待进一步探索。另外,在油茶栽培实践中,产地、气候条件、立地条件、植株年龄、冠幅、叶片特征以及果实采收时期等因子可能影响油茶果实性状[22-23],进而影响其产量,后续可考虑在基因水平分析上述因子对茶油脂肪酸组成和活性成分的影响,进而为筛选或培育优质高产的育种材料提供参考。
