青花菜花蕾转录组测序分析及蜡粉合成相关基因挖掘
2022-06-15张振超陶美奇潘永飞戴忠良姚悦梅
张振超 陶美奇 潘永飞 戴忠良 姚悦梅

摘要:【目的】对青花菜花蕾进行转录组测序分析,并挖掘与蜡粉合成相关基因,为探明青花菜花球表面蜡粉形成的分子机制提供理论参考。【方法】分别提取野生型和蜡粉缺失型青花菜花球总RNA,采用Illumina HiSeqTM2500平台进行转录组测序,获得高质量Clean reads,采用Trinity进行序列组装后获得青花菜Unigene库,将获得的Unigene序列与Nr、Nt、KEGG、Pfam、KOG/COG、Swiss-Prot和GO数据库比对,获得基因功能注释信息;使用DESeq2进行差异表达分析。【结果】共获得44.68 Gb Clean data,De novo组装得到41244条Unigenes,N50长度为1847 bp。从所获得的Unigenes中筛选出8685个差异表达基因(DEGs)(上调基因5747个,下调基因2938个),共有8038个基因被注释到不同数据库,其中,5220个基因注释到Pfam数据库;2066个基因注释到COG数据库,3866个基因注释到KOG数据库;2580个差异表达基因被注释到75个转录因子家族中,注释最多的是MYB家族(235个);GO数据库中6095个差异表达基因注释到细胞组分、分子功能和生物学过程三大类的52个功能分类;KEGG数据库中,1671个差异表达基因富集到138条代谢通路,其中13个差异表达基因与脂肪酸合成有关,7个差异表达基因与蜡粉生物合成途径有关。【结论】转录因子MYB家族在调控青花菜蜡粉合成中发挥重要作用。蜡粉合成过程中相关酶基因的差异表达是调控青花菜蜡粉合成的关键,尤其是野生型和蜡粉缺失突变体中特异性表达的差异表达基因,可作为后续研究青花菜花球表面蜡粉形成分子机制的对象。
关键词: 青花菜;花蕾;蠟粉缺失突变体;转录组;基因功能注释
中图分类号: S635.3 文献标志码: A 文章编号:2095-1191(2022)03-0607-11
Transcriptome analysis and mining of genes related to wax powder synthesis of broccoli flower buds
ZHANG Zhen-chao, TAO Mei-qi, PAN Yong-fei, DAI Zhong-liang, YAO Yue-mei
(Zhenjiang Institute of Agricultural Sciences in Hilly Area of Jiangsu Province, Jurong, Jiangsu 212400, China)
Abstract:【Objective】To perform transcriptome sequencing on broccoli buds, and mine their genes related to wax powder synthesis, so as to lay a foundation for the discovery of the molecular mechanism of wax powder formation on the surface of broccoli spherules. 【Method】Total RNA from wild-type and wax-powder-deficient broccoli flower bulbs was extracted, and Illumina HiSeqTM2500 platform was used for transcriptomes sequencing to obtain high-quality Clean reads, and Trinity software was used for sequence assembly to obtain the broccoli Unigene library. The obtained Unigenes sequence was compared with that in Nr, Nt, KEGG, Pfam, KOG/COG, Swiss-Prot, GO databases to obtain gene function annotation information; DESeq2 software was used for differential expression analysis. 【Result】A total of 44.68 Gb Clean data was generated. And 41244 unigenes with N50 length of 1847 bp, were obtained by De novo assembly method. 8685 differentially expressed genes (DEGs) (5747 up-regulated genes and 2938 down-regulated genes) were obtained in the unigenes, of which 8038 had annotated information. Of these, 5220 genes were annotated to the Pfam database; 2066 genes annotated to COG database and 3866 genes annotated to KOG database annotation analysis showed that 36230 unigenes had homologens in different public protein databases. A total of 2580 DEGs were annotated to 75 transcription factor families, and the most annotated was MYB family (235). In GO database, 6095 DEGs were annotated to 52 functional groups in three categories:cell components, molecular functions and biological processes. In KEGG database, 1671 DEGs were classified into 138 metabolic pathway branches, and 13 DEGs were annotated to fatty acid synthesis related pathway, and 7 DEGs were annotated to wax powder biosynthesis related pathway. 【Conclusion】The MYB family of transcription factors plays an important role in the regulation of wax powder synthesis in broccoli. The differential expression of related enzyme genes during wax powder synthesis is the key to regulating wax powder synthesis in broccoli. In particular, the DEGs in wild-type and wax powder-deficient mutants can be used for further study on the molecular mechanism of wax powder formation on the surface of broccoli.BF9E10E1-A8E8-4AA4-96C2-0B323DA83DF0
Key words: broccoli; flower bud; wax powder deletion mutant; transcriptome; gene function annotation
Foundation items: Jiangsu Key Research and Development Project (BE2021376); Jurong Science and Technology Innovation Fund Project (ZB22105);Youth Fund Project of Zhenjiang Academy of Agricultural Sciences(QNJJ2021003)
0 引言
【研究意义】植物蜡粉,又称蜡质,是覆盖于植物组织外在表皮上脂质成分的统称,由脂肪酸、烷烃、醇类、醛类和酮类等亲脂性化合物组成,一般呈绿灰色或灰白色霜状(Domínguez et al.,2011;张曦等,2013)。蜡粉不仅是植物表皮细胞外的疏水屏障,还是重要的防卫机构,在植物生长发育过程中发挥反射紫外线、抵御病菌入侵、防止食草昆虫侵害等作用(Oliveira et al.,2004;王立山等,2018)。青花菜(Brassica oleracea L. var. italica Plenck)口感清脆、营养价值高且富含抗癌活性成分硫代葡萄糖苷,被誉为“蔬菜皇冠”。青花菜花球表面因覆盖一层蜡粉而呈灰绿色或灰白色,其中以蜡粉含量多、颜色灰绿的花球较受市场青睐(张振超等,2021)。因此,开展青花菜花球表面蜡粉合成代谢的分子机制研究及相关功能基因挖掘,对提高青花菜优异种质资源创制和新品种选育具有重要意义。【前人研究进展】近年来,拟南芥、油菜、大白菜、玉米、大麦等作物的蜡粉缺失突变体均有报道。拟南芥和大麦的蜡粉突变位点被称为eceriferum(cer),油菜和玉米中的突变位点被称为glossy(张曦等,2013;Saet and Mi,2015)。目前从大麦蜡粉突变体中获得了85个cer突变位点(Wettstein and Chua,1987);从拟南芥中筛选出32个突变位点,其中与蜡粉合成相关的基因有20多个,如CER1、CER2、KCS1和WAX2等,这些基因参与蜡粉直接合成、转录和激素水平调控等(Pruitt et al.,2000;Jenks et al.,2002;Chen et al.,2003)。对油菜(Pu et al.,2013)、白菜(Zhang et al.,2013)和结球甘蓝(Liu et al.,2017,2018)中的蜡粉缺失基因进行区间精细定位,并将区间内与拟南芥CER1和CER4基因同源的基因确定为候选目的基因。Pyee和Kolattukudy(1995)以青花菜中编码WAX9蛋白的cDNA序列作为探针从基因文库中分离出青花菜表面蜡粉4个同源基因wax9A、wax9B、wax9C和wax9D,并对蜡粉遗传规律进行了系统研究,结果显示,所有基因均在叶片和花蕾中表达,但均不在根中表达, wax9A、wax9B和wax9D在茎和花中也有表达。近年来,随着高通量测序技术快速发展,该技术被广泛应用于基因表达与植物表型关系的研究。利用该技术对同一物种不同样本进行转录组测序分析,在获得大量功能基因的基础上比较样本间存在表达差异的基因,并根据生物信息学分析探究差异表达基因功能分类,筛选出与差异表型相关的基因群,进而发掘新的功能基因(林珲等,2018;吴宁柔等,2018;张振超等,2018)。【本研究切入点】目前高通量测序技术已成功应用到植物代谢机制和基因调控功能的研究中,如青花菜胚胎发育初期小孢子经高通量测序筛选得到大量与胚胎发育相关的代谢通路和候选基因(张振超等,2018)。但未见利用转录组測序技术对蜡粉合成基因挖掘及功能分析的研究报道。【拟解决的关键问题】以野生型青花菜花球与蜡粉缺失突变体为试材,采用高通量测序技术进行转录组测序分析,对差异表达基因进行功能注释,并对其中的脂肪酸和蜡粉合成相关基因进行KEGG代谢通路富集分析,以筛选出合成途径中的关键酶基因,为深入探究青花菜花球蜡粉生物合成代谢的分子机制、挖掘相关基因及遗传育种应用打下基础。
1 材料与方法
1. 1 试验材料
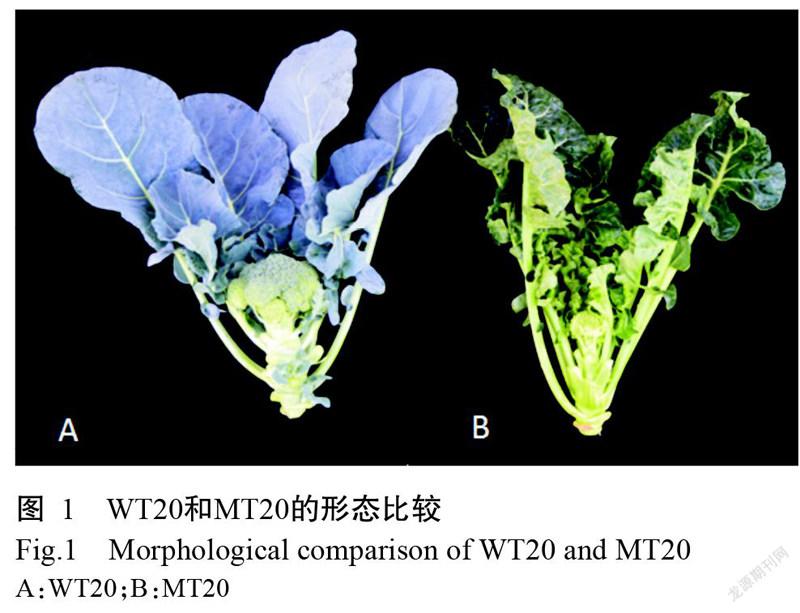
青花菜野生型(WT20)与蜡粉缺失突变体(MT20)(图1)由江苏丘陵地区镇江农业科学研究所提供。植物总RNA提取试剂盒购自宝生物工程(大连)有限公司。主要仪器设备:NanoDrop ND-2000微量紫外分光光度计(NanoDrop technology,美国)、Qubit2.0核酸蛋白荧光定量仪(美国赛默飞世尔科技有限公司)和Aglient 2100芯片生物分析仪(美国安捷科技有限公司)。
1. 2 试验方法
1. 2. 1 总RNA提取 参照植物总RNA提取试剂盒说明提取青花菜总RNA。设置3个生物学重复,每个重复3株。采用1%琼脂糖电泳检测RNA完整性,分别用NanoDrop ND-2000微量紫外分光光度计、Qubit2.0核酸蛋白荧光定量仪和Aglient 2100芯片生物分析仪定量检测RNA样品的浓度、纯度和完整性等,以保证使用合格的样品进行转录组测序。
1. 2. 2 转录组测序及生物信息学分析 转录组测序参照张振超等(2018)的方法。利用Illumina HiSeqTM 2500测序平台进行高通量测序,形成双端测序数据,测序读长为PE150。将获得的原始数据进行过滤,去除其中的接头序列及低质量reads,获得高质量Clean reads,采用Trinity进行序列组装后获得青花菜Unigenes库。利用Blast Version 2.2.26和HMMER 3.1进行测序饱和度、基因覆盖度和冗余序列分析,将获得的Unigenes序列在Nr(NCBI non-redundant protein sequences)、Nt(NCBI non-redundant nucleotide sequence)、KEGG(Kyoto encyclopedia of genes and genomes)、Pfam(Protein family)、KOG/COG(Clusters of orthologous groups of proteins)、Swiss-Prot(A manually annotated and revie-wed protein sequence database)、KEGG、GO(Gene ontology)数据库进行比对,获得基因功能注释信息。BF9E10E1-A8E8-4AA4-96C2-0B323DA83DF0
1. 2. 3 差异表达基因筛选及分析 采用FPKM值(Fragments Per Kilobase of transcript per Million mapped reads)表示基因表达水平,使用DESeq2进行差异表达分析。将|log2FC(Fold change,差异倍数)|≥1且FDR(False discovery rate,错误发现率)<0.05作为差异表达基因(Differentially expressed genes,DEGs)筛选标准。采用Benjamini-Hochberg法对差异显著性P值进行校正,并将FDR作为差异表达基因筛选的关键指标。参照GO数据库,将差异表达基因按照参与的生物学过程、细胞组成和分子功能进行分类;参照KEGG数据库分析差异表达基因参与的代谢途经,并筛选出与蜡粉合成代谢相关的基因;对差异表达基因的转录因子进行预测分析。
2 结果与分析
2. 1 转录组测序数据组装结果
本研究获得的原始数据经过测序质量控制和数据分析后,共得到44.68 Gb Clean data,GC含量在45%~47%,Q30碱基百分比超过92.92%,表明测序质量较好。经De novo拼接组装,共得到164914条Transcripts和41244条Unigenes,N50长度分别为1954和1847 bp(表1),表明数据组装完整性较高,可进行后续生物信息学分析。对组装的基因片段长度进行分析,结果发现Transcripts和Unigenes中1000~2000 bp所占比例最高,分别为37.68%和32.33%。
2. 2 Unigene功能注释结果
使用BLAST和HMMER将获得的Unigenes序列分别与Nr、Swiss-Prot、GO、COG、KOG、KEGG、Pfam数据库进行比对,结果发现有36230条Unigenes(≥300 bp)获得注释信息,占比87.8%。在获得注释的Unigenes中,在Nr数据库注释的Unigenes最多,为35971条,其次是GO数据库,为27966条,再次是SWISS-PROT数据库,为26540条,而在KEGG数据库注释的Unigenes最少,有8566条(表2)。在获得注释的Unigenes中,长度为300~1000 bp的Unigenes数为14999条,长度≥1000 bp的Unigenes有21231条(表2)。
2. 3 差异表达基因筛选及功能分析结果
2. 3. 1 差异表达基因的筛选 采用DESeq2进行样品组间差异表达分析,以|log2FC|≥1且FDR<0.05作为筛选标准获得差异表达基因,并进行层次聚类分析。将表达行为相同或相似的基因进行归类,获得差异基因聚类图谱(图2和图3)。在WT20和MT20样本的41244条Unigenes中,差异表达基因共8685个,其中上调基因5747个,下调基因2938个,这些基因是后续研究的重点。
2. 3. 2 差异表达基因的功能注释 将筛选到的8685个差异表达基因与数据库进行比对,共有8038个基因被注释,其中6095个基因注释到GO数据库,1671个基因注释到KEGG数据库,2066个基因注释到COG数据库,3866个基因注释到KOG数据库,5220个基因注释到Pfam数据库,6059个基因注释到Swiss-Prot数据库,8020个注释到Nr数据库(表3)。
2. 3. 3 差异表达基因转录因子注释 在8038个被注释的差异表达基因中,有2580个被注释为转录因子(表3),分布在75個转录因子家族中(图4),其中,注释为MYB家族的基因最多,为235个;富集基因数为100~200个的转录因子家族有7个:AP2/ERF(162个)、B3(107个)、C2C2(131个)、C2H2(118个)、NAC(137个)、WRKY(105个)和bHLH(119个);富集基因数为50~99个的转录因子家族有6个,富集基因数为0~49个的转录因子家族有61个;注释到其他转录因子家族的基因有120个。
2. 3. 4 差异表达基因的GO功能注释分析结果
6095个差异表达基因注释到细胞组分(Cell component)、分子功能(Molecular function)和生物学过程(Biological process)三大类别的52个功能分类。其中,共有4710个基因富集到细胞组分中,其中富集程度最高的是细胞(Cell),为4576个基因,其次为细胞部件(Cell part),为4562个基因;共有4497个基因富集到分子功能,富集程度最高的是结合(Binding),为3183个基因;共有4771个基因被富集到生物学过程,其中富集程度最高的是细胞过程(Cellular process),为3252个基因,其次是代谢过程(Metabolic process),为3237个基因。
2. 3. 5 差异表达基因的KEGG代谢通路富集分析结果 KEGG是系统分析基因功能和基因组信息的数据库。对差异表达基因的代谢通路进行分析有助于进一步解读基因的功能。将获得的8685个显著差异表达基因与KEGG数据库进行比对,结果(图6)发现,有1671个显著差异表达基因富集到代谢(Meta-bolism)、遗传信息处理(Genetic information proces-sing)、环境信息处理(Environmental information processing)、细胞过程(Cellular processes)、人类疾病(Human diseases)和有机系统(Organismal systems)六大类的21个功能分类,共138条代谢通路。其中显著富集通路有9条(FDR≤0.05),由于P越小,富集程度越高,因此最显著富集通路是硫甙生物合成(Glucosinolate biosynthesis),P为2.3948e-07;富集基因最多的是次生代谢物的生物合成(Biosynthesis of secondary metabolites),为245个基因,占19.25%,其次是植物信号转导(Plant hormone signal transduction),为110个基因,占8.64%(表4)。BF9E10E1-A8E8-4AA4-96C2-0B323DA83DF0
2. 3. 6 脂肪酸和蜡粉合成相关基因表达分析结果
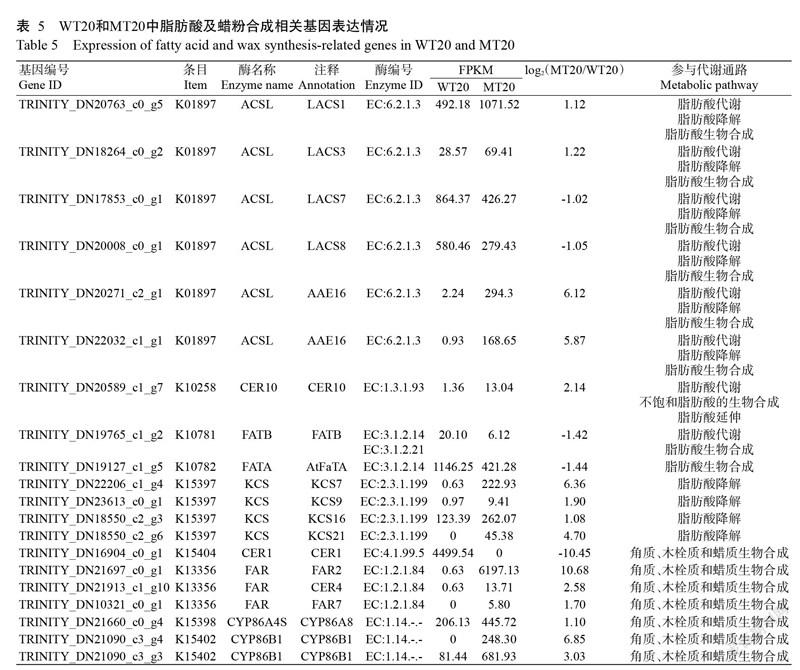
拟南芥蜡质的生物合成一般发生于表皮细胞内,具体过程为含有16~18个C的酰基载体蛋白(C16-C18 acyl-acyl carrier proteins,ACP)前体在质体中被合成后便被酰基载体蛋白硫酯酶(Fatty acyl-ACP thioesterases, FATA and FATB)水解生成含有16~18个C的脂肪酸,后经长链酰基辅酶A合成酶(Long chain acylcoenzyme A synthetases, LACS)催化合成胞质酰基辅酶A后运输到内质网中(Bonaventure et al.,2003)。根据上述蜡粉生物合成过程,本研究重点分析脂肪酸代谢(Fatty acid metabolism)、脂肪酸降解(Fatty acid degradation)、脂肪酸生物合成(Fatty acid biosynthesis)、不饱和脂肪酸生物合成(Biosynthesis of unsaturated fatty acids)、脂肪酸延伸(Fatty acid elongation)及角质、木栓质和蜡质生物合成(Cutin,suberine and wax biosynthesis)通路。根据KEGG数据库的功能释义和已知研究报道对青花菜蜡粉合成相关差异表达基因进行分析,结果如表5所示。有6个基因(TRINITY_DN20763_c0_g5、TRINITY_ DN18264_c0_g2、TRINITY_DN17853_c0_g1、TRINITY_DN20008_c0_g1、TRINITY_DN20271_c2_g1和TRINITY_DN 22032_c1_g1)注释到K01897,对应酶名称为ACSL(酰基辅酶A合成酶长链家族成员),其在数据库中分别注释为LACS1(长链酰基辅酶A合成酶)、LACS3、LACS7、LACS8和AAE16(酰基活化酶16)。TRINITY_DN20589_c1_g7注释到K10258,对应酶为CER10(ECERIFERUM 10)。TRINITY_ DN19765_c1_g2注释到K10781,其在数据库中注释为FATB(脂肪酰基ACP硫酯酶B)。TRINITY_ DN19127_c1_g5注释到K10782,对应的酶为FATA(脂肪酰基ACP硫酯酶A),其在数据库中注释为AtFaTA(拟南芥硫脂酶)。4个差异表达基因(TRINITY_DN22206_c1_g4、TRINITY_DN23613_c0_g1、TRINITY_DN18550_c2_g3和TRINITY_DN18550_ c2_g6)注释为KCS(酮酰辅酶A合酶),其在数据库中注释分别为KCS7、KCS9、KCS16和KCS21。
7个差异表达基因注释到角质、木栓质和蜡质生物合成通路,其中TRINITY_DN16904_c0_g1注释到K15404,注释为无蜡粉基因CER1(ECERIFERUM 1);3个基因(TRINITY_DN21697_c0_g1、TRINITY_ DN21913_c1_g10和TRINITY_DN10321_c0_g1)注释到K13356,均注释为FAR(脂肪酰辅酶A还原酶),但在数据库中分别注释为FAR2、CER4和FAR7;TRINITY_DN21660_c0_g4注释到K15398,注释为CYP86A4S(细胞色素P450 86A8),在数据库中注释为CYP86A8;TRINITY_DN21090_c3_g4和TRINITY_DN21090_c3_g3注释为CYP86B1(细胞色素P450 86B1)(表5)。
根据WT20和MT20的FPKM值(表5)可知,有5个差异表达基因TRINITY_DN17853_c0_g1、TRINITY_ DN20008_c0_g1、TRINITY_DN19765_c1_g2、TRINITY_DN19127_c1_g5和TRINITY_DN16904_c0_g1表达下调,其余均上调表达。在WT20中,有3个差异表达基因TRINITY_DN18550_c2_g6、TRINITY_ DN10321_c0_g1和TRINITY_DN21090_c3_g4的FPKM值为0,在MT20中,TRINITY_ DN16904_c0_g1的FPKM值为0。TRINITY_DN21697_c0_g1的log2 (MT20/WT20)绝对值最高为10.68,其次是TRINITY_DN16904_c0_g1为10.45,最小的是TRINITY_ DN17853_c0_g1为1.02。
3 讨论
本研究将野生型青花菜和蜡粉缺失实变体的转录组测序结果与Nr、Nt、KEGG、Pfam、KOG/COG、Swiss-Prot和GO数据库进行比对,结果发现有36230条(占比87.8%)Unigenes得到注释,未获得注释的基因有5014条,可能是由于测序片段过短、获得的特殊基因在数据库中的注释信息缺乏等原因,与在青花菜(张振超等,2018)、花椰菜(林珲等,2018)、油菜(吴宁柔等,2018)等植物转录组结果相似。对青花菜样本的表达谱进行比对,获得差异表达基因8685个,共有2580个差异表达基因被注释到75个转录因子家族中,其中注释为MYB家族的基因最多,为235个,占9.10%,表明MYB家族转录因子参与了调控蜡粉合成。Dubos等(2010)的研究结果也证实转录因子如MYB可通过独立或协同的方式调控某些基因的表达从而实现蜡粉的生物合成。
植物表皮蜡粉主要是由特长链饱和脂肪酸的衍生物组成,还包括萜类化合物和次生代谢物等(Bourdenx et al.,2011),其合成过程需要多种酶的共同参与,如酰基载体蛋白硫脂酶(FATA和FATB)、长链酰基辅酶A合成酶(LACS)、β-酮脂酰CoA合成酶(KCS)、β-酮脂酰CoA还原酶(KCR)、β-羟酰基辅酶A水解酶(HCD)和烯酰基 CoA还原酶(ECR)、脂肪酸延伸酶(FAE)等(Kunst and Samuels,2003;Bourdenx et al.,2011)。本研究根据青花菜野生型(WT20)和蜡粉缺失突变体(MT20)的转录组信息与KEGG数据库比对结果,重点分析脂肪酸代谢、脂肪酸降解、脂肪酸生物合成、不饱和脂肪酸生物合成、脂肪酸延伸及角质、木栓质和蜡质生物合成6个通路,并参照GO、Nr、Nt、Pfam、KOG/COG、Swiss-Prot、KEGG的注释信息进行分析,结果发现有6个差异表达基因注释为ACSL,其中4条基因在数据库中注释为LACS1、LACS3、LACS7和LACS8,2条基因注释为AAE16。LACS1、LACS3、LACS7和LACS8是长链脂肪酸合成关键酶,但行使的功能各不相同。LACS1编码长链酰基辅酶 A 合成酶,可催化合成具有活性的脂酰辅酶A,并参与蜡粉代谢(Lü et al.,2009);LACS3参与脂质的跨膜运输和细胞内运输(Pulsifer et al.,2012);LACS7编码氧化物酶体LACS蛋白,参与脂肪酸的β-氧化,可催化大多数脂肪酸(Fulda et al.,2002);LACS8激活长链脂肪酸以合成细胞脂质和通过β-氧化降解(田亚英等,2018)。AAE16为酰基活化酶,可能参与脂肪酸向酰基载体蛋白的活化。此外,本研究中有1个差异表达基因(TRINITY_DN20589_c1_g7)在数据库中注释为CER10。CER10为烯酰辅酶 A 还原酶,参与角质层蜡、贮藏脂和鞘脂代谢所需的所有超长链脂肪酸(VLCFA)延伸反应,该基因突变体表现出异常的器官形态和茎光泽度(Rashotte et al.,2001)。本研究中有2个差异表达基因(TRINITY_DN19765_c1_g2和TRINITY_DN19127_c1_g5)分别注释FATB和FATA。FATB和FATA可催化16~18个C的ACP形成16~18个C的脂肪酸,从而参与植物蜡质生物合成过程,FATB功能缺失的拟南芥叶片和茎部的蜡质含量会降低约20%和50% (Bonaventure et al.,2003)。本研究中4个差异表达基因注释为KCS,其在数据库中分别注释为KCS7、KCS9、KCS16和KCS21。前人研究发现,KCS催化丙二酰CoA和长链酰基CoA的聚合反应,具有严格的底物特异性,目前已在拟南芥基因组中注释了21个KCS基因(Joubès et al.,2008),现已有7个KCS基因的功能及其编码蛋白的底物特异性被确认,其中已确认与蜡质的生物合成相关基因包括KCS1、KCS2、KCS6、KCS9和KCS20(Franke et al.,2010;Lee et al.,2010;Kim et al.,2013),其他KCS的功能未见报道。本研究发现,7个差异表达基因注释到角质、木栓质和蜡质生物合成通路,其中与蜡质合成相关的酶包括CER1、FAR、CYP86A4S和CYP86B1。其中,CER1基因编码醛脱羰酶,在烷烃生物合成通路中催化醛脱羰形成烷烃,其突变体中烷烃的含量显著减少,而过表达 CER1 基因时,拟南芥中烷烃的含量会增加,器官呈现蜡粉合成减少(Bernard et al.,2012;Oshima et al.,2013)。目前在擬南芥中已检测出8个FAR编码基因,其中只有CER4基因参与了蜡质的合成(Doan et al.,2009),Liu等(2018)也研究确定甘蓝蜡粉缺失突变体LD10GL是由拟南芥CER4的同源基因Bol013612突变造成的。CYP86A4S和CYP86B1是细胞色素P450单加氧酶CYP第86家族中A和B亚族成员,均在木栓质脂肪族成分的ω-羟基化中发挥重要作用(Vincent et al.,2009;Bruckhoff et al.,2016)。由上述推断蜡粉合成过程中相关酶基因的差异表达是调控青花菜蜡粉合成的关键。BF9E10E1-A8E8-4AA4-96C2-0B323DA83DF0
本研究對WT20和MT20植株中脂肪酸及蜡粉合成相关基因的表达情况进行分析,结果发现20个差异表达基因的FPKM值在WT20和MT20中存在明显差异,其中3个差异表达基因(TRINITY_DN18550_ c2_g6、TRINITY_DN10321_c0_g1TRINITY_DN210 90_c3_g4)的FPKM值在WT20中为0,1个差异表达基因(TRINITY_DN16904_c0_g1)的FPKM值在MT20值为0,差异极显著,存在显著和极显著差异的差异表达基因在野生型和蜡粉缺失突变体中表达具有特异性,可作为下一步研究的重点。
4 结论
转录因子MYB家族在调控青花菜蜡粉合成中发挥重要作用。蜡粉合成过程中相关酶基因的差异表达是调控青花菜蜡粉合成的关键,尤其是野生型和蜡粉缺失突变体中特异性表达的差异表达基因,可作为后续研究青花菜花球表面蜡粉形成分子机制的对象。
参考文献:
林珲,薛珠政,李永平,李大忠,刘建汀,朱海生,温庆放. 2018. 青梗花椰菜和白梗花椰转录组分析[J]. 核农学报,32(9):1708-1720. [Lin H,Xue Z Z,Li Y P,Li D Z,Liu J T,Zhu H S,Wen Q F. 2018. Transcriptome analysis of the green stalk and the white stalk of cauliflower[J]. Journal of Nuclear Agricultural Sciences,32(9):1708-1720.] doi:10.11869/j.issn.100-8551.2018.09.1708.
田亚英,傅念,吴清. 2018. 长链脂酰辅酶A合成酶及其调控因素的研究及进展[J]. 医学研究杂志,47(10):13-15. [Tian Y Y,Fu N,Wu Q. 2018. Research and progress of long-chain acyl-CoA synthase and its regulatory factors[J]. Journal of Medical Research,47(10):13-15.] doi:10.11969/j.issn.1673-548X.2018.10.005.
王立山,丁兵,李玉花,张旸. 2018. 植物表皮蜡质合成转运调控相关基因与干旱响应的研究进展[J]. 园艺学报,45(9):1831-1843. [Wang L S,Ding B,Li Y H,Zhang Y. 2018. Reaserch progress of plant cuticular wax biosynthesis,export and regulation related genes responsed to drought[J]. Acta Horticulturae Sinica,45(9):1831-1843.] doi:10.16420/j.issn.0513-353x.2017-0548.
吴宁柔,韦云婷,官梅. 2018. 甘蓝型油菜不同花色转录组分析[J]. 分子植物育种,16(22):7234-7240. [Wu N R,Wei Y T,Guan M. 2018. Transcriptome analysis of Brassica napus with different flower colors[J]. Molecular Plant Breeding,16(22):7234-7240.] doi:10.13271/j.mpb.016. 007234.
张曦,王秋实,邹春蕾,刘志勇,王一衡,冯辉. 2013. 大白菜花茎蜡粉基因的遗传分析与初步定位[J]. 分子植物育种,11 (6):804-808. [Zhang X,Wang Q S,Zou C L,Liu Z Y,Wang Y H,Feng H. 2013. Genetic analysis and preliminary mapping of wax gene on stem in Chinese cabbage[J]. Molecular Plant Breeding,11(6):804-808.] doi:10. 3969/mpb.011.000804.
张振超,潘永飞,戴忠良,秦文斌,山溪. 2021. 青花菜越冬设施高产栽培技术[J]. 长江蔬菜,(11):24-26. [Zhang Z C,Pan Y F,Dai Z L,Qin W B,Shan X. 2021. High-yield cultivation techniques of broccoli overwintering in facility[J]. Journal of Changjiang Vegetables,(11):24-26.]
张振超,姚悦梅,毛忠良,孙国胜,秦文斌,戴忠良. 2018. 基于高通量测序的青花菜早期发育小孢子转录组分析与基因功能注释[J]. 核农学报, 32(5):848-855. [Zhang Z C,Yao Y M,Mao Z L,Sun G S,Qin W B,Dai Z L. 2018. Transcriptome analysis and gene function annotation of early developmental broccoli microspores based on high-throughput sequencing technology[J]. Journal of Nuclear Agricultural Sciences,32(5):848-855.] doi: 10.11869/j.issn.100-8551.2018.05.0848.BF9E10E1-A8E8-4AA4-96C2-0B323DA83DF0
Bernard A,Domergue F,Pascal S,Jetter R,Renne C,Faure J D,Haslam R P,Napier J A,Lessire R,Joubes J. 2012. Reconstitution of plant alkane biosynthesis in yeast de-monstrates that Arabidopsis ECERIFERUM1 and ECERIFERUM3 are core components of a very-long-chain alkane synthesis complex[J]. The Plant Cell,24(7):3106-3118. doi:10.1105/tpc.112.099796.
Bonaventure B,Salas J J,Pollard M R,Ohlrogge J B. 2003. Disruption of the FATB gene in Arabidopsis demonstrates an essential role of saturated fatty acids in plant growth[J]. The Plant Cell,15(4):1020-1033. doi:10.1105/tpc. 008946.
Bourdenx B,Bernard A,Domergue F,Pascal S,Léger A,Roby D,Pervent M,Vile D,Haslam R,Napier J A,Lessire R,Joubès J. 2011. Overexpression of Arabidopsis ECERIFERUM1 promotes wax very-long-chain alkane biosynthesis and influences plant response to biotic and abiotic stresses[J]. Plant Physiology,156(1):29-45. doi:10.1104/ pp.111.172320.
Bruckhoff V,Haroth S,Feussner K,Konig S,Brodhun F,Feussner I. 2016. Functional characterization of CYP94-genes and identification of a novel jasmonate catabolite in flowers[J]. PLoS One,11(7):e0159875. doi:10.1371/journal.pone.0159875.
Wettstein D V,Chua N H. 1987. Plant molecular biology[M]. New York :Plenum Press,305-314.
Chen X B,Goodwin M,Boroff V L,Liu X L,Jenks M A. 2003. Cloning and characterization of the WAX2 gene of Arabidopsis involved in cuticle membrane and wax production[J]. The Plant Cell,15(5):1170-1185. doi:10. 1105/tpc.010926.
Doan T T,Carlsson A S,Hamberg M,Bülow L,Stymne S,Olsson P. 2009. Functional expression of five Arabidopsis fatty acyl-CoA reductase genes in Escherichia coli[J]. Journal of Plant Physiology,166(8):787-796. doi:10.1016/ j.jplph.2008.10.003.
Domínguez E,Heredia-Guerrero J A,Heredia A. 2011. The biophysical design of plant cuticles:An overview[J]. New Phytologist,189(4):938-949. doi:10.1111/j.1469-8137. 2010.03553.
Dubos C,Stracke R,Grotewold E,Weisshaar B,Martin C,Lepiniec L. 2010. MYB transcription factors in Arabidopsis[J]. Trends in Plant Science,15(10):573-581. doi:10. 1016/j.tplants.2010.06.005.
Franke R,Hfer R,Briesen I,Emsermann M,Schreiber L. 2010. The DAISY gene from Arabidopsis encodes a fatty acid elongase condensing enzyme involved in the biosynthesis of aliphatic suberin in roots and the chalaza-micropyle region of seeds[J]. The Plant Journal,57(1):80-95. doi:10.1111/j.1365-313X.2008.03674.x.
Fulda M,Shockey J,Werber M,Wolter F P,Heinz E. 2002. Two long-chain acyl-CoA synthetases from Arabidopsis thaliana involved in peroxisomal fatty acid beta-oxidation[J]. The Plant Journal,32(1):93-103. doi:10.1046/j.1365- 313x.2002.01405.xBF9E10E1-A8E8-4AA4-96C2-0B323DA83DF0
Jenks M A,Eigenbrode S D,Lemieux B. 2002. Cuticular waxes of Arabidopsis[J]. Arabidopsis Book,1:e0016. doi:10. 1199/tab.0016.
Joubès J,Raffaele S,Bourdenx B,Garcia C,Laroche-Traineau J,Moreau P,Domergue F,Lessire R. 2008. The VLCFA elongase gene family in Arabidopsis thaliana:Phylogene-tic analysis, 3D modelling and expression profiling[J]. Plant Molecular Biology,67(5):547. doi:10.1007/s11103- 008-9339-z.
Kim J,Jung J H,Lee S B,Go Y S,Kim H J,Cahoon R,Markham J E,Cahoon E B,Suh M C. 2013. Arabidopsis 3-ketoacyl-coenzyme a synthase9 is involved in the synthesis of tetracosanoic acids as precursors of cuticular waxes,suberins, sphingolipids,and phospholipids[J]. Plant Physiology,162(2):567-580. doi:10.1104/pp.112.210450.
Kunst L,Samuels A L. 2003. Biosynthesis and secretion of plant cuticular wax[J]. Progress in Lipid Research,42(1):51-80. doi:10.1016/s0163-7827(02)00045-0.
Lee S B,Jung S J,Go Y S,Kim H U,Kim J K,Cho H J,Park O K,Suh M C. 2010. Two Arabidopsis 3-ketoacyl CoA synthase genes, KCS20 and KCS2/DAISY, are functionally redundant in cuticular wax and root suberin biosynthesis,but differentially controlled by osmotic stress[J]. The Plant Journal,60(3):462-475. doi:10.1111/j. 1365-313X.2009.03973.x.
Liu D M,Tang J,Liu Z Z,Dong X,Zhuang M,Zhang Y Y,Lü H H,Sun P T,Liu Y M,Li Z S,Ye Z B,Fang Z Y,Yang L M. 2017. Cgl2 plays an essential role in cuticular wax biosynthesis in cabbage(Brassica oleracea L. var. capitata)[J]. BMC Plant Biology,17(1):223. doi:10. 1186/s12870-017-1162-8.
Liu D M,Dong X,Liu Z Z,Tang J,Zhuang M,Zhang Y Y,Lv H H,Liu Y M,Li Z S,Fang Z Y,Yang L M. 2018. Fine mapping and candidate gene identification for wax biosynthesis locus, BoWax1 in Brassica oleracea L. var. capitata[J]. Frontiers in Plant Science,9:309. doi:10.3389/fpls.2018.00309.
Lü S,Song T,Kosma D K,Parsons E P,Rowland O,Jenks M A. 2009. Arabidopsis CER8 encodes LONG-CHAIN ACYL COA SYNTHETASE 1(LACS1) and has overlapping functions with LACS2 in plant wax and cutin synthesis[J]. The Plant Journal,59(4):553-564. doi:10.1111/ j.1365-313X.2009.03892.
Oliveira A F,Meirelles S T,Salatino A. 2004. Epicuticular waxes from caatinga and cerrado species and their efficiency against water loss[J]. Anais da Academia Brasileira de Ciências,75(4):431-439. doi:10.1590/s0001-3765200 3000400003.
Oshima Y,Shikata M,Koyama T,Ohtsubo N,Ohme-Takagi M M. 2013. MIXTA-Like transcription factors and WAX INDUCER1/SHINE1 coordinately regulate cuticle deve-lopment in Arabidopsis and Torenia fournieri[J]. The Plant Cell,25(5):1609-1624. doi:10.1105/tpc.113.110783.BF9E10E1-A8E8-4AA4-96C2-0B323DA83DF0
Pruitt R E,Vielle-Calzada J P,Ploense S E,Grossniklaus U,Lolle S J. 2000. FIDDLEHEAD, a gene required to suppress epidermal cell interactions in Arabidopsis, encodes a putative lipid biosynthetic enzyme[J]. Proceedings of the National Academy of Sciences,97(3):1311-1316. doi:10.1073/pnas.97.3.1311.
Pu Y Y,Gao J,Guo Y L,Liu T T,Zhu L X,Xu P,Yi B,Wen J,Tu J X,Ma C Z,Fu T D,Zou J T,Shen J X. 2013. A novel dominant glossy mutation causes suppression of wax biosynthesis pathway and deficiency of cuticular wax in Brassica napus[J]. BMC Plant Biology,13:215-228. doi:10.1186/1471-2229-13-215.
Pulsifer I P,Kluge S,Rowland O. 2012. Arabidopsis LONG-CHAIN ACYL-COA SYNTHETASE 1(LACS1), LACS2,and LACS3 facilitate fatty acid uptake in yeast[J]. Plant Physiology and Biochemistry,51:31-39. doi:10.1016/j.plaphy.2011.10.003.
Pyee J,Kolattukudy P E. 1995. The gene for the major cuticular wax-associated protein and three homologous genes from broccoli(Brassica oleracea) and their expression patterns[J]. The Plant Journal,7(1):49-59. doi:10.1046/j.1365-313x.1995.07010049.
Rashotte A M,Jenks M A,Feldmann K A. 2001. Cuticular waxes on eceriferum mutants of Arabidopsis thaliana[J]. Phytochemistry,57(1):115-123. doi:10.1016/s0031-9422(00)00513-6.
Saet B L,Mi C S. 2015. Advances in the understanding of cuticular waxes in Arabidopsis thaliana and crop species[J]. Plant Cell Reports,34(4):557-572. doi:10.1007/s00299-015-1772-2.
Vincent C,Patrik D,Irène B,Denise M,Hubert S,Lukas S,Rochus F,Franck P. 2009. CYP86B1 is required for very long chain ω-hydroxyacidand a,ω-dicarboxylic acid synthesis in root and seed suberin polyester[J]. Plant Physio-logy,150:1831-1843. doi:10.1104/pp.109.141408.
Zhang X,Liu Z,Wang P,Wang Q,Yang S,Feng H. 2013. Fine mapping of BrWax1, a gene controlling cuticular wax biosynthesis in Chinese cabbage(Brassica rapa L. ssp. pekinensis)[J]. Molecular Breeding,32(4):867-874. doi:10.1007/s11032-013-9914-0.
(責任编辑 陈 燕)BF9E10E1-A8E8-4AA4-96C2-0B323DA83DF0
