基于线粒体Cytb和COⅠ基因的洞庭湖区养殖克氏原螯虾遗传多样性分析
2022-06-10欧琳张余陈晓芳周刚黄宇宏陈蕾肖调义刘巧林
欧琳 张余 陈晓芳 周刚 黄宇宏 陈蕾 肖调义 刘巧林
(湖南农业大学 湖南省特色水产资源利用工程技术研究中心,湖南长沙 410128)
克氏原螯虾(Procambarusclarkii)俗称小龙虾,隶属于节肢动物门、甲壳纲、十足目、螯虾科、原螯虾属,原产于美国,现已成为我国重要的淡水养殖虾类[1-5]。该虾肉质鲜美,营养丰富,深受市场欢迎,是近几年夏季消费的“网红”产品[6-7]。湖南省是我国克氏原螯虾的主要养殖省份之一,小龙虾养殖已成为当地产值超百亿元人民币的特色新产业,其中洞庭湖区的益阳、岳阳、常德等3市为主要产区。克氏原螯虾种质资源的研究对其保护和利用、提高养殖产量、良种选育等具有重要意义[8]。开展对克氏原螯虾群体遗传结构及遗传变异的相关研究,有助于丰富其种质资源多样性,保证相关产业的可持续发展。
近年来,分子生物学技术不断发展,不仅被国内广大学者广泛应用于水生生物遗传学分析[9-13],还被国外学者大量应用于水产动物遗传育种研究[14-18]。动物的线粒体DNA分子量较小,结构相对稳定,进化速度较快,遵循严格的母系遗传,是进行群体研究的理想材料[19]。其中,细胞色素b(Cytb)基因和细胞色素C氧化酶亚基Ⅰ(COⅠ)基因的长度和进化速率适宜,常被用于群体遗传学研究[20-23]。本研究基于以上两种基因,对洞庭湖区岳阳华容县和益阳南县2处主养殖区共11个采集点的克氏原螯虾群体进行了遗传多样性分析,以期丰富克氏原螯虾种质资源的研究数据,为洞庭湖区克氏原螯虾的综合养殖和育种研究提供参考。
1 材料和方法
1.1 样本采集
样本采集点参照当地畜牧水产局提供的养殖小龙虾大户或合作社名单确定。在岳阳市华容县选取6个采集点,共计180个样本;益阳市南县选取5个采集点,共计150个样本。样本个体健壮,生长良好,附肢齐全,颜色呈暗红或黑红,体表光泽度好。剪取样本的肌肉组织,置于1.5 mL离心管中,-80 ℃冷冻保存。
1.2 DNA提取
根据Tissue DNA Kit(D3396-01)试剂盒上的说明提取样本DNA。
1.3 Cytb和COⅠ基因扩增与测序
反应引物及序列见表1。PCR反应体系为:1 μL DNA,0.5 μL上游引物,0.5 μL下游引物,10 μL 2×TaqDNA poly-merase Mix,8 μL ddH2O。PCR反应程序:94 ℃预变性5 min;94 ℃变性1 min,57 ℃退火45 s,72 ℃延伸1 min,共35次循环;72 ℃延伸5 min,于4 ℃保存。

表1 基因扩增引物
将得到的产物用加入1%琼脂糖凝胶液的凝胶板通电进行电泳(60~100 V,40 min),电泳完毕后,将凝胶进行染色,用凝胶成像系统拍照保存。
将含有目的条带的产物送到武汉奥科鼎盛生物科技有限公司进行双向测序。
1.4 数据处理
测序所得各片段用GENEIOUS 9.13软件进行序列的双向拼接并校对,排查删除错误片段,截取等长的有效片段,导出fasta文件用于后期单倍型分析。通过DnaSp 5.0软件进行遗传多样性参数分析。通过MEGA 7.0软件采用邻接法(neighbor-joining,NJ)构建系统进化树,同时对11个采集点样本之间的遗传距离进行简要分析。
2 结果
2.1 Cytb基因序列分析
2.1.1 碱基组成
经质量检测,在所提取的克氏原螯虾基因组DNA中,华容县采集点获得179个有效样本,益阳南县采集点获得149个有效样本。PCR扩增后,经双向测序,测序结果经双向拼接并校对,排查删除错误片段后,截取等长的有效Cytb基因序列为762 bp,碱基平均含量由高到低依次为:T(34.08%)、A(31.08%)、C(18.51%)、G(16.33%),其中A+T的含量(65.16%)明显高于G+C的含量(34.84%)。这说明11个采集点的克氏原螯虾在Cytb基因的碱基组成上表现出一定的偏向性。
2.1.2 遗传多样性参数
11个采集点Cytb基因的遗传多样性参数见表2。结果表明,11个采集点的克氏原螯虾的遗传多样性指数略有差异,华容县6个采集点克氏原螯虾的遗传多样性指数要高于南县5个采集点的,总体表现出较低的单倍型多样性和较低的核苷酸多样性。
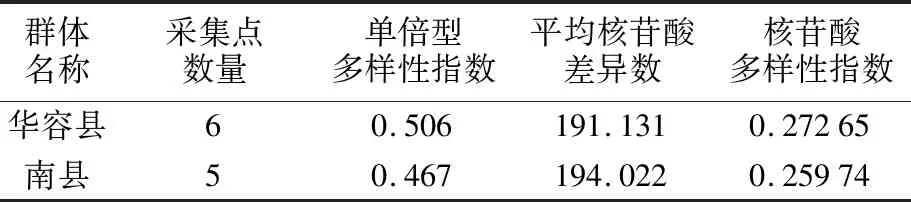
表2 11个采集点Cytb基因的遗传多样性参数
2.1.3 单倍型分布及系统进化树
在不包含空白与缺失位点的检测下,11个采集点共检测出5个单倍型(见表3)。其中,南县5个采集点的克氏原螯虾群体存在3个单倍型,而华容县6个采集点的克氏原螯虾群体存在4个单倍型。结果显示,Hap1、Hap3为11个采集点所共有,Hap2、Hap4为华容县6个采集点所特有,Hap5为南县5个采集点所特有,单倍型以Hap1(62.2%)为主。

表3 11个采集点Cytb基因单倍型在群体中的分布
基于Kimura 2-Parameter模型,以邻接法构建克氏原螯虾11个采集点Cytb基因的单倍型系统进化树(见图1),设置检验次数为1 000次。结果显示,Hap2和Hap3最先聚为1支,然后与Hap1聚为1支,接着与Hap4聚为1支,最后和Hap5聚为1支。结果表明,11个采集点的克氏原螯虾群体聚类在一起,不存在明显的遗传分化。
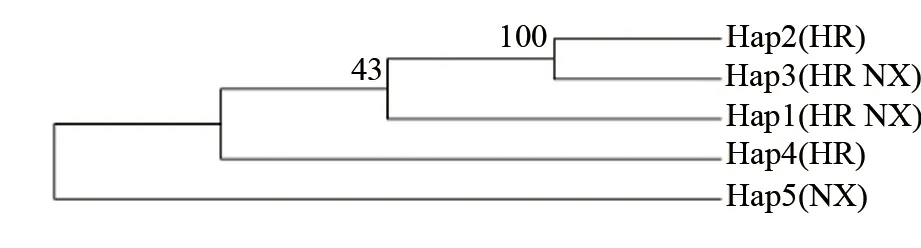
注:HR表示华容县样本,NX表示南县样本。
2.1.4 遗传距离
11个采集点Cytb基因的遗传距离见表4。结果显示,华容县6个采集点的克氏原螯虾的种内遗传距离(0.551)大于11个采集点间的遗传距离(0.535),而南县5个采集点的克氏原螯虾的种内遗传距离(0.519)相较于11个采集点间的遗传距离又偏小。试验表明,11个采集点之间不存在明显的遗传分化(P>0.05)。
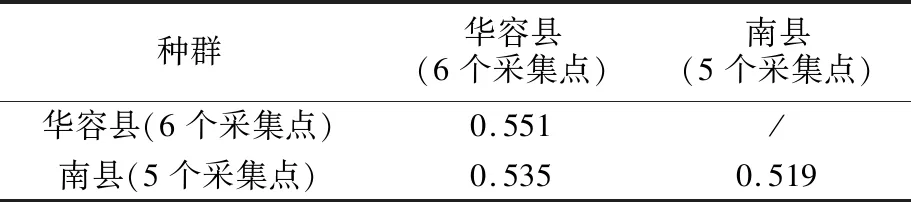
表4 11个采集点Cytb基因的遗传距离
2.2 COⅠ基因序列分析
2.2.1 碱基组成
PCR扩增产物经双向测序、拼接校对,排查删除错误片段后,截取等长有效COⅠ基因序列为847 bp,碱基平均含量由高到低依次为:T(40.00%)、A(26.90%)、G(18.55%)、C(13.50%),其中A+T的含量(66.90%)明显高于G+C的含量(32.05%)。这表明11个采集点的克氏原螯虾COⅠ基因存在着明显的T/A碱基偏向。
2.2.2 遗传多样性参数
11个采集点COⅠ基因的遗传多样性参数见表5。结果表明,11个采集点COⅠ基因的遗传多样性参数略有差异,华容县6个采集点克氏原螯虾的遗传多样性高于南县5个采集点的克氏原螯虾,总体表现出较低的单倍型多样性和较低的核苷酸多样性。

表5 11个采集点COⅠ基因的遗传多样性参数
2.2.3 单倍型分布及系统进化树
在不包含空白与缺失位点的检测下,11个采集点共检测出9个单倍型(见表6)。其中南县5个采集点的克氏原螯虾群体存在5个单倍型,而华容县6个采集点的克氏原螯虾群体存在6个单倍型。结果显示,Hap1、Hap3、Hap7、Hap8、Hap9为11个采集点所共有,Hap4、Hap5为华容县6个采集点所特有,Hap2、Hap6为南县5个采集点所特有,单倍型以Hap1(86.1%)为主。

表6 11个采集点COⅠ基因单倍型在群体中的分布
基于Kimura 2-Parameter模型以邻接法构建克氏原螯虾11个采集点COⅠ基因的单倍型系统进化树(见图2),检验次数设置为1 000次。结果显示,克氏原螯虾分为2大支,Hap1至Hap6聚为1大支,Hap7、Hap8和Hap9形成另1个大分支。结果表明,11个采集点的克氏原螯虾群体之间并无明显的遗传分化。

注:HR表示华容县样本,NX表示南县样本。
2.2.4 遗传距离
11个采集点COⅠ基因的遗传距离见表7。结果表明,华容县6个采集点克氏原螯虾的种内遗传距离(0.122)大于11个采集点间的遗传距离(0.114),而南县5个采集点克氏原螯虾的种内遗传距离(0.099)相较于11个采集点间的遗传距离偏小。结果表明,11个采集点克氏原螯虾群体之间遗传分化不显著。
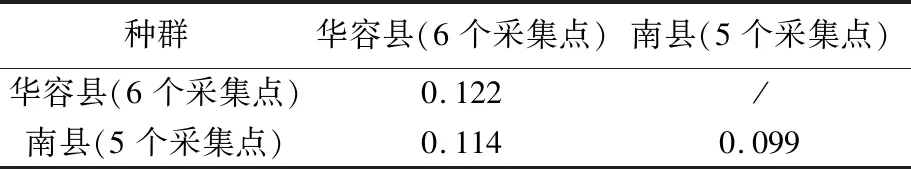
表7 11个采集点COⅠ基因的遗传距离
3 讨论
3.1 序列特征
本研究从线粒体Cytb和COⅠ基因序列分析入手,探讨洞庭湖区养殖克氏原螯虾的遗传多样性。经基因组DNA提取、PCR扩增、序列测定、序列拼接和序列比对分析,最终获得的Cytb和COⅠ基因长度分别为762 bp和847 bp。在以上两种基因的碱基组成中,A和T的总含量均明显高于G和C的总含量。这表明克氏原螯虾线粒体基因序列的碱基组成成分并不均一,具有较强的偏向性。在其他十足目物种中也存在同样的规律[24-26]。导致这一现象的原因可能是基因长度和基因表达水平存在较大的差异。
3.2 遗传多样性
在遗传多样性参数中,单倍型多样性指数和核苷酸多样性指数常常被用来评价群体遗传多样性水平的高低[27-28]。本研究中,基于Cytb基因序列在华容县的6个采集点中,克氏原螯虾的单倍型多样性指数为0.506,核苷酸多样性指数为0.272 65;在南县的5个采集点中,克氏原螯虾的单倍型多样性指数为0.467,核苷酸多样性指数为0.259 74。基于COⅠ基因序列在华容县的6个采集点中,克氏原螯虾的单倍型多样性指数为0.258,核苷酸多样性指数为0.136 1;在南县的5个采集点中,克氏原螯虾的单倍型多样性指数为0.244,核苷酸多样性指数为0.122 3。上述数据表明,11个采集点的克氏原螯虾群体总体呈现出较低的单倍型多样性和较低的核苷酸多样性。这一结果与我国长江中下游流域克氏原螯虾群体遗传多样性的研究结果不一致[29-30],说明洞庭湖区养殖克氏原螯虾的种质来源比较单一。此外,根据Cytb基因序列得到的遗传多样性水平高于根据COⅠ基因序列得到的遗传多样性水平。这一现象的出现可能是因为Cytb基因相较于COⅠ基因变异速率快,这与其他的研究结果较为一致[31-32]。
在本研究中,单倍型系统进化树和遗传距离的结果均表明,洞庭湖区11个采集点克氏原螯虾群体的遗传分化并不显著,其共有单倍型数量较多,存在一定的基因交流。11个采集点克氏原螯虾的遗传结构不同表现在两个方面:(1)单倍型数量不同。基于Cytb基因序列的单倍型南县(5个采集点)有3个,华容县(6个采集点)有4个。基于COⅠ基因序列的单倍型南县(5个采集点)有5个,华容县(6个采集点)有6个;(2)优势单倍型占比差异较大。基于Cytb基因序列的优势单倍型为62.2%,基于COⅠ基因序列的优势单倍型为86.1%,这种差异导致洞庭湖区养殖克氏原螯虾遗传多样性不丰富。
遗传多样性越丰富,物种对环境的适应能力就越强,而遗传多样性降低对物种的生存极有可能造成不良的影响,甚至使其走向灭亡[33-35]。外来物种在入侵的过程中常常会经历遗传漂变或“瓶颈”效应,表现为遗传多样性偏低[36]。对于克氏原螯虾这类外来物种,可以考虑其来源地和引入地,通过多次引种或杂交,提高其遗传多样性。
