化肥减量和有机替代对潮土微生物群落分子生态网络的影响*
2022-06-09赵建宁杨殿林王丽丽修伟明
吴 宪,胡 菏,王 蕊,赵建宁,杨殿林,王丽丽,李 刚,修伟明†
(1. 农业农村部环境保护科研监测所农业农村部产地环境污染防控重点实验室,天津 300191;2. 农业农村部环境保护科研监测所天津市农业环境与农产品安全重点实验室,天津 300191)
华北平原是我国重要的农业耕作区,总占地面积为35 万km2,其中农业用地约为1 800 万hm2[1],占全国耕地面积的27%[2]。该地区常规耕作方式为冬小麦-夏玉米轮作[3],其中小麦产量占全国总产量的60%~80%,玉米产量占35%~40%[2]。作为粮食作物的“粮食”,化肥在粮食增产中的作用占40%~50%[4]。然而,近年来,为了满足日益增长的人口需求,大量化肥被施入农田以获得更高的作物产量[5],而化肥的施用量通常远高于作物的需求[6-7]。合理施肥可促进土壤生态系统的正向发展[8],但化肥的大量施用会使得资源和环境的矛盾日益凸显,致使包括土壤板结、土壤酸化、土壤污染和土壤生物多样性锐减在内的多种环境问题逐渐显现[9]。面对长期过量施用化肥对农业生态系统产生不利影响的严峻形势,减少化肥施用量并配合有机替代成为扭转上述不利局面的重要措施。有机添加不仅可为作物提供养分,而且可通过提高土壤有机质(SOM)含量来维持土壤肥力。作物秸秆和有机肥富含有机碳和作物生长所必需的营养元素,秸秆还田和有机肥部分替代不仅可节省化肥,而且可有效增加土壤有机碳含量,减少农业污染[10]。
化肥的不当施用可能会间接影响土壤微生物群落,这些微生物群落通过改变群落组成和多样性对环境条件的微小变化做出响应。诸多研究[11-12]表明有机物料的施用对土壤微生物群落产生积极作用,增强了相对于单施化肥或不施肥的微生物群落的抗干扰能力。土壤SOM 是评价土壤质量的重要指标,在调节大气CO2浓度和维持土壤生产力方面发挥着重要的作用[13]。为了提高土壤SOM 含量并提升土壤肥力,施用有机物料成为最有效的方法[14]。有机物料对土壤SOM 的积累尤为重要,并影响微生物群落组成。同样,微生物在土壤SOM 周转过程中起着关键作用。有研究[13]表明长期施用猪粪可增加土壤有机质含量,并改变微生物群落结构及关键类群。
近年来,分子生态网络分析因其可直观地展示微生物之间复杂的生态相互作用以及微生物对环境变化的响应而被广泛应用[15],其打破了在物种数量和多样性上进行微生物群落研究的局限,可揭示微生物潜在的互作机制及影响因素[16]。基于拓扑结构的网络分析已被证明是了解微生物共现网络特征的有力工具,为深入了解微生物群落结构和微生物之间的相互作用提供了依据。关于网络分析方法已有很多,其中基于随机矩阵理论的分子生态网络方法能更加准确可靠地进行网络构建和功能预测[17]。目前,针对化肥减施和有机替代的研究主要集中在土壤理化性质、氮素平衡、作物产量和微生物群落等方面,其中对土壤微生物群落的研究以微生物的多样性、组成、结构和功能变化及与环境因子的相关性等方面为主[18],从分子生态网络构建的角度探索化肥减量和有机替代下微生物互作关系变化的研究较少。因此,本研究利用生态网络分析方法构建化肥减量和有机替代等5种施肥处理土壤中的细菌和真菌共现网络,以探究化肥减施结合有机替代所致的微生物互作关系变化以及与SOM 变化有关的微生物种群。
1 材料与方法
1.1 研究区概况与土壤样品采集
本试验始于2015 年10 月,试验基地位于天津市宁河区(39°48′N,117°71′E)。该地区属温带大陆性季风气候,年均气温 11.2 ℃,年均降水量642 mm,全年无霜期210 d。耕作方式为冬小麦-夏玉米轮作,土壤类型为华北平原典型的潮土。本研究共包括5 个处理:矿物氮磷钾肥料(NPK)、矿物氮磷钾减量肥料(NPKR)、矿物氮磷钾减量肥料+玉米秸秆(NPKRS)、矿物氮磷钾减量肥料+有机肥(NPKRO)以及矿物氮磷钾减量肥料+有机肥+玉米秸秆(NPKROS)。各小区具体施肥情况见文献[19]。因不施肥并非当地农田常规施肥制度,故本研究未设置不施肥处理,而以NPK 作为对照。试验中施用的肥料包括无机复合肥(N 含量280 g·kg-1,P2O5含量130 g·kg-1,K2O 含量50 g·kg-1)、磷肥(P2O5含量120 g·kg-1)、钾肥(K2O 含量600 g·kg-1)、尿素(N 含量460 g·kg-1)以及有机肥(NPK 总含量大于等于50 g·kg-1,有机质含量大于等于400 g·kg-1)。纯氮量600 g·kg-1的化肥(无机复合肥)作为基肥在冬小麦和夏玉米播种时一次性施入农田,纯氮量400 g·kg-1的化肥(尿素)在小麦苗期和玉米拔节期分别追施入农田,有机肥仅在小麦季施入。小麦季所有处理产生的小麦秸秆全部粉碎后覆盖地表还田,玉米季NPKRS 和NPKROS 处理产生的玉米秸秆粉碎成小段后随土壤耕翻还田。田间管理按照当地常规生产模式进行。各处理小区面积较大(22 m×22 m),故未设置重复小区,在取样时将小区分为3个区域,即3 次假重复[20]。
分别于2019 年5 月底和9 月底作物收获前,使用直径5 cm 的土壤采样器于每个小区随机采集0~20 cm 耕层土壤,将采集的5 个土芯混合成1 个土壤样品,剔除石砾和植物残体等杂质后装入灭菌自封袋暂存于保温箱中并迅速带回实验室。将每份土壤样品过2 mm 筛后分为2 份子样品,一份于-70°C保存,用于土壤DNA 提取;另一份经风干后用于测定土壤SOM 含量。土壤SOM 含量采用重铬酸钾容量法测定[21]。
1.2 高通量测序及分析
本研究共对30 个土壤样品进行测序分析,其中小麦季5 个处理,每个处理各3 个重复;玉米季5个处理,每个处理各3 个重复。使用Fast DNA®Spin Kit for Soil(MP Biomedicals,美国)从0.5 g 冻存于-70°C 冰箱的土壤提取土壤基因组DNA,按照操作说明书进行,使用 1%的琼脂糖凝胶电泳检测DNA 质量,并使用 NanoDrop2000(NanoDrop Technologies,美国)测定DNA 浓度和纯度。用带有序列标签的引物组338F-806R 和ITS1F-ITS2R 分别扩增细菌16S rRNA 基因的V3~V4 高变区序列和真菌的ITS 区序列[22-23]。参照文献[19]进行PCR扩增并对原始测序序列进行质控和拼接。高通量测序委托上海美吉公司在Illumina MiSeq 平台上完成。使用UPARSE 软件根据97%的相似度对高质量的核酸序列进行聚类到操作分类单元(Operational taxonomic units,OTUs)并剔除嵌合体[24]。利用RDP classifier 对每条序列进行物种分类注释,设置比对阈值为70%。
1.3 分子生态网络分析
采用高通量测序数据在属水平上进行微生物群落的共现网络分析[25],在R 平台中使用‘psych’软件包进行相关性矩阵分析,并在交互平台 Gephi(Version 0.9.2)中绘制网络图。为减少配对比较数量并降低网络复杂性,仅选择相对丰度大于0.1%的细菌属和全部真菌属用于网络分析。选择皮尔森(Pearson)相关性系数r>|0.9|且显著性P<0.01 的数值进行微生物生态网络构建。微生物群落网络构建依照分子生态学网络分析流程(MENAP)进行(http://ieg4.rccc.ou.edu/mena/login.cgi)[26]。用Zi和Pi表示每个节点在拓扑结构中的作用,根据简化网络的Zi和Pi阈值,将整个生态网络分为4 部分,分别是外围节点、模块枢纽、连接器和网络枢纽[26]。
1.4 数据与统计分析
采用SPSS 23.0 软件(SPSS Inc.,Chicago,IL,美国)对不同处理的土壤SOM 含量(同一处理两次取样结果进行加和)进行单因素方差分析(One-way ANOVA),并采用邓肯(Duncan)方法进行显著性检验(P< 0.05)。使用Origin 2019 绘制5 种施肥处理的微生物网络拓扑角色分布图。
2 结 果
2.1 不同施肥处理下土壤有机质含量变化
如图1 所示,经过4 年的施肥试验,有机替代处理(NPKRS、NPKRO 和NPKROS)的SOM 含量发生显著变化。与 NPK 处理相比,NPKRS 和NPKROS 处理均显著提高了SOM 含量(P< 0.05)。NPKROS 处理的SOM 含量较NPKR 处理显著提高了15.52%(P< 0.05)。与化肥处理(NPK 和NPKR)相比,NPKRO 处理对SOM 含量的增加效果均达到了极显著水平(P< 0.01)。
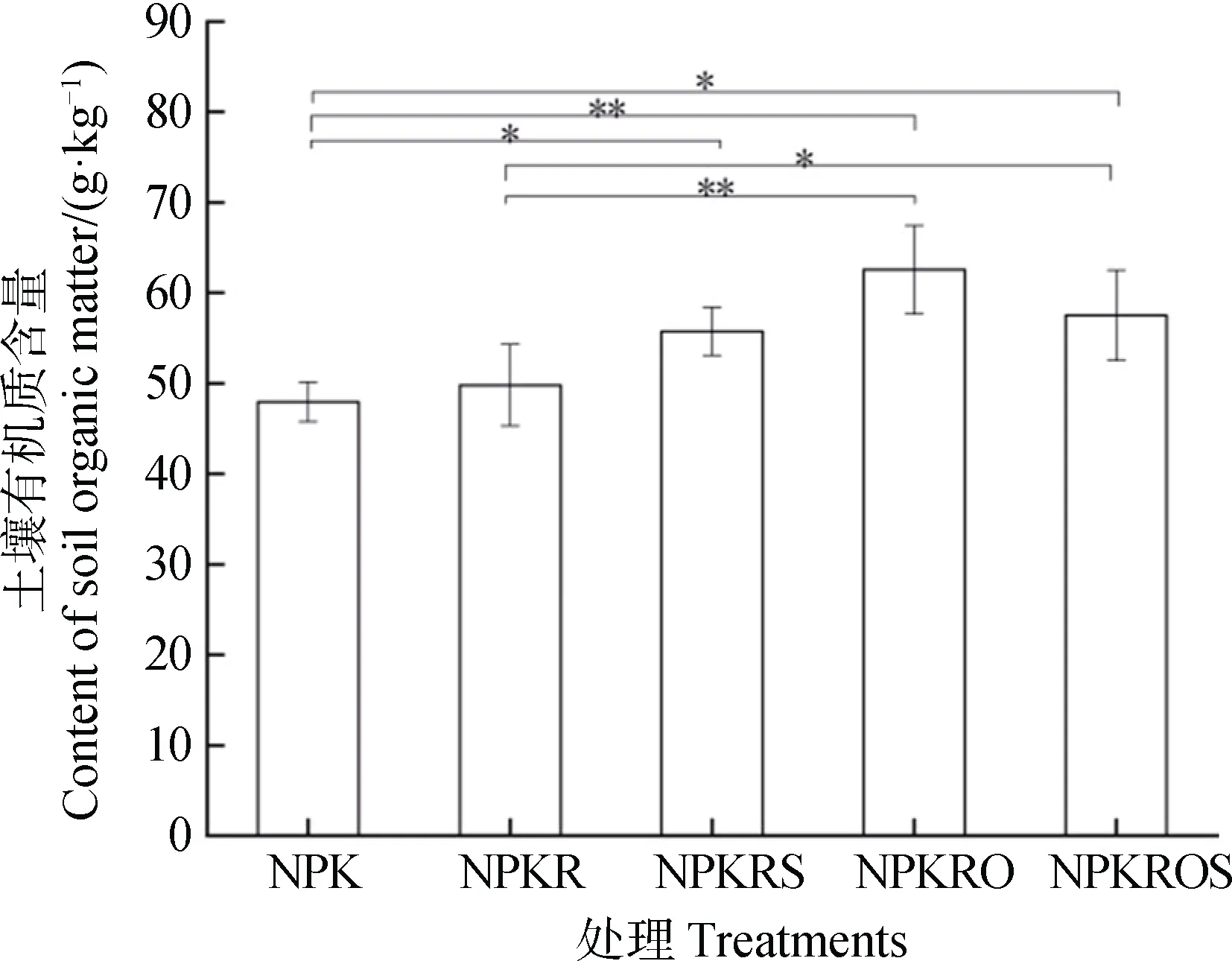
图1 不同施肥处理的土壤有机质含量Fig. 1 The content of soil organic matter of different treatments
2.2 不同施肥处理下土壤微生物群落组成与结构
经过质量筛选后,共获得1 639 138 条高质量的16S rRNA 基因序列和1 977 576 条高质量的ITS 序列。当序列数达到28 697 时,细菌稀释曲线基本趋于缓和;当序列数达到38 949 时,真菌稀释曲线基本趋于缓和。基于97%的相似性阈值进行聚类,分别得到6 859 个和934 个OTUs。聚类分析结果表明,细菌菌群分属46 个门、120 个纲、247 个目、474个科、951 个属和1 975 个种,真菌菌群分属10 个门、28 个纲、74 个目、158 个科、277 个属和453个种。细菌和真菌群落在门水平上的物种组成如图2 所示。各处理中土壤优势细菌门为放线菌门(Actinobacteria,平均相对丰度为25.87%)、变形菌门(Proteobacteria,24.80%)、绿弯菌门(Chloroflexi,18.48%)和酸杆菌门(Acidobacteria,13.61%)。子囊菌门(Ascomycota)为各处理中优势真菌门,平均相对丰度为90.88%。

图2 土壤细菌(a)和真菌(b)群落组成(门水平)Fig. 2 Compositions of soil bacterial (a) and fungal (b) communities (phylum level)
NMDS(非度量多维尺度)分析表明(图3),土壤细菌群落结构更易受取样时间的影响,小麦季和玉米季土壤细菌群落结构显著区分。土壤真菌群落结构受施肥处理影响更大。与NPK 处理相比,NPKR和NPKRS 处理下真菌群落结构有一定的迁移。添加有机肥处理(NPKRO 和NPKROS)的真菌群落结构显著区分于未配施有机肥处理(NPK、NPKR和NPKRS)。
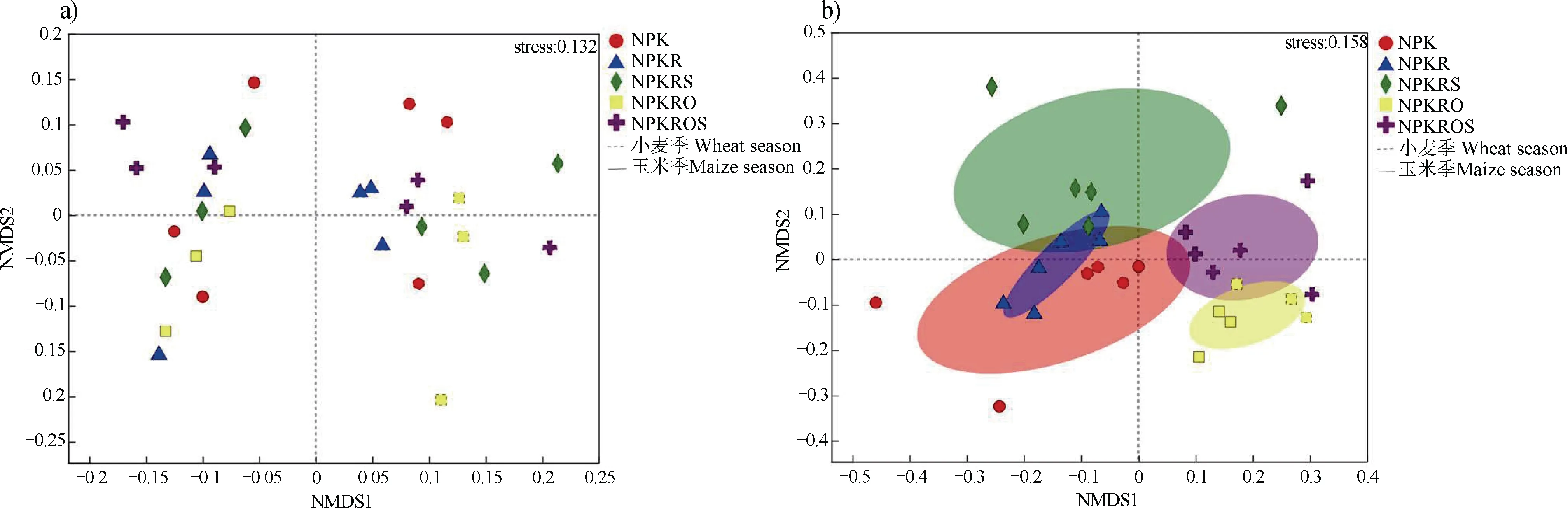
图3 土壤细菌(a)和真菌(b)群落非度量多维尺度(NMDS)分析Fig. 3 NMDS (non-metric multidimensional scaling) analysis of soil bacterial (a)and fungal (b) communities
2.3 微生物网络互作关系
分别基于5 种施肥模式微生物的测序结果构建分子生态网络(图4 和图5),网络拓扑参数见表1。细菌群落中,与NPK 处理相比,化肥减量和有机替代处理(NPKR、NPKRS、NPKRO 和NPKROS)提高了网络节点数和边数,降低了平均路径长度;NPKRS 和NPKROS 处理的平均聚类系数和网络密度较高;NPKRO 和NPKROS 处理的模块性较高;5个细菌网络均以正相关关系占主导地位,各处理细菌菌群的正相关比例大小顺序为:NPKRS>NPK>NPKR>NPKRO>NPKROS。真菌群落中,与NPK 处理相比,NPKR 处理提高了网络节点数和边数;有机替代处理(NPKRS、NPKRO 和NPKROS)增加了平均路径长度,降低了平均聚类系数;NPKR 处理的网络密度较高,而模块性较低;5 个真菌网络均同样以正相关关系占主导地位,各处理真菌菌群的正相关比例大小顺序为:NPKROS>NPK>NPKR>NPKRS>NPKRO。
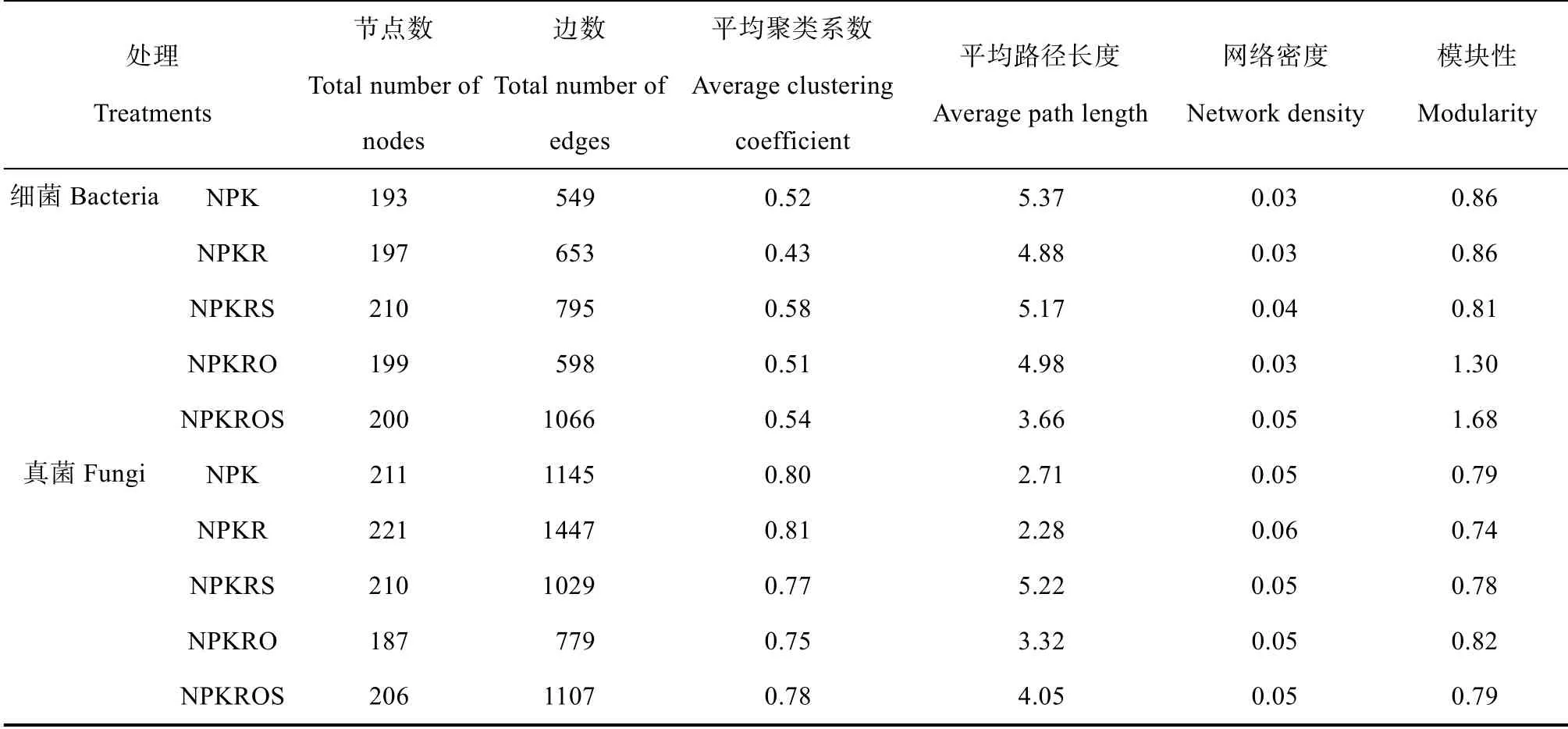
表1 不同处理微生物互作网络拓扑性质Table 1 Topological properties of the microbial interactions network relative to treatment
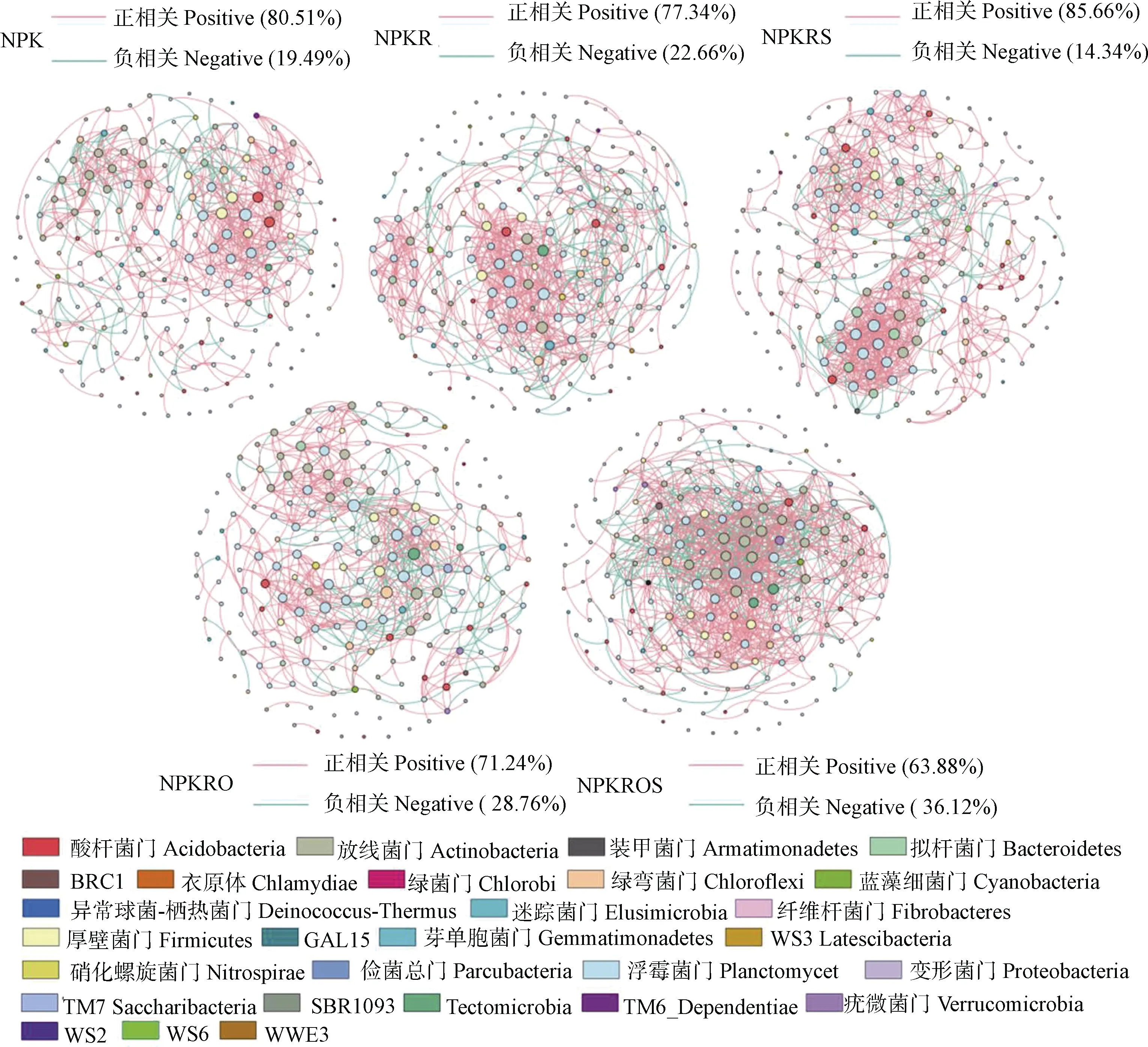
图4 细菌群落共现性网络图谱Fig. 4 Network co-occurrence analysis of the bacterial communities in the soil
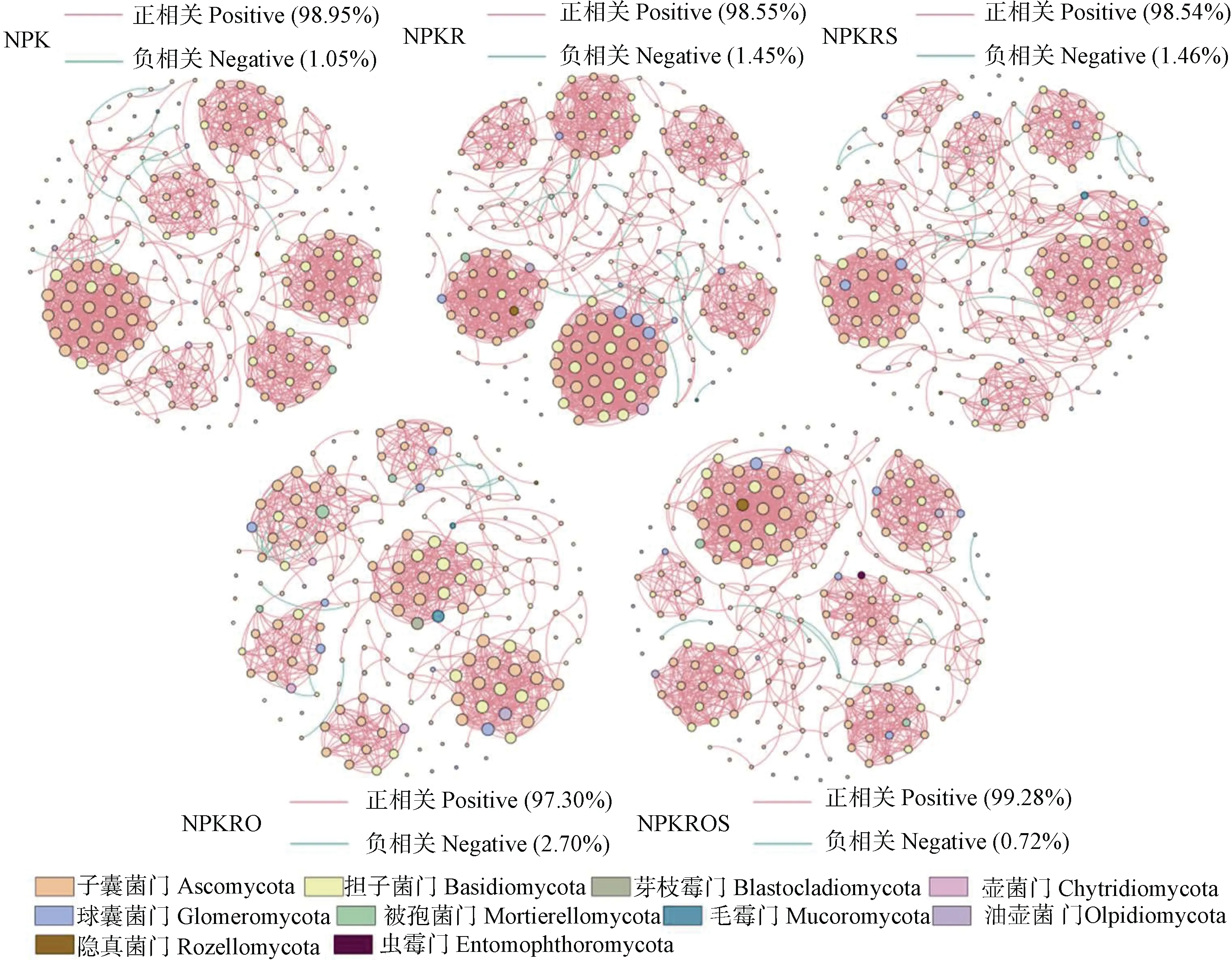
图5 真菌群落共现性网络图谱Fig. 5 Network co-occurrence analysis of the fungal communities in the soil
2.4 微生物群落与SOM 含量的关系
细菌和真菌群落与土壤SOM 含量的关系分别见图6 和图7。与NPK 处理相比,化肥减量和有机替代处理(NPKR、NPKRS、NPKRO 和NPKROS)提高了与土壤 SOM 含量显著正相关的细菌菌群占比,但降低了与土壤SOM 含量显著正相关的真菌菌群占比。在化肥处理(NPK 和NPKR)细菌群落中,芽胞杆菌属(Bacillus)、类芽孢杆菌属(Paenibacillus)、芽孢八叠球菌属(Paenisporosarcina)和G55属相对丰度较高,且均与SOM 含量显著负相关。Iamia属、壤霉菌属(Agromyces)、贫养杆菌属(Modestobacter)和德沃氏菌属(Devosia)与SOM含量显著正相关,Variibacter属与SOM 含量呈显著负相关,上述细菌菌群主要存在于有机替代处理(NPKRS、NPKRO 和NPKROS)中。真菌群落中,油壶菌属(Olpidium)、管柄囊霉属(Funneliformis)、腐质霉属(Humicola)、淡紫拟青霉(Purpureocillium)、拟盘多毛孢属(Neopestalotiopsis)和周刺座霉属(Volutella)仅存在于NPK 处理,且均与SOM 含量显著正相关;Westerdykella属、Zopfiella属、Tetracladium属、杯梗孢属(Cyphellophora)、Meyerozyma属和光柄菇属(Pluteus)仅存在于NPKR处理中,除Westerdykella属与SOM 含量显著负相关外,其他5 个真菌属均与SOM 含量显著正相关;青霉属(Penicillium)、帚枝霉属(Sarocladium)、裂壳属(Schizothecium)、赤霉属(Gibberella)、粉褶菌属(Entoloma)、锥毛壳属(Coniochaeta)和Colacogloea属仅存在于 NPKRS 处理中,除Colacogloea属与SOM 含量显著负相关外,其他6 个真菌属均与 SO M 含量显著正相关;链格孢属(Alternaria)和沃德霉属(Wardomyces)仅存在于NPKRO 处理,其中链格孢属(Alternaria)与SOM 含量显著正相关,沃德霉属(Wardomyces)与SOM 含量显著负相关;镰刀菌属(Fusarium)、拟棘壳孢属(Pyrenochaetopsis)和柄孢壳菌属(Podospora)仅存在于NPKROS 处理,镰刀菌属(Fusarium)和拟棘壳孢属(Pyrenochaetopsis)与SOM 含量显著负相关,柄孢壳菌属(Podospora)与SOM 含量显著正相关。
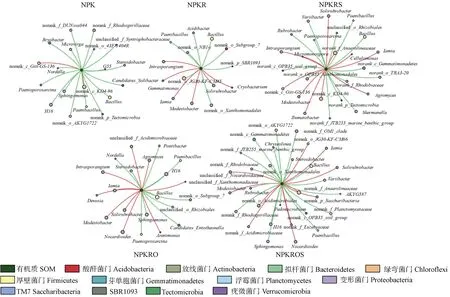
图6 土壤有机质(SOM)含量与细菌群落网络关系Fig. 6 Network relationship between soil organic matter content and bacterial community
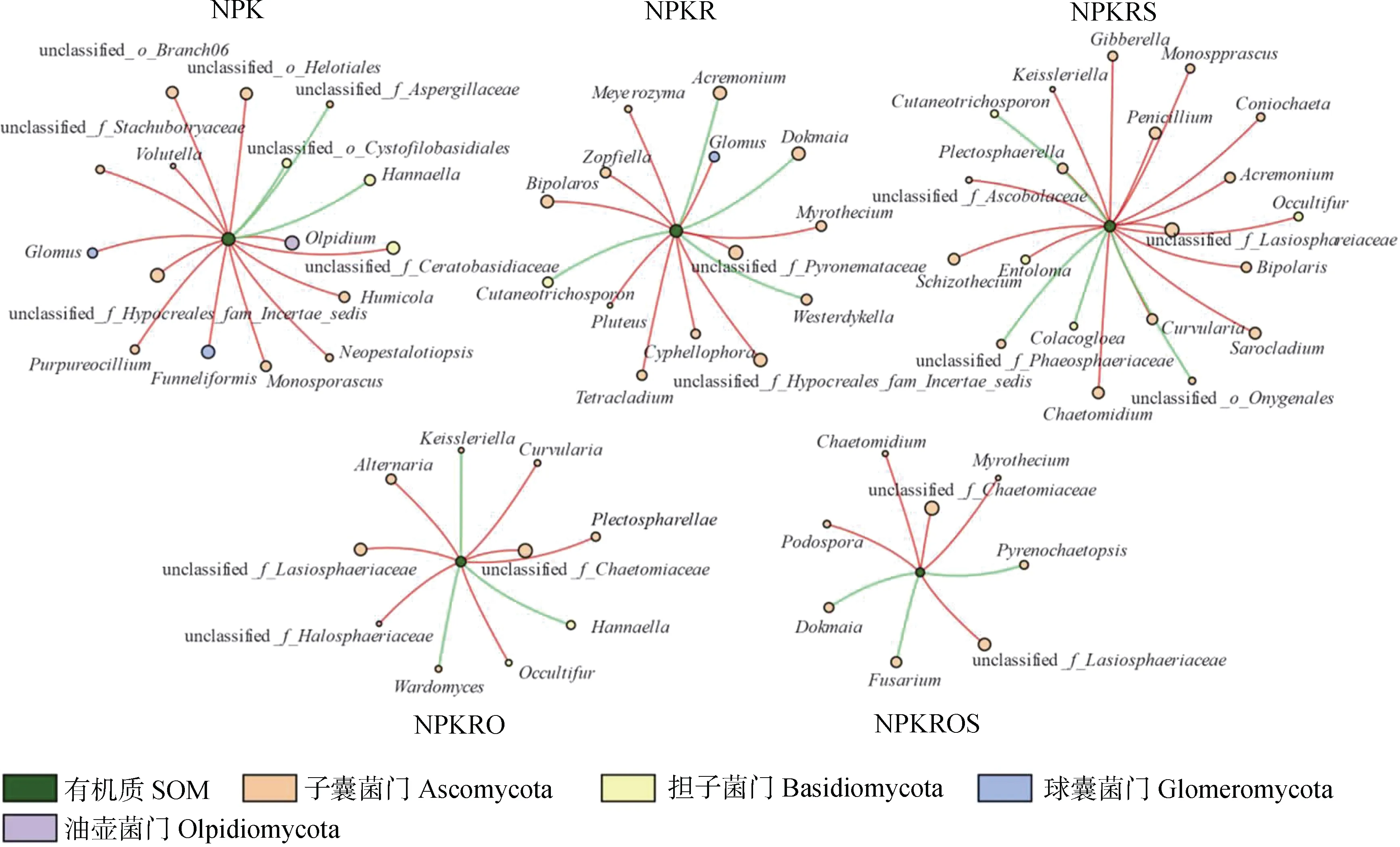
图7 SOM 含量与真菌群落网络关系Fig. 7 Network relationship between soil organic matter content and fungal community
2.5 微生物分子生态网络关键节点特征
经过对原始OTU 的筛选后,小麦季和玉米季5 个施肥处理的所有OTU 通过随机矩阵理论构建细菌和真菌群落网络结构模型(图8)。本研究所构建的细菌分子生态网络中,共有368 个节点,其中:(1)94.29%的节点属于外围节点,其中79.62%的节点仅与各自模块内部的某些节点之间有联系,即Pi=0;(2)2.99%的节点属于模块枢纽,共11 个OTUs;(3)2.72%的节点属于连接器,共10 个OTUs。真菌分子生态网络中,共有168 个节点,其中:(1)98.21%的节点属于外围节点,其中74.40%的节点仅与各自模块内部的某些节点之间有联系,即Pi=0;(2)1.19%的节点属于模块枢纽,共2 个OTUs;(3)0.60%的节点属于连接器,共1 个OTU。网络枢纽在本研究的细菌和真菌群落中均未出现。5 种不同施肥模式的关键细菌菌群主要分布在变形菌门、绿弯菌门、放线菌门、芽单胞菌门、拟杆菌门、厚壁菌门和酸杆菌门;关键真菌菌群主要分布在子囊菌门、担子菌门和球囊菌门。

图8 细菌(a)和真菌(b)网络模块中各OTU 的拓扑结构作用Zi-Pi 散点图Fig. 8 Topological Zi-Pi scattering map of each OUT in the bacterial (a) and fungal (b) network modules relative to their function
3 讨 论
3.1 化肥减量和有机替代对土壤有机质及微生物网络互作关系的影响
研究结果表明,化肥减量和有机替代显著提高了土壤有机质含量(图1),这与前人的研究结果[6,24]相一致。秸秆进入土壤后在其表面形成微生物活动层并加速其分解[27]。有机肥为土壤提供大量有机碳,外源碳输入可通过刺激微生物矿化来提高有机质含量[6]。本研究结果显示,配施有机肥和秸秆的NPKROS 处理的有机质含量并未达到最高值(图1),这可能与外源有机物料的施入在经过一定时间后会对土壤有机碳产生正激发效应,促进了土壤原有有机碳的分解,使土壤有机质含量有所下降有关[28-29]。网络节点数和边数可以表示分子生态网络的规模大小[29]。本研究中,化肥减量和有机替代处理的细菌分子生态网络节点数和边数均高于NPK处理,但仅有NPKR 处理的真菌分子生态网络节点数和边数与NPK 处理相比有所提高(表1),表明化肥减量和有机替代能扩大土壤细菌网络规模,而真菌网络规模的扩大则主要依赖于单独进行化肥减量。网络中物种间的正负相关性分别可表示菌群之间的协同合作和竞争捕食关系[29]。由微生物菌群之间正负相关比例(图4)可见,NPKRS 处理的细菌菌群正相关比例最高,添加有机肥处理(NPKRO 和NPKROS)的正相关比例较低,说明秸秆的添加增强了细菌菌群之间的协同作用,而在有机肥添加的情况下细菌菌群之间形成了较强的竞争关系。这可能与有机肥中含有大量更易被细菌群落分解利用的半纤维素和蛋白质等简单有机物有关[30]。真菌群落各处理之间显著正相关比例远高于显著负相关比例(图5),表明在本研究的土壤中真菌群落之间有更为紧密的协同合作关系,竞争关系较弱。与NPK 处理相比,化肥减量和有机替代均降低了细菌分子生态网络的平均路径长度,而在真菌群落中,仅NPKR处理的平均路径长度低于NPK 处理,表明化肥减量和有机替代均提高了细菌网络物种间物质循环、能量流动和信息传递的效率[31],而真菌网络中这一现象的发生主要依赖于化肥减量,这一点与微生物群落网络节点数和边数的结果相一致。平均聚类系数越高,表明微生物对外界环境因子的扰动更加敏感,响应迅速[31]。本研究表明,单独进行化肥减量的细菌群落结构更加稳定,而秸秆的添加则加剧了土壤细菌群落互作和对外界环境的敏感程度。对真菌群落而言,除化肥减量的平均聚类系数略有增加外,有机替代处理的平均聚类系数均有所下降(表1),这表明有机替代后土壤真菌响应速度较慢,外界环境对其扰动性并不会在较短时间内影响整个真菌生态网络。真菌分子生态网络密度仅在NPKR 处理表现出增加,表明化肥减量会增加真菌群落互作。细菌和真菌群落模块性(Modularity)数值均大于0.4,故可判断该网络具有模型结构[25]。添加有机肥处理(NPKRO 和NPKROS)的细菌和真菌模块性数值均高于其他3 个处理,表明有机肥可能是微生物模块化增强的重要原因。
3.2 化肥减量和有机替代对微生物群落与土壤有机质含量关系的影响
化肥减量和有机替代处理中与SOM 含量显著正相关的细菌菌群占比均高于NPK 处理(图6),表明化肥减量和有机替代可能更利于SOM 的形成。有机替代处理中Iamia属、壤霉菌属(Agromyces)、贫养杆菌属(Modestobacter)和德沃氏菌属(Devosia)与SOM 含量显著正相关,说明上述微生物偏爱SOM 含量高的生境。丁柳屹[32]的研究发现Iamia属与土壤有机碳含量呈正相关;徐丽萍等[33]通过Illumina MiSeq 高通量测序技术预测德沃氏菌属(Devosia)可降解玉米秸秆。与细菌群落不同,有机替代处理中与SOM 含量显著正相关比例的真菌菌群比例低于NPK 处理(图7),这可能是由于特定真菌类群更喜欢在有机肥施入后形成的土壤环境中生存,其消耗大量营养物质,从而抑制了其他类群的生长[34]。赤霉属(Gibberella)和链格孢属(Alternaria)是小麦、玉米等农作物的主要致病菌之一[35-36],但链格孢菌(Alternaria Nees)具有生物资源的应用潜力,在杀菌、杀虫等方面起重要作用[37]。上述研究均表明不同施肥会引起SOM 的变化,进而影响微生物群落的相互作用。
细菌网络中变形菌门是占关键节点最多的物种,说明变形菌门是研究区潮土中普遍存在的重要细菌种群。这可能与变形菌门作为富营养细菌,适宜生长在含碳量较高地环境有关[38]。真菌网络中子囊菌门占关键节点的1/3,这可能是由子囊菌门作为一种腐生真菌适宜生存在干旱环境中所决定的。
4 结 论
化肥减量和有机替代能显著提高土壤有机质含量,同时提高细菌分子生态网络规模和物种间传递物质、能量和信息的效率。秸秆的添加能增加细菌群落互作,使细菌对外界环境因子的扰动更加敏感。化肥减量能提高真菌分子生态网络规模和群落互作,有机替代使真菌群落网络结构更加稳定。在化肥减量的基础上进行秸秆还田能够促进细菌菌群之间协同合作关系,有机肥的施入会加剧其竞争关系;真菌菌群之间具有较为紧密的协同合作关系,竞争关系较弱。化肥减量和有机替代丰富了与SOM 显著正相关的细菌类群,但简化了与SOM 显著正相关的真菌类群。变形菌门、绿弯菌门、放线菌门、芽单胞菌门、拟杆菌门、厚壁菌门和酸杆菌门中的部分菌群在土壤细菌分子生态网络中起着重要的连接作用;子囊菌门、担子菌门和球囊菌门中的部分菌群在土壤真菌分子生态网络中起着重要的连接作用。
