空间转录组学技术及其应用的研究进展
2022-05-20张心雅马伟燕柯荣秦
林 辰,张心雅,马伟燕,柯荣秦
(华侨大学医学院,福建 泉州 362021)
基因表达是一个动态的过程,具有广泛的时空异质性.传统的转录组测序将RNA的表达量平均化,忽略了细胞群体内不同细胞之间基因表达的异质性[1].单细胞RNA测序技术的出现解决了这一问题,它能够独立地提供每个细胞的RNA表达谱,区分出细胞之间的基因表达差异,并鉴定出异质细胞群中的稀有细胞[2-3].尽管单细胞RNA测序技术极大地扩展了人们对细胞群体异质性的认识,但是进行单细胞测序需要将细胞从组织中解离,从而导致细胞空间位置信息的丢失[4].而细胞的空间位置信息预示着细胞之间可能存在相互作用,而这又与生理和病理功能紧密相关,因此有必要将基因表达与空间位置信息联系在一起[5].在这种需求的驱动下,Joakim Lundeberg课题组于2016年首次提出了空间转录组学的概念,发表了第一个基于原位捕获RNA的空间转录组学技术[6].此后,一系列能够进行高通量原位RNA检测分析的技术都被归为空间转录组学技术的范畴.尽管这些技术的原理不尽相同,但都有一个共同点,即记录所检测RNA分子的空间位置信息.本文对空间转录组学技术的发展进行介绍,总结并比较不同技术之间的优缺点,举例说明空间转录组学技术在不同研究领域的应用,最后讨论其所面临的挑战并展望其发展趋势.
1 空间转录组学技术的分类
现有的空间转录组学技术在可检测的基因数量和可测定的组织大小方面差异很大,可以分为两大类:一类是基于原位捕获和测序的方法,在测序之前通过原位捕获将空间位置信息编码到转录本上,之后通过高通量测序获得转录本及其空间位置信息;另一类是基于成像的方法,即在RNA的原始位置对其进行染色编码,实现多重检测.
1.1 基于原位捕获和测序的空间转录组学技术
为了解决单细胞RNA测序丢失细胞空间位置信息的问题,2016年,Joakim Lundeberg课题组展示了一种利用引物微阵列进行原位RNA表达分析的空间转录组学技术(下文简称ST技术)[6].该文首次介绍了利用带有空间标签序列的引物微阵列原位捕获转录本,从而保留转录本位置信息的方法.其基本原理如图1所示:将组织切片贴于带有空间位置信息标签序列的多聚T碱基引物微阵列芯片上,通过苏木精-伊红染色法对组织进行染色并拍照记录组织的形态;接着对组织进行透化处理及原位逆转录,获得带有空间位置信息的互补DNA(complementary DNA,cDNA);最后通过二代测序获得基因序列并解码空间位置信息标签,通过与组织形态联合分析就可以得到该组织的空间转录组数据.利用该技术,他们对小鼠嗅球组织中的RNA进行测序,获得了组织切片上完整的基因空间表达图谱.2018年,10x Genomics收购了该技术,并推出Visium芯片技术,与最初的方案相比,进一步提高了空间分辨率和灵敏度,并且成为一种被广泛接受的商品化空间转录组学技术,应用于生物学不同领域.
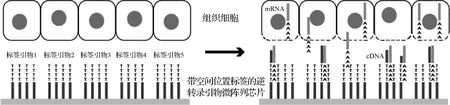
图1 基于原位捕获和测序的空间转录组学技术的基本原理
通过图1所示的空间转录组学技术基本原理可知,实现空间转录组测序的关键在于对空间位置信息的保留.因此,利用不同的方法构建带有空间位置信息的引物微阵列芯片是此类技术的核心步骤.Slide-seq是继ST技术之后发表的第二个基于原位捕获和测序的高通量空间转录组学技术[7-8].在Slide-seq中,大量直径为10 μm的带有DNA标签引物的微球首先被平铺于芯片的表面,通过二代测序方法SoLiD(sequencing by oligonucleotide ligation and detection)技术原位获得每个微球的空间DNA标签序列;然后将组织切片贴附于带有这些微球的芯片表面,采用组织透化技术释放样品中的RNA进行原位逆转录,在芯片表面获得带有空间位置标签序列的cDNA;这些cDNA再通过建库和二代测序可以推断出所检测RNA的空间位置信息,从而获得组织的空间基因表达图谱.与ST技术中引物微阵列的引物点相比,Slide-seq的微球直径更小,因此大大提高了分辨率和灵敏度,达到对单细胞转录组进行空间定位的水平.为了达到更高的分辨率以实现对单细胞的定位和分析,Vickovic等[9]提出了高分辨率空间转录组学(high-definition spatial transcriptomics,HDST)技术.HDST技术使用类似于Slide-seq的方法来提高分辨率,不同的是该技术将带有空间位置标签的微球放置于芯片表面的微孔内;而这些微球直径只有2 μm,理论上来说能够提供比Slide-seq更高的空间分辨率.Seq-Scope技术是一种基于Illumina测序平台的空间转录组学技术,该技术通过桥式PCR在捕获芯片的表面生成一系列带有随机标签的引物簇[10].通过成像和测序获取引物簇的序列及空间位置信息后,利用这些引物簇捕获组织中的RNA并转录为待测序的cDNA.该方法可获得0.5~0.8 μm分辨率的引物簇,进一步提高了空间分辨率,达到单细胞甚至是亚细胞的分辨率水平.除此之外,Pixel-seq[11]和Stereo-seq[12]是最近报道的两种基于捕获的空间转录组学技术.其中Pixel-seq是利用类似于Seq-Scope技术的原理,通过桥式PCR获得具有空间分辨率和空间标签的引物簇[11].Stereo-seq是华大基因所开发的技术,该方法基于华大基因所特有的DNA纳米球(DNA nanoball,DNB)测序芯片,将带有随机序列的DNB置于芯片上测序获得位置及标签序列后,这些DNB再接上多聚T碱基尾巴作为引物用于捕获信使RNA(messenger RNA,mRNA),后续测序步骤与其他方法类似[12].Stereo-seq的特点在于利用DNB芯片的规则阵列以及DNB的微小尺寸实现高分辨率的空间RNA捕获,并且拥有较大的整体捕获面积,达到厘米级别的大视野空间转录组分析水平.
上述方法都是基于原位转录本捕获并测序获得组织的空间转录图谱,其标记空间信息的方法均是基于芯片表面的标签引物.2020年,Liu等[13]则采用了一种名为组织层面的条形码编码的空间组测序(deterministic barcoding in tissue for spatial omics sequencing,DBiT-seq)的微流控空间组测序方法.与其他方法不同,该技术并不将引物固定于芯片表面,而是通过两个正交方向的微流控将引物引入组织表面,每个方向50个微通道,每个通道里引入不同的寡聚核苷酸序列进行组合,从而形成2 500个标签编码点.通过这种方式获得的每个标签编码点的大小约10 μm,具有较高的空间分辨率.同时,他们利用带有寡聚核苷酸标签的抗体对不同蛋白质进行空间定位和定量分析,实现了多组学基因表达空间图谱的构建.
总之,利用具有空间位置信息的DNA标签引物捕获并标记转录本的空间位置信息,是基于原位捕获和测序的空间转录组学技术的核心所在.
1.2 基于成像的空间转录组学技术
基于成像的空间转录组学技术的本质是RNA原位杂交,具体地说就是多重RNA原位杂交技术.严格意义上,这一系列技术的起源都可以追溯到单分子RNA原位杂交技术.早在1998年,Femino等[14]就利用标记有多个荧光基团的DNA探针与RNA分子进行原位杂交,实现了对单分子RNA的原位检测.不仅如此,Levsky等[15]还在后续研究中利用颜色组合的方式,突破了光谱分辨的限制,实现了对11个基因的同时检测.Raj等[16]对该方法进行改进,通过将24~48条的单标记探针杂交于同一个RNA分子上,实现了更加高效灵敏的单分子RNA原位杂交,并将此方法正式命名为单分子荧光原位杂交(single-molecule fluorescenceinsituhybridization,smFISH).Lubeck等[17]通过改变杂交于RNA分子上标记探针的荧光标签的顺序或者比例,利用高分辨显微镜进行分辨,从而实现了对多种RNA分子的同时检测.以上方法均在同一轮成像中进行不同种类RNA分子的原位多重检测,因此能够实现的检测通量比较有限.2013年,Ke等[18]首次发表了原位测序(insitusequencing,ISS)技术,提供了一种通过多轮成像实现颜色序列编码的方法实现对不同种类的RNA进行高度多重原位检测的方案.其基本原理如图2所示,即对同一个RNA分子在其原始位置进行信号放大后利用不同的标记探针进行多次标记成像,并通过高级图形分析算法,将每一轮的颜色进行配准和标注,得到一串颜色编码标签,达到利用不同颜色顺序检测不同基因的目的,从而不受光谱和显微镜检测通道数量的限制达到高度多重的RNA原位检测.

图2 基于成像的空间转录组学方法的基本原理
ISS的具体做法是在固定的细胞或者组织中将RNA原位逆转录为cDNA,再将带有4种碱基组成的基因特异DNA标签的锁式探针与其目标基因杂交并连接成环,通过滚环扩增(rolling circle amplification,RCA)获得信号放大的RCA产物.为了解析不同RCA产物上的标签序列,Drmanac等[19]采用边连接边测序(sequencing by ligation,SBL)的二代测序技术对DNA标签的4种碱基进行测序,结合图形信息得到每个扩增产物的标签序列,从而可以得知该扩增产物所检测的RNA种类.利用该方法,Ke等[18]展示了在乳腺癌冰冻切片组织中识别31个基因的空间表达图谱.2016年,基于ISS技术成立了Cartana公司,正式将该技术商业化.2019年,10x Genomics公司收购了Cartana公司.
ISS技术基于锁式探针特异识别和RCA进行信号放大,并通过二代测序化学实现原位测序,由于其具有操作简易和信号稳定等特点,所以此后有多种技术基于锁式探针和RCA发展而来.Wang等[20]报道了一种能够实现三维原位测序的STARmap技术,该技术能够对160~1 020个基因实现同时检测.STARmap技术也是基于锁式探针来实现靶向原位测序的,与ISS技术不同的是其探针直接与RNA杂交,并通过辅助的领位杂交引物进行成环步骤,省去了原始ISS技术中的逆转录步骤,这种成环模式的探针也称为蜗牛探针.在RCA完成后,通过对扩增产物上的氨基与N-羟基丁二酰亚胺反应修饰后与丙烯酰胺一起聚合形成水凝胶,之后使用蛋白酶消化一些自发荧光的蛋白等使组织透明化,以降低测序中的荧光背景.STARmap还设计了一种纠错的连接测序方法以增加测序的准确率,在多重改进条件的保证下,实现了对0.1 mm厚的小鼠大脑皮层中上千个基因的原位检测,展示了其作为三维原位测序技术的潜力.Liu等[21]报道的条形码寡核苷酸多重原位分析(barcoded oligonucleotides ligated on RNA amplified for multiplexed and parallelinsituanalyses,BOLORAMIS)技术则直接以RNA为模板杂交,并利用能够在RNA上连接DNA探针的连接酶SplintR连接锁式探针成环,在RCA后采用SBL方法测得标签序列,可实现多重RNA原位检测.利用该方法在细胞系和共培养的神经元细胞与小胶质细胞上对多达96个基因的空间表达进行了分析,然而却缺少组织基因表达分析的数据.
2013年,Ke等[18]除利用带有标签的锁式探针实现多重RNA原位检测外,还介绍了一种利用带有几个碱基缺口的特殊锁式探针(gap-fill padlock probe)来实现对RNA序列本身进行测序的方法,并将该方法用于不同RNA序列的测序和稀有突变的检测.Chen等[22]对该方法进行了优化,开发了BaristaSeq技术,利用Phusion DNA聚合酶代替ISS中的Stoffel片段聚合酶,提高了缺口的补充效率,并利用和Illumina测序平台相同的边合成边测序方法获得了更稳定的测序荧光信号,从而实现更长的测序长度.与其他基于锁式探针的空间转录组学技术不同的是,BaristaSeq技术并非用来检测单个转录本的表达位置,而是检测表达同一个由病毒引入细胞的RNA标签,从而实现对整个细胞的编码检测.此后,Chen等[23]在此基础上又发展了BARseq技术,通过对不同细胞的标签进行原位测序,获得细胞的空间位置信息,实现了对小鼠大脑听觉皮层中3 579个神经元的连接图谱绘制.
以上基于锁式探针的原位测序技术可以认为是靶向空间转录组学方法.在2014年,Lee等[24]就开发了一种高通量原位测序方法,即荧光原位RNA测序(fluorescentinsituRNA sequencing,FISSEQ),能够非靶向地对转录组水平的RNA表达同时进行定位分析.FISSEQ技术之所以能够实现非靶向RNA表达高通量原位分析,是因为其直接将逆转录获得的所有cDNA通过单链DNA环化酶CircLigase进行环化,并通过RCA信号放大后直接对RNA本身进行测序,获取RNA序列信息来判定不同的基因类型.然而,非靶向检测与RCA信号放大会导致扩增产物过多而产生光学拥挤效应,并导致无法正确区分信号点,降低了检测效率.最近开发的扩展测序(expansion sequencing,ExSeq)技术使用扩展显微镜解决了这个问题[25],该技术利用可膨胀的水凝胶吸水膨胀后将含有扩增产物的组织物理放大,从而增加信号点之间的距离,进而提高了分辨率和检测效率.
第二类基于成像的方法是在原位杂交技术基础上,通过互补荧光探针的杂交来检测目标序列.2014年,Lubeck等[26]首次提出了连续单分子荧光原位杂交技术的概念,并采用该技术在固定的细胞中利用4种颜色编码的荧光探针组进行两轮杂交,同时检测多个基因,在两轮杂交之间利用光漂白和DNA酶对上一轮的检测探针进行消化剥离及新一轮的探针杂交.2015年,Chen等[27]发表了同样基于单分子荧光原位杂交的多重抗误差荧光原位杂交(multiplexed error-robust fluorescenceinsituhybridization,MERFISH)技术,该技术可以鉴定单个细胞中数千种RNA的拷贝数和空间定位.MERFISH利用组合探针与连续成像等技术来提高检测通量,并通过汉明码纠错的二进制条形码来抵消单分子标记检测错误.目前,MERFISH 已广泛应用于多个领域,从单个细胞内的转录本位置到组织水平的空间转录组学均展示出其独特的优势.2016年,Shah等[28]结合杂交链式反应(hybid chain reaction,HCR)进行信号放大,正式将其连续单分子RNA荧光原位杂交技术命名为seqFISH(sequential fluorescenceinsituhybridization).该方法实现了对小鼠海马体中249个基因的同时检测,并通过基因表达聚类分析实现了对不同类型细胞的原位鉴别.2019年,Xia等[29]则提出了与支链DNA技术相结合对MERFISH进行信号放大的方法,证明在基因杂交探针较少的情况下信号放大具有一定的优势,不过使用更多对探针也能替代信号放大的效果.同年,Eng等[30]发表了seqFISH+技术,该技术采用和MERFISH相似的探针设计策略和超分辨成像,不同的是该技术使用了3个成像通道,经过20次反复杂交获得60个假单色进行编码.由于每个通道的20种假单色经过3轮解码可以获得203(8 000)种基因编码,所以3个通道就可以编码24 000种基因.通过该方法,他们在亚细胞分辨率下实现了对1万个基因的空间表达分析.
2020年,Goh等[31]提出改良型RNA单分子荧光原位杂交技术并命名为split-FISH.该技术主要是在RNA与检测探针之间引入了一对编码探针,当编码探针与目标基因杂交后靠近,桥接探针与两条编码探针的3’-端和5’-端杂交结合后再与检测探针结合,保证了特异性.通过与MERFISH相同的杂交编码方法,split-FISH实现了对317个基因的同时检测.与通过编码方式实现多重检测的方法不同,Codeluppi等[32]开发的循环单分子荧光原位杂交技术(cyclic single-molecule fluorescentinsituhybridization,osmFISH)技术是直接在每一轮杂交中利用不同的荧光标记和检测通道只检测3种基因,利用多轮杂交来实现更多的基因检测;他们经过13轮杂交,检测了小鼠躯体感觉皮质层中33种基因的表达,通过基因表达模式区分并定位了不同类型的细胞.组织上的单细胞分辨率的原位杂交(single cell resolutioninsituhybridization on tissues,SCRINSHOT)技术[33]则是先通过RCA对RNA信号进行放大后,采用类似osmFISH的多轮不相关杂交方法,实现了多达29种基因的同时检测分析.由于osmFISH和SCRINSHOT技术对不同杂交轮次之间的信号不进行关联配准分析,所以它们之间互不影响,避开了由于光学拥挤产生的信号识别问题,对高表达基因具有更好的检测效果.
综上所述,现有的空间转录组学技术泛指一类能够实现高度多重RNA原位检测的新型转录组分析技术,通过将基因表达信息与组织形态学信息联系在一起,为准确地预测基因功能和调控网络提供更多的支持信息.然而,这些技术往往具有不同的检测通量和分辨率等,研究人员需要根据不同的目标进行合理的选择.表1对一些主要的空间转录组学技术的特点进行了总结.
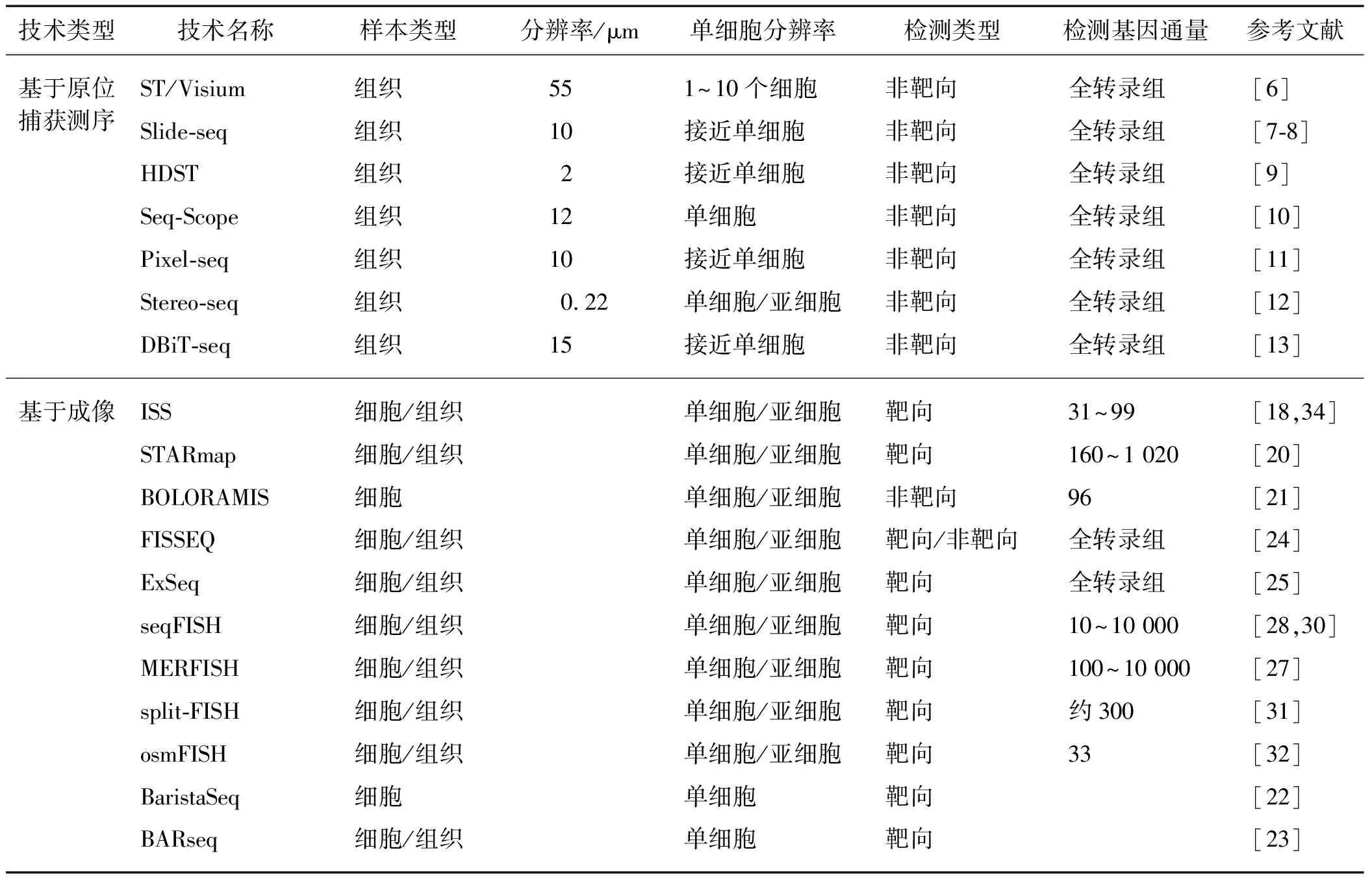
表1 不同空间转录组学技术的特点
1.3 其他的空间转录组学技术
除利用上述方法能够实现对基因表达位置信息的记录外,还可以利用切割的方法对组织进行空间基因表达分析.激光捕获显微切割(laser capture microdissection,LCM)[35]可以对组织切片中感兴趣的区域进行切割捕获,分离得到的小组织块可以进行包括转录组测序在内的多组学分析,获得其含有空间位置信息的基因表达谱等.在Tomo-seq技术[36]中,冰冻的组织被连续切片后对每个切片进行转录组测序,得到具有空间位置信息的基因表达图谱.Geo-seq技术[37]则是将组织切割成小块,对每个小块进行转录组测序,利用小组织块的位置信息重构三维基因表达图谱.虽然Tomo-seq和Geo-seq技术可以获得三维空间表达图谱,但是空间分辨率较低;而LCM虽然能够获得高分辨率区域基因表达谱,但是其分析通量很低.与其他空间转录组学技术相比,基于切割的空间转录组学技术都存在步骤繁琐、不易操作的问题.
2 空间转录组学技术的应用
空间转录组技术提供的基因表达的空间位置信息能够用于建立组织细胞的空间图谱,具有重要的参考价值.经过短短几年的发展,现有的空间转录组学技术已经被广泛应用于神经生物学、发育生物学、肿瘤生物学等领域[38-40].
2.1 在神经生物学中的应用
神经科学中的一个主要挑战是如何系统地了解脑细胞类型及其在组织中的定位,并解析其工作机制.空间转录组学可以将脑细胞类型与形态学、生理学和连接性相关的功能学联系起来,从而全面了解脑神经回路[41].虽然利用单细胞测序能够对脑组织中不同类型的细胞进行分型和定义,但是由于获得的单细胞已经从组织解离,所以无法准确定位这些细胞的微环境,不利于后续的细胞功能分析.Codeluppi等[32]将小鼠细胞体感区神经元细胞标志基因结合osmFISH技术对这些细胞类型进行空间定位.Qian等[34]采用在原有ISS技术基础上开发的pciSeq技术,通过检测已有单细胞数据筛选出来的标志基因,对小鼠海马区CA1中的抑制性神经元以及不同类群的锥体细胞进行定位.Moffitt等[42]结合单细胞RNA测序和MERFISH技术创建了小鼠下丘脑视前区的空间转录细胞图谱,绘制了约100万个细胞的轮廓,鉴定了约70个具有不同神经调节特征和空间组织特征的神经元群体,该方法为在不同组织和生物体中构建细胞图谱开辟了一条新的途径.Chen等[43]同样运用单细胞RNA测序与MERFISH结合的方法,系统地解析了小鼠伏隔核脑区的细胞类型,区分出D1中型多棘GABAergic神经元、D2中型多棘GABAergic神经元和中间神经元等主要细胞类型,并将其分为多种亚型;接着利用MERFISH技术确定了这些神经元亚型在伏隔核脑区不同位置的空间分布,构建了小鼠伏隔核细胞空间图谱,为伏隔核结构与功能的研究奠定了良好的基础.Yu等[44]利用单细胞测序以及ISS技术对人脑发育过程中的中间神经元起源和多样性进行探究,系统地揭示了人类胚胎大脑不同发育阶段中间神经元的起源和特异分子标记;其中ISS技术对基因表达的定位起关键作用.Rodriques等[7]利用其开发的Slide-seq技术将单细胞RNA测序确定的细胞类型在小脑和海马体内进行定位,表征小鼠小脑浦肯野层的基因空间表达模式,并展示了创伤性脑损伤小鼠模型中细胞类型特异性反应的时间演变.Maynard等[45]选择人背外侧前额叶皮层(dorsolateral prefrontal cortex,DLPFC)中一个与许多神经精神疾病有关的大脑区域进行Visium空间转录组测序,绘制DLPFC中基因表达的层状图谱,并利用已发表的大规模snRNA-seq(single nuclei RNA sequencing)研究的数据进行验证,确定了已知的精神分裂症和自闭症相关基因的空间分布模式,从而提出了精神分裂症遗传易感性的机制.
利用单独的空间转录组学技术也能对神经系统中的不同细胞类型进行鉴定和定位.Yao等[46]使用MERFISH技术分析了小鼠初级运动皮层中的约30万个细胞,识别了95个神经元和非神经元细胞簇,发现不仅是兴奋性神经元簇,大多数抑制性神经元簇也都有分层组织,构建了一个复杂的空间细胞图谱.Eng等[30]采用seqFISH+技术对小鼠脑室下皮层和嗅球中的1万个基因进行检测,对其中2 963个细胞进行了空间定位.不同空间转录组学技术的组合也有独特的优势,例如Chen等[47]将ST与ISS技术相结合,研究阿尔茨海默病(Alzheimer’s disease,AD)淀粉样斑块周围直径100 μm的组织结构域的基因表达变化,揭示了富含髓鞘和少突胶质细胞基因的基因共表达网络的早期改变,以及斑块诱导基因的多细胞基因共表达网络,为AD和其他脑部疾病的研究提供了新的思路.
2.2 在发育生物学中的应用
构成复杂生命体组织或器官的细胞都是由多功能干细胞或者祖细胞分化而来的,在胚胎发育的早期,细胞根据生长发育的需要分化为特定细胞,这些细胞的分化轨迹和发育谱系及其在组织中的空间定位对组织器官的正常发育至关重要[48-49].van den Brink等[50]使用单细胞RNA测序和ST技术比较了小鼠原肠胚和胚胎,在原肠胚中鉴定了以前未知的胚胎细胞类型,并发现体细胞发生的关键调节因子在胚胎和原肠胚之间具有许多相似的地方.Fawkner-Corbett等[51]通过对来自17例胚胎的77个样本进行单细胞RNA测序,并对来自5个样本的8张切片进行空间转录组测序,探究肠道发育的分子生物学基础,通过一系列的生物信息学分析,鉴定了肠道发育过程中的101种细胞类型,绘制了其发育的空间图谱.该研究揭示了肠道发育的一些关键事件,如上皮隐窝-绒毛的形成原理,确定了发育中的成纤维细胞和肌成纤维细胞亚型的分化层次和功能、血管的扩张、免疫定植和肠道相关淋巴样组织的形成等.Asp等[52]采集了妊娠早期3个阶段(孕4.5~5.0周,6.5周和9.0周)的心脏组织样本,采用单细胞测序结合ST和ISS技术绘制了心脏发育过程中基因表达的时空图谱.ST与ISS技术联合确定了发育过程中不同的解剖区域特异的基因表达谱,而单细胞RNA测序则帮助鉴定了细胞的类型,并通过联合分析映射到特定的解剖区域,最终构建了妊娠早期3个阶段心脏发育的三维可视化转录图谱.这些研究均展示了空间转录组学技术对发育过程中基因表达的时空特异性分析和细胞的定位具有独特的优势,能够为其分子机制的研究提供关键信息.
2.3 在肿瘤生物学中的应用
异质性是恶性肿瘤的主要特征之一,与癌症的发生发展、转移侵袭以及预后治疗等密切相关.空间转录组学技术能够直接观测到不同组织区域基因表达的异质性,因此非常适用于肿瘤组织的异质性研究.Ke等[18]最早利用ISS技术观测到乳腺癌组织中31个基因表达的异质性;Svedlund等[53]在此基础上,利用ISS技术对乳腺癌组织中91个基因的表达进行分子与形态学的联合分析,构建出肿瘤地图,揭示了与肿瘤亚型关联的肿瘤内部异质性并观测到含有少数细胞亚群的区域;此外,ISS技术还被应用于稀有突变以及基因融合的原位检测[54].Berglund等[55]利用ST技术对前列腺癌的6 750个区域进行分析,利用反卷积方法提取特异的基因表达谱,揭示了在前列腺癌中健康与肿瘤组织基因表达的变化,并在肿瘤区域周围发现了与肿瘤微环境重分层相关的基质区基因表达的梯度.Sun等[56]利用ST技术研究缺氧状态下胰腺导管癌的异质性,发现缺氧使得肿瘤细胞类群从对照组中的9个减少为7个,并对其微环境中的特异基因表达进行了分析.
此外,单细胞测序与空间转录组学技术相结合也是揭示肿瘤异质性的重要手段.Wu等[57]开发了一种固有亚型分类SCSubtype的单细胞分析方法,揭示了复发性乳腺癌肿瘤细胞的异质性,结合ST技术的分析表明不同临床亚型的相关细胞簇在空间上相互排斥.Moncada等[58]将来自胰腺导管癌患者的同一肿瘤组织的样本分布进行单细胞测序和空间转录组学分析,利用多模态交叉分析将癌细胞状态映射到不同的空间组织区域,解析它们与其他类型细胞的相互作用.Ji等[59]将单细胞转录组测序和空间转录组学技术进一步结合多路复用离子束成像技术,对一系列人皮肤鳞状细胞癌(cutaneous squamous cell carci-nomad,cSCC)与匹配的正常皮肤组织进行比较分析,鉴定了cSCC的4种肿瘤细胞亚群,包括1种癌症特有的肿瘤特异角化细胞(tumor specific keratinocytes,TSK)和3种与正常组织相同的细胞群,并且TSK细胞是细胞通讯的中心.
2.4 在其他方面的应用
除了神经科学、发育生物学、肿瘤生物学领域之外,空间转录组学在其他多个领域的研究中也开始崭露头角.Carlberg等[60]利用Visium空间转录组学技术对类风湿性关节炎(rheumatoid arthritis,RA)和脊柱关节炎(spinal arthritis,SpA)患者的滑膜组织进行建库测序,通过对差异表达基因的功能和通路进行分析,揭示了RA与适应性免疫应答相关,而SpA与细胞外基质及软骨损伤修复过程相关.Boyd等[61]利用ST技术对肺损伤的相关基因进行空间定位,为研究病毒感染导致肺损伤的机制提供了新的视角.Desai等[62]利用 NanoString 公司的 GeoMx空间转录组学技术对新冠病毒感染者的肺部组织进行分析,揭示了病毒感染过程中不同部位受到的不同影响,剖析了新冠病毒在肺部感染组织中引起的免疫反应存在异质性.在肿瘤免疫学以及免疫治疗方面,空间转录组学技术也是一种具有很大潜力的分析手段.例如Wu等[63]使用Visium平台对7名患者的21个组织标本进行空间转录组测序,结果表明不同肿瘤区域(癌区、癌旁和前沿)的免疫细胞具有高度的多样性和异质性,为肿瘤免疫治疗的个性化设计提供了参考信息.空间转录组学技术除应用于动物组织外,还在植物组织上进行了尝试[64];但是植物组织与动物组织的细胞在形态结构上有很大的差异,因此需要更多的优化和处理以保证更好的实验结果[65].可以预见,随着时间的推移空间转录组学技术将会在不同的领域继续扩大应用.
3 空间转录组学存在的挑战和机遇
空间转录组学的出现让人们以一种前所未有的视角观察基因的表达和调控网络,使人们对其功能机制有了更深入的了解[66].空间转录组学技术已经逐渐成为一项被广泛接受的组学研究新手段,然而当前空间转录组学仍然面临许多挑战.尤其是在实验技术方面,空间转录组学仍然有待改进.
首先,目前的空间转录组学技术大多数是基于薄的组织切片,获得的是基因在二维空间的表达情况,仍然无法实现真正的三维空间转录组.近期Bergenstråhle等[67]在BMCGemoimcs杂志上发表了一篇探索性文章,利用连续组织切片进行二维空间基因表达分析,然后采用生物信息学工具STUtility,将获得的二维基因表达图谱进行重构得到三维基因表达图谱.然而该方法仍然存在过程复杂、无法保证切片质量以及难以获得完美三维结构等问题,因此是一种折中的办法.由此可见,基于原位捕获和测序的空间转录组学技术存在如何捕获三维位置信息的问题,而这个问题可能需要革命性技术才能够真正解决.而基于成像的空间转录组学技术由于具有真正在原位进行检测的特点,所以在三维空间转录组学方面有一定的优势和潜力.理论上来说,只要能够对信号点进行三维原位成像,获得其位置信息并进行解码,便可以获得三维转录图谱.然而,有几个主要的技术性问题需要解决,包括如何能够使组织透明化以对组织内部的信号进行成像[68],如何使成像探针和一些方法所需要的酶分子等进入组织内部进行反应,以及如何有效地进行三维成像的大数据分析等.STARmap技术就是利用水凝胶和组织透明化技术,实现了在0.1 mm厚度的小鼠脑组织切片上的三维基因表达分析[20].虽然与常规的显微切片几微米级的厚度相比已经有数量级的增加,并且提供了第三维度的空间信息,但是相对于全脑的尺寸仍然有很大的差距.ExSeq技术也利用组织透明化技术[25],并同时引入扩展显微技术,进一步提高了空间分辨率,实现了三维空间转录组图谱的构建.
其次,目前的空间转录组学技术在单细胞层面的分辨率和基因检测效率仍然不如普通的单细胞RNA测序[69].总体来说,基于原位捕获和测序的空间转录组学技术拥有较高的基因数目检测通量,但单细胞或者亚细胞的空间分辨率较低;而基于成像的空间转录组学技术理论上来说具有更高的空间分辨率,但只有较低的基因数目检测通量(表1).然而,两者的单细胞基因数目检测效率和分辨率与普通的单细胞RNA测序相比仍然存在差距.这是因为普通的单细胞RNA测序具有更高的基因捕获效率,几乎可以无偏差捕获不同基因的转录本,并且比较容易实现单细胞之间的真正分离和分析;而基于原位捕获和测序的空间转录组学技术存在捕获点分辨率不同以及潜在的转录本扩散等问题,难以很好地区分单细胞,基于成像的空间转录组学技术则由于光学拥挤等因素导致所能分辨的基因数量有限[32].因此,这些技术性问题仍然是现有空间转录组学技术需要进一步克服的.
再者,空间转录组学数据的分析以及空间转录组数据库的建立仍然有待开发和完善.空间转录组学数据分析的主要目的是将带有位置信息的基因表达原始数据转化为有用的生物学信息,如细胞的空间定位和细胞之间的通讯等[70].空间转录组学中最重要的数据是基因表达的位置信息,而将这些基因表达数据定位到组织中的单细胞水平是目前研究者最关注的问题,因为这是实现对组织中不同类型的细胞进行分型和定位的基础.然而,现有的商业化空间转录组学技术如10x Genomics的Visium以及NanoString的GeoMx技术均未能达到单细胞的分辨率,因此只能依靠生物信息学算法来实现对部分区域内细胞类型的预测[70].而在基于成像的空间转录组学技术中,还可以利用标志基因来确定细胞类型[71].基于成像的空间转录组学技术依赖于对细胞的正确划分,即如何找到细胞边界去确定RNA分子归属的细胞.对于高表达的基因或者检测效率较高的成像技术来说,比较容易实现细胞的划分[32],而对于一些检测效率较低的技术(如ISS),则需要通过单细胞测序获得的标志物基因作为参考,并通过概率算法确定RNA分子归属的细胞,再依据表达谱确定细胞类型[34].除此之外,一些新的算法也有助于分辨图形中的单细胞及其相关的基因表达信号[72].一般来说,当细胞被正确定位和分型后,下游的分析相对来说比较容易.尽管当前空间转录组学数据在快速地增长,但尚未有对应的数据库来对这些数据进行汇总集成.为了方便研究者的使用,这样的数据库应该具备交互式访问界面以及较为全面的数据等.然而,目前这样的网站并不多,且现有网站也存在数据有限等问题[73].
总之,经过短短几年的发展,空间转录组学已经发展成一个具有多种技术解决方案且能够提供不同精度基因表达空间信息的新的组学研究手段.空间转录组学与其他组学技术相结合,将有助于人们更好地了解组织结构的细胞群体之间的位置信息及其交流通讯网络,为更好地理解组织器官甚至整个生命体如何工作提供了有力的工具.未来的空间转录组学技术将朝着更高的空间分辨率、更好的自动化实验流程以及数据分析流程发展,并且实验成本有望进一步下降.
