奶山羊ATGL基因启动子鉴定与转录活性分析
2022-05-12闫志浩贾万里许蒙蒙张景锋徐秋良韩浩园
李 君,闫志浩,贾万里,许蒙蒙,张景锋,徐秋良,韩浩园,权 凯
(1.河南牧业经济学院 动物科技学院,河南 郑州 450046;2.河南省反刍动物营养与饲料资源开发国际联合实验室,河南 郑州 450046;3.西峡县农业农村局,河南 西峡 474550)
脂肪甘油三酯水解酶(Adipose triglyceride lipase,ATGL)是脂肪水解过程中的主要限速酶,它可以特异性地水解甘油三酯为甘油二酯和游离脂肪酸,是调控细胞中甘油三酯和脂肪酸之间平衡的关键因子[1]。ATGL在成熟的棕色脂肪组织中表达量最高,主要存在于细胞的胞质内,少量分布于脂滴中。除脂肪组织外,其在肝脏、心脏、肠胃、大脑和肾脏中也有少量表达[2]。在COS-7细胞中过表达ATGL,检测到培养基中游离脂肪酸含量增加,细胞内甘油三酯储存减少,表明ATGL在甘油三酯水解过程中发挥重要作用[3]。在妊娠和泌乳这种特殊的生理阶段,乳腺具有强大的短期合成和分解脂质的能力,是机体最活跃的代谢器官。研究发现,ATGL在奶山羊的各组织中均有表达,且在脂肪组织、肺脏和乳腺组织中表达量高于其他组织;同时还发现,ATGL在泌乳的乳腺组织中表达量高于干奶期乳腺组织,且干扰ATGL显著增加乳腺上皮细胞中甘油三酯含量[4]。因此推测,ATGL在奶山羊乳腺泌乳过程中发挥重要的生物学功能。
ATGL作为脂肪第一步水解过程中的主要限速酶,控制着整个水解过程的进展速率。ATGL的表达量及活性高低对甘油三酯和脂肪酸含量的影响具有关键作用。启动子是控制基因转录活性的重要元件,研究表明,ATGL的转录活性受到营养和激素水平的调控,饥饿会激活ATGL转录、喂食后则降低其转录水平[5]。多个转录因子可调控ATGL转录,Roy等[6]研究发现,过氧化物酶体增殖物激活受体(Peroxisome proliferators-activated receptors,PPARG)与特异性蛋白1(Specificity protein 1,Sp1)相互作用共同调控ATGL的转录。Kaltenecker等[7]在小鼠脂肪细胞中研究发现,生长激素通过信号转导与转录活化蛋白5(Signal transducers and activators of transcription 5,STAT5)促进ATGL的转录,从而促进脂解。P53上调ATGL转录活性并且在脂肪细胞中促进脂解[8]。研究还表明,ATGL基因的转录受到干扰素调节因子4(Interferon regulatory factor 4,IRF4)和叉头蛋白O1(Forkhead box protein O1,FOXO1)转录因子的调节,生物信息学分析发现,ATGL的启动子区域含有至少2个FOXO1结合位点[9-10]。因此,加强对ATGL基因启动子的深入研究,将有助于掌控ATGL的转录表达。山羊奶脂肪酸含量丰富,乳脂滴小,利于消化吸收。
为了进一步提高羊乳品质,本研究在对奶山羊ATGL基因功能研究的基础上,通过对其启动子进行克隆与转录活性分析,对ATGL转录调控机制进行分析,旨在为进一步研究该基因的表达调控机制以及改善羊乳品质提供基础资料。
1 材料和方法
1.1 试验材料与试剂
血液基因组DNA提取试剂盒和DNA回收试剂盒购自北京艾德莱生物科技有限公司;LATaqDNA聚合酶、克隆载体pMD19-T、限制性内切酶BglⅡ和MluⅠ均购自TaKaRa公司;DNA Marker、感受态细胞TOP10和高纯度质粒小提中量试剂盒等购自北京天根生化科技有限公司;双荧光素酶报告系统购自Promega公司;X-tremeGENE HP DNA转染试剂购自罗氏公司;DMEM/F-12培养基、表皮生长因子、氢化可的松、胰岛素、催乳素、胎牛血清等购自Invitrogen公司。奶山羊血液和奶山羊乳腺上皮细胞由河南牧业经济学院羊生物育种实验室提供。
1.2 ATGL基因启动子的克隆
利用血液基因组DNA提取试剂盒提取奶山羊基因组DNA。参照GenBank中牛(Bostaurus)的基因组序列,利用Primer Premier 5.0设计合成山羊(Caprahircus)ATGL基因启动子克隆引物APF和APR,启动子缺失片段引物APS1、APS2、APS3、APS4、APS5、APA(表1)。以奶山羊基因组DNA为模板,PCR扩增ATGL基因启动子序列,反应体系:DNA模板(100 ng/μL)0.5 μL,上、下游引物(10 μmol/L)各1 μL,2×GC Buffer Ⅱ 10 μL,dNTP Mix(100 μmol/L)2 μL,LATaq酶0.2 μL,ddH2O补齐至20 μL;反应程序:95 ℃预变性4 min;95 ℃变性 30 s,56 ℃退火30 s,72 ℃延伸 2 min,33 个循环;72 ℃ 再次延伸10 min,12 ℃保存。利用1%琼脂糖凝胶电泳对PCR产物进行检测,将预期目的条带进行凝胶回收。将回收产物与pMD19-T于16 ℃连接4 h,然后转化入感受态细胞TOP10,挑取单克隆菌落于LB液体培养基内培养12~16 h,扩繁后用高纯度质粒提取试剂盒提取质粒,经BglⅡ和MluⅠ双酶切鉴定后筛选阳性克隆,送至上海生工生物工程有限公司测序。
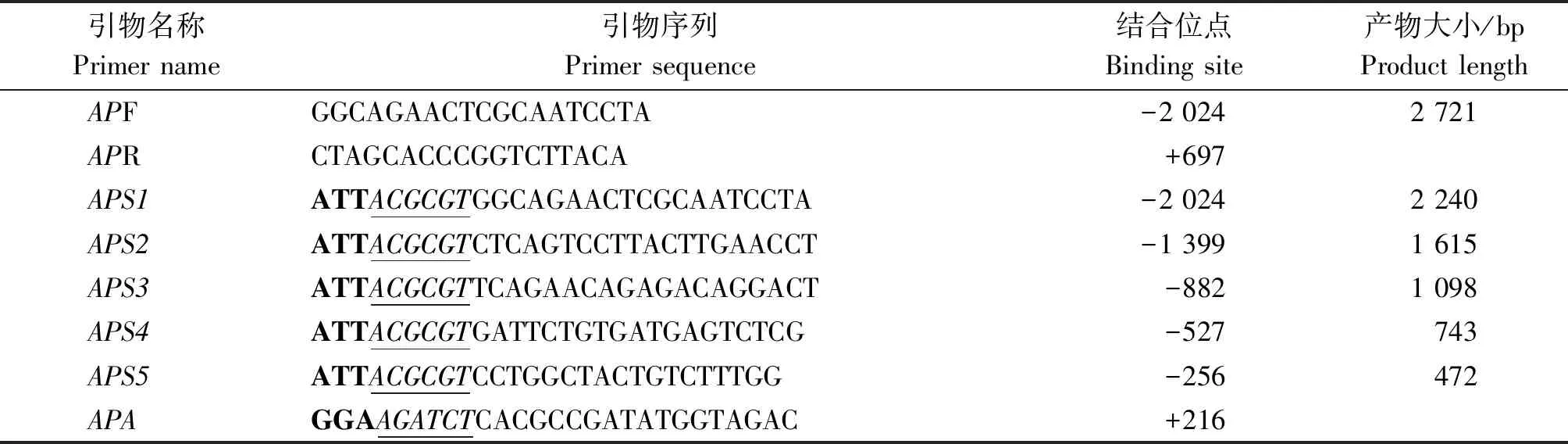
表1 奶山羊ATGL基因启动子克隆引物序列Tab.1 The sequences of primers used for cloning ATGL gene promoter of dairy goat
1.3 ATGL基因启动子的生物信息学分析
应用Promoter 2.0预测ATGL基因的启动子核心区域;ATGL基因启动子CpG岛采用CpG Islands(2004版,http://www.ebi.ac.uk/emboss/cpgplot/)在线软件进行分析;ATGL基因启动子上潜在的转录因子结合位点应用Gene Regulation(2015版,http://www.gene regulation.coml)和JASPAR(2020版,http://jaspar.genereg.net/)数据库进行预测,综合比较预测分值较高的结合位点,以确保其具有较高的特异性[11]。
1.4 ATGL启动子报告基因载体构建
根据设计的启动子缺失片段引物(表1),以含有ATGL启动子的阳性质粒为模板进行PCR扩增,得到一系列带有BglⅡ和MluⅠ酶切位点的启动子缺失片段,分别将其与pGL3-Basic载体(Promega)经双酶切后进行连接,最后得到含有ATGL启动子不同缺失片段的报告基因重组质粒,对酶切鉴定正确的质粒进一步测序以鉴定其准确性,用于后续试验。
1.5 细胞培养与转染
奶山羊乳腺上皮细胞培养采用含10% FBS、100 U/mL青霉素、100 μg/mL链霉素、10 ng/mL表皮生长因子、5 μg/mL氢化可的松和5 μg/mL胰岛素的DMEM/F12细胞培养基,于37 ℃,5% CO2培养箱中培养24 h,更换新鲜培养基,倒置显微镜下观察细胞汇合度达90%以上时,即可进行传代培养。
细胞于转染前一天铺48孔板,待细胞密度达到70%~80%时,参照转染试剂操作说明进行DNA转染。48孔板每孔转染复合物体系为:X-tremeGENE HP 1 μL,质粒 0.3 μg(pGL3-ATGL:pRL-TK =25∶1),DMEM/F12补齐至30 μL。每个处理重复3次。
将罗格列酮和T0901317溶解于DMSO中,分别配制成10 mol/L的处理液。DNA转染细胞12 h后,将配制好的罗格列酮或T0901317加入细胞中至最终的试验浓度分别为50,1 μmol/L,同时对照组用相同浓度的DMSO进行处理。培养36 h后收集细胞,用于后续双荧光素酶活性检测。
1.6 双荧光素酶活性检测
乳腺上皮细胞转染48 h后,收集细胞,按照双荧光素酶报告基因检测试剂盒操作说明测定其活性。先弃去培养基,用PBS清洗细胞2次以去除残留培养基,之后加入细胞裂解液于摇床上孵育30 min以充分裂解细胞,最后于多功能酶标仪上检测萤火虫荧光素酶活性(F值)与海肾荧光素酶活性(R值),以萤火虫与海肾的比值代表ATGL启动子的相对活性。每孔进行3次测定,取平均值。
1.7 数据分析
对于启动子活性检测结果,利用数据分析软件SPSS 22.0进行统计学分析,每个处理设置3个生物学重复,采用t检验和单因素方差分析进行显著性检验,P<0.05为差异显著,P<0.01为差异极显著。
2 结果与分析
2.1 奶山羊ATGL启动子的克隆与序列鉴定
利用引物APF和APR进行PCR扩增,成功得到约2 700 bp的ATGL基因5′侧翼序列(图1)。连接克隆载体,将鉴定为阳性的重组质粒进行测序,结果显示,成功克隆得到奶山羊ATGL基因5′侧翼序列2 721 bp。

图1 ATGL基因启动子的PCR扩增产物Fig.1 The PCR amplification products of ATGL gene promoter
2.2 ATGL启动子的生物信息学分析
经分析发现,扩增得到的ATGL基因5′侧翼序列,转录起始位点(+1)上游2 024 bp及下游697 bp,包含外显子1和部分内含子1(图2-A)。CpG岛分析结果显示,其序列中存在1个CpG岛,位置为-253—+1位,且ATGL启动子具有典型的TATA-box。经在线软件预测发现,ATGL启动子上存在多个重要的调控元件,包括FOXO1、FOXO3、SREBF1、PPARA、PPARG、C/EBPα和E2F1等转录因子结合位点(图 2-B)。
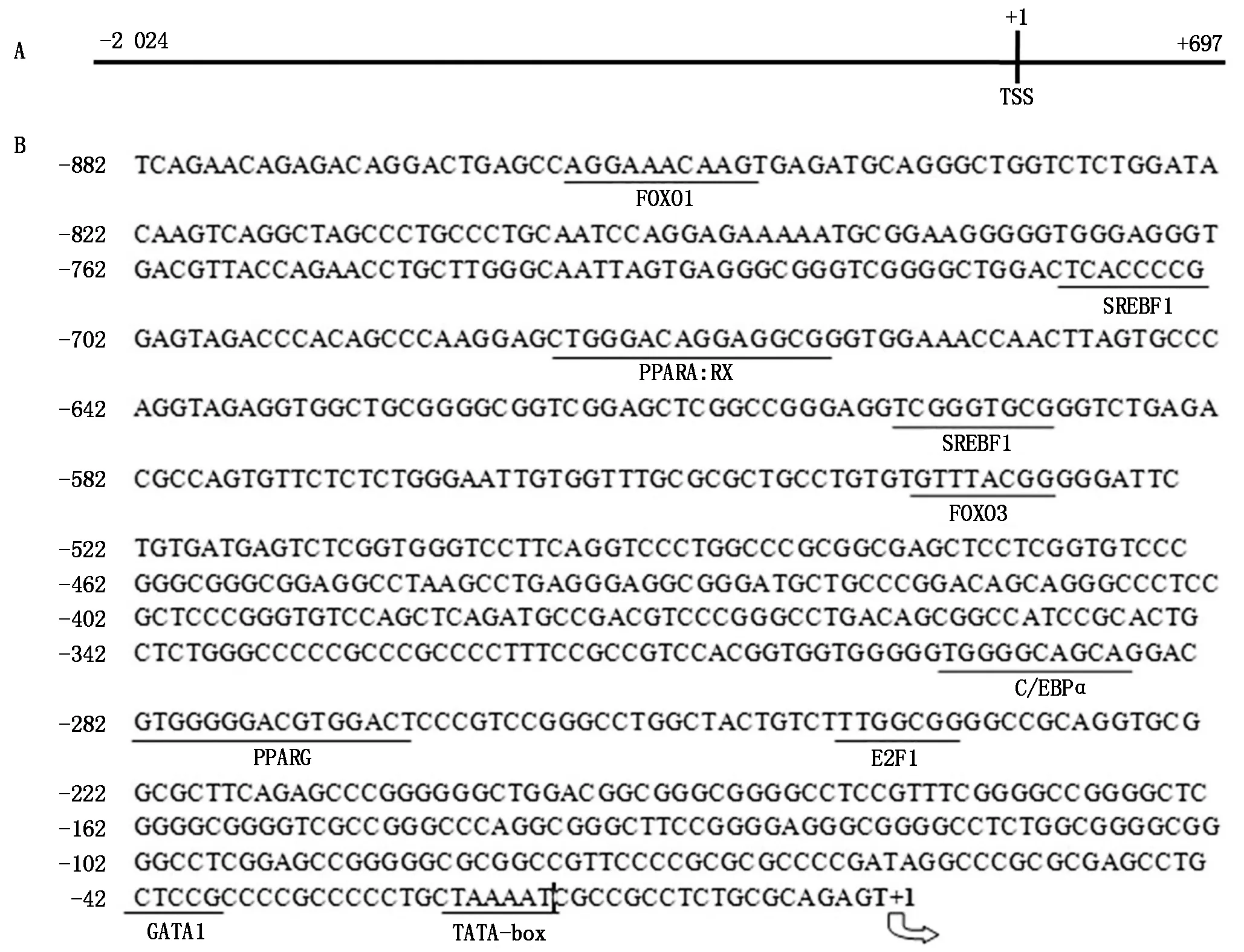
A.ATGL基因5′侧翼序列图示;B.ATGL启动子潜在的转录因子结合位点分析(+1为转录起始位点)。A.Schematic representation of ATGL 5′flanking region;B.Analysis of the putative transcription factor binding sites of the ATGL promoter(+1 is the first base at transcription start site).
2.3 ATGL启动子缺失片段载体构建
以ATGL基因5′侧翼序列全长为模板,利用一系列缺失引物进行PCR扩增,得到5个不同长度的启动子截短片段(图3-A)。通过连接pGL3-Basic报告基因载体,得到启动子缺失片段报告基因重组质粒,通过酶切鉴定(图3-B)和测序,结果证实重组质粒构建成功。
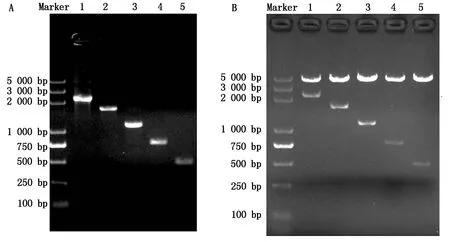
A.不同缺失片段PCR扩增产物:1.-2 024—+216位;2.-1 399—+216位;3.-882—+216位;4.-527—+216位;5.-256—+216位;B.不同缺失片段重组质粒酶切鉴定:1.pGL-APS1(-2 024—+216位);2.pGL-APS2(-1 399—+216 位);3.pGL-APS3(-882—+216位);4.pGL-APS4(-527—+216位);5.pGL-APS5(-256—+216位)。
2.4 ATGL基因启动子核心区域确定
将ATGL缺失片段报告基因载体和内参质粒(海肾荧光素酶载体)共转染奶山羊乳腺上皮细胞,48 h后收集细胞检测相对荧光素酶活性。结果表明,ATGL启动子区域由-2 024位缺失到-882位时,启动子活性无显著变化(P>0.05);由-882位缺失到-527位时,其活性显著降低(P<0.05);由-527位缺失到-256位时,启动子活性又显著上升(P<0.05)(图4)。说明启动子区域-527—-256位可能存在抑制元件,推测转录起始位点上游-256 —+1位为ATGL基因启动子转录核心区域。

pGL3-Basic.荧光素酶报告基因空载体;LUC.不同缺失片段的荧光素酶报告基因载体;不同字母表示差异显著(P<0.05)。图5—6同。pGL3-Basic.Luciferase reporter gene empty vector;LUC.Luciferase reporter gene vector with various deletion fragments;Different letters indicate significant differences(P<0.05).The same as Fig.5—6.
2.5 罗格列酮对ATGL启动子活性的影响
对乳腺上皮细胞转染pGL-APS1重组质粒,用PPARG特异性激动剂罗格列酮(ROSI)处理细胞,结果发现,罗格列酮显著上调ATGL启动子活性(P<0.05)(图5-A)。接着将活性变化较为显著的pGL-APS3、pGL-APS4和pGL-APS5重组质粒转染奶山羊乳腺上皮细胞,并用罗格列酮处理,结果发现,APS3和APS4均能响应罗格列酮信号(图5-B),表明PPARG调控奶山羊ATGL启动子活性,且作用区域位于-527—-256位。
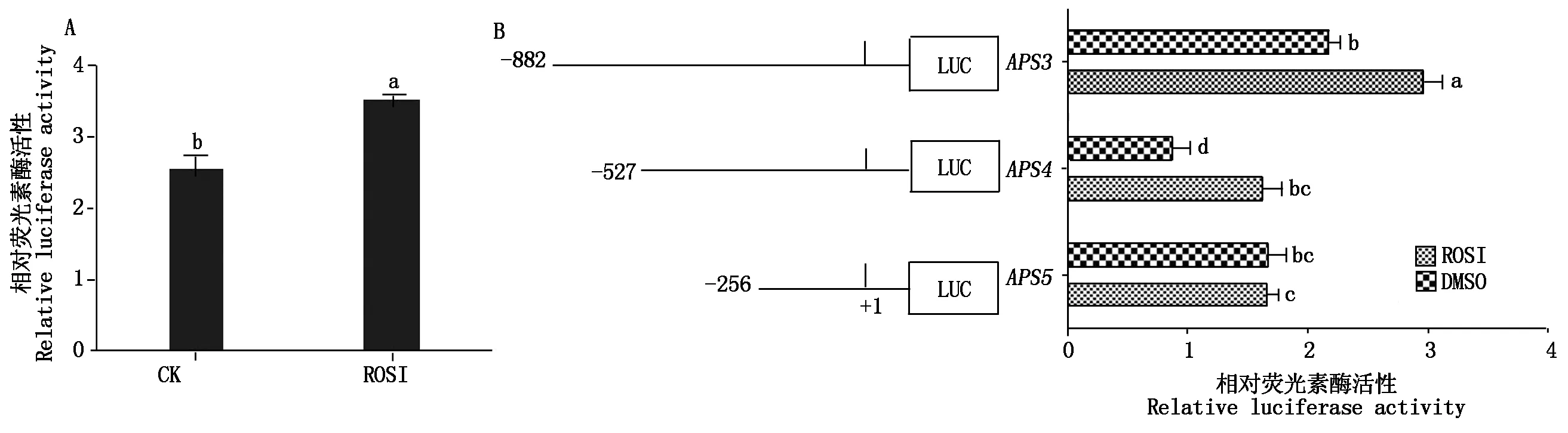
A.罗格列酮对ATGL启动子全长活性的影响;B.罗格列酮对ATGL启动子不同片段的影响。A.Effect of ROSI on the activity of ATGL promoter in full length;B.Effect of ROSI on the activity of various deletion fragments of ATGL promoter.
2.6 T0901317对ATGL启动子活性的影响
以同样的方法在乳腺上皮细胞中转染pGL-APS1重组质粒,用SREBP1激动剂T0901317处理细胞发现,T0901317显著上调ATGL启动子活性(P<0.05)(图6-A);而在不同缺失片段中,T0901317仅对APS3有显著上调作用(P<0.05),对APS4和APS5没有显著影响(P>0.05)(图6-B),表明SREBP1参与调控奶山羊ATGL基因转录,且其作用区域位于-882—-527位。生物信息学分析发现,该片段中-710—-703位和-600—-592位存在预测的SREBP1结合位点,T0901317可能通过这2个位点发挥调控作用。
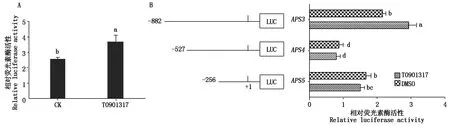
A.T0901317对ATGL启动子全长活性的影响;B.T0901317对ATGL启动子不同片段的影响。A.Effect of T0901317 on the activity of ATGL promoter in full length;B.Effect of T0901317 on the activity of various deletion fragments of ATGL promoter.
3 结论与讨论
ATGL作为甘油三酯水解的限速酶,可以催化动物脂肪组织和非脂肪组织的甘油三酯水解[12],前期的研究发现,ATGL在奶山羊乳腺组织中高表达,且在乳腺上皮细胞中发挥重要作用,ATGL影响细胞中甘油三酯含量和游离脂肪酸含量,同时影响脂肪酸代谢相关基因表达水平[4]。基于前期的这些研究成果,对奶山羊ATGL基因启动子的结构与转录活性进行分析,有助于阐明ATGL基因在乳腺细胞中的转录调控机制,对于研究羊奶中乳脂和乳脂肪酸的调控作用具有重要意义。
不同的组织和细胞含有相同的DNA序列,而组织和细胞功能的多样性则是由基因的差异表达实现的,因此,研究基因的转录调控对理解生命活动具有重要意义。转录调控主要是通过转录因子结合靶基因启动子上特定DNA序列来实现,启动子上存在多种调控元件,这些元件的数量、种类以及它们之间的距离与顺序都有可能影响基因的转录,它们通过激活或抑制基因转录,对基因的表达差异发挥作用[13]。ATGL基因在脂肪细胞中受到多种转录因子调控,其5′调控区含有E-box、TATA box、CAAT box等结合位点[14],多种转录因子可以识别基因5′调控区的特异位点并与之作用调控ATGL基因的表达[15]。本研究以河南奶山羊基因组DNA为模板,扩增出ATGL基因5′侧翼序列2 721 bp,参考其他物种的ATGL启动子序列和生物信息学分析发现,转录起始位点上游序列包含典型的启动子元件(TATA-box),这些结果与在猪上的研究结果一致[16]。在线软件预测发现,奶山羊ATGL启动子中还含有FOXO1、FOXO3、SREBF1、PPAR和CEBPα等转录因子结合位点,这些结合位点和猪上ATGL启动子预测结果相一致[17]。通过缺失分析发现,从-882位缺失到-527位时启动子活性显著下降,表明此区域含有正调控元件,而从-527位缺失到-256位时,启动子活性反而上调,表明此区域含有抑制其活性的元件。经过分析确定其核心区域在-256—+1位,这与孙健[16]在草鱼上和王小宇[17]在猪上的研究报道结果基本一致,但是二者报道的ATGL启动子活性区域更小。
PPARG已被证实在奶牛和奶山羊的乳腺脂肪酸调控网络中发挥重要作用,其影响脂肪酸的从头合成、甘油三酯的合成和分泌相关基因的表达[18-20],PPARG通过结合在PPAR 反应元件上对下游基因表达进行调控[21]。Shi等[22]研究发现,在奶山羊乳腺上皮细胞中,罗格列酮和PPARG过表达影响奶山羊ATGL基因mRNA水平,在多个物种的ATGL启动子上均存在PPARγ结合位点[23]。有研究发现,在脂肪细胞中PPARG抑制ATGL基因的转录,但是在脂肪细胞分化后期,PPARG促进ATGL基因的转录,这种促进作用是通过与转录因子Sp1 的共同作用完成,说明PPARG对ATGL的调控具有时空特异性[6]。本试验中,通过在线软件预测到在ATGL启动子的-282—-267位存在潜在的PPARG结合位点,通过PPARG特异性激动剂处理细胞发现,罗格列酮在截短片段的-527—-256位发挥作用,表明PPARG调控ATGL基因转录,这与前人的研究结果一致。
SREBP1是调控脂肪合成的主要转录因子,SREBP1核蛋白在运输进入细胞核后与靶基因启动子上的位点结合从而激活靶基因的转录[24-25]。研究证实,LXRα可直接结合在SREBP1的启动子元件上,对其进行转录调控[26]。而LXRα的人工合成激动剂T0901317又被广泛地作为SREBP1的激动剂应用于SREBP1的功能研究中[27]。Xu等[28]研究发现,过表达SREBP1影响ATGL基因mRNA表达。本研究通过T0901317处理细胞,发现其显著上调ATGL启动子活性,且作用区域位于-882—-527位;生物信息学分析发现,奶山羊ATGL启动子-710—-703位和-600—-592位存在预测的SREBP1结合位点。表明SREBP1可能是ATGL基因的转录因子,促进ATGL基因转录,但是关于SREBP1对ATGL启动子的调控作用及其作用位点目前还未见报道,在奶山羊上还需进一步的试验验证。
本研究成功克隆奶山羊ATGL启动子序列,利用生物信息学分析了ATGL启动子序列中的调控元件结合位点,构建了不同片段长度的ATGL启动子报告基因载体,启动子活性分析表明其核心区域位于-256—+1位,且PPARG和SREBP1能够促进ATGL基因的启动子活性,且作用区域分别位于-527—-256位和-882—-527位。
