早发冠心病血瘀证DNA羟甲基化相关差异基因研究
2022-04-28江雨洁张湘卓陈悦张书萌于子璇刘佳陈伶利李杰
江雨洁 张湘卓 陈悦 张书萌 于子璇 刘佳 陈伶利 李杰


〔摘要〕 目的 探討早发冠心病(premature coronary heart disease, PCHD)血瘀证DNA羟甲基化相关差异基因表达,以期寻找PCHD血瘀证诊断和治疗的新靶点。方法 病例来自湖南中医药大学第二附属医院PCHD患者,分为PCHD血瘀证组(A组)和PCHD非血瘀证组(B组),各6例,采用DNA羟甲基化免疫共沉淀芯片技术分析两组的相关羟甲基化差异基因表达情况,并对其进行聚类分析、KEGG通路富集分析及GO功能富集分析。结果 A组与B组相比,高CpG密度启动子差异羟甲基化基因数目343个,低CpG密度启动子差异羟甲基化基因数目144个,通路富集分析得出PCHD血瘀证羟甲基化差异基因主要与细胞周期、心肌能量代谢、炎症、内皮损伤有关,GO功能富集分析得出PCHD血瘀证生物学功能多与细胞发育和分化、蛋白酶活性、细胞内结构等有关。A组的CDKN1B、HDAC2、MDM2羟甲基化表达程度均较高。结论 PCHD血瘀证差异基因羟甲基化表达程度较高,这些差异基因主要与转录调控心肌细胞周期有关,可能影响心肌细胞周期阻滞、凋亡、衰老等,导致疾病的发生发展,为PCHD血瘀证诊断和治疗提供一定的参考。
〔关键词〕 早发冠心病;血瘀证;DNA羟甲基化;差异基因;表观遗传学
〔中图分类号〕R25 〔文献标志码〕A 〔文章编号〕doi:10.3969/j.issn.1674-070X.2022.04.022
Study on the DNA hydroxymethylation related difference genes in premature coronary heart
disease with blood stasis syndrome
JIANG Yujie, ZHANG Xiangzhuo, CHEN Yue, ZHANG Shumeng, YU Zixuan, LIU Jia, CHEN Lingli*, LI Jie*
(Hunan University of Chinese Medicine, Changsha, Hunan 410208, China)
〔Abstract〕 Objective To explore the differential expression of DNA hydroxylmethylation related genes in premature coronary heart disease (PCHD) with blood stasis syndrome, in order to find a new target for diagnosis and treatment of PCHD with blood stasis syndrome. Methods The patients were from PCHD patients in The Second Affiliated Hospital of Hunan University of Chinese Medicine. They were divided into PCHD with blood stasis syndrome group (group A) and PCHD with non-blood stasis syndrome group (group B), with 6 patients in each group. DNA hydroxymethylated immunoprecipitation chip technique was used to analyze the differential gene expression of related hydroxymethylation between the two groups. Cluster analysis, KEGG pathway enrichment analysis and GO function enrichment analysis were performed. Results Compared with group B, group A had 343 differential hydroxymethylation genes with high CpG density promoter and 144 differential hydroxymethylation genes with low CpG density promoter. Pathway enrichment analysis showed that the differential genes of hydroxymethylation in PCHD with blood stasis syndrome were mainly related to cell cycle, myocardial energy metabolism, inflammation and endothelial injury. Go function enrichment analysis showed that the biological functions of PCHD with blood stasis syndrome were mostly related to cell development and differentiation, protease activity, intracellular structure and so on. The degree of hydroxymethylation of CDKN1B, HDAC2, MDM2 in group A was higher than that in group B. Conclusion The expression degree of hydroxymethylation of differential genes in PCHD with blood stasis syndrome is higher. These differential genes are mainly related to transcriptional regulation of myocardial cell cycle, which may affect cardiomyocyte cycle arrest, apoptosis, senescence and so on, leading to the occurrence and development of the disease, which provides a reference for the diagnosis and treatment of PCHD with blood stasis syndrome.
〔Keywords〕 premature coronary heart disease; blood stasis syndrome; DNA hydroxymethylation; differential gene; epigenetics
冠心病(coronary heart disease, CHD)已成为全球重大公共卫生问题之一[1],其发病及病理机制尚未阐述清楚,但已知CHD是一种典型的多因素复杂疾病,由遗传因素和环境因素相互作用而引起[2-3],而表观遗传机制很好地体现了环境和基因的交互作用[4]。DNA羟甲基化修饰是表观遗传学中重要修饰形式之一,参与了多种疾病的病理生理改变,目前在心血管领域日益成为研究的热点[5-6]。早发冠心病(premature coronary heart disease, PCHD)是指冠心病发病年龄男性<55岁和女性<65岁的冠心病[7],已有文献报道PCHD与脂质代谢基因密切相关[8],本课题组前期研究表明PCHD血瘀证与CCNA2、ARF6、PSD2基因启动子羟甲基化水平密切相关[9-10],CHD中医病名为胸痹,血瘀证是胸痹最常见的证型之一[11],进一步深入研究羟甲基化修饰PCHD血瘀证相关差异基因有利于该疾病的防治。本研究以PCHD血瘀证及非血瘀证患者为研究对象,应用DNA羟甲基化免疫共沉淀芯片技术分析PCHD血瘀证相关差异基因表达,探讨疾病与基因之间的关系,以期寻找PCHD血瘀证诊断和治疗的新靶点。
1 资料与方法
1.1 诊断标准
(1)PCHD诊断标准[7]:CHD发病年龄男<55岁,女<65岁。(2)CHD诊断标准[12]:冠脉造影提示至少有1支冠状动脉直径狭窄≥50%。(3)中医辨证标准:血瘀证辨证标准参照《中华人民共和国中医药行业标准·中医病症诊断疗效标准》[13]。心胸阵痛,如刺如绞,固定不移,入夜为甚,伴有胸闷心悸,面色晦暗;舌质紫暗,或有瘀斑,舌下络脉青紫,脉沉涩或结代为心血瘀阻证。(4)CHD血瘀证评分标准:血瘀证评分参照中国中西医结合学会活血化瘀专业委员会制定的《冠心病血瘀证诊断标准》[14]。①主要指标:胸痛位置固定,舌质色紫暗,舌有瘀斑瘀点,冠脉造影显示至少一支冠状动脉狭窄≥75%,超声或造影显示有冠状动脉血栓或心腔内附壁血栓;②次要指标:胸痛夜间加重,口唇或齿龈紫暗,舌下静脉曲张或色紫暗,冠脉造影显示至少一支冠状动脉狭窄≥50%且<75%,部分凝血活酶时间(acivated partial thromboplastin time, APTT)或凝血酶原时间(prothrombin time, PT)缩短;③辅助指标:面色黧黑,脉涩,冠脉非创伤性血管成像技术或冠脉造影显示血管明显钙化或弥漫病变,纤维蛋白原升高。诊断须包含主要指标、次要指标中至少1项宏观指标,CHD血瘀证计分≥19分可诊断为血瘀证,计分高低可用于评价CHD血瘀证的程度,不符合血瘀证标准即为非血瘀证。
1.2 纳入标准
(1)符合上述PCHD患者年龄、CHD诊断标准和中医辨证分型标准;(2)参与研究者知情同意。
1.3 排除标准
CHD患者兼有以下各种疾病者:(1)甲状腺功能亢进性心肌病、糖尿性心肌病、肺源性心肌病、高血压性心肌病、风湿性心肌病、贫血性心肌病;(2)恶性肿瘤、甲状腺疾病、严重肝肾疾病、血液系统疾病、结缔组织疾病、传染病;(3)精神系统疾病;(4)受检者依从性差,或不能完成全部临床检测,资料不全者。
1.4 一般资料
所有的样本来自2019年10月13日至2020年1月10日湖南中医药大学第二附属医院心血管内科患者,并通过湖南中医药大学第二附属医院医学伦理审批委员会的审批,根据纳排标准从中选取PCHD血瘀证组(A组)和PCHD非血瘀证组(B组),各6例。两组一般资料比较,差异无统计学意义(P>0.05),具有可比性,见表1。
1.5 标本采集
每例受试者空腹8 h,留取肘静脉血10 mL,用EDTA二钠抗凝,室温静置2 min,-80 ℃储存备用。
1.6 DNA羟甲基化免疫共沉淀芯片分析
使用DNeasy血液组织试剂盒提取全血中基因组DNA,然后用NanoDrop 1000超微量分光光度计对DNA进行定量,再通过琼脂糖凝胶电泳分析DNA的完整性和质量。DNA质检合格后,参照芯片标准流程进行样本的标记、芯片杂交及清洗。首先,本研究选用的芯片为Arraystar Human 4x180K RefSeq Promoter Microarray,是一款在RefSeq基因启动子区域内检测表观遗传修饰和转录因子结合位点的芯片,芯片以210 bp的间隙用大约180 000根探针覆盖了23 148个基因启动子区域。然后,用Cy5-9mer、Cy3-9mer引物对样本进行标记,标记好的DNA纯化后再进行芯片杂交,芯片洗涤后使用Agilent微阵列扫描仪对芯片进行扫描,该过程均由上海康成生物有限公司完成。
1.7 统计学分析
采用SPSS 25.0软件统计分析数据。計量资料以“x±s”表示,符合正态分布、方差齐者,采用t检验,反之则用秩和检验;计数资料用“%”表示,采用卡方检验。以P<0.05为差异具有统计学意义。
1.8 生物信息学分析
1.8.1 羟甲基化富集峰分析 使用Nimblescan v2.5提供的滑动窗口(1500 bp)峰值算法从标准化的log2-ratio数据开始查找分析MeDIP芯片数据。并确定这些探针的log2-ration值分布是否符合正态性,然后通过Kolmogorov-Smirnov检测得到的-log10 p-value值,均来自特定探针滑动窗口。若发现相邻探针的-log10 p-value值明显高于设定的阈值,那么这几个探针区域就是一个富集峰(enrichment peak, EP)。EP必须是至少大于两个探针最小p-value的阈值。
1.8.2 羟甲基化差异富集峰分析 当进行两组样品比较来筛选差异富集峰(differential enrichment peak, DEP)即差异羟甲基化区域时,使用每组探针的log2-ratio的均值来计算探针的M'值。M'=PCHD血瘀证组的Average(log2 MeDIPE/Input)-PCHD非血瘀证组的Average(log2 MeDIPC/Input),然后基于M'值再次运行NimbleScan的peak-finding程序来搜索羟甲基化peaks。当两组中至少有一组的median(log2 MeDIP/Input)≥0.3且median(M')>0并在两组中任意一个peak对应的探针至少有一半变异系数≤0.8即筛选为差异羟甲基化区域。启动子在转录起始位点的上游0.7 kb至下游0.2 kb有一个500 bp的区间,该区间的(G+C)-fraction≥0.55,并且CpG的观测/期望比≥0.6,则为高CpG密度启动子,说明羟甲基化程度高。启动子不包含CpG的观测/期望比≥0.4的500 bp长的区域,则为低CpG密度启动子,说明羟甲基化程度低。通过聚类分析直观展示DEP探针在样本中的DNA羟甲基化情况,然后对差异基因进行通路富集分析和基因本体(gene ontology, GO)富集分析,以探讨羟甲基化差异基因主要影响的通路和生物学功能。
2 结果
2.1 启动子羟甲基化差异基因数目
A组与B组相比,高CpG密度启动子差异羟甲基化基因数目343个,低CpG密度启动子差异羟甲基化基因数目144个。可见PCHD血瘀证与非血瘀证相比差异基因羟甲基化修饰程度较高。
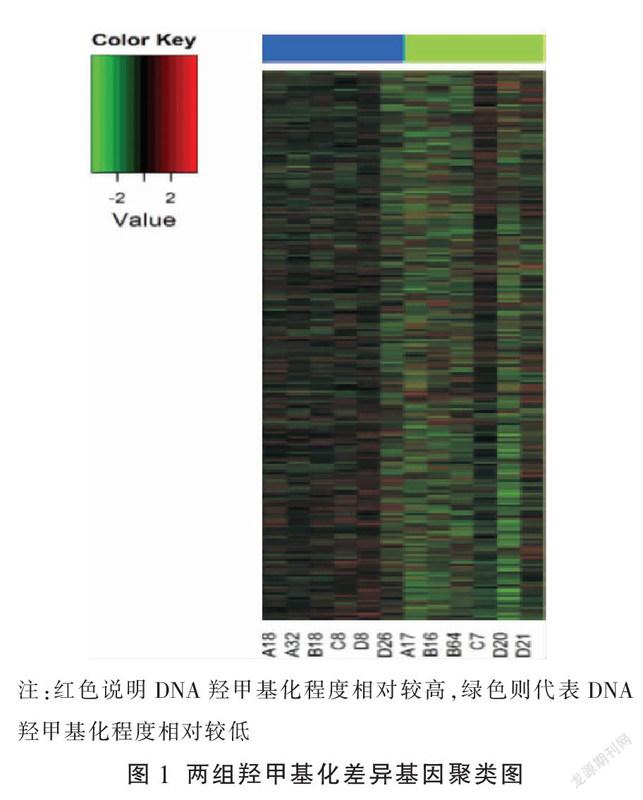
2.2 差异羟甲基化DEP聚类分析
对DEP的结果做聚类分析,聚类图以直观的展示DEP的探针在各个样本中的DNA羟甲基化情况,B组羟甲基化程度低于A组,见图1。
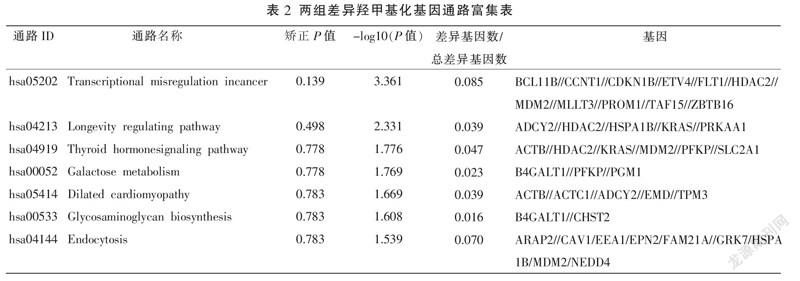
2.3 羟甲基化差异基因通路分析
使用KEGG数据库分析两组羟甲基化差异基因所在显著性(P<0.05)富集通路,主要与癌症转录失调、长寿调节途径、甲状腺激素信号通路、半乳糖代谢、扩张型心肌病、黏多糖生物合成、内吞作用等通路相关,见表2。
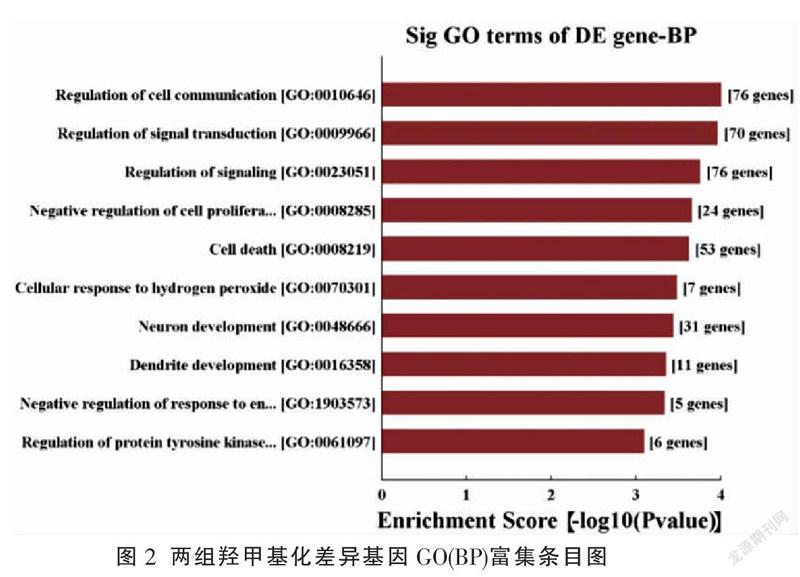
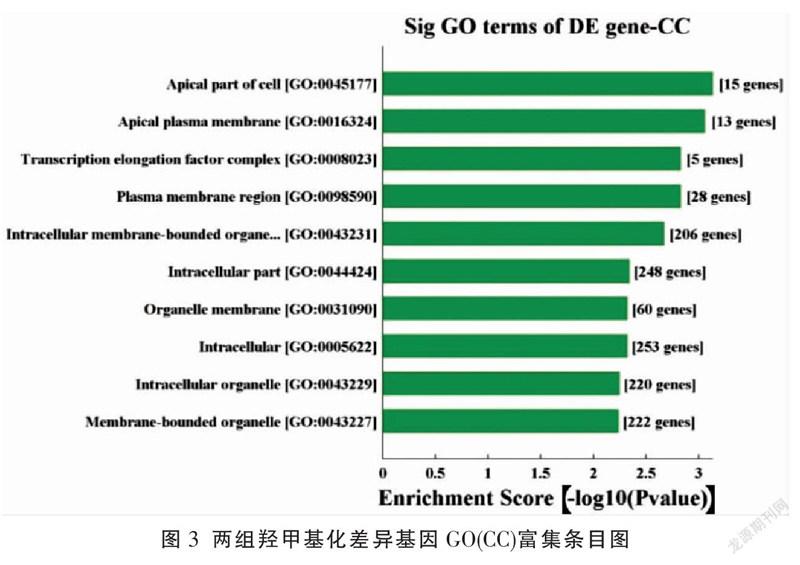
2.4 羟甲基化差异基因GO富集分析
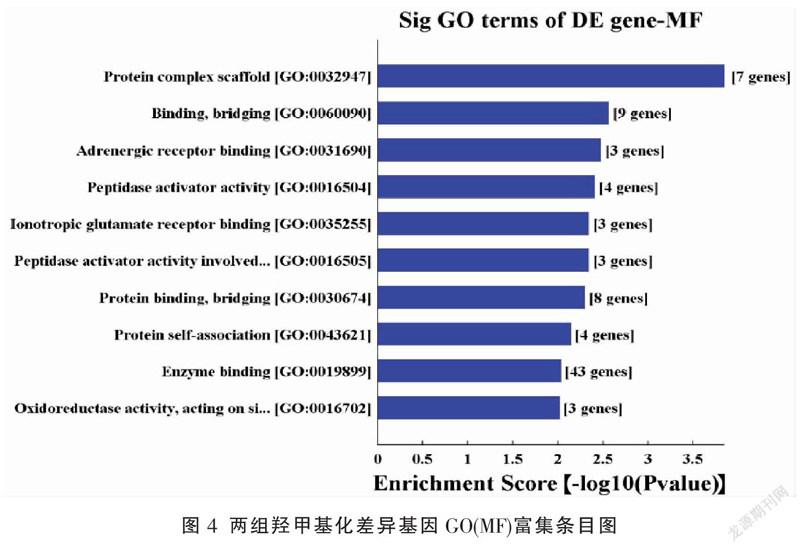
根据芯片检测结果,对羟甲基化差异基因进行GO功能分析。GO数据库总共有三大类,分别是生物学过程(biological process, BP)、细胞组分(cellular component, CC)和分子功能(molecular function, MF),各自描述了基因产物可能行使的分子功能,所处的细胞环境,以及参与的生物学过程。A组与B组相比,羟甲基化差异基因BP差异具有统计学意义(P<0.05),主要与细胞通讯调节、信号转导调节、信号调节、细胞增殖的负调控、细胞死亡、细胞对过氧化氢的反应、神经元发育、枝晶发育、内质网应激反应的负调控、蛋白酪氨酸激酶活性的调节等相关;CC差异具有统计学意义(P<0.05),主要与细胞顶端、顶端质膜、转录延伸因子复合物、质膜区、细胞内膜结合细胞器、细胞外、细胞器膜、细胞内、细胞内细胞器、膜结合细胞器等相关;MF差异具有统计学意义(P<0.05),主要与蛋白质复合物支架、肾上腺素能受体结合、肽酶激活剂活性、肽酶激活剂活性参与细胞凋亡过程、离子型谷氨酸受体结合、蛋白质结合、蛋白质自缔合、酶结合、氧化还原酶活性等相关。见图2-4。
2.5 启动子羟甲基化差异基因筛选
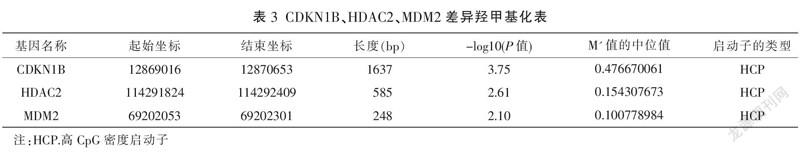
根据通路富集分析、GO功能分析,结合P值、M'值的中位值等指标,筛选CDKN1B、HDAC2、MDM2 3个差异基因,见图5、表3。
3 讨论
中医学认为,CHD与外邪侵袭、七情内伤、膏粱厚味、年老久病等因素相关。该病病机复杂,总属本虚标实,心脉痹阻。胸痹与血瘀证息息相关,中医古籍对此有大量的记载,如《素问·脉要精微论》“夫脉者,血之府也……涩则心痛”,指出瘀血阻滞、心脉不通,可导致心痛。《素问·调经论》曰:“厥气上逆,寒气积于胸中而不泻,不泻则湿气去,寒独留,则血凝泣,凝则脉不通”,指出寒凝血瘀,凝滞心脉。《肘后备急方》曰:“胸痹之病,令人心中坚辟急痛,绞急如刺,不得仰俯,或血脉瘀阻”,可见血瘀证为CHD发生发展的主要病因。 DNA羟甲基化是近几年来发现的一种DNA修饰方法,在生物学上的重要性及其作为表观遗传标记的作用近年来才得到重视。在染色体10/11易位(ten-eleven translocation, TET)家族蛋白酶催化下,DNA CpG岛的5-甲基胞嘧啶(5-methylcytosine, 5mC)氧化成5-羟甲基胞嘧啶(5-hydroxymethylcytosine, 5hmC)这一过程称为DNA羟甲基化[15]。越来越多的证据表明,作为TET家族成员的5hmC及其TET2酶在动脉粥样硬化中起着重要作用,不僅参与血管平滑肌细胞表型的调节,而且与内皮功能障碍和炎症免疫反应密切相关[16-19]。有研究发现,冠心病患者与对照组基因体中5hmC的富集有显著差异,用5hmC标记可以很好地将冠心病患者与对照组个体区分,循环游离DNA中的5hmC标记对心肌梗死的预测潜力优于心肌肌钙蛋白和肌红蛋白[20]。DNA基因体或启动子区等部位发生羟甲基化修饰后能促进基因的表达,直接影响基因组的结构和功能,从而调节细胞发育和干细胞的多能性,而它的异常改变是许多疾病发生基础[21]。故而本研究以PCHD血瘀证患者为研究对象,利用DNA羟甲基化免疫共沉淀芯片技术分析其差异基因的表达,以探讨其发病及病理机制。结果显示,共有487个羟甲基化差异基因与PCHD血瘀证相关,其中,高CpG密度启动子差异羟甲基化基因数目343个,低CpG密度启动子差异羟甲基化基因数目144个。
通路富集分析得出PCHD血瘀证与细胞周期、心肌能量代谢、炎症、内皮损伤关系密切。内吞作用是细胞从细胞表面除去配体、营养物质、质膜蛋白和脂质,并将其带入细胞内部的一种机制。有研究表明,心肌梗死后心脏内蛋白复合体2被磷酸化,提示缺氧条件可能触发网格蛋白介导的内吞作用,网格蛋白介导的心肌细胞内吞将循环miRNA转运到细胞中起关键作用,从而发挥抗心肌细胞凋亡作用[22]。亦有研究表明,小窝蛋白(caveolin)参与低密度脂蛋白内吞作用,介导动脉粥样硬化[23]。本研究结果发现,内吞作用富集显著且位于该通路的小窝蛋白呈高羟甲基化状态可能通过参与内吞作用介导心肌细胞周期及内皮损伤有关。长寿调节通路是介导衰老的重要通路,衰老是分子、细胞和器官损伤累积的复杂过程,可导致功能丧失,增加疾病与死亡的易感性。研究发现,与衰老相关的遗传基因与冠心病风险关系密切[24]。本研究结果发现,长寿调节途径中的富集显著且位于该通路的HDAC2呈高羟甲基化状态,可能与该通路参与调节衰老有关。GO功能富集分析得出,PCHD血瘀证生物学功能多与细胞发育和分化、蛋白酶活性、细胞内结构等有关。
细胞周期蛋白依赖激酶抑制剂1B(cyclin depe?鄄
ndent kinase inhibitor 1B, CDKN1B),别名KIP1、MEN4、CDKN4、MEN1B、P27,该基因编码的蛋白与细胞周期素E-CDK2或细胞周期素D-CDK4复合物结合并阻止其激活,从而控制细胞周期在G1期的进程[25]。心肌细胞周期的改变可导致诸多心血管疾病,故而通过调节CDKN1B的表达可能会成为治疗心血管疾病的关键介质[26]。研究发现,CDKN1B的过度表达能选择性抑制巨噬细胞的增殖,从而阻止动脉粥样硬化的进展[27]。本研究CDKN1B在PCHD血瘀证中呈高羟甲基化状态,故推测CDKN1B可能导致细胞凋亡或自噬缺陷,控制G1细胞周期进程,从而影响PCHD血瘀证进程。
组蛋白脱乙酰酶2 (histone deacetylase 2, HDAC2),负责核心组蛋白N端赖氨酸残基的脱乙酰化,它在转录调控、细胞周期进程和发育事件中起着重要作用[28]。越来越多的证据表明,该基因调控心肌肥厚、纤维化、凋亡和能量代谢等过程,是内皮依赖性血管舒张和动脉粥样硬化的关键调节因子,抑制HDAC2被认为是抗纤维化、抗肥厚的有效方法[29-31]。Zhang 等[32]研究发现,TET2通过HDACs介导IL-6的活性抑制,从而在炎症消退过程中清除组蛋白乙酰化,表明在炎症消退过程中,HDAC2是组蛋白去乙酰化介导的抑制IL-6的关键酶。本研究中发现该基因呈高羟甲基化水平,可能与其转录调控心肌细胞周期和介导炎症有关。
MDM2癌基因(murine double minute 2, MDM2),别名ACTFS、HDMX、HDM2,该基因编码核定位的E3泛素连接酶,編码的蛋白可通过靶向肿瘤抑制蛋白以促进蛋白酶体降解来促进肿瘤形成。目前,关于MDM2羟甲基化在PCHD血瘀证方面的研究尚未报道,相关报道主要集中在癌症方面,Ye等[33]研究发现,MDM2在肝细胞癌中呈低羟甲基化水平,细胞周期通路富集显著。该基因受肿瘤蛋白p53(tumor protein p53, TP53)转录调控,调节靶基因的表达,诱导细胞周期阻滞、凋亡、衰老、DNA修复或代谢改变[34]。TP53为平滑肌细胞进入增殖期的负性生长调节剂,影响心脏结构、收缩力、线粒体生物发生、糖酵解和氧化磷酸化能力,是心脏转录组的主要调节因子[35-36],并通过调控MDM2的表达影响心肌细胞凋亡、衰老等方面在心血管疾病中发挥重要作用[37-38]。同时,CDKN1B、HDAC2均与TP53相关,CDKN1B表达受TP53密切调控,并介导TP53依赖的G1细胞周期在多种应激刺激下的阻滞,HDAC2为TP53的共激活剂,参与DNA损伤后的早期分子事件[39]。其关系如图5所示,提示这3个差异基因在PCHD血瘀证中呈高羟甲基化水平与调控心肌细胞周期密切相关。
综上所述,CDKN1B、HDAC2、MDM2可能直接或间接转录调控心肌细胞周期,在PCHD血瘀证中均呈高羟甲基化水平,可能影响心肌细胞周期阻滞、凋亡、衰老等,导致疾病的发生发展。但其具体的作用机制仍需深入探索,因此,后续将进一步通过RT-PCR验证本研究筛选出的差异基因,同时也会扩大样本量及实验组别获取更多的实验数据,提高实验的科学性、精确性、严谨性,进一步利用中医药多靶点、多途径的干预以达到精准治疗的目的。
参考文献
[1] VIRANI S S, ALONSO A, APARICIO H J, et al. Heart disease and stroke statistics-2021 update: A report from the American heart association[J]. Circulation, 2021, 143(8): e254-e743.
[2] ORHO-MELANDER M. Genetics of coronary heart disease: Towards causal mechanisms, novel drug targets and more personalized prevention[J]. Journal of Internal Medicine, 2015, 278(5): 433-446.
[3] KHERA A V, EMDIN C A, DRAKE I, et al. Genetic risk, adherence to a healthy lifestyle, and coronary disease[J]. The New England Journal of Medicine, 2016, 375(24): 2349-2358.
[4] LIPPI G, CERVELLIN G. The interplay between genetics, epigenetics and environment in modulating the risk of coronary heart disease[J]. Annals of Translational Medicine, 2016, 4(23): 460.
[5] JIANG D, SUN M, YOU L N, et al. DNA methylation and hydroxymethylation are associated with the degree of coronary atherosclerosis in elderly patients with coronary heart disease[J]. Life Sciences, 2019, 224: 241-248.
[6] GRECO C M, KUNDERFRANCO P, RUBINO M, et al. DNA hydroxymethylation controls cardiomyocyte gene expression in development and hypertrophy[J]. Nature Communications, 2016, 7: 12418.
[7] TONSTAD S, WESTHEIM A. Implementation of guidelines to screen relatives of patients with premature coronary heart disease in a hospital setting[J]. The American Journal of Cardiology, 2002, 90(11): 1211-1214.
[8] 彭 琴,周琴怡,黄 柯,等.早发冠心病相关脂质代谢基因变异的研究进展[J].中国动脉硬化杂志,2021,29(3):264-270.
[9] 陈 悦.基于羟甲基化修饰探讨早发冠心病血瘀证与ARF6、PSD2易感基因的相关性研究[D].长沙:湖南中医药大学,2019.
[10] 吴喜庆.早发冠心病血瘀证外周血CCNA2、TBC1D1基因启动子羟甲基化水平研究[D].长沙:湖南中医药大学,2019.
[11] 骆始华,李 易,赵丽娟,等.冠心病临界病变患者的中医证候分布规律[J].中国实验方剂学杂志,2020,26(9):53-57.
[12] 柯元南,陈纪林.不稳定性心绞痛和非ST段抬高心肌梗死诊断与治疗指南[J].中华心血管病杂志,2007,35(4):295-304.
[13] 国家中医药管理局.中华人民共和国中医药行业标准:中医病症诊断疗效标准[S].南京:南京大学出版社,1994.
[14] 中國中西医结合学会活血化瘀专业委员会.冠心病血瘀证诊断标准[J].中国中西医结合杂志,2016,36(10):1162.
[15] ITO S, D’ALESSIO A C, "TARANOVA O V", et al. Role of Tet proteins in 5mC to 5hmC conversion, ES-cell self-renewal and inner cell mass specification[J]. Nature, 2010, 466(7310): 1129-1133.
[16] FUSTER J J, MACLAUCHLAN S, ZURIAGA M A, et al. Clonal hematopoiesis associated with TET2 deficiency accelerates atherosclerosis development in mice[J]. Science (New York, N Y), 2017, 355(6327): 842-847.
[17] LIU R J, JIN Y, TANG W H, et al. Ten-eleven translocation-2 (TET2) is a master regulator of smooth muscle cell plasticity[J]. Circulation, 2013, 128(18): 2047-2057.
[18] PENG J, YANG Q, LI A F, et al. Tet methylcytosine dioxygenase 2 inhibits atherosclerosis via upregulation of autophagy in ApoE-/-mice[J]. Oncotarget, 2016, 7(47): 76423-76436.
[19] DENG Q Y, HUANG W, PENG C Y, et al. Genomic 5-mC contents in peripheral blood leukocytes were independent protective factors for coronary artery disease with a specific profile in different leukocyte subtypes[J]. Clinical Epigenetics, 2018, 10: 9.
[20] DONG C R, CHEN J M, ZHENG J L, et al. 5-Hydroxymethylcytosine signatures in circulating cell-free DNA as diagnostic and predictive biomarkers for coronary artery disease[J]. Clinical Epigenetics, 2020, 12(1): 17.
[21] RAJNEESH R, SINHA R P. Hydroxymethylation of DNA: An epigenetic marker[J]. EXCLI Journal, 2014, 13: 592-610.
[22] EGUCHI S, TAKEFUJI M, SAKAGUCHI T, et al. Cardiomyocytes capture stem cell-derived, anti-apoptotic microRNA-214 via clathrin-mediated endocytosis in acute myocardial infarction[J]. The Journal of Biological Chemistry, 2019, 294(31): 11665-11674.
[23] GERBOD-GIANNONE M C, DALLET L, NAUDIN G, et al. Involvement of caveolin-1 and CD36 in native LDL endocytosis by endothelial cells[J]. Biochimica et Biophysica Acta General Subjects, 2019, 1863(5): 830-838.
[24] HAHN J, FU Y, BROWN M R, et al. Genetic loci associated with prevalent and incident myocardial infarction and[J]. PloS one, 2020, 15(11): e230035.
[25] WANG Z, ZHU H, SHI H T, et al. Exosomes derived from M1
macrophages aggravate neointimal hyperplasia following carotid artery injuries in mice through miR-222/CDKN1B/CDKN1C pathway[J]. Cell Death & Disease, 2019, 10(6): 422.
[26] GAO J, ZHU M, LIU R F, et al. Cardiac hypertrophy is positively regulated by microRNA24 in rats[J]. Chinese Medical Journal, 2018, 131(11): 1333-1341.
[27] YAMADA S, SENOKUCHI T, MATSUMURA T, et al. Inhibition of local macrophage growth ameliorates focal inflammation and suppresses atherosclerosis[J]. Arteriosclerosis, Thrombosis, and Vascular Biology, 2018, 38(5): 994-1006.
[28] GEDIYA P, PARIKH P K, VYAS V K, et al. Histone deacetylase 2: A potential therapeutic target for cancer and neurodegenerative
disorders[J]. European Journal of Medicinal Chemistry, 2021, 216: 113332.
[29] YAN H, YI S W, ZHUANG H, et al. Sphingosine-1-phosphate ameliorates the cardiac hypertrophic response through inhibiting the activity of histone deacetylase-2[J]. International Journal of Molecular Medicine, 2018, 41(3): 1704-1714.
[30] SCHOLZ B, SCHULTE J S, HAMER S, et al. HDAC (histone deacetylase) inhibitor valproic acid attenuates atrial remodeling and delays the onset of atrial fibrillation in mice[J]. Circulation Arrhythmia and Electrophysiology, 2019, 12(3): e007071.
[31] HORI D, NOMURA Y, NAKANO M, et al. Endothelial-specific overexpression of histone deacetylase 2 protects mice against endothelial dysfunction and atherosclerosis[J]. Cellular Physiology and Biochemistry, 2020, 54(5): 947-958.
[32] ZHANG Q, ZHAO K, SHEN Q C, et al. Tet2 is required to resolve inflammation by recruiting Hdac2 to specifically repress IL-6[J]. Nature, 2015, 525(7569): 389-393.
[33] YE C, TAO R, CAO Q Y, et al. Whole-genome DNA methylation and hydroxymethylation profiling for HBV-related hepatocellular carcinoma[J]. International Journal of Oncology, 2016, 49(2): 589-602.
[34] MIRYALA S K, ANBARASU A, RAMAIAH S. Gene interaction network to unravel the role of gut bacterial species in cardiovascular diseases: E. coli O157: H7 host-bacterial interaction study[J]. Computers in Biology and Medicine, 2021, 133: 104417.
[35] KOLOVOU V, TSIPIS A, MIHAS C, et al. Tumor protein p53 (TP53) gene and left main coronary artery disease[J]. Angiology, 2018, 69(8): 730-735.
[36] VEEROJU S, MAMAZHAKYPOV A, RAI N, et al. Effect of p53 activation on experimental right ventricular hypertrophy[J]. PLoS One, 2020, 15(6): e0234872.
[37] WANG Q S, ZHOU J, LI X. LncRNA UCA1 protects cardiomyocytes against hypoxia/reoxygenation induced apoptosis through inhibiting miR-143/MDM2/p53 axis[J]. Genomics, 2020, 112(1): 574-580.
[38] SAMARAKOON R, HIGGINS S P, HIGGINS C E, et al. The TGF-β1/p53/PAI-1 signaling axis in vascular senescence: Role of caveolin-1[J]. Biomolecules, 2019, 9(8): E341.
[39] SUN D, YU M, LI Y Y, et al. Histone deacetylase 2 is involved in DNA damage-mediated cell death of human osteosarcoma cells through stimulation of the ATM/p53 pathway[J]. FEBS Open Bio, 2019, 9(3): 478-489.
