基于环境DNA的进口凡纳滨对虾携带水传播虾肝肠胞虫风险评估
2022-04-21张娜刘荭廖金湾郑晓聪郑枢刘骁谢艳辉李家侨李红权
张娜 刘荭 廖金湾 郑晓聪 郑枢 刘骁 谢艳辉 李家侨 李红权


摘要:【目的】基于环境DNA(eDNA)分析进口凡纳滨对虾携带水传播和运输虾肝肠胞虫(EHP)的风险性,为环境水中疫病监测和风险评估提供参考依据。【方法】取60批进口亲虾携带水用0.45 µm硝酸纤维素膜过滤并提取eDNA,挑取10个携带水样滤膜eDNA用于凡纳滨对虾物种特异性鉴定,采用实时荧光定量PCR对所有携带水样滤膜进行EHP检测;同时以EHP阳性进口亲虾携带水开展人工感染试验,感染10 d后分别取养殖水样及虾体进行EHP检测;对进口亲虾携带水EHP阳性滤膜进行宏基因组测序分析,采用Krona對物种注释结果进行可视化展示,并将宏基因组测序分析结果与NCBI已公布的EHP氨基酸序列进行BLAST比对分析。【结果】进口亲虾携带水检测结果显示,成功从10个携带水滤膜eDNA扩增出凡纳滨对虾347 bp的特异目的片段,与原始序列片段的相似性达100%;在60批进口亲虾携带水样品中有2批携带水呈EHP阳性。EHP阳性进口亲虾携带水能成功感染虾苗组,且在虾苗肝胰腺组织分离获得成熟的EHP孢子,但亲虾组的虾体EHP检测呈阴性。宏基因组测序得到EHP的物种相对丰度值占真核动物物种的0.7%;与NCBI已公布的EHP氨基酸序列进行BLAST比对,结果获得213个同源EHP氨基酸序列;从进口亲虾携带水样品中分离鉴定获得的EHP与已报道物种黄道蟹肠孢虫(Enterospora canceri)和毕氏肠胞虫(Enterocytozoon bieneusi)的亲缘关系较近,对应的氨基酸序列相似性为别为83%和81%。【结论】进口凡纳滨对虾亲虾携带水具有传播EHP的可能性,即仅针对亲虾虾体进行EHP检测具有不完全性。eDNA检测可用来补充传统的虾体疫病监测方法,作为水环境疫病检测和风险评估的重要手段。
关键词: 凡纳滨对虾;亲虾;携带水;虾肝肠胞虫(EHP);环境DNA(eDNA);风险评估
中国分类号: S945.49 文献标志码: A 文章编号:2095-1191(2022)01-0219-10
Risk analysis of imported live shrimp carrying water trans-mitted Enterocytozoon hepatopenaei by environmental
DNA method
ZHANG Na1, LIU Hong2, LIAO Jin-wan3, ZHENG Xiao-cong2, ZHENG Shu3,
LIU Xiao1, XIE Yan-hui1, LI Jia-qiao1, LI Hong-quan1*
(1Zhanjiang Customs Technology Center, Zhanjiang, Guangdong 524000, China; 2Animal and Plant Inspection and Quarantine Technical Centre, Shenzhen, Guangdong 518045, China; 3Zhanjiang Customs, Zhanjiang,
Guangdong 524000, China)
Abstract:【Objective】The purpose of this study was to analyze the risks of water-borne Enterocytozoon hepatopenaei (EHP) in imported live shrimp using environmental DNA(eDNA) method, to provide a reference basis for environmental water disease monitoring and risk assessment. 【Method】In this study, 60 batches of imported parent shrimp carrying water were filtered by 0.45 μm nitrocellulose membrane, eDNA of 10 filter membranes were amplified byspecific primers of Litopenaeus vannamei. All 60 batches of filter membranes were tested for EHP via real-time fluorescence quantitative PCR. At the same time, EHP positive carrying water was used for artificial infection test. After 10 d of infection, environmental water and shrimp body were taken from each group for EHP detection. The metagenome of EHP positive filter membrane was sequenced and analyzed,Krona was used to visualize the annotation results, and the results of metagenome sequencing were compared with the EHP amino acid sequences registered in NCBI by EHP. 【Result】The results showed that a 347 bp specific target fragment of L.vannamei was successfully amplified from 10 filter membranes,the sequencing results showed that the homology with the original sequence was 100%. Two batches of EHP were positive in 60 batches of imported prawns. The results of artificial infection test showed that EHP positive carrying water successfully infected the post-larvae group, and mature EHP spores were isolated from the hepatopancreas of shrimps, but the shrimps in parent group body were negative in EHP detection. The species relative abundance of EHP accounted for 0.7% of eukaryotic, the results compared with the amino acid sequences registered in NCBI, 213 homologous EHP amino acid sequences were obtained, the amino acid sequences of EHP obtained were similar to those of other reported evolutionary trees:Enterospora canceri (amino acid similarity 83%), Enterocytozoon bieneusi (amino acid similarity 81%). 【Conclusion】It is possible for imported animals to carry water to spread EHP. EHP detection on parent shrimp only is incomplete. eDNA method can be used to supplement the traditional shrimp disease monitoring method as an important means of water disease detection and risk assessment.
Key words: Litopenaeus vannamei;parent shrimp; carrying water; Enterocytozoon hepatopenaei(EHP); environmental DNA(eDNA); risk analysis
Foundation items:Science and Technology Project of General Administration of Customs(2021HK007,2019HK038);Science and Technology Project of Guangdong Entry Exit Inspection and Quarantine Bureau(2018GDK35)
0 引言
【研究意义】随着水产养殖业的不懂发展,水生动物疫病发生率也呈逐年上升趋势。据报道,我国的水产养殖病害种类超过200多种,尤其是一些危害严重的病毒病每年均会引起巨大的经济损失。在水生动物疫病中,对虾白斑综合征病毒(WSSV)、桃拉综合征病毒(TSV)、急性肝胰腺坏死病致病性副溶血弧菌(VPAHPND)、虾肝肠胞虫(EHP)等均由境外入侵,进而对我国对虾养殖业造成严重威胁(孙卫芳等,2019;陈洁等,2021)。进口鲜活水生动物是外来病原体在全球范围内肆意传播的主要途径之一,各种病原体及其感染宿主被引入本地环境后,可对生物多样性、生态系统及养殖业等造成严重危害。为了杜绝活体动物贸易中病原体的传播,各国政府根据科学的风险分析,兼顾水生动物种群的起源和历史、宿主易感性、传播风险及检测方法可靠性等,制定了相应的检疫管理措施(Hood et al.,2019)。因此,从鲜活水生动物及其携带水等多维度加强疫病检疫的方法研究和评估分析,对确保我国水生动物养殖业的持续健康发展具有重要意义。【前人研究进展】目前,我国对于进口鲜活水生动物检疫的主要目标是动物活体,按照抽样标准抽取一定数量的样品,由于其样品的代表性具有挑战性,导致检测结果有可能存在偏差;对于高价值且数量极少的进口活动物而言,活体抽样数量不足及检疫成本过高的缺点越来越凸显(Barnes and Turner,2016)。环境DNA(eDNA)是由生物体自然排出的DNA,如通过表皮脱落、代谢废物排泄物及死后代谢至环境中(Doi et al.,2017;张娜等,2020)。寄生虫的卵、孢子、包囊、幼虫、幼体和成体在整个生命周期各阶段均可从宿主活体脱离而存在于水体环境中(Bass et al.,2015),因此寄生虫基因组DNA可基于eDNA通过分子生物学方法进行捕捉、提取和筛选,进而在水生寄生虫学中实现物种水平的检测及监测(Lymbery et al.,2014;Trujillo-González et al.,2018,2019a;张娜等,2021b)。从水样中捕获并提取的eDNA已被证明可准确检测感染野生两栖动物的致病性吸虫,或监测感染养殖和野生鱼类的寄生虫(Tomsen and Wil-lerslev,2015)。eDNA已被提议作为一种非破坏性和敏感性的生物安全检测工具(Goldberg et al.,2016;Gomes et al.,2017),无需任何靶向PCR,能克服传统物种鉴定方法耗时费力的缺点,可高通量、快速地鉴定物种(Deiner et al.,2017)。孙晶莹等(2018)采用宏条形码技术对太湖流域常见的5种枝角类浮游动物物种进行鉴定和生物量监测,并与实时荧光定量PCR相比,结果表明扩增引物对eDNA宏条形码技术影响显著,检出的每个物种序列数与实时荧光定量PCR定量拷贝数高度一致,可实现对浮游动物物种的半定量检测。Rusch等(2018)对挪威德位门河流的9个位置进行取样,通过eDNA监测大西洋三代虫(Gyrodactylus salaris)及其2个宿主[大西洋鲑鱼(Salmo salar)和虹鳟鱼(Oncorhynchus mykiss)存在情况,结果在受大西洋三代虫污染的水域检测到寄生虫eDNA,并在河流上游检测到虹鳟鱼。Trujillo-González等(2019b)使用eDNA对进口观赏鱼进行5种寄生虫检测,以防止外来寄生虫和疫病的传入。此外,张娜等(2021a)基于eDNA优化了检测环境水中WSSV的切向流浓缩方法;Wang等(2022)依据eDNA原理实现对环境水中传染性皮下和造血器官坏死病毒(IHHNV)進行浓缩和检测。可见,eDNA对于水中病毒检测具有较高的应用价值。【本研究切入点】虾肝肠胞虫(Enterocytozoon hepatopenaei,EHP)是近几年新发现寄生于对虾肝胰腺组织的病原(Tourtip et al.,2009;Tangprasittipap et al.,2013;宋居易等,2020),在我国、印度尼西亚、马来西亚、越南、印度和泰国的对虾中均有检出(Biju et al.,2016;Jaroenlak et al.,2018)。调查发现,在孵育的1日龄凡纳滨对虾虾苗中即检出EHP阳性,故推测其SPF种虾是高风险性传染源,提示一些拥有SPF标签的进口凡纳滨对虾也可能携带EHP,而进口对虾携带水也有可能是传染源。目前,针对虾肝肠胞虫的流行病学监测多以虾体为主,利用eDNA方法开展入境水生生物携带水风险评估的研究鲜见报道。因此,急需建立进口亲虾携带水中肝肠胞虫检疫方法,以确保我国对虾养殖业的健康发展。【拟解决的关键问题】以进口亲虾携带水eDNA为研究对象进行EHP检测,并对携带水过滤膜进行宏基因组测序,采用多种检测方法分析进口亲虾携带水传播和运输EHP的风险性,为环境水中疫病监测和风险评估提供参考依据。
1 材料与方法
1. 1 试验材料
感染EHP的凡纳滨对虾阳性样品由湛江海关技术中心动物检疫实验室保存提供;DNeasy Blood and Tissue Kit购自德国Qiagen公司;Ex Taq DNA聚合酶购自宝生物工程(大连)有限公司;PCR管等购自美国Axygen公司。主要仪器设备:实时荧光定量PCR仪(美国ABI StepOne Plus)、高速冷冻离心机(Eppendorf Centrifuge 5417R)、真空泵(津腾,GM-0-33A)。
1. 2 试验方法
1. 2. 1 携带水样品过滤处理及凡纳滨对虾物种特异性鉴定 以进口亲虾携带水样为研究对象,共60批样品,每个水样取1 L,使用孔径0.45 µm的硝酸纤维素膜进行无菌真空抽滤,每次过滤前均用无菌水、0.5 mol/L氢氧化钠和3%次氯酸钠冲洗过滤漏斗,防止交叉污染。过滤完成后将每张滤膜单独放入1.5 mL离心管中,用剪刀将滤膜剪成细条状,以DNeasy Blood and Tissue Kit试剂盒提取滤膜eDNA(具体提取方法见说明书)。在GenBank检索凡纳滨对虾EF584003.1线粒体基因序列,使用Primer Premier 6.0设计引物(F2:5'-AACAGCCCTCACTATTTCAA GACC-3';R2:5'-ATCGTTGCGTTGGATAAAATGG C-3'),在NCBI 网站上进行引物特异性测试后委托宝生物工程(大连)有限公司合成,预期扩增片段长度为347 bp。PCR扩增程序:94 ℃预变性3 min,94 ℃ 1 min,58 ℃ 1 min,72 ℃ 1 min,进行35个循环;72 ℃延伸10 mim。PCR扩增产物委托生工生物工程(上海)股份有限公司进行测序分析。
1. 2. 2 携带水样品EHP检测 参考张娜等(2017)的方法设计引物和探针序列,采用建立好的实时荧光定量PCR对滤膜eDNA进行EHP检测。EHP阳性样品使用套式PCR进行确定(Jaroenlak et al.,2016)。第一步扩增引物F:5'-TTGCAGAGTGTTGTTAAG GGTTT-3',R:5'-CACGATGTGTCTTTGCAATTTT C-3'。扩增程序:94 ℃预变性5 min;95 ℃ 30 s,58 ℃ 30 s,72 ℃ 30 s,进行35个循环;72 ℃延伸5 min。第二步扩增引物F:5'-TTGGCGGCACAATTCTCAA ACA-3',R:5'-GCTGTTTGTCTCCAACTGTATTTG A-3'。扩增程序:94 ℃预变性5 min;94 ℃ 30 s,55 ℃ 30 s,72 ℃ 40 s,进行35个循环;72 ℃延伸5 min。
1. 2. 3 人工感染动物试验 选取凡纳滨对虾亲虾及10日龄虾苗,经EHP、WSSV、TSV、VPAHPND、IHHNV、偷死野田村病毒(CMNV)、黄头病毒(YHV)及传染性肌坏死病毒(IMNV)等主要病原检测均呈阴性,暂养于长100 cm、宽30 cm、高50 cm的水槽中,以不含任何抗病药物的饲料喂养7 d后备用。感染试验共分为4组,每组均设2个平行。T1组:阳性对照组,对虾肝胰腺组织感染EHP的虾苗组;T2组:阴性对照组,为非攻毒组;T3组:虾苗感染试验组(每组300尾虾苗),以检测呈阳性的进口对虾携带水进行饲养;T4组:亲虾感染试验组(每组4尾亲虾),以检测呈阳性的进口对虾携带水进行饲养。攻毒感染前停食1 d。
1. 2. 4 人工感染试验后养殖水样和虾体检测 每日投喂饲料1次,每天观察凡纳滨对虾死亡情况,及时清理死亡对虾。饲养期间不换水,饲养10 d后全部扑杀和取样,同时对养殖水样进行抽滤取过滤,采用DNeasy Blood and Tissue Kit试剂盒提取eDNA,并进行EHP检测。
1. 2. 5 EHP孢子分离与染色鉴定 取所有虾苗及亲虾的肝胰腺,混合置于玻璃研磨器中加入液氮研磨,反复冻融5次,组织匀浆液中加入3倍体积的PBS稀释,先通过4层200目筛绢过滤除去较粗的组织碎片,过滤液再通过6层200目筛绢过滤除去较粗的组织碎及和黏液。滤出液13000 r/min离心30 min,弃上清液,加入PBS重悬沉淀,重复操作2次;2000 r/min离心10 min,取上清液;13000 r/min离心30 min,弃上清液;沉淀加入500 μL PBS重悬。取300 μL重悬液缓慢注入到预先铺制蔗糖密度梯度(由上至下浓度依次为30%、35%、40%和50%)的30 mL试管表面,水平方向5000 r/min离心1 h。每层蔗糖面有黄色可见物,EHP主要分布在35%~40%的蔗糖面,小心吸出各层可见物,以PBS稀释10倍后14000 r/min离心10 min,弃上清液,沉淀用少量PBS悬浮后4 ℃保存(乔毅等,2018;Aldama-Cano et al.;2018;常晓晴等,2019)。取1滴纯化后的孢子悬液均匀涂布于载玻片上,滴加1滴2%荧光桃红B,缓慢摇匀,室温放置3 min,置于100倍油镜下观察。
1. 2. 6 进口亲虾携带水滤膜宏基因组测序分析
对检测呈阳性的进口亲虾携带水滤膜eDNA使用Quibt进行浓度测定,以1.0%琼脂糖凝胶电泳检测eDNA完整性及纯度,取质量合格的样品构建基因文库。利用超声波仪Covaris M220(Woburn,MA,USA)将各基因组eDNA样本随机打断成300 bp左右的片段,随后利用NEBNext® UltraTM DNA Library Prep Kit for Illumina®(NEB,USA)构建基因文库,采用Agencourt AMPure XP(Beckman,USA)进行磁珠纯化,经GenNextTM NGS Library Quantification Kit(Toyobo,Japan)定量分析后以Illumina Novaseq 6000 PE150進行测序分析。然后对原始序列进行去接头、质量剪切及去除污染等优化处理,使用优质序列进行拼接组装和基因预测,并对得到的基因进行物种和功能注释及分类。
1. 2. 7 进口亲虾携带水样品EHP氨基酸序列比对分析 使用BEDTools的GenomeCoverageBed计算基因丰度,从不同分类层级(门、纲、母、科、属、种)的相对丰度出发,在非冗余蛋白的氨基酸序列(nr)数据库使用DIAMOND进行分析,分析获得的EHP氨基酸序列与NCBI已公布的EHP氨基酸序列进行BLAST比对分析。
2 结果与分析
2. 1 携带水样品中凡纳滨对虾物种鉴定结果
为有效鉴定eDNA的提取有效性,对宿主凡纳滨对虾的物种特异性进行鉴定。由图1可看出,成功从10个进口亲虾携带水样滤膜eDNA中扩增出347 bp的特异目的片段,且目的条带单一明亮,与预期结果相符。在NCBI网站上经BLAST比对分析,结果显示扩增获的目的片段与原始序列片段的相似性达100%。
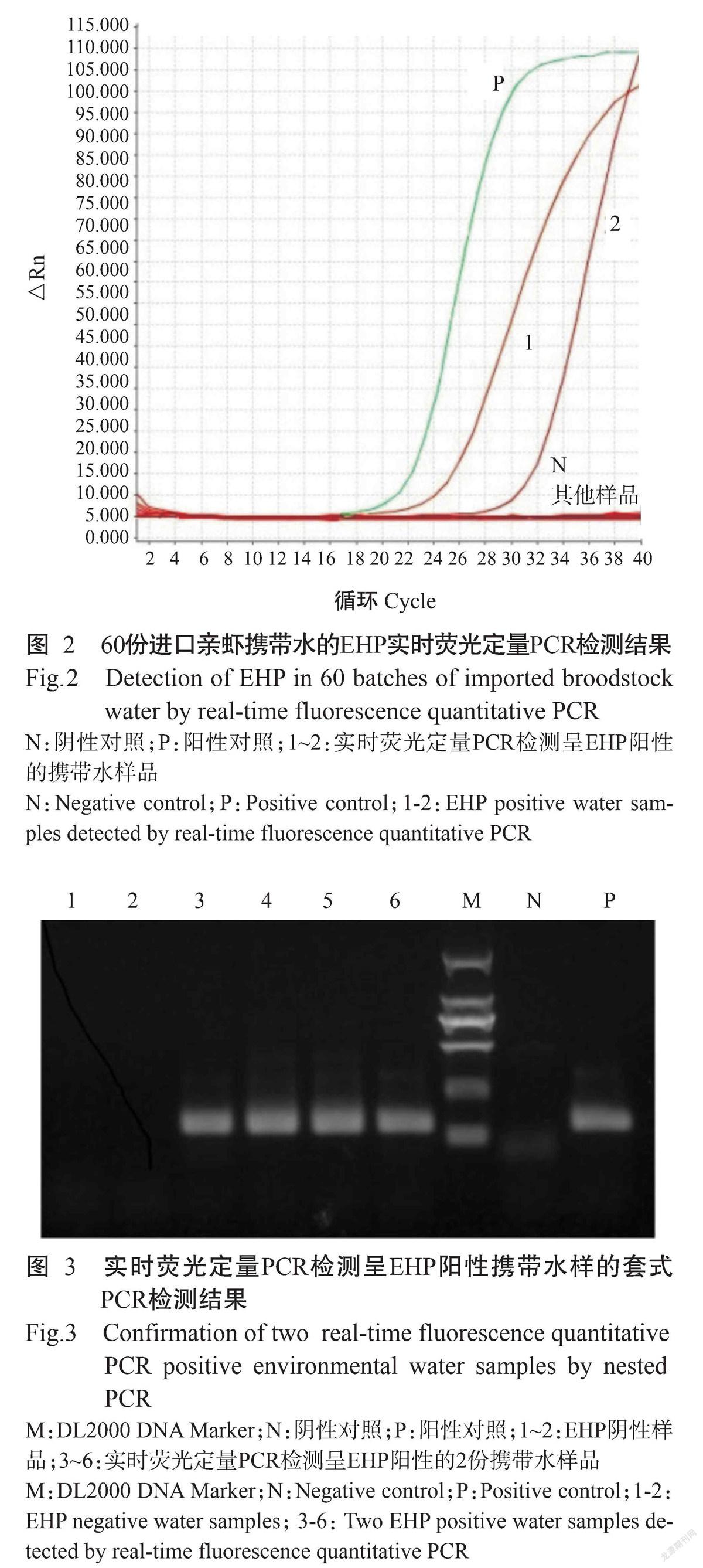
2. 2 携带水样品中EHP检测结果
利用实时荧光定量PCR对60份进口亲虾携带水样品进行检测,结果(图2)显示有2批携带水呈EHP阳性;然后对这2批携带水进行套式PCR扩增,扩增获得的条带(图3)与预期结果相符。经测序及比对分析,发现与EHP序列的相似性达99%,确定为EHP阳性。
2. 3 人工感染试验结果
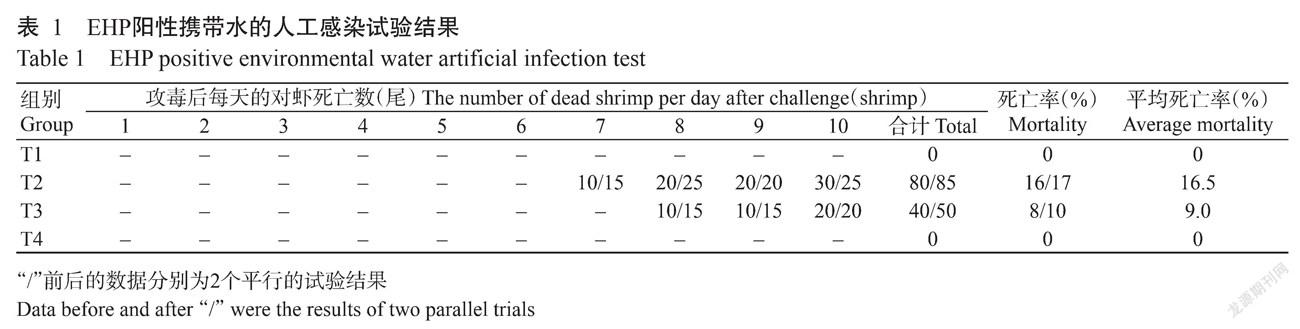
由表1可看出,T1组(阴性对照组)无死亡对虾;T2组(阳性对照组)从换水感染后第7 d开始有虾苗死亡,平均死亡率为16.5%;T3组(虾苗感染试验组)在换水感染后第8 d开始有虾苗死亡,平均死亡率为9.0%;T4组(亲虾感染试验组)在进行换水感染后无死亡虾。经统计学方差分析,发现各组间的差异均达显著水平(P<0.05)。可见,EHP检测呈阳性的进口亲虾携带水对虾苗具有感染性,但对亲虾的感染力较弱,究其原因可能是EHP的孢子数量较少,尚未达到足够感染量,或亲虾的抵抗力较强。
2. 4 人工感染试验后养殖水样及虾体的EHP检测结果
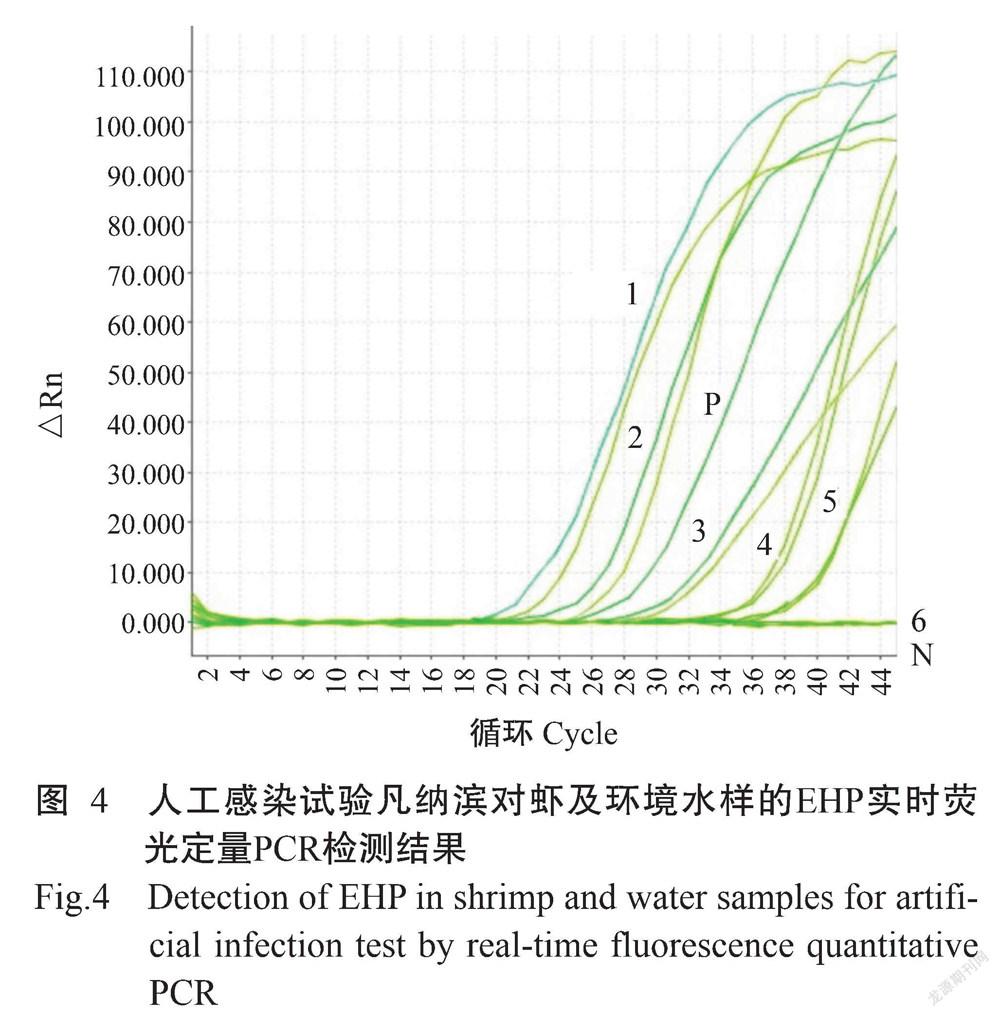
人工感染10 d后对所有凡纳滨对虾进行扑杀和取样,同时抽滤养殖水样取过滤膜,采用实时荧光定量PCR进行EHP检测。各组虾体检测结果(图4)显示,T1组(阳性对照组)和T3组(虾苗感染试验组)虾体EHP检测均呈阳性,T2组(阴性对照组)和T4组(亲虾感染试验组)虾体EHP检测均呈阴性。各组的养殖水样过滤膜检测结果显示,T1组(阳性对照组)、T3组(虾苗感染试验组)和T4组(亲虾感染试验组)养殖水样过滤膜EHP检测均呈阳性,以T4组(亲虾感染试验组)养殖水样过滤膜的核酸检测浓度最低;T2组(阴性对照组)养殖水样过滤膜EHP检测呈阴性。对实时荧光定量PCR检测呈阳性的样品再进行套式PCR扩增,结果(图5)显示扩增获得的条带(图3)与预期结果相符,经测序及比对分析,确定为EHP阳性。
2. 5 人工感染试验后凡纳滨对虾肝胰腺EHP分离及镜检结果
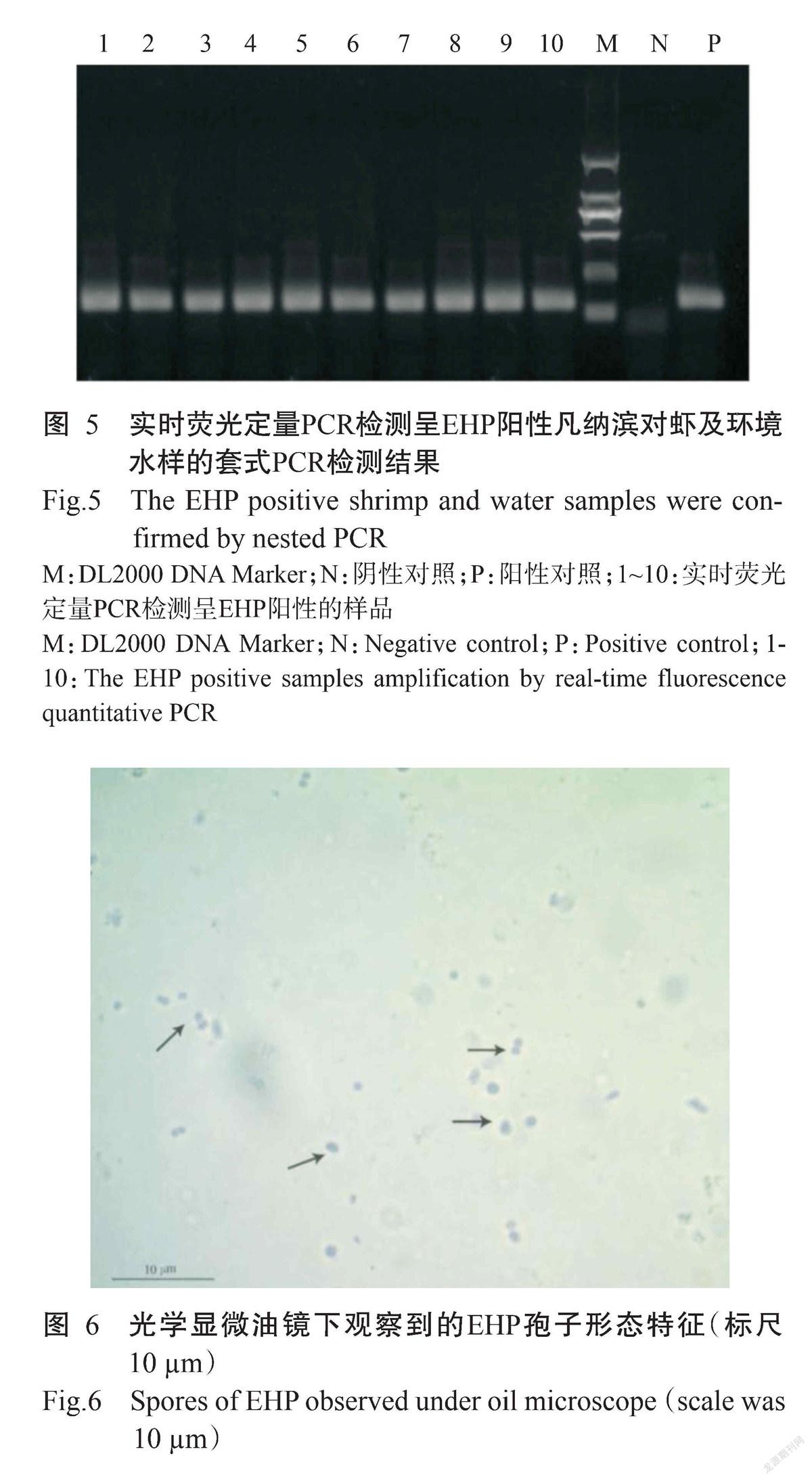
取T3组(虾苗感染试验组)虾苗肝胰腺进行匀浆,经过滤、洗涤、离心后采用蔗糖密度梯度离心得到EHP孢子悬液,取1滴纯化后的孢子悬液均匀涂布于载玻片上,滴加1小滴2%荧光桃红B,缓慢摇匀,室温放置3 min后置于100倍油镜下进行镜检,结果(图6)显示EHP孢子大小约1.2±0.2 µm×1.6±0.2 µm,呈卵圆形、球形或椭圆形。T4组(亲虾感染试验组)亲虾肝胰腺未分离获得EHP孢子。
2. 6 进口亲虾携带水样eDNA宏基因组测序分析结果
对进口亲虾携带水过滤膜进行宏基因组测序,使用DIAMOND将UniqGeneSet与NCBI已公布的氨基酸序列进行BLAST比对分析(期望值E-value设为1E-5);取相似性最高的比对序列作为该序列的物种注释信息,计算该物种的基因丰度总和,并从门(Phylum)、纲(Class)、目(Order)、科(Family)、属(Genus)、种(Species)各分类学水平统计物种在各样品中的相对丰度,构建相应分类学水平上的丰度谱(Abundance profile)。为直观展示各样品中不同分类层级的物种相对丰度,采用Krona对物种注释结果进行可视化展示,结果(图7)显示,EHP的物种相对丰度值占真核动物物种的0.7%。
2. 7 进口亲虾携带水样品EHP氨基酸序列比对分析结果
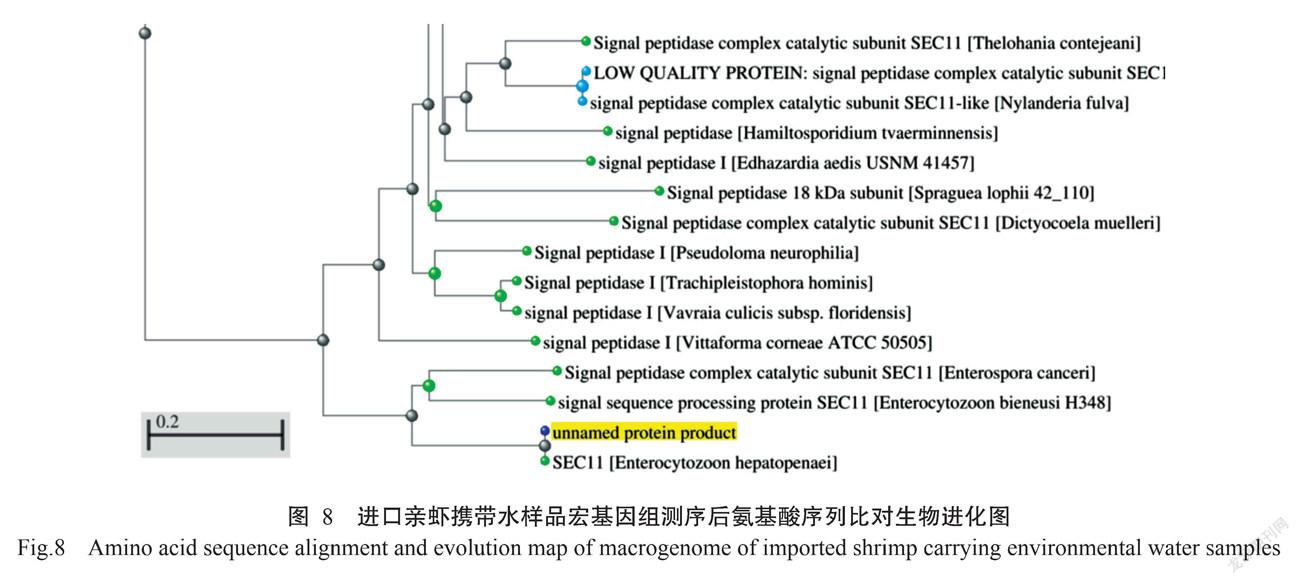
宏基因组测序分析获得的EHP氨基酸序列与NCBI已公布的EHP氨基酸序列进行BLAST比对,结果获得213个同源EHP氨基酸序列。以NCBI已公布的EHP氨基酸序列(OQS53361.1)为参考,基于EHP氨基酸序列相似性构建生物进化图,结果(图8)显示,从进口亲虾携带水样品中分离鉴定获得的EHP与已报道物种黄道蟹肠孢虫(Enterospora canceri)和毕氏肠胞虫(Enterocytozoon bieneusi)的亲缘关系较近,对应的氨基酸序列相似性为别为83%和81%。
3 讨论
EHP隶属于微孢子虫科(Microsporidia)肠胞虫属(Enterocytozoon),是一种专性细胞内寄生虫,主要寄生在虾类肝胰腺肝小管上皮细胞内,凡纳滨对虾、脊尾白虾和斑节对虾等主要经济养殖虾类均为易感宿主(Jaroenlak et al.,2018)。尽管虾肝肠胞虫不会引起对虾很高的死亡率,甚至有时不表现出症状,但会导致对虾生长缓慢,尤其与其他疫病混合感染时能加重对虾疫情及增加致死率。EHP感染在苗种培育和成虾养殖阶段均有出现,严重影响对虾的生长速度及成虾品质,给对虾产业带来较大经济损失(张娜等,2020)。目前,实验室检测EHP的常用方法有套式PCR、实时荧光定量PCR、环介导等温扩增(LAMP)及原位雜交技术(胡吉卉等,2020),鲜见基于eDNA对环境水进行EHP检测的研究报道。Jaroenlak等(2016)研究发现轮虫和卤虫均可携带EHP,将增加对虾感染EHP的可能性,因此建议在养殖生产中应对投喂的鲜活饵料进行巴氏消毒处理,以预防对虾感染EHP。丁慧昕等(2018)采集养殖虾塘排水渠的环境样品,包括虾、蟹及鱼类等水生动物进行EHP检测,结果从这些环境样品中均检测到EHP。本研究基于eDNA直接对进口亲虾携带水进行EHP检测,并对携带水样过滤膜进行宏基因组测序分析,通过多维度分析进口亲虾携带水传播和运输EHP的风险,结果表明进口亲虾环境水携带感染性EHP的可能性非常大,也进一步佐证仅针对亲虾虾体进行EHP检测具有不完全性。
宏基因组(Metagenomics)是指特定环境下所有生物遗传物质的总和,其决定生物群体的生命现象。宏基因组是以生态环境中全部DNA作为研究对象,通过克隆和异源表达来筛选有用基因及其产物,分析其功能及彼此间的关系和相互作用(Tisthammer et al.,2016)。目前,宏基因组测序是eDNA研究的重要方法。在序列运行中,序列质量并不均匀,会出现一些序列读取的平均质量较其他序列差的序列,而QIME(Caporaso et al.,2012)和OBITOLS程序(Boyer et al.,2016)等方法可实现DNA宏条形码分析。本研究通过对进口亲虾携带水进行宏基因组测序,并与NCBI已公布的EHP氨基酸序列进行BLAST比对,结果表明,从进口亲虾携带水样品中分离鉴定获得的EHP与黄道蟹肠孢虫和毕氏肠胞虫的物种进化树非常接近,且聚为一支。毕氏肠胞虫是重要的人源微孢子虫,其宿主包括人类及家畜、野生动物和鸟类;而黄道蟹肠孢虫的感染宿主是黄道蟹(Palenzuela et al.,2014;颜远义等,2018)。故推测携带水滤膜eDNA宏基因组测序的氨基酸序列比对结果存在偏差,因此后续需要对序列比对数据库进一步更新,同时对宏基因组编码及序列比对标准化进行规范。
eDNA检测是一种最具应用前途的工具,可用来补充甚至取代传统的动物个体取样检测和生物监测(Ardura et al.,2015;Coble et al.,2019),无需杀死大量动物或进行非常耗时的人工检查,而实现快速检测。目前,已有关于寄生虫eDNA检测的研究报道,涉及六齿轮虫、新本尼登虫、阿片虫、间日疟原虫及粘液虫等(Robson et al.,2016;Pochon et al.,2017)。eDNA监测方法不仅缩短了采样时间和降低了检测成本,还提高了动物福利,且能同时检测寄生虫和宿主,因而具有极大的应用潜力。利用本研究制定的检测方案不仅可在单个样本上检测到EHP,还能检测到其感染宿主基因。通过综合使用其他检测方法,即可检测或监测几乎所有水生寄主病原体复合体的存在,但其前提是滤膜规格适合从目标生物体中捕获eDNA。
4 结论
进口凡纳滨对虾亲虾携带水具有传播EHP的可能性,即仅针对亲虾虾体进行EHP检测具有不完全性。eDNA检测可用来补充传统的虾体疫病监测方法,作为水环境疫病检测和风险评估的重要手段。
参考文献:
常晓晴,王元,英娜,李楠英,黄倞,李新苍,周俊芳,房文红. 2019. 8种染色方法对组织切片中虾肝肠胞虫染色效果的比较[J]. 海洋渔业,41(1):91-99. [Chang X Q,Wang Y,Ying N,Li N Y,Huang L,Li X C,Zhou J F,Fang W H. 2019. Comparison of 8 different special staining techniques of Enterocytozoon hepatopenaei in histologic section[J]. Marine Fishries,41(1):91-99.] doi:10.3969/j.issn.1004-2490.2019.01.011.
陈洁,廖国礼,吴玉娇,张庆,杨小娟,范晓东,龙梦娴,潘国庆,周泽扬. 2021. 虾肝肠胞虫的3种快速顯微镜检查法[J]. 西南大学学报(自然科学版),43(3):17-23. [Chen J,Liao G L,Wu Y J,Zhang Q,Yang X J,Fan X D,Long M X,Pan G Q,Zhou Z Y. 2021. Three methods for light microscopic detection of Enterocytozoon hepatopenaei[J]. Journal of Southwest University (Natural Science),43(3):17-23.] doi:10.13718/j.cnki.xdzk.2021.03.003.
丁慧昕,施慧,谢建军,王庚申,汪玮,何杰,许文军. 2018. 虾肝肠胞虫(EHP)在凡纳滨对虾养殖水环境中的分布情况及传播途径初步研究[J]. 浙江海洋大学学报(自然科学版),37(1):14-19. [Ding H X,Shi H,Xie J J,Wang G S,Wang W,He J,Xu W J. 2018. Preliminary study on distribution and transmission ways of Enterocytozoo hepa-topenaei in Litopenaeus vannamei’s culture environment[J]. Journal of Zhejiang Ocean University (Natural Scien-ce),37(1):14-19.] doi:10.3969/j.issn.1008-830X.2018. 01.003.
胡吉卉,李正民,段健诚,高焕. 2020. 虾类肝肠胞虫流行病学研究进展[J]. 水产养殖,41(1):1-4. [Hu J H,Li Z M,Duan J C,Gao H. 2020. Advances in epidemiological studies of Enterocytozoon hepatopenaei cysts in shrimp[J]. Journal of Aquaculture,41(1):1-4.] doi:10.3969./j.issn.1004-2091.2020.11.001.
乔毅,沈辉,万夕和,范贤平,蒋葛,黎慧,王李宝,史文军,成婕. 2018. 南美白对虾肝肠胞虫的分离及形态学观察[J]. 中国水产科学,25(5):1051-1058. [Qiao Y,Shen H,Wan X H,Fan X P,Jiang G,Li H,Wang L B,Shi W J,Cheng J. 2018. Preliminary isolation and morphological ovservation of Enterocytozoon hepatopenaei infecting the shrimp Penaeus vannamei[J]. Journal of Fishery Scien-ces of China,25(5):1051-1058.] doi:10.3724/SP.J.1118. 2018.17430.
宋居易,刘昱辰,张苏楠,江高伟,杨华卫. 2020. 南美白对虾4种病原体可视化快速检测试剂盒(生物芯片法)的研发[J]. 江苏农业学报,36(3):656-665. [Song J Y,Liu Y C,Zhang S N,Jiang G W,Yang H W. 2020. Development of a rapid visualization detection kit (biochip method) for four pathogens in Penaeus vannamei[J]. Jiangsu Journal of Agricultural Sciences,36(3):656-665.] doi:10.3969/j.issn.1000-4440.2020.03.018.
孙晶莹,杨江华,张效伟. 2018. 环境DNA(eDNA)宏条形码技术对枝角类浮游动物物种鉴定及其生物量监测研究[J]. 生态毒理学报, 13(5):76-86. [Sun J Y,Yang J H,Zhang X W. 2018. Identification and biomass monitoring of zooplankton cladocera species with eDNA metabarco-ding technology[J]. Asian Journal of Ecotoxicology,13(5):76-86.] doi:10.7524/AJE.1673-5897.20180108001.
孙卫芳,黄小帅,胡晓娟,文国樑,曹煜成,张建设. 2019. 广东沿海地区凡纳滨对虾EHP、VPAHPND和SHIV感染情况调查与分析[J]. 南方农业学报,50(10):2343-2349. [Sun W F,Huang X S,Hu X J,Wen G L,Cao Y C,Zhang J S. 2019. Detection and analysis of Enterocytozoon hepatopenaei (EHP),vibrio parahaemolyticus acute hepatopancrea-tic necrosis disease(VPAHPND) and shrimp hemocyte iridescent virus(SHIV) from Litopenaeus vannamei in coastal areas of Guangdong Province[J]. Journal of Southern Agri-culture,50(10):2343-2349.] doi:10.3969/j.issn. 2095-1191.2019.10.27.
颜远义,刘新华,徐力文,章晋勇. 2018. 南海石斑鱼苗种肠道微孢子虫病病原的鉴定[J]. 水生生物学报,42(5):942-949. [Yan Y Y,Liu X H,Xu L W,Zhang J Y. 2018. The taxonomic positon of causative agent of enteric microsporidiosis of hatchery-bred juvenile grouper,Epinephelus spp.,cultured in the area off coast of South China Sea[J]. Acta Hydrobiologica Sinica,42(5):942-949.] doi:10.7541/2018.116.
張娜,刘荭,谢艳辉,黄磊,孙思阳,斯泽恩,李家侨. 2021a. 用优化的切向流超滤法提高环境水中对虾白斑综合症病毒的浓度及检测效率[J]. 中国兽医杂志,57(10):104-110. [Zhang N,Liu H,Xie Y H,Huang L,Sun S Y,Si Z E,Li J Q. 2021a. Usr of optimized tangential flow ultrafiltration for improved concentration and detection of white spot syndrome virus in environmental water[J]. Chinese Journal of Veterinary Medicine,57(10):104-110.]
张娜,仇保丰,谢艳辉,李家侨,斯泽恩. 2021b. 水环境生态系统中环境DNA脱落、衰变和运输过程的研究进展[J]. 海洋湖沼通报,43(3):129-135. [Zhang N,Qiu B F,Xie Y H,Li J Q,Si Z E. 2021b. Research progress of environmental DNA shedding,decay and transport in aquatic ecosystem[J]. Transactions of Oceanology and Limno-logy,43(3):129-135.] doi:10.13984/j.cnki.cn37-1141. 2021.03.017.
张娜,谢艳辉,陈进会,李家侨,斯泽恩,黄磊. 2017. 虾肝肠胞虫TaqMan荧光PCR方法的建立与应用[J]. 中国动物检疫,34(10):98-103. [Zhang N,Xie Y H,Chen J H,Li J Q,Si Z E,Huang L. 2017. Establishment and application of TaqMan fluorescent PCR method for detection of Enterocytozoon hepatopenaei[J]. China Animal Health Inspection,34(10):98-103.] doi:10.3969/j.issn.1005-944X. 2017.10.026.
張娜,谢艳辉,李家侨,斯泽恩. 2020. 环境DNA 应用研究进展[J]. 中国动物检疫,37(11):68-75. [Zhang N,Xie Y H,Li J Q,Si Z E. 2020. Research progress on the application of environmental DNA[J]. China Animal Health Inspection,37(11):68-75.] doi:10.3969/j.issn.1005-944X. 2020.11.014.
Aldama-Cano D J,Sanguanrut P,Munkongwongsiri N,Ibarra-Gámez J C,Itsathitphaisarn O,Vanichviriyakit R,Flegel T W,Sritunyalucksana K,Thitamadee S. 2018. Bioassay for spore polar tube extrusion of shrimp Enterocytozoon hepatopenaei (EHP)[J]. Aquaculture,490:156-161. doi:10.1016/j.aquaculture.2018.02.039.
Ardura A,Zaiko A,Martinez J L,Samulioviene A,Semenova A,Garcia-Vazquez E. 2015. eDNA and specificprimers for early detection of invasive species—A case study on the bivalve Rangia cuneata, currently spreading in Europe[J]. Marine Environmental Research,112(Part B):48-55. doi:10.1016/j.marenvres.2015.09.013.
Barnes M A,Turner C R. 2016. The ecology of environmental DNA and implications for conservation genetics[J]. Conservation Genetics,17:1-17. doi:10.1007/s10592-015- 0775-4.
Bass D,Stentiford G D,Littlewood D T J,Hartikainen H. 2015. Diverse applications of environmental DNA me-thods in parasitology[J]. Trends in Parasitology,31(10):499-513. doi:10.1016/j.pt.2015.06.013.
Biju N,Sathiyarj G,Raj M,Shanmugan V,Baskaran B,Go-vindan U,Kumaresan G,Kasthuriraju K K,Chellamma T S R Y. 2016. High prevalence of Enterocytozoon hepatopenaei in shrimps Penaeus monodon and Litopenaeus vannamei sampled from slow growth ponds in India[J]. Diseases of Aquatic Organisms,120(3):225-230. doi:10. 3354/dao03036.
Boyer F,Mercier C,Bonin A,Le Bras Y,Taberlet P,Coissac E. 2016. OBITOOLS:A unix-inspired software package for DNA metabarcoding[J]. Molecular Ecology Resour-ces,16(1):176-182. doi:10.1111/1755-0998.12428.
Caporaso J G,Lauber C L,Walters W A,Berg-Lyons D,Huntley J,Fierer N,Owens S M,Betley J,Fraser L,Bauer M,Gormley N,Gilbert J A,Smith G,Knight R. 2012. Ultrahigh-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms[J]. The ISME Journal,6:1621-1624. doi:10.1038/ismej.2012.8.
Coble A A,Flinders C A,Homyack J A,Penaluna B E,Cronn R C,Weitemier K. 2019. eDNA as a tool for identifying freshwater speciesin sustainableforestry:A criticalreviewand potential futureapplications[J]. Science of the total Environment,649:1157-1170. doi:10.1016/j.scitotenv. 2018.08.370.
Deiner K,Bik H M,Machler E,Seymour M,Lacoursière-Roussel A,Altermatt F,Creer S,Bista I,Lodge D M,de Vere N,Pfrender M E,Bernatchez L. 2017. Environmental DNA metabarcoding:Transforming how we survey animal and plant communities[J]. Molecular Ecology,26(21):5872-5895. doi:10.1111/mec.14350.
Doi H,Inui R,Akamatsu Y,Kanno K,Yamanaka H,Takahara T,Minamoto T. 2017. Environmental DNA analysis for estimating the abundance and biomass of stream fish[J]. Freshwater Biology,62(1):30-39. doi:10.1111/fwb.12846.
Goldberg C S,Turner C R,Deiner K,Klymus K E,Thomsen P F,Murphy M A,Spear S F,McKee A,Oyler-McCance S J,Cornman R S,Laramin M B,Mahon A R,Lance R F,Pilliod D S,Strickler K M,Waits L P,Fremier A K,Takahara T,Herder J E,Taberlet P. 2016. Critical considerations for the application of environmental DNA me-thods to detect aquatic species[J]. Methods in Ecology and Evolution,7(11):1299-1307. doi:10.1111/2041-210X. 12595.
Gomes G B,Hutson K S,Domingos J A,Chung C,Hayward S,Miller T L,Jerry D R. 2017. Use of environmental DNA (eDNA) and water quality data to predict protozoan parasites outbreaks in fsh farms[J]. Aquaculture,479:467-473. doi:10.1016/j.aquaculture.2017.06.021.
Hood Y,Sadler J,Poldy J,Starkey C S,Robinson A P. 2019. Biosecurity system reforms and the development of a risk-based surveillance and pathway analysis system for ornamental fish imported into Australia[J]. Preventive Veterinaty Medicine,167:159-168. doi:10.1016/j.prevetmed.2018.11.006.
Jaroenlak P,Boakye D W,Vanichviriyakit R,Williams B A P,Sritunyalucksana K,Itsathitphaisarn O. 2018. Identification,characterization and heparin binding capacity of a sporewall,virulence protein from the shrimp microspori-dian,Enterocytozoon hepatopenaei(EHP)[J]. Parasites and Vectors,11(1):177. doi:10.1186/s13071-018-2758-z.
Jaroenlak P,Sanguanrut P,Williams B A P,Stentifor G D,Flegel T W,Sritunyalucksana K,Itsathitphaisrn O. 2016. A nested PCR assay to avoid false positive detection of the microsporidian Enterocytozoon hepatopenaei (EHP) in environmental samples in shrimp farms[J]. PLoS One,11(11):e0166320. doi:10.1371/journal.pone.0166320.
Lymbery A J,Morine M,Kanani H G,Beatty S J,Morgan D L. 2014. Coinvaders:The effects of alien parasites on native hosts[J]. International Journal for Parasitology:Parasites and Wildlife,3(2):171-177. doi:10.1016/j.ijppaw. 2014.04.002.
Palenzuela O,Redondo M J,Cali A,Takvorian P M,Alonso-Naveiro M,Alvarez-Pellitero P,Sitjà-Bobadilla A. 2014. A new intra nuclear microsporidium,Enterospora nucleophila n. sp.,causing an emaciative syndrome in a piscine host(Sparus aurata),prompts the redescription of family Enterocytozoonidae[J]. International Journal of Pa-rasitology,44(3-4):189-203. doi:10.1016/j.ijpara.2013. 10.005.
Pochon X,Zaiko A,Fletcher L M,Laroche O,Wood S A. 2017. Wanted dead or alive? Using metabarcoding of environmental DNA and RNA to distinguish living assemblages for biosecurity applications[J]. PLoS One,12:e0187636. doi:10.1371/journal.pone.0187636.
Robson H L A,Noble T H,Sanders R J,Robson S K A,Burrows D W,Jerry D R. 2016. Fine-tuning for the tropics:Application of eDNA technology for invasive fish detection in tropical freshwater ecosystems[J]. Molecular Eco-logy Resources,16(4):922-932. doi:10.1111/1755-0998. 12505.
Rusch J C,Hansen H,Strand D A,Markussen T,Hytterød S,Vrålstad T. 2018. Catching the fish with the worm:A case study on eDNA detection of the monogenean parasite Gyrodactylus salaris and two of its hosts,Atlantic salmon(Salmo salar) and rainbow trout(Oncorhynchus mykiss)[J]. Parasites and Vectors,11(1):333. doi:10.1186/ s13071-018-2916-3.
Tangprasittipap A,Srisala J,Chouwdee S,Somboon M,Chuchird N,Limsuwan C,Srisuvan T,Flegel T W,Sritunyalucksana K. 2013. The microsporidian Enterocytozoon hepatopenaei is not the cause of white feces syndrome in whiteleg shrimp Penaeus(Litopenaeus) vannamei[J]. BMC Veterinary Research,9(1):139. doi:10.1186/1746-6148-9-139.
Tisthammer K H,Cobian G M,Amend A S. 2016. Global biogeography of marine fungi is shaped by the environment[J]. Fungal Ecology,19:39-46. doi:10.1016/j.funeco. 2015.09.003.
Tomsen P,Willerslev E. 2015. Environmental DNA—An emerging tool in conservation for monitoring past and present biodiversity[J]. Biological Conservation,183:4-18. doi:10.1016/j.biocon.2014.11.019.
Tourtip S,Wongtripop S,Stentiford G D,Bateman K S,Sriurairatana S,Chavadej J,Sritunyalucksana K,Withyachumnarnkul B. 2009. Enterocytozoon hepatopenaei sp. nov. (Microsporida:Enterocytozoonidae),a parasite of the black tiger shrimp Penaeus monodon(Decapoda:Penaeidae):Fine structure and phylogenetic relationships[J]. Journal of Invertebrate Pathology,102(1):21-29. doi:10. 1016/j.jip.2009.06.004.
Trujillo-González A,Becker J A,Hutson K S. 2018. Chapter seven—Parasite dispersal in the goldfsh trade[J]. Advances in Parasitology,100:239-281. doi:10.1016/bs.apar.2018. 03.001.
Trujillo-González A,Becker J A,Vaughan D B,Hutson K S. 2019a. Correction to:Monogenean parasites infect ornamental fish imported to Australia[J]. Parasitology Research,118(1):383-384. doi:10.1007/s00436-018-6156-4.
Trujillo-González A,Edmunds R C,Becker J A,Hutson K S. 2019b. Parasite detection in the ornamental fsh trade using environmental DNA[J]. Scientific Reports,9:5173. doi:10.1038/s41598-41517-2.
Wang N,Yin W L,Zhang N,Yang J X,Jia P,Fang B H,Yue Z Q,Xie Y H,Li J Q,Zhao J L,Liu H. 2022. Establishment and clinical application of a detection method for the infectious hypodermal and hematopoietic necrosis virus in water[J]. Aquaculture,546:737228. doi:10.1016/j.aquaculture.2021.737228.
(責任编辑 兰宗宝)
