同源臂长度对CRISPR/Cas9介导hLF基因打靶山羊β-乳球蛋白位点效率的影响
2022-04-21李丹周鸣鸣何正义吴赵曼秋宋绍征
李丹 周鸣鸣 何正义 吴赵曼秋 宋绍征


摘要:【目的】探究同源臂長度对CRISPR/Cas9系统介导人乳铁蛋白基因(hLF)打靶山羊β-乳球蛋白基因(BLG)座位点效率的影响,为今后体细胞核移植制备BLG -/hLF +基因打靶山羊提供科学依据,也为CRISPR/Cas9基因编辑系统介导BLG基因或其他基因座位点定向精准分子修饰的遗传育种提供借鉴。【方法】针对山羊BLG基因第一外显子区域设计构建sgBLG/Cas9载体,电转染山羊胎儿成纤维细胞,PCR验证BLG基因座位点致突变活性;以BLC14乳腺特异性表达载体为基础构建3种同源臂长度(6.0、3.5和1.2 kb)的hLF基因打靶载体,分别与sgBLG/Cas9载体共转染山羊胎儿成纤维细胞,经500 µg/mL G418筛选后,采用PCR检测基因打靶情况。【结果】sgBLG/Cas9载体在山羊胎儿成纤维细胞BLG基因座附近切割DNA双链的致突变活性效率在30%~35%。构建获得3种同源臂长度的hLF基因打靶载体(BLC14-1、BLC14-2和BLC14-3),对应的同源臂长度分别为6.0、3.5和1.2 kb;将3种hLF基因打靶载体与sgBLG/Cas9载体共转染山羊胎儿成纤维细胞,经5次电转染和G418筛选,分别获得83、77和86株药物抗性细胞,经PCR同源重组检测最终获得42、38和44株BLG -/hLF +基因打靶细胞株,即hLF基因在山羊胎儿成纤维细胞BLG基因座的平均打靶效率分别为50.6%(42/83)、49.4%(38/77)和51.2%(44/86)。3种不同长度同源臂构建的hLF基因打靶载体在山羊BLG基因座位点的打靶效率在统计学上无显著差异(P>0.05),表明同源臂长度对CRISPR/Cas9介导hLF基因打靶山羊BLG基因座位点无显著影响。【结论】利用CRISPR/Cas9系统介导hLF基因打靶山羊胎儿成纤维细胞BLG基因座位点能成功获得多株hLF +/BLG -基因打靶细胞株(BLG基因座定点打靶hLF基因),但打靶载体同源臂长度对CRISPR/Cas9系统介导BLG位点定向整合hLF基因的打靶效率无明显影响。
关键词: 山羊;CRISPR/Cas9;基因打靶;同源臂长度;β-乳球蛋白基因(BLG);人乳铁蛋白基因(hLF)
中图分类号: S814.8 文献标志码: A 文章编号:2095-1191(2022)01-0182-09
Effects of homologous arm length on the efficiency of CRISPR/Cas9 mediated hLF gene knock-in at goat β-lactoglobulin locus
LI Dan1, ZHOU Ming-ming1, HE Zheng-yi2, WU Zhao-man-qiu1, SONG Shao-zheng1*
(1Department of Basic Medicine, School of Health and Nursing, Wuxi Taihu University, Wuxi, Jiangsu 214000, China; 2College of Veterinary Medicine, Yangzhou University/Jiangsu Provincial Research Center for Animal
Transgenesis and Biopharming, Yangzhou , Jiangsu 225009, China)
Abstract:【Objective】To explore the effect of homology arm length on the efficiency of CRISPR/Cas9 system-me-diated human lactoferrin gene (hLF) targeting goat β-lactoglobulin gene (BLG) locus, and to provide a scientific basis for the preparation of BLG -/hLF + gene target cells by somatic cell nuclear transfer in the future. It also provided a reference for genetic breeding of BLG gene or other gene locus directed by precise molecular modification mediated by CRISPR/Cas9 gene editing system. 【Method】According to the first exon region of the goat BLG gene, the sgBLG/Cas9 vector was designed and constructed, and it was electrotransfected goat fetal fibroblasts. The site-mutagenic activity of the BLG gene locus was verified by PCR. Based on BLC14 mammary gland-specific expression vector, three hLF gene targeting vectors with homology arm length (6.0, 3.5 and 1.2 kb) were constructed, and it was cotransfected into goat fetal fibroblasts with sgBLG/Cas9 vector respectively. After 500 µg/mL G418 screening, the gene targeting was detected by PCR. 【Result】The mutagenic activity efficiency of cleaving DNA double strands with BLG locus in goat fetal fibroblasts was 30%-35% by sgBLG/Cas9 vector. The hLF gene targeting vectors (BLC14-1, BLC14-2 and BLC14-3) were constructed to obtain three homology arm lengths, corresponding to 6.0, 3.5 and 1.2 kb. Three hLF gene targeting vectors and sgBLG/Cas9 vector were cotransfected into goat fetal fibroblasts. After 5 times of electrotransfections and G418 screening, 83, 77 and 86 drug-resistant cells were obtained respectively. Finally, 42, 38 and 44 BLG -/hLF + gene targeting cell lines were obtained by PCR homologous recombination detection. The average targeting efficiencies of hLF gene at the BLG locus of goat fetal fibroblasts were 50.6% (42/83), 49.4 % (38/77) and 51.2% (44/86). There was no statistically significant difference in the targeting efficiency of hLF gene targeting vectors constructed with three different lengths of homology arms at the BLG gene locus in goats (P>0.05), indicating that the homology arm length had no significant effect on CRISPR/Cas9-mediated hLF gene targeting at the BLG gene locus in goats. 【Conclusion】A number of BLG -/hLF + gene targeting cell lines(BLG loci for site-directed target hLF genes) have been successfully obtained by CRISPR/Cas9 system-mediated hLF gene targeting at BLG gene locus in goat fetal fibroblasts. However, the length of the homology arm of the targeting vector has no significant effect on the targeting efficiency of the CRISPR/Cas9 system-mediated BLG site-directed integration of the hLF gene.
Key words: goat; CRISPR/Cas9; gene targeting; homology arm length; β-lactoglobulin gene(BLG); human lactoferrin gene(hLF)
Foundation items: National Key Research and Development Program of China(2016YFE0126000);Science and Technology Project of Jiangxi Provincial Health Commission(202130627);Natural Science Foundation of Colleges and Universities in Jiangsu (19KJB180030);Excellent Young Backbone Teacher Project of “Qinglan Project” in Colleges and Universities of Jiangsu (Sujiaoshihan〔2021〕11)
0 引言
【研究意义】基因打靶是指利用同源重组的原理,通过精准的碱基插入、缺失或突变等方式改变生物体基因组中的某一特定基因,从而实现对生物遗传信息的定向改造(Dever et al.,2016)。CRISPR/Cas9是繼人工锌指核酸酶(ZFNs)和转录激活因子样效应物核酸酶(TALENs)之后新兴起的一种基因打靶编辑技术,主要由sgRNA序列引导Cas9蛋白核酸酶识别切割特异性DNA,使其双链断裂引发同源重组或非同源末端连接机制进行修复(Grunwald et al.,2019;朱丽颖等,2020;王妍鳕等,2021),具有编辑效率高、精准性高、构建序列简单等特点,已广泛应用于功能基因学、人类疾病动物模型及畜禽遗传育种改良等研究领域(Kleinstiver et al.,2016;Zhou et al.,2017;刘雷雷等,2019;Char et al.,2020)。因此,利用CRISPR/Cas9系统介导在山羊β-乳球蛋白(BLG)基因座位点打靶人乳铁蛋白(hLF)基因,可在敲除羊乳中致敏原BLG基因的同时插入hLF基因,有望实现羊乳人源化改造(Oliveira et al.,2019),有效提高羊乳品质,使其更具营养价值。【前人研究进展】以体细胞核移植和基因编辑为基础的家畜基因打靶技术在转基因新品种培育中具有重要意义,以动物体细胞为宿主细胞,打靶载体进入细胞核内,载体的同源序列与宿主基因组序列发生同源重组,进而完成基因的插入或敲除。为了便于同源重组检测,打靶载体通常包含长、短两个同源臂,在传统的基因打靶中同源臂长度是影响基因同源重组效率的关键因素(Shulman et al.,1990)。Shulman等(1990)研究发现,基因打靶效率随着同源臂长度的增加而呈现指数性增长,且同源臂序列最长可达14 kb;Thomas等(1992)研究表明,当同源臂长度低于1 kb时,会导致接头区重组的保真度降低;McCreath等(2000)通过基因打靶技术在绵羊COL1A1基因3'非翻译区引入hAAT基因,是最早的家畜基因打靶研究案例。随后,Shen等(2007)成功在山羊胎儿成纤维细胞中敲除β-酪蛋白基因,以期获得低致敏原性的转基因山羊新品系;王向鹏等(2012)利用正负筛选策略,通过构建2种不同长度(1.9和5.7 kb)的同源臂,成功获得基因打靶细胞株;刘婉霞等(2015)利用TALENs与多位点打靶载体设计,在293T细胞中进行高效基因打靶;Zhu等(2016)利用TALENs介导山羊BLG基因座定点打靶人α-乳清白蛋白基因,构建获得可用于转基因动物研究的打靶细胞系;Hryhorowicz等(2017)研究证实CRISPR/Cas9基因编辑技术可介导动物基因打靶,从而引入不同的外源打靶载体基因;广璐等(2019)利用CRISPR/Cas9介导hFAD3基因在牛胎儿成纤维细胞中开展基因打靶研究,通过构建5'和3'端不同长度的同源臂高效率获得中靶细胞株;王文文等(2020)基于CRISPR/Cas9基因编辑系统分别构建多个不同的MUC4和SLC12A8基因敲除载体,并探究不同载体的打靶效率。【本研究切入点】近年来,已有研究利用CRISPR/Cas9基因编辑系统分别在山羊成纤维细胞的β-酪蛋白和BLG基因位点进行打靶(刘畅,2016;Zhou et al.,2017),为羊乳人源化改造的分子遗传育种奠定了基础,但有关同源臂长度对CRISPR/Cas9系统介导山羊基因打靶效率的相关研究国内外鲜见报道。【拟解决的关键问题】构建3种不同同源臂长度的hLF基因打靶载体,利用CRISPR/Cas9基因编辑系统介导在山羊胎儿成纤维细胞的BLG基因座位点打靶hLF基因,探究同源臂长度对打靶效率的影响,为今后体细胞核移植制备BLG -/hLF +基因打靶细胞株提供科学依据,也为CRISPR/Cas9基因编辑系统介导BLG基因或其他基因座位点定向精准分子修饰的遗传育种提供借鉴。
1 材料与方法
1. 1 试验材料
妊娠30~35日龄的萨能奶山羊由扬州大学兽医学院提供,常规饲养于扬州大学农牧实验场。CRISPR/Cas9二合一质粒构建试剂盒(AxyPrep PCR Clea-nup Kit,AP-PCR-250)购自南京尧顺禹生物科技有限公司,BLC14质粒和菌种(哺乳动物乳腺特异性表达载体含山羊BLG基因5'端调控序列、山羊BLG基因3'端调控序列、hLF基因cDNA序列、NEO基因及CMV基因增强子,已通过小鼠、兔和山羊细胞进行表达验证)(An et al.,2012;Cheng et al.,2012)及大肠杆菌(Escherichia coli)DH5α感受态细胞均由扬州大学江苏省转基因动物制药工程研究中心保存提供;Trypsin(Amresco,0458)、DMEM/F12(Hyclone,D2906)、FBS(Hyclone,SH30070.03)、G418(Amesco,0344)、青链霉素(Sigma,J091911)及DNA胶回收纯化试剂盒购自QIAGEN公司;DNA聚合酶、连接酶及各种限制性内切酶购自宝生物工程(大连)有限公司;其他试剂均为国产分析纯,购自生工生物工程(上海)股份有限公司。
1. 2 引物设计与合成
根据GenBank已公布的山羊BLG基因序列和hLF基因序列,利用Primer Premier 5.0设计4对不同的扩增引物序列(表1),所有PCR引物均委托上海英骏生物技术有限公司合成。
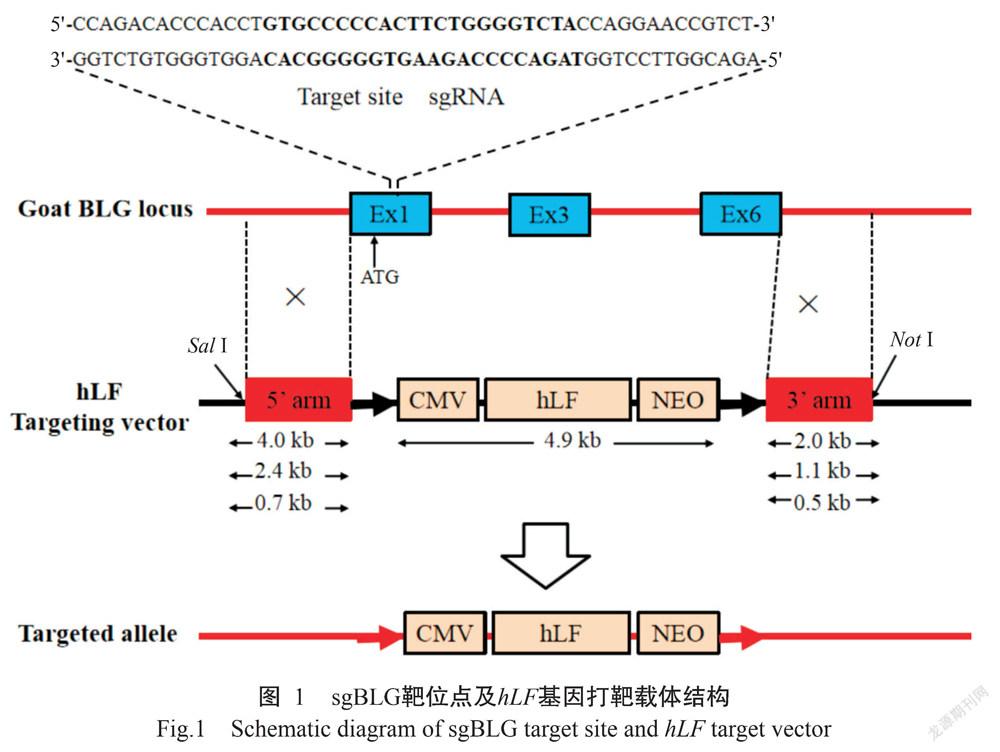
1. 3 sgRNA设计及sgBLG/Cas9载体和hLF基因打靶载体构建
根据山羊BLG基因座(Z33881.1,8088 bp)第一外显子序列设计sgRNA,使用gRNA(https://zlab.bio/ guide-design-resources)设计sgRNA引导序列(长度为22 nt),靶位点序列为GTGCCCCCACTTCTGGGGT CTA,通过酶切、连接及转化等常规分子生物学方法构建表达载体,并命名为sgBLG/Cas9。以山羊基因组DNA为模板,PCR扩增不同长度的同源臂;以BLC14乳腺特异性表达载体为基础,通过常规分子生物学技术构建3种不同同源臂长度的hLF基因打靶载体,分别命名为BLC14-1、BLC14-2和BLC14-3。
1. 4 山羊胎儿成纤维细胞分离培养
通过无菌剖宫产手术获取35日龄山羊胎儿,置于洁净培养平皿内,以D-Hank’s缓冲液洗涤3次;去除四肢、内脏和头部后,剪碎剩余组織(约1 mm3),经D-Hank’s缓冲液洗涤3次后,置于洁净离心管内。添加5 mL消化液(0.05%胰酶+0.04% EDTA-Na2),使用移液管反复吹打消化直至浑浊液完全产生后静置2 min,移液管吸取上层浑浊液置于另一洁净离心管内;1500×g离心5 min,D-Hank’s缓冲液重悬洗涤,重复2次,添加DMEM/F12+10% FBS细胞培养液重悬细胞,并调整密度为5×105个/mL(细胞计数板);然后接种于6孔细胞板内,置于CO2培养箱(37 ℃,5% CO2,饱和湿度)中静置培养,隔天换液。上述未完全消化的剩余组织块继续重复消化1次,收集细胞。
1. 5 CRISPR/Cas9编辑山羊BLG基因位点致突变活性检测
收集生长汇合度约80%的山羊胎儿成纤维细胞,电转染液重悬细胞并调整密度至1×106个/mL,sgBLG/Cas9质粒经回收纯化后调整终浓度至20 μg/mL,然后与细胞悬液混合进行电转染。转染条件:2 mm间隙电极杯,2.0 kV/cm、250 µs,电击2次,静置5 min。使用DMEM/F12+10% FBS培养液轻轻混匀转染后的细胞,接种至6孔细胞板内,置于CO2培养箱中静置培养(37 ℃,5% CO2,饱和湿度),48 h后换液。同时设未转染的正常细胞为阴性对照。待细胞生长汇合度至50%~60%时,胰蛋白酶消化收集细胞,提取基因组DNA,以sg-F/sg-R为引物(表1)PCR检测靶标区域,并对扩增产物进行测序,比对峰图突变情况。
1. 6 CRISPR/Cas9系统介导hLF基因打靶载体的转染与筛选
hLF基因打靶载体经Sal I/Not I双酶切线性化处理后,与纯化的sgBLG/Cas9质粒混合进行电转染,基因终浓度均为20 μg/mL,电转染方法与条件同1.5。48 h后添加500 µg/mL G418进行筛选,每隔48 h换1次液。同时设未经转染的正常山羊胎儿成纤维细胞为阴性对照。连续筛选培养10~14 d后,当阴性对照组细胞全部死亡时,换成正常培养液(DMEM/F12+10% FBS),挑取单克隆细胞株接种于48孔细胞板内继续培养,72 h后传代于12孔细胞板内进行扩大培养。
1. 7 BLG-/hLF+基因打靶细胞株检测与冻存
待细胞生长汇合度为80%~90%时,收集部分细胞,提取细胞基因组DNA用于PCR检测,以hLF-F/hLF-R为引物对hLF基因进行整合检测,以5'-BLG-F/5'-BLG-R为引物对5'端靶位点同源重组进行检测,以3'-BLG-F/3'-BLG-R位引物对3'端靶位点同源重组进行检测。剩余细胞继续培养48 h后,使用DMEM/F12+10% DMSO+20% FBS冻存液进行稀释,液氮速冻保存。
1. 8 统计分析
采用SPSS 22.0对获得的BLG -/hLF +基因打靶细胞株进行统计分析,比较3种不同长度同源臂打靶载体对基因打靶效率的影响。P >0.05时表明同源臂长度对基因打靶无显著影响,P<0.05时表明同源臂长度对基因打靶有显著影响。
2 结果与分析
2. 1 sgBLG/Cas9载体与hLF基因打靶载体构建情况
针对山羊BLG基因座设计构建的sgBLG/Cas9载体靶标位点位于第一外显子区域,如图1所示。在BLC14载体的基础上,根据山羊BLG基因组序列,经PCR扩增、酶切、连接及转化等分子生物学方法构建3种不同长度的同源臂作为打靶载体同源重组(表2),同源臂长度分别为6.0 kb(BLC14-1,5'端长臂4.0 kb+3'端短臂2.0 kb)、3.5 kb(BLC14-2,5'端长臂2.4 kb+3'端短臂1.1 kb)、1.2 kb(BLC14-3,5'端长臂0.7 kb+3'端短臂0.5 kb),其打靶载体结构如图1所示。
2. 2 山羊胎儿成纤维细胞的培养形态特征
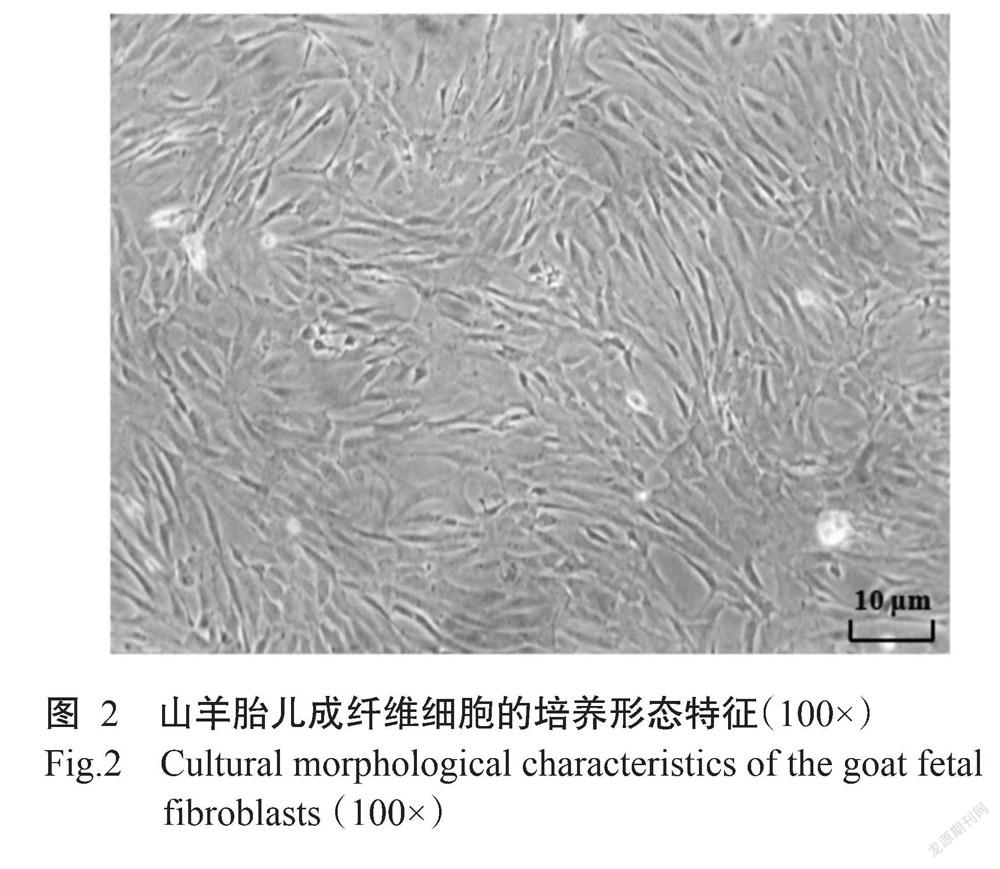
经胰蛋白酶消化法(0.05%胰酶+0.04% EDTA-Na2)体外分离获得的山羊胎儿成纤维细胞属于一种贴壁细胞,光学显微镜下观察其形态较小,典型的成纤维细胞呈细长条状、梭形或不规则形状。经传代培养,细胞生长汇合度为80%~90%时,细胞形成集落,细胞群呈涡旋状、火焰状或放射状排列,偶尔可见细胞群内有部分白色集落存在,如图2所示。
2. 3 CRISPR/Cas9编辑山羊胎儿成纤维细胞BLG基因座位点活性检测结果
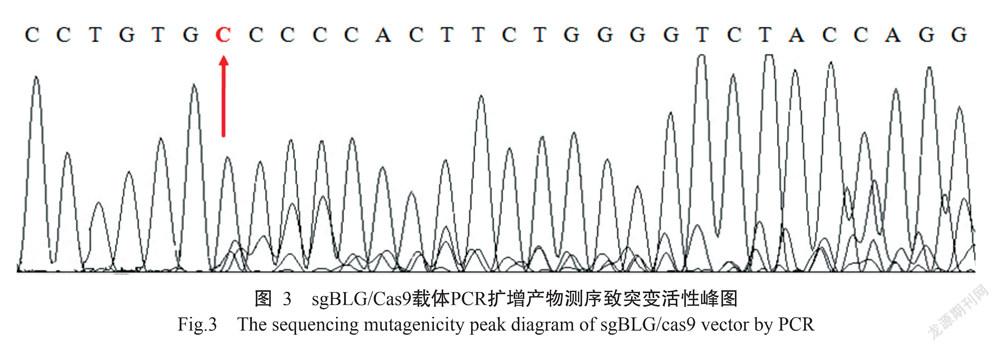
sgBLG/Cas9载体经纯化回收后,电转染山羊胎儿成纤维细胞,收集细胞后提取基因组DNA进行PCR扩增,PCR扩增产物经测序峰图验证突变情况,若峰图出现重叠的套峰,则表明该细胞系发生一定程度的突变,且根据重叠套峰面积所占比例可初步判断致突变活性(图3)。从图3中箭头所指处开始出现重叠套峰,即此处开始出现突变(山羊BLG基因座第一外显子中C碱基为sgBLG/Cas9特异性识别位点)。根据对重叠套峰面积的分析,可初步判断sgBLG/Cas9载体在山羊胎儿成纤维细胞BLG基因座附近切割DNA双链的致突变活性效率在30%~35%。
2. 4 hLF基因打靶细胞系构建及其效率分析结果
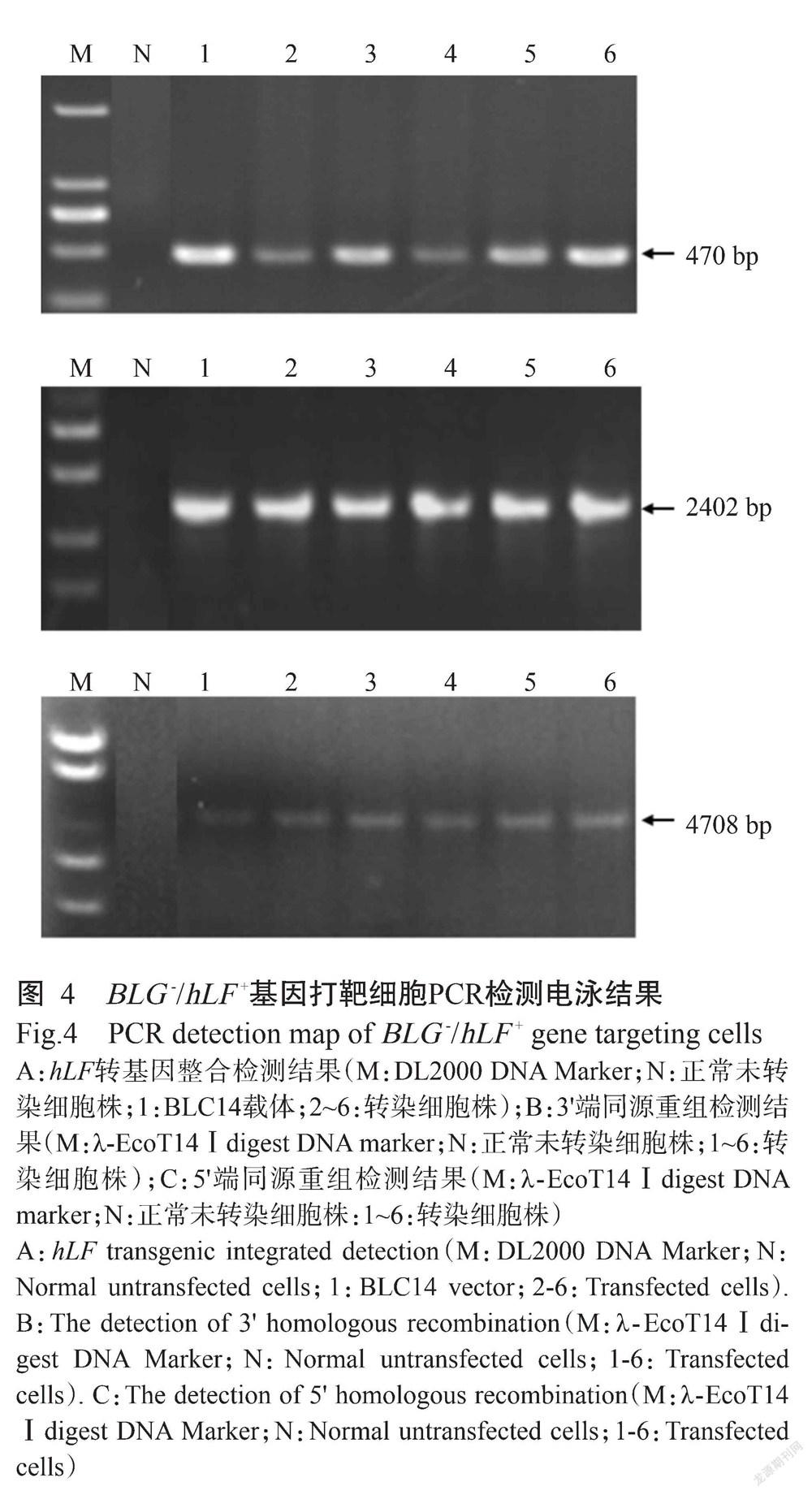
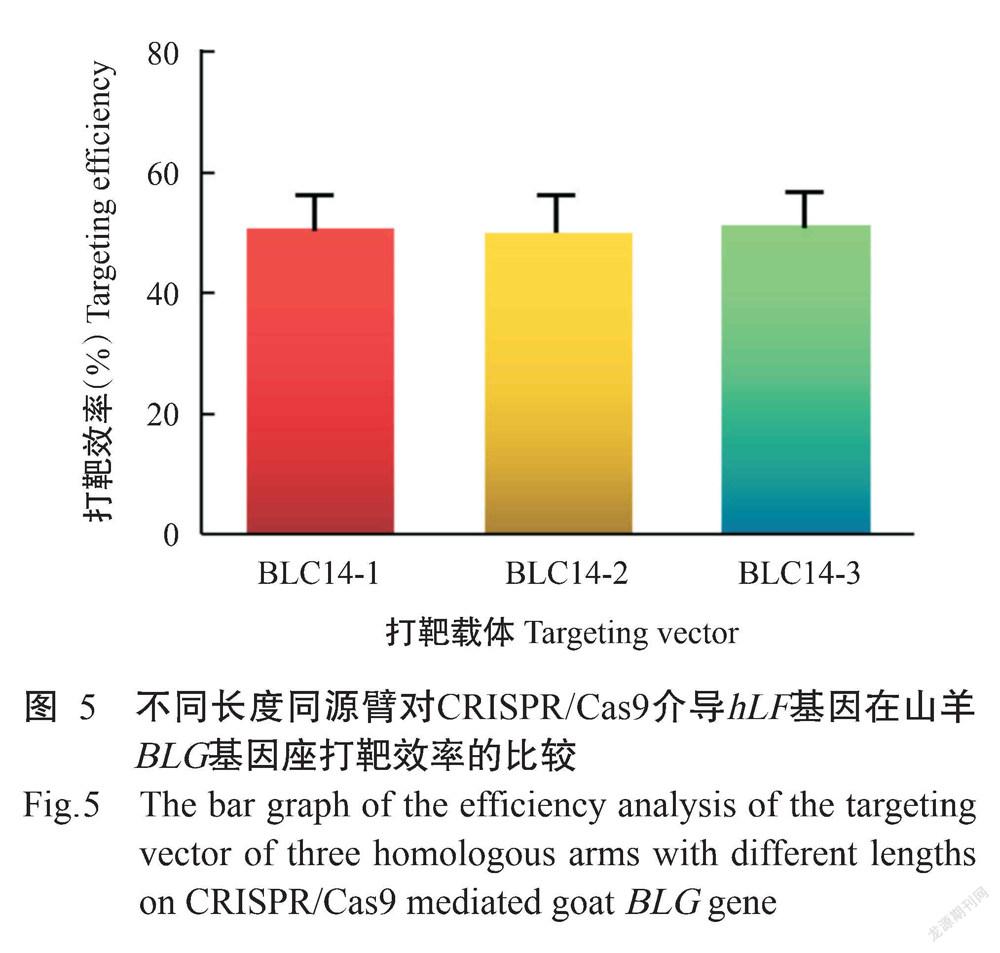
在相同的条件下,分别将3种hLF基因打靶载体(BLC14-1、BLC14-2和BLC14-3)与sgBLG/Cas9载体共转染山羊胎儿成纤维细胞,每个打靶载体转染5次,每次使用不同细胞系(5个细胞系转染),每次转染细胞数为1×106个。以hLF-F/hLF-R为引物对hLF基因进行整合检测,PCR扩增产物长度为470 bp(图4-A);以3'-BLG-F/3'-BLG-R为引物对hLF转基因细胞株进行3'端同源重组检测,PCR扩增产物长度为2402 bp(图4-B);以5'-BLG-F/5'-BLG-R为引物对hLF转基因细胞株进行5'端同源重组检测,PCR扩增产物长度为4708 bp(图4-C)。BLC14-1、BLC14-2和BLC14-3等3种hLF基因打靶载体(同源臂长度分别为6.0、3.5和1.2 kb)经5次电转染和G418筛选,分别获得83、77和86株药物抗性细胞株(表2)。其中,经PCR整合检测分别有76、72和80株hLF转基因细胞株,经PCR同源重组检测发现有42、38和44株为BLG -/hLF +基因打靶细胞株,hLF基因在山羊胎儿成纤维细胞BLG基因座的平均打靶效率分别为50.6%(42/83)、49.4%(38/77)和51.2%(44/86)。SPSS 22.0统计分析结果(图5)显示,3种不同长度同源臂构建的hLF基因打靶载体在山羊BLG基因座位点的打靶效率在统计学上无显著差异(P>0.05),表明同源臂长度对CRISPR/Cas9介导hLF基因在山羊BLG基因座附近的平均打靶效率无显著影响。
3 讨论
自20世纪初通过基因打靶和体细胞核移植技术成功获得多种基因打靶家畜以来,关于基因打靶的研究报道从未间断(Becher et al.,2018)。基因打靶是根据同源重组的原理,通过外源DNA与宿主染色体DNA的同源序列发生重组,从而将外源基因精准定点整合至宿主基因组上的特定位置,实现对生物体基因组定点精确修饰及改造的目的(Zhou et al.,2017),现已广泛应用于生物医药蛋白生产、器官移植、生物医学研究及动物品种改良分子遗传育种等领域。在家畜遗传育种方面,通过基因打靶技术获得转基因动物能实现短期内改造动物生产性能,赋予动物优良的性状,且外源基因的整合与表达不受宿主基因的“位置效应”影响,也不会干扰宿主其他基因的正常生理功能,具有遗传稳定、背景清楚等优点,更有利于转基因新品种的培育(Kim et al.,2016)。基因打靶技术历经了构建长同源臂、ZFNs、TALENs和CRISPR/Cas9等几代的发展,尤其是近年来CRISPR/Cas9系统介导的生物体基因组编辑,主要是利用单链引导序列sgRNA与Cas9核酸内切酶复合物,特异性切割断裂基因组DNA双链的特定位点,有效诱导同源重组或非同源末端连接机制进行修复,从而实现对生物体基因组定向精准编辑(黄娟等,2018;Grunwald et al.,2019)。本研究利用CRISPR/Cas9系统介导hLF基因打靶山羊BLG基因座位点,成功获得多株BLG -/hLF +基因打靶细胞株。
随着生活水平的不断提高和饮食结构的改变,人们对食用乳品的要求也越来越高(Sackesen et al.,2011)。BLG是牛乳和羊乳中主要的致敏原,可引起哮喘、呕吐及腹泻等过敏症状(Ehn et al.,2005;Oliveira et al.,2019;Varlamova and Zaripov,2020)。hLF是人乳中的主要乳清蛋白,具有抗菌、抗癌及增强免疫力等生物学活性,是理想的乳品添加剂(Wang et al.,2019;Shao et al.,2020)。由于山羊乳成分最接近于人乳,因此对山羊乳进行人源化改造,去除致敏原BLG基因的同时添加功能营养蛋白基因,赋予其人乳的特征,是一种理想的选择。本研究利用CRISPR/Cas9系统介导在山羊BLG基因座位点打靶hLF基因,能有效去除致敏原BLG并增加功能营养成分hLF,使山羊乳具有更高的营养价值和生物学功能。至今,已有关于CRISPR/Cas9系统介导山羊乳蛋白基因编辑的研究报道。刘畅(2016)应用CRISPR/Cas9系统介导在山羊胎儿成纤维细胞β-酪蛋白基因第二外显子序列中进行打靶,其打靶效率为74.29%;Zhou等(2017)利用CRISPR/Cas9系统介导山羊耳成纤维细胞BLG基因座位点打靶hLF基因,其打靶效率为36.69%。这些研究为羊乳蛋白基因编辑和转基因羊乳人源化改造的精准分子育种提供了科学依据,但主要集中于靶标位点的选择和设计,针对打靶载体同源臂构建,尤其是同源臂长度对打靶效率的影响鲜见报道。
本研究根据山羊BLG基因组序列,在BLC14乳腺特异性表达载体的基础上分别构建3种同源臂长度的hLF基因打靶载体,并系统比较分析不同长度同源臂在CRISPR/Cas9介导山羊乳蛋白基因座中的编辑效率。早期的基因打靶研究发现,打靶载体的同源臂长度是影响载体同源序列与宿主基因组序列发生同源重组的重要因素,因此,试图通过增加打靶载体同源臂的长度以提高重组打靶效率(聂宇等,2016)。Shulman等(1990)将打靶载体的同源臂长度增加至14.0 kb时,其打靶效率呈指数型增长趋势。An等(2012)直接构建长同源臂打靶载体应用于山羊BLG基因座打靶人乳白蛋白基因,结果发现打靶载体同源臂总长度低于5.5 kb时未筛选到打靶细胞株,而将同源臂延长至8.8 kb时其打靶效率明显提高至5.3×10-5。聂宇等(2016)对ZFNs介导基因打靶载体同源臂长度的研究结果表明,打靶载体单同源臂長度至少在1.0 kb以上才具有较高的重组效率,且证实当同源臂延长251 bp时,其重组效率可提高12.5%。本研究中,3种不同长度(6.0、3.5和1.2 kb)同源臂经CRISPR/Cas9介导的打靶效率分别为50.6%、49.4%和51.2%,即同源臂长度并未对hLF基因打靶载体在山羊BLG基因座位点的打靶效率造成显著影响,究其原因可能是CRISPR/Cas9基因编辑靶标位点的识别是由crRNA引导的RNA与DNA碱基配对过程,Cas9蛋白负责DNA切割,相对于TALENs蛋白对DNA序列的识别更加精准,且识别序列仅为几十个碱基,简单便捷(Curtin et al.,2018;Zhang et al.,2019),也进一步证实CRISPR/Cas9系统无需较长的同源臂打靶载体即可实现高效的基因打靶。该结论为羊乳中低致敏原性和富含功能营养成分的人源化改造及转基因动物精准分子遗传育种打下了理论基础。
4 结论
利用CRISPR/Cas9系统介导hLF基因打靶山羊胎儿成纤维细胞BLG基因座位点能成功获得多株hLF +/BLG -基因打靶細胞株(BLG基因座定点打靶hLF基因),但打靶载体同源臂长度对CRISPR/Cas9系统介导BLG位点定向整合hLF基因的打靶效率无明显影响。
参考文献:
广璐,张英,郭晶,白春玲,魏著英,于超然,扈廷茂,李光鹏. 2019. CRISPR/Cas9介导hFAD3基因在牛NCAPG-LCORL位点的定点整合[J]. 农业生物技术学报,27(1):12-22. [Guang L,Zhang Y,Guo J,Bai C L,Wei Z Y,Yu C Y,Hu T M,Li G P. 2019. Site-specific integration of hFAD3 gene in bovine (Bos taurus) NCAPG-LCORL locus mediated by CRISPR/Cas9[J]. Journal of Agricultu-ral Biotechnology,27(1):12-22.] doi:10.3969/j.issn.1674-7968.2019.01.002.
黄娟,邓国富,高利军,高菊,卿冬进,朱昌兰. 2018. CRISPR/Cas9系统及其在作物育种中的应用[J]. 南方农业学报,49(1):14-21. [Huang J,Deng G F,Gao L J,Gao J,Qing D J,Zhu C L. 2018. CRISPR/Cas9 system and its application in crop breeding[J]. Journal of Southern Agriculture,49(1):14-21.] doi:10.3969/j.issn.2095-1191.2018. 01.03.
刘畅. 2016. CRISPR/Cas9与TALENs介导奶山羊β-酪蛋白位点基因打靶效率的比较研究[D]. 呼和浩特:内蒙古大学. [Liu C. 2016. The study on efficiency of CRISPR/Cas9 and TALENs mediated gene targeting at dairy goat β-casein loucs[D]. Hohhot:Inner Mongolia University.] doi:10.7666/d.Y3027840.
刘雷雷,贾启鹏,李宗帅,杨洋,李海江,赵兴绪,张勇. 2019. 利用CRISPR/Cas9基因编辑技术靶向敲除奶山羊胎儿成纤维细胞SCD1基因[J]. 甘肃农业大学学报,54(6):30-38. [Liu L L,Jia Q P,Li Z S,Yang Y,Li H J,Zhao X X,Zhang Y. 2019. Targeted knockout of SCD1 gene in dairy goat fetal fibroblasts by CRISPR/Cas9 gene editing technology[J]. Journal of Gansu Agricultural University,54(6):30-38.] doi:10.13432/j.cnki.jgsau.2019.06.005.
刘婉霞,严爱芬,刘芳,蒋泓,冯翠兰,刘靖,唐冬生,张雅洁. 2015. 人工转录激活子样效应因子核酸酶与多位点基因打靶载体的构建与鉴定[J]. 中华生物医学工程杂志,21(5):400-406. [Liu W X,Yan A F,Liu F,Jiang H,Feng C L,Liu J,Tang D S,Zhang Y J. 2015. Artificial construction and identification of transcription activator-like effector nucleases and multi-loci gene targeting vector[J]. Chinese Journal of Biomedical Engineering,21(5):400-406.] doi:10.3760/cma.j.issn.1674-1927.2015.05.003.
聂宇,乔艳乐,陈瑶生,何祖勇. 2016. 供体同源臂长度对ZFN介导的同源重组效率的影响[J]. 中山大学学报(自然科学版),55(4):100-107. [Nie Y,Qiao Y L,Chen Y S,He Z Y. 2016. The effect of the length of donor homologous arm on the efficiency of ZFN-induced homologous recombination[J]. Acta Scientiarum Naturalium Universitatis Sunyatseni,55(4):100-107.] doi:10.13471/j.cnki.acta.snus.2016.04.017.
王文文,俞英,张勤. 2020. 利用CRISPR/Cas9系统构建猪MUC4和SLC12A8基因打靶载体[J]. 农业生物技术学报,28(7):1306-1313. [Wang W W,Yu Y,Zhang Q. 2020. Construction of gene targeting vectors for MUC4 and SLC12A8 in pig(Sus scrofa) by CRISPR/Cas9 system[J]. Journal of Agricultural Biotechnology,28(7):1306-1313.] doi:10.3969/j.issn.1674-7968.2020.07.017.
王向鹏,肖一红,刘园园,杜永坤,马玉萍,周恩民. 2012. 猪MSTN基因双筛选标记打靶载体的构建及其功能鉴定[J]. 西北农林科技大学学报(自然科学版),40(8):47-53. [Wang X P,Xiao Y H,Liu Y Y,Du Y K,Ma Y P,Zhou E M. 2012. Construction and functional analysis of a pig myostatin gene targeting vector containing double-selection markers[J]. Journal of Northwest A & F University (Natural Science Edition),40(8):47-53.] doi:10.13207/j.cnki.jnwafu.2012.08.033.
王妍鱈,任亭亭,孙跃峰,刘磊. 2021. 利用CRISPR/Cas9系统构建SBNO2基因敲除细胞系及其功能研究[J]. 甘肃农业大学学报,56(1):22-28. [Wang Y X,Ren T T,Sun Y F,Liu L. 2021. Construction of SBNO2 knockout cell lines using CRISPR/Cas9 system and its function evaluation[J]. Journal of Gansu Agricultural University,56(1):22-28.] doi:10.13432/j.cnki.jgsau.2021.01.004.
朱丽颖,郑月萍,徐雪珍,段芊芊,韩妮莎. 2020. 一种准确、简便测定CRISPR/Cas9基因编辑效率的方法[J]. 江苏农业学报,36(2):299-305. [Zhu L Y,Zheng Y P,Xu X Z,Duan Q Q,Han N S. 2020. A convenient and accurate method for determining the efficiency of CRISPR/Cas9-based gene editing[J]. Jiangsu Journal of Agricultural Sciences,36(2):299-305.] doi:10.3969/j.issn.1000-4440. 2020.02.007.
An L Y,Yuan Y G,Yu B L,Yang T J,Cheng Y. 2012. Gene-ration of human lactoferrin transgenic cloned goats using donor cells with dual markers and a modified selection procedure[J]. Theriogenology,78(6):1303-1311. doi:10. 1016/j.theriogenology.2012.05.027.
Becher B,Waisman A,Lu L F. 2018. Conditional gene-targe-ting in mice:Problems and solutions[J]. Immunity,48(5):835-836. doi:10.1016/j.immuni.2018.05.002.
Char S N,Wei J L,Mu Q,Li X R,Zhang Z J,Yu J M,Yang B. 2020. An agrobacterium-delivered CRISPR/Cas9 system for targeted mutagenesis in sorghum[J]. Plant Biotechnology Journal,18(2):319-321. doi:10.1111/pbi. 13229.
Cheng Y,An L Y,Yuan Y G,Wang Y,Du F L,Yu B L,Zhang Z H,Huang Y Z,Yang T J. 2012. Hybrid expression cassettes consisting of a milk protein promoter and a cytomegalovirus enhancer significantly increase mammary-specific expression of human lactoferrin in transgenic mice[J]. Molecular Reproduction and Development,79(8):573-585. doi:10.1002/mrd.22063.
Curtin S J,Xiong Y,Michno J M,Campbell B W,Stec A O,Čermák T,Starker C,Voytas D F,Eamens A L,Stupar R M. 2018. CRISPR/Cas9 and TALENs generate heritable mutations for genes involved in small RNA processing of Glycine max and Medicago truncatula[J]. Plant Biotechnology Journal,16(6):1125-1137. doi:10.1111/pbi.12857.
Dever D P,Bak R O,Reinisch A,Camarena J,Washington G,Nicolas C E,Pavel-Dinu M,Saxena N,Wilkens A B,Mantri S,Uchida N,Hendel A,Narla A,Majeti R,Weinberg K I,Porteus M H. 2016. CRISPR/Cas9 β-globin gene targeting in human haematopoietic stem cells[J]. Nature,539(7629):384-389. doi:10.1038/nature20134.
Ehn B M,Allmere T,Telemo E,Bengtsson U,Ekstrand B. 2005. Modification of IgE binding to beta-lactoglobulin by fermentation and proteolysis of cow’s milk[J]. Journal of Agricultural and Food Chemistry,53(9):3743-3748. doi:10.1021/jf048121w.
Grunwald H A,Gantz V M,Poplawski G,Xu R X S,Bier E,Cooper K L. 2019. Super-Mendelian inheritance media-ted by CRISPR-Cas9 in the female mouse germline[J]. Nature,566(7742):105-109. doi:10.1038/s41586-019-0875-2.
Hryhorowicz M,Lipiński D,Zeyland J,Słomski R. 2017. CRISPR/Cas9 immune system as a tool for genome engineering[J]. Archivum Immunologiae et Therapiae Experimentails(Warsz),65(3):233-240. doi:10.1007/s00005-016-0427-5.
Kim Y,Cheong S A,Lee J G,Lee S W,Lee M S,Baek I J,Sung Y H. 2016. Generation of knockout mice by Cpf1-mediated gene targeting[J]. Nature Biotechnology,34(8):808-810. doi:10.1038/nbt.3614.
Kleinstiver B P,Pattanayak V,Prew M S,Tsai S Q,Nguyen N T,Zheng Z,Joung J K. 2016. High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects[J]. Nature,529(7587):490-495. doi:10.1038/nature16526.
McCreath K J,Howcroft J,Campbell K H,Colman A,Schnieke A E,Kind A J. 2000. Production of gene-targeted sheep by nuclear transfer from cultured somatic cells[J]. Nature,405(6790):1066-1069. doi:10.1038/35016604.
Oliveira J P B,Candreva A M,Rizzo G,Ramos M V,Oliveira J S,Oliveira H D,Ary M B,Docena G,Freitas C D T. 2019. Allergenicity reduction of cow’s milk proteins using latex peptidases[J]. Food Chemistry,284:245-253. doi:10.1016/j.foodchem.2019.01.123.
Sackesen C,Assa'ad A,Baena-Cagnani C,Ebisawa M,Fiocchi A,Heine R G,von Berg A,Kalayci O. 2011. Cow’s milk allergy as a global challenge[J]. Current Opinion Allergy and Clinical Immunology,11(3):243-248. doi:10.1097/ACI.0b013e328346566f.
Shao Y H,Zhang Y,Liu J,Tu Z C. 2020. Influence of ultrasonic pretreatment on the structure,antioxidant and IgG/IgE binding activity of β-lactoglobulin during digestion in vitro[J]. Food Chemistry,312:126080. doi:10.1016/j.foodchem. 2019.126080.
Shen W,Lan G C,Yang X Y,Li L,Min L J,Yang Z T,Tian L Y,Wu X J,Sun Y J,Chen H,Tan J H,Deng J X,Pan Q J. 2007. Targeting the exogenous htPAm gene on goat somatic cell beta-casein locus for transgenic goat production[J]. Molecular Reproduction and Development,74(4):428-434. doi:10.1002/mrd.20595.
Shulman M J,Nissen L,Collins C. 1990. Homologous recombination in hybridoma cells:Dependence on time and fragment length[J]. Molecular and Cellular Biology,10(9):4466-4472. doi:10.1128/mcb.10.9.4466-4472.1990.
Thomas K R,Deng C,Capecchi M R. 1992. High-fidelity gene targeting in embryonic stem cells by using sequence replacement vectors[J]. Molecular and Cellular Biology,12(7):2919-2923. doi:10.1128/mcb.12.7.2919-2923. 1992.
Varlamova E G,Zaripov O G. 2020. Beta-lactoglobulin-nutrition allergen and nanotransporter of different nature ligands therapy with therapeutic action[J]. Research in Veterinary Science,133:17-25. doi:10.1016/j.rvsc.2020. 08.014.
Wang B,Timilsena Y P,Blanch E,Adhikari B. 2019. Lactoferrin:Structure,function,denaturation and digestion[J]. Critical Reviews in Food Science and Nutrition,59(4):580-596. doi:10.1080/10408398.2017.1381583.
Zhang H X,Zhang Y,Yin H. 2019. Genome editing with mRNA encoding ZFN,TALEN,and Cas9[J]. Molecular Therapy,27(4):735-746. doi:10.1016/j.ymthe.2019.01.014.
Zhou W J,Wan Y J,Guo R H,Deng M T,Deng K P,Wang Z,Zhang Y L,Wang F. 2017. Generation of beta-lactoglo-bulin knock-out goats using CRISPR/Cas9[J]. PLoS One,12(10):e0186056. doi:10.1371/journal.pone.0186056.
Zhu H M,Liu J,Cui C C,Song Y J,Ge H T,Hu L Y,Li Q,Jin Y P,Zhang Y. 2016. Targeting human α-lactalbumin gene insertion into the goat β-lactoglobulin locus by TALEN-mediated homologous recombination[J]. PLoS One,11(6):e0156636. doi:10.1371/journal.pone.0156636.
(責任编辑 兰宗宝)
