基于同源双交换的聚球藻PCC 7942基因组大片段无标记删除
2022-04-13柯竹芳徐旭东
柯竹芳 徐旭东 高 宏
(1. 中国科学院水生生物研究所中国科学院藻类生物学重点实验室, 武汉 430072; 2. 中国科学院大学, 北京 100049)
蓝藻是一类具植物型放氧光合系统的原核生物, 被广泛用于光合作用[1]、原核细胞分化和图式形成[2]、固氮作用和昼夜节律[3]等研究。同时, 蓝藻还可能作为微藻细胞工厂, 合成多种天然活性物质和工业化学品, 极具开发利用潜力[4]。在蓝藻合成生物学中, 基因组简化有利于提高底盘基因组的可控性和可预测性, 避免无关基因的干扰, 为蓝藻合成生物学的研究和应用提供一个优化的底盘细胞。通过自上而下的基因组简化是构建底盘细胞的重要途径[5], 借助基因组编辑技术删除冗余序列可以获取基因组简化藻株。若是对基因组中非必需基因逐个地删除, 工作量大且耗时久。因此, 发展高效的大片段无标记删除技术是基因组简化的关键。无标记遗传操作技术在大肠杆菌和枯草芽孢杆菌基因组简化研究中已取得较大的进展[6—9],而蓝藻基因组简化的研究发展相对较晚, 在蓝藻细胞中开发基因组大片段无标记删除技术进行基因组简化对于构建高效的合成生物学平台很有必要。理论上, 传统的同源重组技术可以用于无标记的基因删除, 但是应用此技术在蓝藻中进行大片段的无标记删除目前尚没有报道。为此, 本研究尝试使用传统的同源重组和筛选技术删除蓝藻中的大片段, 为进一步开展基因组简化研究打下基础。
聚球藻(Synechococcus elongatus)PCC 7942是一种单细胞淡水蓝藻, 最早被称为Anacystis nidulansR2, 也是最早发现可以进行自然转化的蓝藻[10],实际上也可以通过接合转移开展遗传操作。由于生长快速、结构简单和具有高效的遗传操作系统[11—13]等特点, 是研究光合作用和昼夜节律的重要模式生物, 并且正被开发为用作生产可再生燃料、化学品和药物的细胞工厂。2015年, Rubin等[14]完成对聚球藻PCC 7942全基因组的饱和诱变, 利用转座插入突变和测序方法, 发现其基因组的2723个基因中有1748个为非必需基因。聚球藻PCC 7942基因组中的非必需基因的确定为我们进行大片段删除提供了重要信息。本研究以聚球藻PCC 7942基因组中3个大于10 kb的非必需区域的无标记删除为例, 证明利用传统技术在蓝藻中进行无标记删除大片段的可行性。
1 材料与方法
1.1 菌株(藻株)、质粒和培养条件
聚球藻PCC 7942系中国科学院青岛生物能源与过程研究所吕雪峰实验室提供, 在30℃, 30 μE/(m2·s)连续光照条件下以BG11液体培养基静置培养。通过测定OD730来检测藻株生长情况, 待藻细胞生长达到对数期时用于各项实验。E. coliDH5a、E.coliHB101在37℃以LB振摇培养。含有质粒的大肠杆菌根据其携带的抗性基因补加相应抗生素: 50 μg/mL壮观霉素 (Sp)、100 μg/mL氨苄青霉素 (Ap) 或10 μg/mL四环素(Tc)、20 μg/mL氯霉素 (Cm), 使用2种及以上抗生素时浓度减半。所有BG11或LB固体培养基中补加1.5%琼脂。接合转移时向BG11固体培养基添加10 μg/mL硫酸链霉素 (Smr) 或25 μg/mL壮观霉素 (Spr), 筛选双交换转化子时向BG11固体培养基中添加5%的蔗糖。质粒pRL443[15]、pRL623[15]和pRL277[16]来自美国密西根州立大学Peter Wolk实验室。
1.2 DNA操作方法
蓝藻基因组的提取参照Cai和Wolk[17]所描述的方法进行, 稍加修改。50 μL PCR反应体系中含25 μL 2×Phanta Max Buffer, 1 μL dNTP Mix (10 mmol/L),上下游引物(10 μmol/L)各2 μL(表 1), 1 μL Phanta Max Super-Fidelity DNA Polymerase(1U), 模板DNA适量, 其余用ddH2O补至50 μL。PCR反应程序: 首先, 95℃预变性3min; 然后, 按照95℃变性15s, 58℃退火15s, 72℃延伸1min 30s进行30个循环反应; 最后, 72℃终延伸5min。融合PCR反应参照文献[18]进行。限制性酶切、连接和转化等DNA重组操作按Molecular Cloning[19]描述的方法进行。PstⅠ和SacⅠ限制性内切酶、T4 DNA连接酶购自宝生物工程(大连)有限公司。PCR产物经琼脂糖凝胶电泳检测后用乙醇沉降并洗涤纯化。之后纯化的PCR产物经PstⅠ和SacⅠ酶切并克隆至经相同酶切的pRL277中, 所得克隆均经过测序验证确保没有发生突变。

表1 本研究中用到的引物Tab. 1 Primers used in this study
1.3 蓝藻和大肠杆菌的接合转移
聚球藻PCC 7942接合转移参照文献[20]进行,略有改动。待藻细胞生长至OD730为0.8—1.2时取10 mL藻液离心收集, 用BG11洗3次, 重悬于1 mL的BG11中。同时收集10 mLE. coliHB101 (pRL443+pRL623+运载质粒), 用LB洗涤3次并重悬于1 mL LB中。将藻液和菌液混合, 弱光孵育4h后涂布到覆盖有硝酸纤维素滤膜的BG11平板上, 每个平板涂布200 μL菌藻混合液。光照24h后将膜转到含有10 μg/mL硫酸链霉素的BG11平板上继续培养至接合子长出。
1.4 蓝藻突变株的筛选
突变株的筛选参考在鱼腥藻PCC7120中描述的方法[20], 但筛选双交换子时不使用任何抗生素标记。先将接合子在含硫酸链霉素平板上划线培养,待长出之后转接至20 mL含相应抗生素的BG11中传代培养并提取基因组DNA, 进行PCR检测筛选完全分离的单交换子。然后, 涂布到含5%蔗糖的BG11固体培养基进行双交换子的筛选。待双交换子长出后, 划线于含5%蔗糖的BG11固体培养基, 再转接到BG11中进行培养并提取其基因组进行PCR检测。
2 结果
2.1 用于大片段删除的质粒构建
利用传统同源重组技术删除非必需区域大片段所用的质粒携带有两侧同源臂、oriT位点、抗性选择标记和sacB致死基因, 但不能在蓝藻细胞中自主复制。利用接合转移将构建的质粒转移到聚球藻PCC 7942中, 质粒通过其中一个同源臂与基因组DNA发生单交换整合到基因组上; 单交换子基因组中的另一同源片段再发生同源重组, 经过含5%蔗糖的BG11的筛选, 杀死携带sacB的细胞, 就可实现非必需区域的无标记删除(图 1)。

图1 两步同源重组删除大片段的示意图Fig. 1 A schematic diagram showing the deletion of a large DNA fragment via two-step homologous recombinations将带有两侧同源序列的质粒通过单交换整合到聚球藻PCC7942基因组中, 获得带有Spr/Smr抗性的单交换子; 之后, 在蔗糖平板上筛选发生第二次交换的藻落The plasmid with two homologous DNA fragments was integrated into the genome of Synechochoccus sp. PCC 7942 via single crossover; colonies with double crossover were selected on sucrose-containing plates
为删除聚球藻PCC 7942基因组中3个大于10 kb的非必需区域, 即Synpcc7942_0050~Synpcc7942_0064(简称0050-0064)、Synpcc7942_0233~Synpcc 7942_0253(简称0233-0253)、Synpcc7942_1391~Synpcc 7942_1400(简称1391-1400), 作者利用pRL277构建了用于相应基因组区段删除的质粒。以0050-0064为例, 用基因组DNA为模板、以引物0050-0064-F1/R1和0050-0064-F2/R2(表 1)进行PCR扩增,获得待删除区域两侧的同源臂, 将二者通过融合PCR获得的融合片段经PstⅠ和SacⅠ酶切并克隆至质粒pRL277, 得到用于0050—0064基因组区段删除的质粒pHB6442。用同样的方法克隆得到用于0233—0253和1391—1400基因组区段删除的质粒pHB6522和pHB6615。
2.2 单交换子的获得
接合转移的完成需要运载质粒、辅助质粒和接合质粒的参与。本实验中用到的接合质粒为pRL443, 辅助质粒为pRL623。通过转化将构建的pHB6442、pHB6522 和pHB6615三个运载质粒分别转入E. coliHB101(pRL443+pRL623), 得到用于接合转移的3个菌株E. coliHB101(pRL443+pRL623+pHB6442)、E. coliHB101(pRL443+pRL623+pHB6522)
和E. coliHB101(pRL443+pRL623+pHB6615)。
将3个E. coliHB 101(运载质粒+pRL443+pRL623)菌株分别与聚球藻PCC 7942进行接合转移, 运载质粒转移到藻细胞中。由于没有蓝藻复制子, 运载质粒不能在蓝藻中自主复制, 必须通过其携带的同源臂与基因组非必需区域的上游或者下游同源部分发生单交换(图 2)。通过同源臂发生整合, 运载质粒稳定存在于PCC 7942的基因组上。所有藻株经过多轮的筛选, 以基因特异的引物(表 1)进行PCR检测。
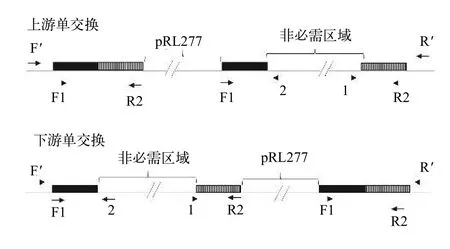
图2 单交换子的PCR检测引物位置示意图Fig. 2 A schematic diagram showing the location of primers used for PCR detection of single crossover recombinantsF′和R′为两侧同源臂以外序列的引物, F1和R2为用于扩增同源臂的4条引物中的2条, 1和2为待删除非必需区域内侧的引物F′ and R′ are primers beyond the two homologous arms, F1 and R2 are 2 of 4 primers for PCR amplification of the homologous arms,1 and 2 are primers located within the large DNA fragment to be deleted
在Synechococcus7942::pHB6442接合子的PCR检测中, 发现随机筛选的接合子中有一半的概率为同时发生上游和下游的单交换整合的突变株(结果未附)。图 3中泳道1—12为Synechococcus7942::pHB6442同时发生上游和下游单交换整合的突变株检测。挑选Synechococcus7942::pHB6522和Synechococcus7942::pHB6615的接合子进行PCR检测时, 未观察到这种情况。图 3中泳道13—18为Synechococcus7942::pHB6522 发生下游单交换整合的突变株检测, 泳道19—24为Synechococ-cus7942::pHB6615发生下游单交换整合的突变株检测。
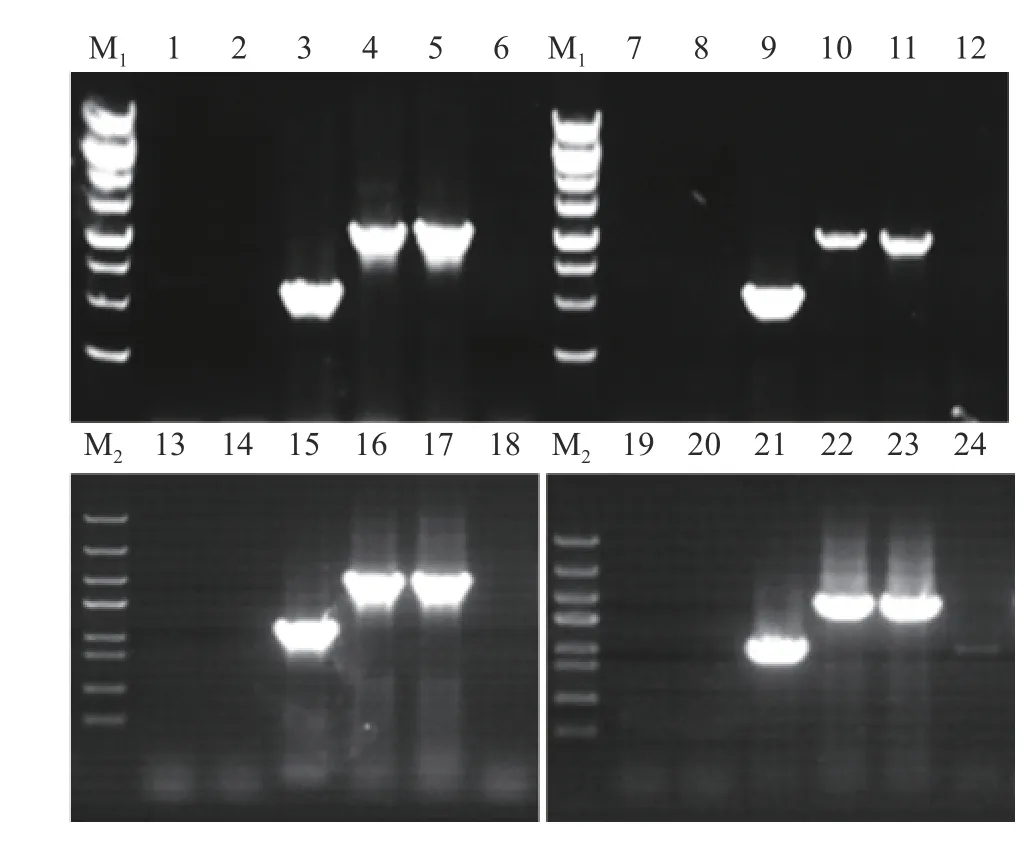
图3 分离完全的单交换突变株的PCR检测Fig. 3 PCR examination of completely segregated singlecrossover mutants泳道M1为1 kb DNA Ladder, 条带大小从上而下依次是10、8、6、5、4、3、2、1.5、1.0和0.5 kb; 泳道M2为Trans 5K DNA Marker, 条带大小从上而下依次是5、3、2、1.5、1、0.8、0.5和0.3 kb; 泳道1和2为pHB6442单交换接合子, 泳道3为野生型, 引物为0050-0064-Fˊ/2; 泳道4和5为pHB6442单交换接合子,泳道6为野生型, 引物为0050-0064-Fˊ/R2; 泳道7和8为pHB6442单交换接合子, 泳道9为野生型, 引物为0050-0064-1/Rˊ; 泳道10和11为pHB6442单交换接合子, 泳道12为野生型,引物为0050-0064-F1/Rˊ; 泳道13和14为pHB6522单交换接合子,泳道15为野生型, 引物为0233-0253-1/Rˊ; 泳道16和17为pHB6522单交换接合子, 泳道18为野生型, 引物为0233-0253-F1/Rˊ; 泳道19和20为pHB6615单交换接合子, 泳道21为野生型,引物为1391-1400-1/Rˊ; 泳道22和23为pHB6615单交换接合子,泳道24为野生型, 引物为1391-1400-F1/RˊLane M1, 1 kb DNA ladder (from top to bottom: 10, 8, 6, 5, 4, 3, 2,1.5, 1.0, 0.5 kb); lane M2, Trans 5K DNA Marker (from top to bottom: 5, 3, 2, 1.5, 1, 0.8, 0.5, 0.3 kb). Lanes 1 and 2 show PCR detection of Synechococcus PCC 7942::pHB6442 singlerecombinants, lane 3 shows detection of the wild type (WT)control, where primers 0050-0064-Fˊ/2 were used. Lanes 4 and 5,Synechococcus PCC 7942::pHB6442; lane 6, WT; primers: 0050-0064-Fˊ/R2. Lanes 7 and 8, Synechococcus PCC 7942::pHB6442;lane 9, WT; primers: 0050-0064-1/Rˊ. Lanes 10 and 11,Synechococcus PCC 7942::pHB6442; lane 12, WT; primers: 0050-0064-F1/Rˊ. Lanes 13 and 14, Synechococcus PCC 7942::pHB 6522; lane 15, WT; primers: 0233-0253-1/Rˊ. Lanes 16 and 17,Synechococcus PCC 7942::pHB6522; lane 18, WT; primers: 0233-0253-F1/Rˊ. Lanes 19 and 20, Synechococcus PCC 7942::pHB 6615; lane 21, WT; primers: 1391-1400-1/Rˊ. Lanes 22 and 23,Synechococcus PCC 7942::pHB6615; lane 24, WT; primers: 1391-1400-F1/Rˊ
2.3 无标记删除
由于运载质粒上含有蔗糖致死基因sacB, 所以单交换突变株不能在含5%蔗糖的BG11培养基中生长。利用蔗糖筛选第二步交换时, 可能获得定向突变株, 也可能获得回复的野生型(决定于哪一侧发生交换), 概率各占50%, 因此需要从多个蔗糖抗性的藻落中挑选出无标记删除突变株。挑取蔗糖平板上获得的藻落, 扩大培养, 反复筛选, 并进行两对PCR检测, PCR扩增30个循环。图 4显示最终获得的无标记删除突变株ΔSynpcc 0050-0064、ΔSynpcc 0233-0253和ΔSynpcc 1391-1400, 所删除片段均在10 kb以上, 其细胞中完全没有野生型基因组存在。
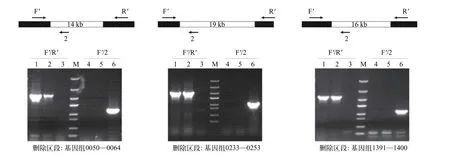
图4 分离完全的双交换突变株的PCR检测Fig. 4 PCR examination of completely segregated double-crossover mutants泳道M为Trans 5K DNA Marker, 条带大小从上而下依次是5、3、2、1.5、1、0.8、0.5和0.3 kb。泳道1和2为双交换突变株, 泳道3为野生型, 引物为Fˊ/Rˊ; 泳道4和5为双交换突变株, 泳道6为野生型, 引物为Fˊ/2Lane M, Trans 5K DNA Marker (from top to bottom: 5, 3, 2, 1.5, 1, 0.8, 0.5, 0.3 kb). Lanes 1 and 2 show PCR detection of double-crossover recombinants, lane 3 shows detection of the wild type (WT) control using primers of Fˊ/Rˊ. Lanes 4 and 5 were double-crossover recombinants; lane 6 was WT using primers of Fˊ/2
3 讨论
原核生物中已有若干种报道了基因组的简化研究。譬如, 在大肠杆菌(Escherichia coli)K-12中删除不稳定片段和非必需基因, 获得了基因组减少15%的菌株[21]。该菌株生长能力和蛋白表达特性依旧良好, 并且电击转化效率和质粒稳定性等性状有所提高。再如, 在枯草芽孢杆菌(Bacillus subtilis)中删除非必需基因, 获得了一系列基因组删减程度不同的菌株。这些菌株的生长速率和转化效率变低, 但细胞产量提高[22]。蓝藻在微生物合成生物学研究中具有独特的价值, 基因组简化也是在蓝藻构建良好的底盘细胞所需要的。聚球藻PCC 7942基因组较小, 而且其基因组中的非必需基因已经系统地鉴定, 为此, 我们选用该藻株开展基因组的简化工作。
借助于同源重组和条件致死基因sacB可在某些细菌获得无标记的删除突变株, 应用于基因组简化[22]。这一过程既可以利用线性DNA转化细胞来实现, 也可以通过质粒的接合转移来实现。聚球藻PCC 7942的遗传操作通常是借助于DNA的转化作用, 但实际上也完全可以利用接合转移将质粒导入其细胞, 而对于实现单交换来说, 接合转移的效率要高得多。本研究结果显示, 接合转移可以有效地将质粒导入该藻株, 并获得单交换子。
在以往的报道中, 利用传统的同源重组和sacB的条件致死作用可以在蓝藻筛选获得一些小片段DNA删除的突变株[23]。而且, 由于蓝藻细胞中具有多拷贝基因组[24], 使用传统同源重组技术删除基因组片段, 在第二步重组筛选时没有抗性选择标记, 往往会筛选到未分离完全的突变株, 也就是仍携带野生型基因组的突变株。虽然如此, 我们发现利用这一方法可以在聚球藻PCC 7942实现10 kb以上区域的删除, 并且在3个非必需区域的删除中都获得了成功。因此, 应用传统的方法也可以对蓝藻基因组进行大片段删除, 进而实现简化。一般来说,第一步同源重组时, 质粒会整合到基因组的上游或者下游, 但是在我们的实验中也观察到质粒同时整合到相应位置的上游和下游的情形。选取上下游同时发生单交换的单交换子进行第二步重组筛选,更容易获得完全分离的缺失突变株。譬如, 在删除Synpcc7942_0050-0064基因组区段时, 选取上下游同时发生双交换的藻株进行第二步重组筛选, 所检测的15个克隆全部为分离完全的缺失突变株。
蓝藻的接合转移和基于sacB的筛选系统最早是在丝状固氮蓝藻发展起来的, 实际上对于单细胞蓝藻也是适用的。所以, 我们有理由相信, 本研究展示的大片段基因组删除方法在可遗传操作的蓝藻中是广泛适用的。尽管本研究证明传统的同源重组和筛选技术可以对蓝藻基因组大片段进行无标记删除, 但是其两步筛选一般耗时较长, 有时还难以获得完全分离的突变株。已有研究报道,在集胞藻 (Synechocystissp.) PCC 6803、聚球藻(Synechococcussp.) UTEX 2973和鱼腥藻 (Anabaenasp.) PCC 7120等3种蓝藻中借助Cpf1可实现基因敲入、敲除和定点突变[25]。还有报道显示,在鱼腥藻PCC7120中利用CRISPR/Cpf1基因编辑系统可删除118 kb大片段[26]。因此, 进一步引入Cpf1等特异的DNA剪切系统或可显著加速无标记删除突变株的筛选过程。
