灵芝转录因子同源蛋白生物信息学分析及转录表达特性研究
2022-04-06刘笑天仇昊田莉师亮任昂赵明文谢荣
刘笑天,仇昊,田莉,师亮,任昂*,赵明文,谢荣
(1.南京农业大学生命科学学院,江苏 南京 210095;2.西藏自治区农牧科学院蔬菜研究所,西藏 拉萨 850032)
转录因子(transcription factor,TF)是一类能够调控基因在不同时间和空间表达的重要蛋白,在调节生物体各种生理活动的过程中发挥重要作用。随着不同物种的全基因组测序工作相继完成,对于深入理解转录因子调控基因表达的机制愈发重要。
在已有的研究中,灵芝(Ganodermalucidum)不同家族转录因子在其生长代谢过程中扮演着不同的角色。来自GATA家族的转录因子AreA在调节灵芝酸(ganoderic acid,GA)生物合成的过程中具有双重功能:一方面,AreA可以通过结合相关基因的启动子来增强GA生物合成;另一方面,AreA被激活以促进氮的吸收,但此期间NO含量的增加会抑制GA的生物合成[1]。来自APSES家族的转录因子GlSwi6在灵芝的生长发育及GA生物合成中起着重要作用,进一步的分析表明,胞内ROS水平也影响由GlSwi6调节的GA生物合成过程[2]。来自C2H2家族的转录因子GlPacC能够响应环境pH的变化,其在灵芝的菌丝体生长、子实体发育和GA的生物合成中具有重要作用[3]。然而,这些工作仅仅局限于少部分转录因子,尚有数量众多的转录因子未被发掘并验证其功能。对于灵芝而言,这一工作的缺失,显然限制了对其基因表达及次级代谢调控的进一步研究。
本研究通过检索真菌模式物种的转录因子公共数据库,结合灵芝蛋白库和全基因组数据,经过比对,将灵芝中可能存在的各转录因子家族及其成员系统地归纳筛选,并对这些转录因子同源蛋白在乙酸处理下的转录组进行表达分析,以期为后续新的转录因子的发掘研究奠定基础。
1 材料与方法
1.1 数据来源
从TRANSFAC®Public转录因子数据库(http://gene-regulation.com/pub/databases/transfac)和FTFD真菌转录因子数据库(http://ftfd.snu.ac.kr)中筛选出源自于酿酒酵母(Saccharomycescerevisiae)、粗糙脉孢菌(Neurosporacrassa)、裂殖酵母(Schizosaccharomycespombe)、双色蜡蘑(Laccariabicolor)等真菌中常见的若干转录因子家族,并以它们的蛋白序列作为参考序列,在本地建立BLASTp;此外,基于隐马尔可夫模型(hidden markov model,HMM),利用HMMER 3.0程序(E-value≤10-5),分别与Chen等[4]公布的灵芝蛋白数据库交叉比对,从中获得含有保守结构域的同源序列。
1.2 灵芝转录因子同源蛋白的生物信息学分析
使用ClustalW软件对所获得的序列进行多重比较分析,并利用MEGA 7.0软件中的邻接法(neighbor-joining method)绘制系统进化树,bootstrap值设置为1 000。将筛选到的灵芝转录因子同源蛋白按照家族进行分类,并与灵芝基因组数据库[4]进行BLASTn比对,确定各转录因子同源蛋白在灵芝染色体上的分布。通过ExPASy(https://web.expasy.org/compute_pi/)分析各转录因子同源蛋白的氨基酸残基数、相对分子质量及等电点。利用在线预测工具BUSCA(http://busca.biocomp.unibo.it/)进行亚细胞定位预测。
1.3 乙酸处理下灵芝转录因子同源蛋白的表达分析
参考Ren等[5]的方法使用乙酸对灵芝HG野生型菌株的菌丝进行处理。灵芝HG野生型菌株由上海市农业科学院提供。于PDB培养基中进行种子培养,28 ℃、150 r·min-1摇床振荡培养7 d。以5%(体积分数)的接种量进行发酵培养,28 ℃、150 r·min-1振荡培养5 d(CYM培养基:10 g·L-1麦芽糖、20 g·L-1葡萄糖、2 g·L-1酵母提取物、2 g·L-1蛋白胨、0.5 g·L-1MgSO4·7H2O、4.6 g·L-1KH2PO4)。培养6 d时,添加乙酸至终浓度为5 mmol·L-1。6 h后收集菌丝,提取RNA。未添加乙酸的对照组及乙酸处理组均设置3次重复。Illumina MiSeq Reagent Kit反转录后构建测序文库,借助Illumina MiSeq平台进行高通量测序。以 |log2(Fold Change)|>1 且-lg(P-value)>1.301表示显著性的表达差异。
2 结果与分析
2.1 灵芝转录因子家族同源蛋白的筛选
从TRANSFAC®和FTFD数据库中获取属于真菌的若干转录因子家族,建立本地BLASTp,并利用基于Pfam数据库的HMMER工具对灵芝的蛋白数据库进行比对。BLASTp工具筛选到分属于若干家族的217个转录因子同源蛋白,HMMER筛选出152个。通过交叉比对,去除重复值,合并比对结果,共筛选出来自19个家族的261个转录因子同源蛋白(表1)。其中,含有OB-fold、Zn2Cys6和C2H2的成员数量较多,分别为61、55和40个。而含有铜拳蛋白(copper fist proteins)、着丝粒蛋白B(centromere protein B)和TATA结合(TATA binding)结构域的转录因子同源蛋白数量较少,均只有1个。此外,有9个同源蛋白含有2种转录因子保守结构域:GL31391_R1、GL24980_R1、GL31253_R1和GL29539_R1都含有Jumonji与C2H2;GL26798_R1、GL18755_R1、GL27347_R1和GL26130_R1都含有APSES与bZIP;GL24061_R1含有bHLH与C2H2。
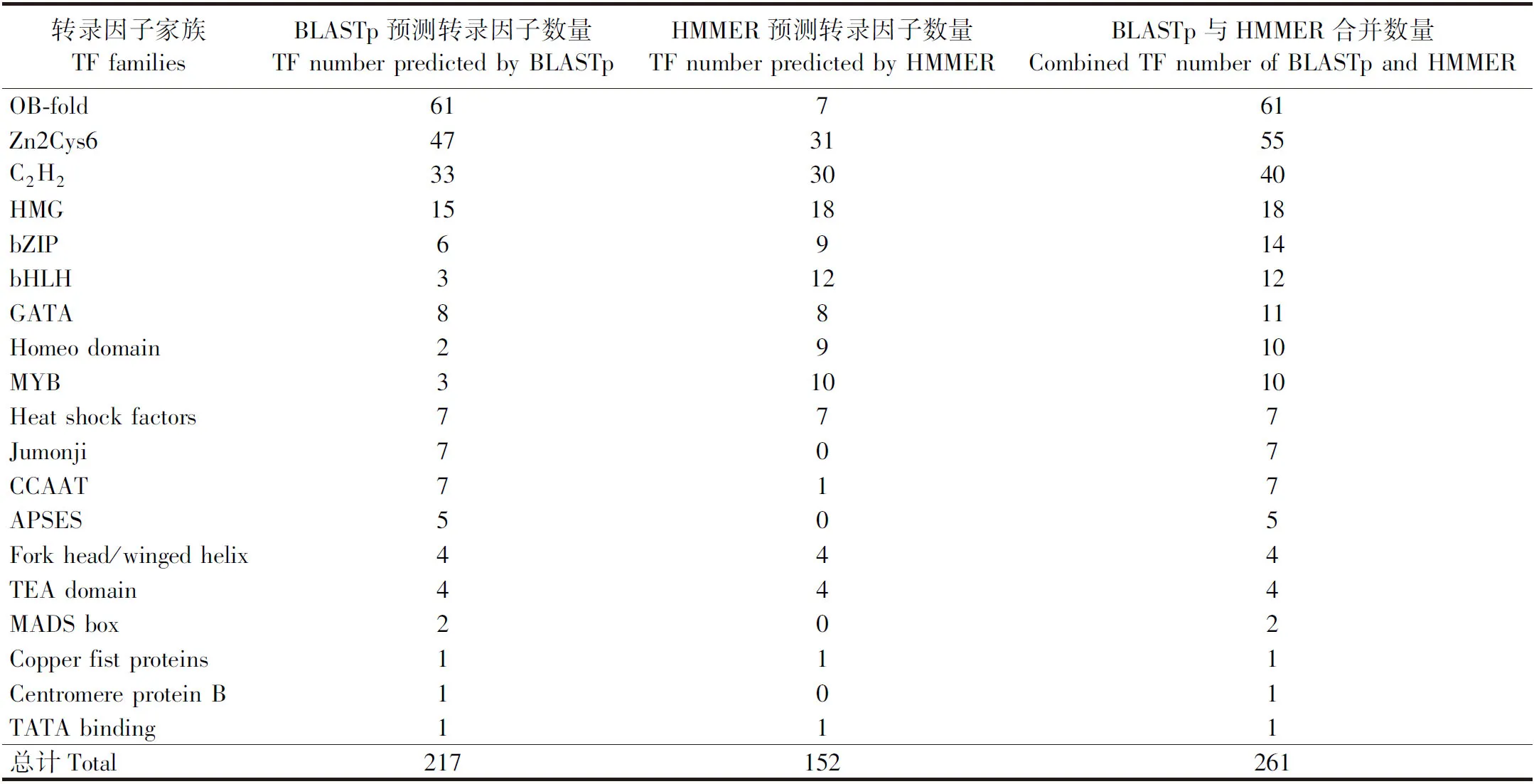
表1 灵芝中主要转录因子家族的同源蛋白筛选结果
2.2 灵芝转录因子家族同源蛋白的基本特性
将261个转录因子同源蛋白按照家族名加序号的命名方式进行编号,分析它们的序列特性。ExPASy 的预测结果表明,这些同源蛋白相对分子质量最小为8.65×103,最大为168.83×103,约94.2%的蛋白相对分子质量在120×103以下(图1-A)。它们的理论等电点为4.06~12.16,多数小于11.5(图1-B)。
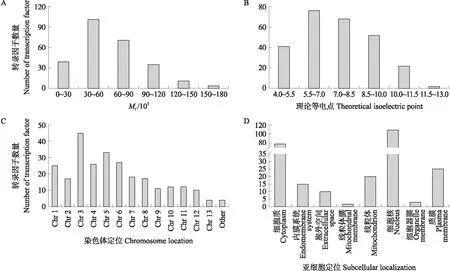
图1 灵芝转录因子家族同源蛋白的基本特性
根据灵芝染色体基因组信息,运行本地BLASTn程序,分析这些同源蛋白在染色体上的分布情况。结果表明,各个染色体上所包含的转录因子同源蛋白数量存在差异。染色体Chr 3上数量最多,共有45个,Chr 13则最少,仅有4个,其余染色体上的转录因子同源蛋白数量分布较为均匀(图1-C)。
使用BUSCA在线预测工具,基于GO注释对261个转录因子同源蛋白进行亚细胞定位预测(图1-D)。结果显示,有108个同源蛋白定位于核仁,78个定位于细胞质。约71.3%的同源蛋白定位于细胞核与细胞质,其他则均匀分布于质膜、线粒体和内膜系统等。
2.3 灵芝与真菌模式物种中主要转录因子家族的比较
以研究较为深入的3个转录因子家族,即C2H2、Zn2Cys6、HMG家族为例,将其与真菌模式物种酿酒酵母(S.cerevisiae),以及同为担子菌的双孢蘑菇(Agaricusbisporus)和双色蜡蘑(L.bicolor)进行对比,计算各家族转录因子数占总基因数的比例(表2)。在3个转录因子家族中,HMG家族在各自物种中所占总基因数的比例最小。根据先前的报道,HMG转录因子家族能够与线性DNA结合,在转录、复制等过程中起到关键作用[6]。而C2H2与Zn2Cys6转录因子则主要参与了生长发育、代谢调控和胁迫响应等过程[7-8]。此外,从灵芝中筛选到的这3种转录因子家族同源蛋白所占总基因数的比例较另外几个物种低。
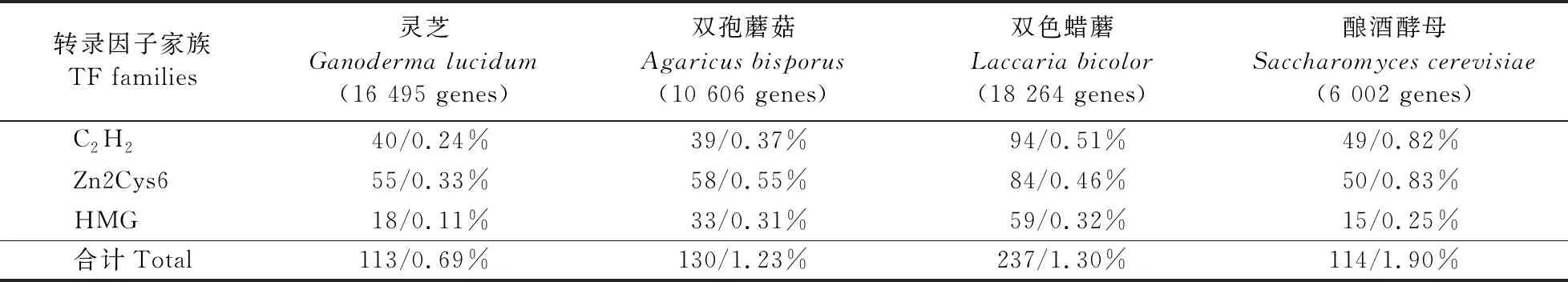
表2 灵芝与真菌模式物种中的3个转录因子家族对比
2.4 灵芝bZIP转录因子同源蛋白的进化分析
在真核生物中,碱性亮氨酸拉链(basic leucine zipper,bZIP)转录因子是较为庞大和多样的转录因子家族之一。真菌中的bZIP转录因子参与菌丝生长发育[9]、氨基酸生物合成[10-11]、次级代谢产物的产生[12]和胁迫反应[13-14]等生理过程。我们整理了11个灵芝bZIP转录因子同源蛋白和来自构巢曲霉(Aspergillusnidulans)、稻瘟病菌(Magnaportheoryzae)和粗糙脉孢菌(N.crassa)的bZIP转录因子,使用MEGA 7.0软件中的ClustalW对它们的氨基酸序列进行多重比较分析,采取邻接法绘制系统进化树。结果表明,灵芝中bZIP转录因子同源蛋白可以分为4个亚组:GL30843、GL24297、GL24299为第1亚组;GL21653、GL25297、GL25947、GL26688、GL29448为第2亚组;GL17608、GL28195为第3亚组;GL23461为第4亚组(图2)。第2亚组内共有5个bZIP转录因子同源蛋白,表明它们可能来源于同一个祖先,且在进化上呈现出更高的分化程度,分工更细致。除此之外,其他几个亚组均匀散布在不同的分支中。
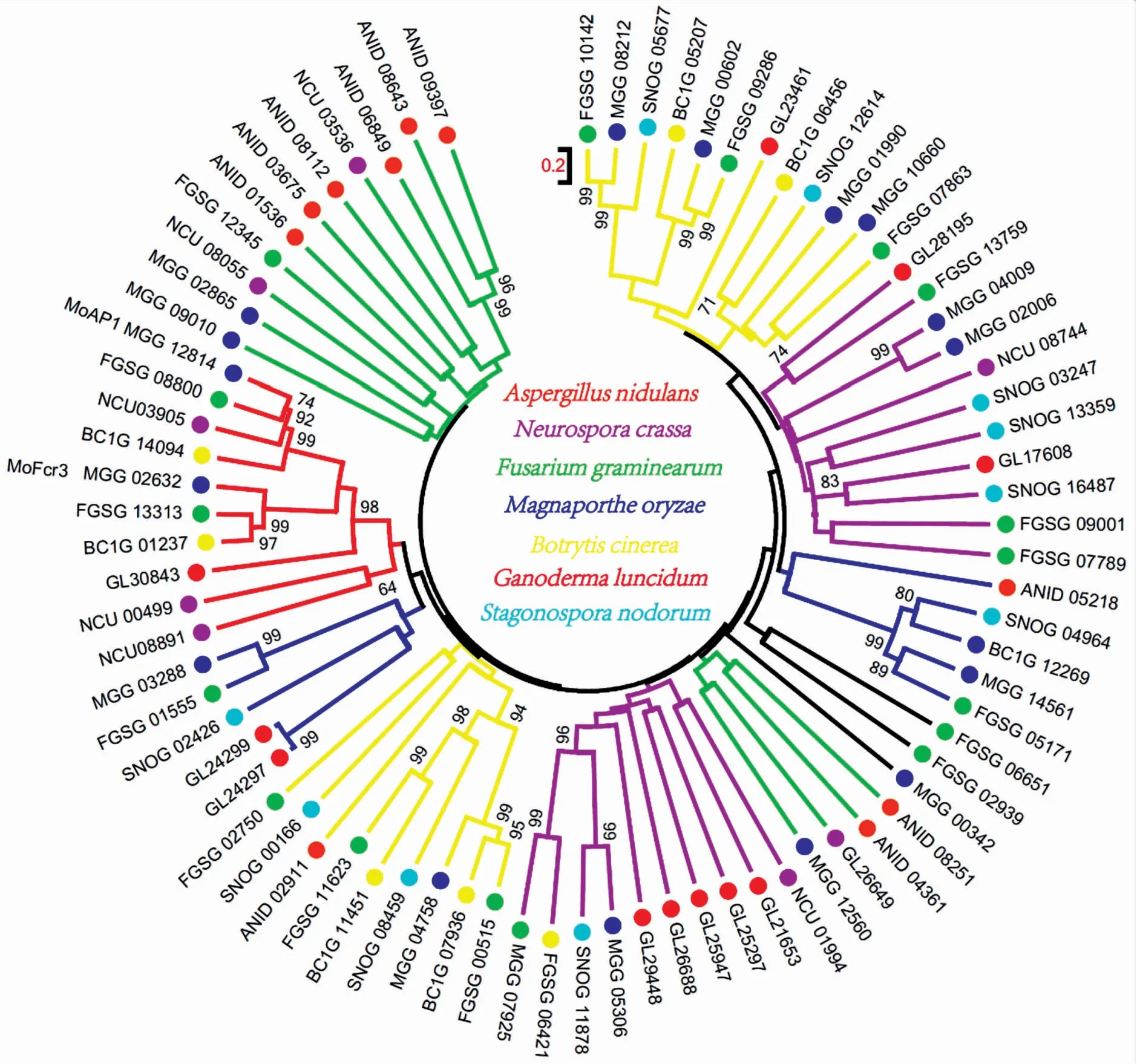
图2 灵芝bZIP转录因子同源蛋白进化树
2.5 乙酸处理的灵芝菌丝体转录因子同源蛋白表达分析
乙酸处理能够诱导灵芝酸合成[5],该过程与乙酸激活灵芝胞内活性氧(reactive oxygen species,ROS)信号相关[15]。乙酸处理的灵芝菌丝体转录组数据分析表明,在我们比对到的这些假定的转录因子中,有5个转录水平发生显著变化。其中,含有TEA结构域的GlTEA-4(GL22568_R1)在乙酸处理后转录水平显著下调了52%。而含有C2H2结构域的GlC2H2-4(GL16029_R1),含有Zn2Cys6结构域的GlZn2Cys6-5(GL16724_R1)、GlZn2Cys6-7(GL17627_R1)和GlZn2Cys6-15(GL21326_R1)在乙酸处理后转录水平显著上调,分别增加了119%、312%、129%和205%,但它们的log2(CPM)并不高,表明它们在灵芝中的分布丰度相对较低(图3)。
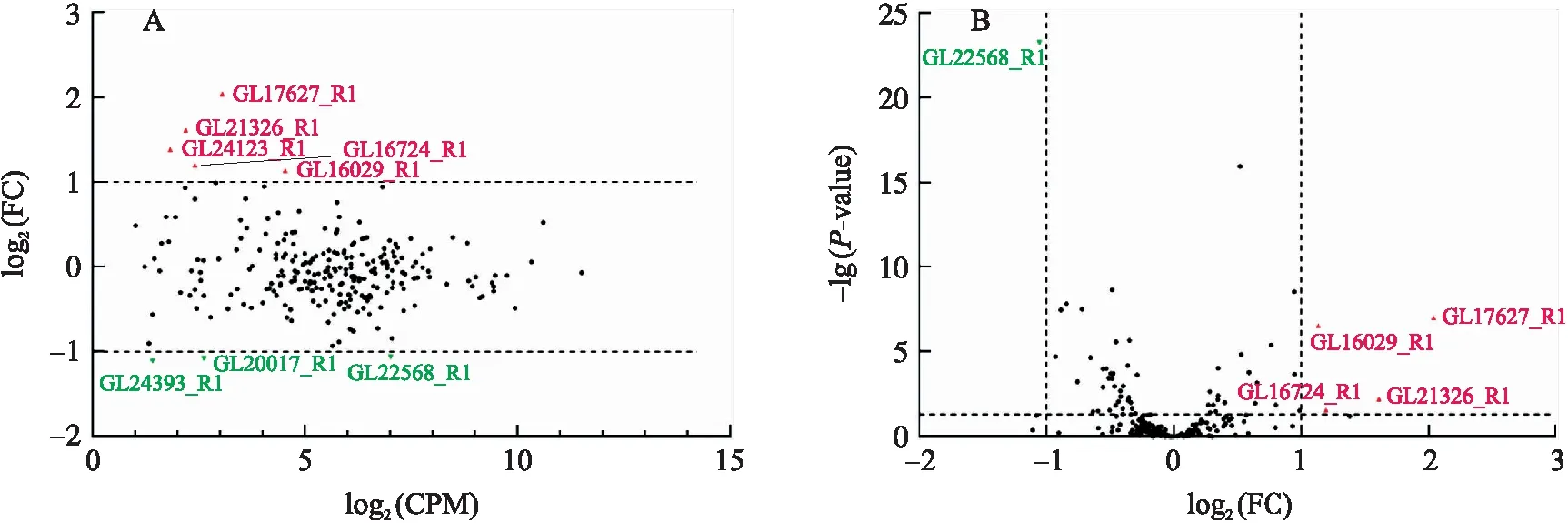
图3 乙酸处理下灵芝转录因子同源蛋白表达分析
3 讨论
转录因子作为一类能够调控基因在不同时间和空间表达的蛋白,在生物的生长发育与代谢调控中具有重要作用。近年来随着基因组学(genomics)、蛋白组学(proteinomics)、代谢组学(metabolomics)和转录组学(transcriptomics)等学科快速发展,我们可以从整体角度去认识基因、蛋白及其分子间相互作用的机制。然而,这些研究大多集中在少数模式物种中,对于非模式物种,例如灵芝,仍然缺少对其转录因子家族系统而全面的分析。
本研究通过检索公共转录因子数据库,将真菌模式物种的转录因子与Chen等[4]公布的灵芝蛋白数据库进行交叉比对,筛选到来自19个家族的261个转录因子同源蛋白。目前,已经公开的数据库中对于灵芝转录因子的收录并不多。在我们前期的尝试性工作中,利用在线BLASTp对NCBI中的灵芝条目进行对比,仅检索到23个,利用在线HMMER筛选出的灵芝转录因子同源蛋白也只有20个,这也说明对于灵芝中转录因子系统而全面的筛选工作较少,大量预测的蛋白功能需要进一步试验的验证。
在261个假定的转录因子中,含有OB-fold、C2H2和Zn2Cys6的家族成员数目最多。现有的研究表明,OB-fold参与维持基因组的稳定性[16],包括DNA复制[17]、DNA修复[18]、细胞周期调节[19]和端粒的维持[20]等;C2H2在真菌的生长发育[7,21-22]、孢子产生[7]、致病力[21-23]以及胁迫响应[24]等方面起到突出的作用;Zn2Cys6转录因子在代谢调控[25]、生长发育[8,26-27]、孢子萌发[8,26]、附着胞形成[8]、致病力[8,26,28]与胁迫应激[8,29]等过程中发挥关键作用。
乙酸处理条件下,灵芝菌丝体有5个转录因子同源蛋白的转录水平发生显著变化,分属于TEA、C2H2和Zn2Cys6三个转录因子家族。据报道,含TEA结构域的转录因子在控制真菌细胞分裂周期、发育[30-32]等方面具有重要作用。酿酒酵母中,含TEA的转录因子Tec1能够激活G1细胞周期蛋白,在酵母交配期间则通过降解而被下调,从而实现酵母不同发育阶段的动态平衡。乙酸处理下,灵芝菌丝体中由于ROS的积累从而导致灵芝酸含量增加[15],在此过程中可能响应胁迫的C2H2和Zn2Cys6转录因子同源蛋白的转录水平增加,TEA同源蛋白转录水平降低。这5个能够显著响应乙酸处理的转录因子同源蛋白,它们可能在乙酸诱导灵芝酸合成,激活灵芝胞内ROS信号等过程中具有重要作用,值得进一步开展研究。
