利用RNAi技术研究基因PDIDSM_85260和PDIDSM_08010表达对柠檬烯转化生成α-松油醇的影响
2022-03-30张璐璐任婧楠潘思轶
张璐璐,范 刚,李 晓,任婧楠,潘思轶,黄 文,何 进
(1.河南工业大学粮油食品学院,河南 郑州 450001;2.华中农业大学食品科学技术学院,环境食品学教育部重点实验室,湖北 武汉 430070;3.华中农业大学生命科学技术学院,湖北 武汉 430070)
随着人们健康意识的增强,天然香料的应用日益广泛,而通过微生物生物转化作用生产的香料已经被认为是天然的[1]。微生物转化能够将品质不高的柠檬烯转化生成α-松油醇等香气更浓、应用价值更高的香料[2-5],然而目前微生物转化柠檬烯生产α-松油醇过程中的关键酶或基因还不清楚,转化途径和转化机制仍不明确,生成的产物成分复杂,所得到的转化产物产率仍然很低。因此,柠檬烯微生物转化的分子机制研究对香精香料的发展以及工业化生产有重要的推动作用。
在本实验室前期研究中,以指状青霉(Penicillium digitatum)DSM62840为研究对象,通过基因组测序,转录组分析以及蛋白分离纯化和生物信息学分析等手段挖掘柠檬烯微生物转化生成α-松油醇过程中的关键基因。其中通过基因组和转录组分析,筛选得到一个候选基因PDIDSM_85260,该基因在柠檬烯转化的过程中上调了3 000多倍[6]。对该基因进行BLAST分析,发现该基因被注释为假定蛋白,含有一个GAL4-like Zn2Cys6 binuclear cluster DNA-binding domain(GAL4)和一个fungal transcription factor regulatory middle homology region(fungal_TF_MHR)。在蛋白水平上,PDIDSM_85260与卡门培尔青霉(Penicillium camemberti)FM013中的真菌转录调控蛋白具有高度相似性(相似度95%)。以上结果说明基因PDIDSM_85260可能是一个转录因子。研究表明大多数锌指蛋白是参与碳/氮源的利用或氨基酸/次级代谢物的生物合成,比如橘青霉菌(P. citrinum)中参与美伐他汀生物合成的MlcR[7]、黄曲霉菌(Aspergillus flavus)中参与黄曲霉毒素生物合成的AflR[8]、酿酒酵母(Saccharomyces cerevisiae)中参与半乳糖代谢的Gal4[9]以及构巢曲霉菌(A. nidulans)中与硝酸盐利用有关的NirA[10]。基因PDIDSM_85260的高表达说明它在柠檬烯生物转化过程中可能起重要作用。此外,通过超速离心、30%~60%硫酸铵沉淀、羟基磷灰石层析和凝胶过滤层析对指状青霉DSM62840转化柠檬烯生成α-松油醇过程中的关键酶进行分离纯化,电泳结果显示在分子质量约为50 kDa处有一条明显的蛋白条带。经过质谱鉴定,该条带中的蛋白可能是二氢硫辛酰胺脱氢酶(dihydrolipoyl dehydrogenase,DLD,PDIDSM_08010),推测其可能参与调控柠檬烯的微生物转化过程(文章未发表)。
为了进一步验证基因PDIDSM_85260和PDIDSM_08010在柠檬烯微生物转化过程中的功能,本研究利用RNAi技术构建PDIDSM_85260和PDIDSM_08010的干涉载体pCAMBIA1303-TrpC-HygrogpdA-85260-TEF1和pCAMBIA1303-TrpC-Hygro-gpdA-08010-TEF1,经农杆菌介导转化到指状青霉DSM62840中。筛选得到阳性转化子之后,通过实时聚合酶链式反应(real-time polymerase chain reaction,real-time PCR)测定基因的表达量以获得干涉菌株,比较干涉菌株与野生型菌株转化柠檬烯的能力,以研究基因PDIDSM_85260和PDIDSM_08010在柠檬烯微生物转化过程中的作用,这不仅将为指状青霉DSM62840生物转化柠檬烯生产α-松油醇的研究提供一定的参考,还为后续其他基因的研究提供思路。
1 材料与方法
1.1 材料与试剂
1.1.1 菌种、质粒与试剂
指状青霉DSM62840购自德国微生物菌种保藏中心;质粒pCAMBIA1303-TrpC-Hygro-gpdA-GFP和pFC332淼灵质粒平台;Trans1-T1 phage resistant感受态细胞北京全式金生物技术有限公司;农杆菌AGL-1 上海唯地生物技术公司。
R-(+)-柠檬烯 东京化工有限公司;R-(+)-α-松油醇美国Sigma公司;卡那霉素(kanamycin,Kan)、潮霉素(hygromycin,Hyg)、利福平(rifampicin,Rif)、头孢噻肟霉素(cefotaxime,Cefo)、2-(N-吗啡啉)乙磺酸(2-(N-morpholino)ethanesulfonic acid,MES)、乙酰丁香酮(acetosyringone,AS) 美国Sigma-Aldrich公司;普通琼脂糖凝胶DNA回收试剂盒(离心柱型)、质粒小抽提试剂盒(离心柱型) 天根生化科技有限公司;SYBR Green Premix ProTaqHS qPCR Kit、Evo M-MLV RT Kit艾科瑞生物工程有限公司。
1.1.2 培养基
麦芽酵母肉汤(malt yeast brot,MYB)培养基:酵母提取物3 g/L,麦芽提取物20 g/L,蛋白胨10 g/L,葡萄糖10 g/L。
Luria-Bertani(LB)培养基:蛋白胨10 g/L,酵母5 g/L,NaCl 10 g/L。
强化分解代谢阻抑肉汤(super optimal broth with catabolite repression,SOC)培养基:在950 mL水中添加蛋白胨20 g、酵母提取物5 g和NaCl 0.5 g,使其完全溶解之后添加10 mL 250 mmol/L KCl溶液,调节pH 7.0,然后定容至1 L,高压灭菌。在使用之前在添加20 mL 1 mol/L葡萄糖溶液(过滤除菌)。
基本培养基(minimal medium,MM):K-buffer 10 mL,M-N buffer 20 mL,20%葡萄糖10 mL,0.01% FeSO410 mL,20% (NH4)2SO42.5 mL,1%CaCl2·2H2O 1 mL,补H2O至1 L,调节pH 6.7~7.0。
诱导培养基(induction medium,IM):K-buffer 10 mL,M-N buffer 20 mL,20%葡萄糖5 mL,0.01% FeSO410 mL,20% (NH4)2SO42.5 mL,1%CaCl2·2H2O 1 mL,50%甘油10 mL,MES 7.808 g,补H2O至1 L,调节pH 5.6。
共培养诱导培养基(cocultivation induction medium,Co-IM):K-buffer 10 mL,M-N buffer 20 mL,20%葡萄糖2.5 mL,0.01% FeSO410 mL,20% (NH4)2SO42.5 mL,l% CaCl2·2H2O 1 mL,50%甘油10 mL,MES 7.808 g,琼脂20 g,补H2O至1 L,调节pH 5.6。
K-buffer:109 g K2HPO4溶于500 mL水中,用磷酸调节pH 4.9,灭菌后室温保存;M-N buffer:15 g MgSO4·7H2O和7.5 g NaCl溶于500 mL水中,灭菌后室温保存。
1.2 仪器与设备
SW-CJ-1FD单人单面净化工作台 苏州净化设备有限公司;LDZX-50KBS立式高压蒸汽灭菌锅 上海申安医疗器械厂;6890N-5975B气相色谱-质谱联用仪美国Agilent公司;THZ-C-1全温振荡器 太仓市实验设备厂;HPX-9082MBE电热恒温培养箱 上海博讯实业有限公司医疗设备厂;ViiA7 System荧光定量PCR仪 美国ABI公司;凝胶成像系统、PCR仪 美国Bio-Rad公司。
1.3 方法
1.3.1 基因PDIDSM_85260和PDIDSM_08010的生物信息学分析
基因PDIDSM_85260和PDIDSM_08010的氨基酸序列来源于指状青霉DSM62840基因组数据(指状青霉DSM62840基因组数据已经被上传至DDBJ/ENA/GenBank,收录号为JAAQRF000000000)。蛋白质的理化性质分析通过在线程序Pro Param(http://web.expasy.org/protparam/)预测分析;蛋白质的二级结构利用在线程序SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)进行预测分析;蛋白质三级结构利用在线程序SWISS-MODEL(https://swissmodel.expasy.org/)进行预测分析[11]。
1.3.2 沉默载体的构建
1.3.2.1 目的基因RNAi序列和TEF1启动子的扩增
以TEF1F/R为引物、pFC332为模版,进行PCR扩增获得pFC332上的TEF1启动子序列。以85260 F/R和08010 F/R为引物,指状青霉DSM62840基因组DNA为模版[12],进行PCR扩增获得基因PDIDSM_85260和PDIDSM_08010的RNAi序列。引物序列见表1。PCR扩增体系:Q5 0.5 µL,Q5 Buffer 10 µL,dNTP(10 mmol/L)2 µL,引物各2 µL,H2O 33 µL,模板(1 ng/µL 质粒,基因组为50 ng/µL)0.5 µL。PCR扩增程序:98 ℃预变性30 s;98 ℃变性10 s,50 ℃退火20 s,72 ℃延伸30 s(循环5 次);98 ℃变性10 s,60 ℃退火20 s,72 ℃延伸30 s(循环30 次);72 ℃延伸5 min。反应结束后进行琼脂糖凝胶电泳检测PCR产物。

表1 载体构建及real-time PCR验证所用引物序列Table 1 Primer sequences used for vector construction and real-time PCR verification
1.3.2.2 质粒双酶切处理
pCAMBIA1303-TrpC-Hygro-GPD使用50 μg/mL Kan抗性的LB培养基进行培养,然后使用质粒小抽提试剂盒进行质粒提取。使用EcoRI和BstEII对质粒pCAMBIA1303-TrpC-Hygro-GPD进行双酶切处理,37 ℃完全酶切3 h后,用1%琼脂糖凝胶电泳检测酶切结果,并对酶切产物进行切胶回收。酶切体系:10×Buffer 5 μL,EcoRI 1.5 μL,BstEII 1.5 μL,载体37 μL,H2O 5 μL。
1.3.2.3 重组反应
将目的基因RNAi序列、TEF1启动子的PCR回收产物与质粒pCAMBIA1303-TrpC-Hygro-GPD的酶切片段进行重组反应,重组体系:pCAMBIA1303-TrpC-Hygro-GPD酶切产物0.5 μL、TEF1启动子1 μL、85620/08010 PCR产物片段1 μL、Clonexpress II 1 μL、Buffer 2 μL、H2O 4.5 μL。将连接体系于37 ℃反应30 min后立刻放置在冰上,随后进行转化反应。将连接产物热激转入Trans1-T1 phage resistant感受态细胞,挑选阳性转化子进行菌落PCR验证及测序,挑选正确的阳性转化子接种于LB培养基中进行扩大培养,提取质粒pCAMBIA1303-TrpCHygro-gpdA-85260-TEF1和pCAMBIA1303-TrpC-HygrogpdA-08010-TEF1。
1.3.3 农杆菌介导的遗传转化
将质粒pCAMBIA1303-TrpC-Hygro-gpdA-85260-TEF1和pCAMBIA1303-TrpC-Hygro-gpdA-08010-TEF1热激转入农杆菌AGL-1感受态细胞[13-14],挑选阳性转化子进行菌落PCR验证及测序,筛选阳性克隆。
挑取验证成功的农杆菌单菌落于1 mL LB(含有50 μg/mL Kan和20 μg/mL Rif)培养液中,200 r/min、28 ℃培养24 h。然后将农杆菌菌液加入到100 mL MM(含有50 μg/mL Kan)液体培养基中,200 r/min、28 ℃培养48 h。收集菌液,5 000×g、4 ℃离心10 min,去上清液后,用等体积IM培养液重悬,并用IM稀释至OD600nm为0.4,添加AS至终浓度为200 μmol/L,于200 r/min、28 ℃培养至OD600nm为0.8。
指状青霉菌丝在PDA固体培养基上活化后,用打孔器打取直径相同的菌块,浸入上述IM农杆菌菌液孵育20 min,每隔10 min轻轻摇匀一次。之后,将菌块用灭菌滤纸稍微吸干菌液后,转入含有200 μmol/L AS的Co-IM固体培养基中,28 ℃正置培养。2 d后,将菌丝块从共培养基上挑取下来,浸入含有400 μg/mL Cefo无菌水中孵育20 min,每隔10 min轻轻摇匀一次。菌块用灭菌滤纸稍微吸干水分后,转移至含有100 μg/mL Hyg和400 μg/mL Cefo的PDA固体培养基上,28 ℃培养7 d,观察菌丝生长情况。
将上步获得的新生菌丝用2 mg/mL纤维素酶消化2 h,然后再接种于新的含有100 μg/mL Hyg和400 μg/mL Cefo的PDA固体培养基上,进行第2次筛选。5~7 d后,得到的再生菌丝即为转化子。
1.3.4 干涉转化子的验证
提取转化子的RNA,以野生型菌株为对照,进行real-time PCR验证实验。使用Trizol法提取RNA[15],cDNA的合成以及real-time PCR实验参照Zhang Lulu等[6]的方法。每个基因均设置3 个技术重复和3 个生物学重复。β-actin作为内参基因。real-time PCR引物见表1。
1.3.5 干涉菌株的微生物转化实验
用1~2 周的野生型菌株指状青霉DSM62840和基因PDIDSM_85260和PDIDSM_08010干涉菌株的斜面培养物制取孢子悬浮液,调整孢子悬浮液的浓度为1.5×107spores/mL。取1 mL孢子悬浮液接种于100 mL MYB培养基中,24 ℃、150 r/min振荡培养48 h。之后加入840 mg/L柠檬烯(0.22 µm过滤除菌)进行微生物转化实验[16]。转化12 h之后,采用固相微萃取-气相色谱-质谱(solid phase microextraction-gas chromatographymass spectrometry,SPME-GC-MS)联用测定产物α-松油醇的含量[17]。实验重复3 次。以野生型菌株的转化产物α-松油醇含量为100%计。
1.4 统计学分析
2 结果与分析
2.1 基因PDIDSM_85260和PDIDSM_08010的生物信息学分析
根据指状青霉DSM62840基因组测序所得到的基因PDIDSM_85260和PDIDSM_08010氨基酸序列,对其理化性质及蛋白结构进行分析。结果显示,PDIDSM_85260编码732 个氨基酸,分子质量约为80 kDa,pI约为6.56。对其二级结构进行预测,结果显示含有40.44%的α-螺旋、4.92%的延伸链、1.91%的β-转角、52.73%的无规卷曲。图1A是PDIDSM_85260编码氨基酸的三维结构预测图,可以看出该蛋白含有大量的α-螺旋和无规卷曲。
PDIDSM_08010编码512 个氨基酸,分子质量约为54 kDa,pI约为8.10。其二级结构预测结果显示含有33.98%的α-螺旋、21.68%的延伸链、9.77%的β-转角、34.57%的无规卷曲。PDIDSM_08010编码氨基酸的三维结构预测图显示该蛋白含有大量的α-螺旋、无规卷曲和延伸链结构,含有较少的β-转角(图1B)。

图1 PDIDSM_85260(A)和PDIDSM_08010(B)编码蛋白的三维结构预测Fig. 1 Three-dimensional structure prediction of PDIDSM_85260 (A) and PDIDSM_08010 (B)
2.2 沉默载体的构建
2.2.1 目的基因RNAi序列和TEF1启动子的扩增
使用TEF1F/R为引物,以模板pFC332为模版,进行PCR扩增获得pFC332上的TEF1启动子。使用85260F/R和08010F/R为引物,以指状青霉DSM62840基因组DNA为模版,进行PCR扩增获得基因PDIDSM_85260和PDIDSM_08010的RNAi序列。其中TEF1启动子大小为890 bp,PDIDSM_85260和PDIDSM_08010的RNAi序列的大小分别为283 bp和306 bp。PCR扩增结果见图2。扩增得到的条带均为单一条带,TEF1启动子的条带在750~1 000 bp之间,PDIDSM_85260和PDIDSM_08010的RNAi序列的条带在250~500 bp之间,靠近250 bp,与预期结果符合。
2.2.2 pCAMBIA1303-TrpC-Hygro-GPD质粒双酶切
以EcoRI和BstEII对质粒pCAMBIA1303-TrpC-Hygro-GPD进行双酶切处理,37 ℃完全酶切3 h之后,用1%琼脂糖凝胶电泳检测酶切结果,结果如图3所示。质粒酶切之后条带清晰,说明酶切彻底,并且大小符合预期结果。
图3 pCAMBIA1303-TrpC-Hygro-GPD酶切产物琼脂糖凝胶电泳图Fig. 3 Agarose gel electrophoresis of enzyme-digested products of pCAMBIA1303-TrpC-Hygro-GPD
2.2.3 沉默载体的构建
将pCAMBIA1303-TrpC-Hygro-GPD酶切骨架、TEF1启动子以及PDIDSM_85620/08010的RNAi片段进行重组反应,并转化至Trans1-T1 Phage Resistant感受态细胞中构建沉默载体pCAMBIA1303-TrpC-Hygro-gpdA-85260-TEF1和pCAMBIA1303-TrpC-Hygro-gpdA-08010-TEF1(图4)。然后挑选阳性转化子进行菌落PCR验证。
以85260 F/08010 F、TEF1F为引物进行菌落PCR验证,实验结果如图5所示。PCR扩增所得到的条带均为单一条带,大小约为1 200 bp,与预期符合。说明双启动子沉默载体pCAMBIA1303-TrpC-Hygro-gpdA-85260-TEF1和pCAMBIA1303-TrpC-Hygro-gpdA-08010-TEF1构建成功。挑选多个阳性克隆测序,对比测序结果,挑选不存在碱基突变和缺失的阳性克隆用于后续研究。

图4 pCAMBIA1303-TrpC-Hygro-gpdA-85260-TEF1(A)和pCAMBIA1303-TrpC-Hygro-gpdA-08010-TEF1(B)载体构建图Fig. 4 Construction of vectors pCAMBIA1303-TrpC-Hygro-gpdA-85260-TEF1 (A) and pCAMBIA1303-TrpC-Hygro-gpdA-08010-TEF1 (B)

图5 菌落PCR琼脂糖凝胶电泳图Fig. 5 Agarose gel electrophoresis of colony PCR products
2.3 沉默菌株的筛选与鉴定
选取在含Hyg平板上可以稳定生长的转化子,并以野生型菌株指状青霉DSM62840作为对照,进行基因PDIDSM_85260和PDIDSM_08010的real-time PCR验证。如图6所示,对于基因PDIDSM_85260,与野生型菌株相比,筛选得到的转化子的表达量均有一定程度的下调,其中转化子85260i-4、85260i-8、85260i-10的表达量分别下调了42.2%、42.1%、48%,因此,选择这3 个转化子进行进一步研究。
对于基因PDIDSM_08010,与野生型菌株相比,筛选得到的转化子的表达量均有一定程度的下调,其中转化子08010i-1、08010i-9、08010i-10、08010i-14的表达量分别下调了41.4%、58.3%、51%、49.7%,因此,选择这4 个转化子进行进一步研究。
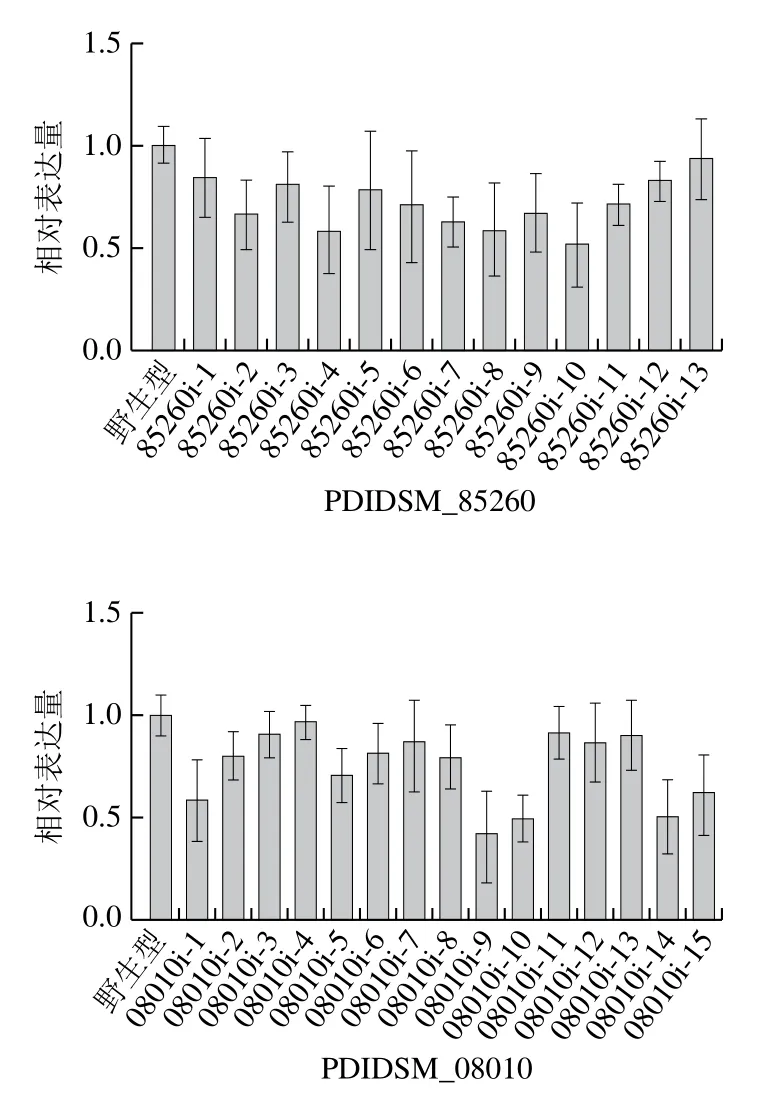
图6 干涉转化子中基因相对表达量的分析Fig. 6 Relative expression levels of genes in RNAi transformants
2.4 干涉菌株的微生物转化实验
为进一步研究基因PDIDSM_85260和PDIDSM_08010的功能,以野生型菌株为对照,进行干涉菌株的微生物转化实验,结果如图7所示。对于基因PDIDSM_85260,实验结果表明,与野生型菌株相比,转化子85260i-4、85260i-8、85260i-10经过微生物转化得到的α-松油醇含量均有不同程度的下降,分别下降了17.44%、16.35%、47.27%,说明基因PDIDSM_85260可能参与调控柠檬烯的微生物转化过程。
对于基因PDIDSM_08010,与野生型菌株相比,转化子08010i-1、08010i-9、08010i-10、08010i-14通过微生物转化产生的α-松油醇的含量减少,尤其是08010i-9,含量下降了22.52%,说明基因PDIDSM_08010也可能参与调控柠檬烯的生物转化过程。
3 讨 论
目前,对于基因功能的研究主要有两种方法,即增强基因的表达量和减弱或终止基因的表达。其中RNAi是最常用的降低基因表达量的技术。RNAi技术可以作为组学测序等技术筛选功能基因的工具,为真菌基因功能的研究提供新的见解[18-19]。对于丝状真菌,使其自身产生dsRNA从而导致基因沉默的策略主要有两种:发夹结构RNA(hairpin RNA,hpRNA)表达系统和双向启动子系统[20]。目前,已经有大量的研究使用RNAi技术诱导基因沉默,从而研究基因的功能[21-22]。Isabelle等[23]利用hpRNA表达系统对烟曲霉(Aspergillus fumigatus)中的ALB1和FKS1基因进行了沉默,与野生型菌株相比,ALB1和FKS1基因的表达量下降了4倍。Wang Lei等[24]也利用hpRNA表达系统对里氏木霉(Trichoderma reesei)中的pyr4基因进行了沉默。Nguyen等[25]利用gpd和TrpC启动子在稻瘟病菌(Magnaporthe oryzae)中构建双启动子系统,在这个系统中有37 个基因是被靶向,从而引起不同程度的沉默。并且在该系统中引入了绿色荧光蛋白eGFP,可以通过荧光信号检测菌株的沉默效率。本研究使用双启动子系统构建沉默载体,以指状青霉DSM62840的基因组为模板扩增目的基因的RNAi序列,以质粒pFC332为模板扩增TEF1启动子,然后对载体pCAMBIA1303-TrpC-Hygro-gpdA-GFP进行酶切处理,在gpdA启动子后连接RNAi序列片段和TEF1启动子片段,构建双启动子沉默载体pCAMBIA1303-TrpC-Hygro-gpdA-85260-TEF1和pCAMBIA1303-TrpC-Hygro-gpdA-08010-TEF1,用以研究基因PDIDSM_85260和PDIDSM_08010在柠檬烯转化过程中的作用。
微生物细胞中的物质转化过程错综复杂,在转化过程中可能会受各种外界因素的影响。当微生物受到外界环境的刺激时,可以通过基因调控表达相关基因改变自身的生长和代谢方式,从而减少外界因素对自身的不利影响[26]。在这个基因调控的过程中,转录因子发挥着不可或缺的作用。在真菌中最常见的转录因子是Zn2Cys6类锌指蛋白,锌指蛋白是一个庞大的转录因子家族,该家族含有手指状结构域,主要功能是调控基因的表达。研究表明Zn2Cys6类锌指蛋白能够参与调控真菌的基础代谢、次级代谢以及药物抗性等过程,在真菌的生长和代谢过程中发挥着重要的调控作用[27-28]。在本研究中,PDIDSM_85260作为一个转录因子,其干涉菌株转化柠檬烯得到α-松油醇的含量,与野生型菌株相比有明显下降,说明该基因可能参与调控柠檬烯的生物转化过程。转录因子PDIDSM_85260可能通过结合柠檬烯转化生成α-松油醇过程中的重要基因的启动子区域调控这些基因的表达,从而直接或间接影响柠檬烯的微生物转化过程。未来需要对PDIDSM_85260的靶标基因、其他潜在的基因功能以及可能存在的调控通路进行进一步的探索。
PDIDSM_08010,即DLD是一种NAD+依赖的氧化还原酶,参与三羧酸循环。在受到外界因素的刺激下,DLD能够影响活性氧自由基(reactive oxygen species,ROS)的生成和清除以及三羧酸循环[29]。Jaewang等[30]研究发现DLD能够诱导ROS、脂质过氧化和亚铁的积累。研究指出DLD能够通过调节细胞内ATP的生成、第二信使分子cAMP的生成以及ROS的生成和清除调控细胞内类固醇激素的合成[31]。研究发现与野生型菌株相比,PDIDSM_08010的干涉菌株转化柠檬烯得到α-松油醇的含量显著下降,这说明该基因可能参与调控柠檬烯的生物转化过程。推测DLD可能通过调控细胞内的ATP含量、ROS等调节柠檬烯的微生物转化过程。
4 结 论
利用RNAi技术构建了PDIDSM_85260和PDIDSM_08010的干涉载体pCAMBIA1303-TrpC-HygrogpdA-85260-TEF1和pCAMBIA1303-TrpC-Hygro-gpdA-08010-TEF1,经农杆菌介导转化到指状青霉DSM62840中。通过Hyg抗性筛选得到阳性转化子,real-time PCR结果显示与野生型菌株相比,基因PDIDSM_85260和PDIDSM_08010干涉转化子的表达量均有所下降,说明干涉菌株构建成功。此外,干涉菌株的微生物转化实验结果表明,与野生型菌株相比,干涉菌株转化柠檬烯得到α-松油醇含量均有不同程度的下降,说明基因PDIDSM_85260和PDIDSM_08010可能参与调控柠檬烯生物转化生成α-松油醇的过程。
