3D U-Net深度学习模型基于盆腔T2WI自动分割盆腔软组织结构
2022-03-01朱丽娜陈卫东黄嘉豪王祥鹏张晓东王霄英
刘 想,韩 超,高 歌,朱丽娜,陈卫东,黄嘉豪,王祥鹏,张晓东,王霄英*
(1.北京大学第一医院医学影像科,北京 100034;2.北京赛迈特锐医学科技有限公司,北京 100011)
多参数MRI(multiparametric MRI, mpMRI)是影像学检查前列腺癌的重要手段[1],不仅可用于临床诊断,且有助于指导放射治疗(简称放疗)前列腺癌[2]。精确勾画靶区是保证放疗疗效的关键因素之一。前列腺癌临床靶区包括整个前列腺和精囊腺,可能受到照射损伤的邻近器官包括直肠、膀胱、尿道及股骨等[3],而勾画上述结构较耗时、费力,且易受主观因素影响。3D U-Net是一种卷积神经网络结构,可用于分割3D图像。本研究评估采用3D U-Net深度学习(deep learning, DL)模型基于盆腔T2WI自动分割盆腔组织结构的可行性。
1 资料与方法
1.1 一般资料 回顾性分析2020年6月—12月147例于北京大学第一医院就诊的前列腺疾病患者,年龄28~86岁,平均(66.3±11.9)岁。纳入标准:①病理证实前列腺癌,或经病理/盆腔MRI随访确诊良性前列腺增生;②资料完整且图像清晰。排除标准:①前列腺癌根治术后;②前列腺周围组织存在原发性或继发性病变;③MR扫描范围不全。147例中,108例良性前列腺增生,39例前列腺癌,经病理证实肿瘤分期≤T2期;其中28例因随访需要接受2次盆腔MR扫描,121例接受单次盆腔MR扫描;共获得175组盆腔T2WI。本研究方案符合院人工智能(artificial intelligence, AI)模型训练规范要求。
1.2 仪器与方法 采用GE Discovery HD 750 3.0 T MR仪或Siemens Avanto 1.5 T MR仪,按照统一盆腔mpMR扫描方案[4]采集轴位T2WI,TR 4 000 ms/7 950 ms,TE 100 ms/112 ms,层厚3 mm/3.5 mm,层间距0,FOV 200 mm×200 mm,矩阵224×224。
1.3 手动标注盆腔组织结构 将DICOM格式T2WI转换为Nifty格式。由1名具有3年影像学诊断经验的住院医师采用ITK-SNAP软件(Version 3.6.0,http://www.itksnap.org/)手动调整窗宽及窗位,逐层标注前列腺、双侧精囊腺、膀胱、直肠、尿道、双侧闭孔内肌和双侧耻骨直肠肌;再由1名具有15年以上影像学诊断经验的主任医师对标注进行修改后确认,获得重建3D图像(图1)。

图1 手动标注T2WI所示盆腔组织示意图 A~C.分别于轴位不同层面T2WI上标注各组织结构; D~F.分别为重建的组织结构正面观、下面观及背面观3D图
1.4 3D U-Net模型分割盆腔组织结构 采用3D U-Net DL网络[5]分割盆腔各组织结构。按8∶1∶1比例将数据分为训练集(n=137)、调优集(n=21)和测试集(n=17)。将训练集T2WI和手动标注的各盆腔结构标签导入3D U-Net模型进行训练。采用Opencv 3.4.0.12(https://opencv.org/)、Numpy 1.16.2(https://numpy.org/)及Simple ITK .2.0(https://simpleitk.org/)软件包对175组T2WI数据进行预处理,使其素数大小为256×256×64(X,Y,Z),自动窗宽、窗位;以Adam(https://arxiv.org/abs/1412.6980)为训练优化器,批量大小1,学习率0.000 1,循环次数200;DL框架为PyTorch 0.4.1(Python 3.6),训练使用显存为GPUNVIDIA Tesla P100 16G(https://www. nvidia.com/en-us/data-center/tesla-p100/),运行系统为ubuntu16.04。完成训练后,采用ITK-SNAP软件将3D U-Net模型分割区域叠加至手动标注区域及T2WI。
1.5 评价分割效能 以手动标注的标签为评价模型分割性能的标准,定量评估3D U-Net模型对盆腔内结构的分割性能,评价指标包括Dice相似系数(Dice similarity coefficient, DSC)、Jaccard相似系数(Jaccard similarity coefficient, JSC)、精确率(precision, PRE)、召回率(recall, REC)和准确率(accuracy, ACC)[6]。计算公式如下:
DSC=2·|X∩Y|/|X+Y|,
JSC=|X∩Y|/|X∪Y|,
ACC=|VTP+VTN|/|VTP+VFN+VFP+VFN|,
PRE=VTP/|VTP+VFP|,
REC=VTP/|VTP+VFN|,
其中,X为手动标注的体素数,Y为模型分割的体素数;VTP代表真阳性部分体素数,即模型分割结果与手动标注一致;VTN代表真阴性部分体素数,模型分割与手动标注一致;VFP为假阳性部分体素数;VFN为假阴性部分体素数。应用最小体积包围盒算法[7]测量并比较各结构的手动标注体积及模型分割体积(图2),评价模型测量盆腔组织结构的体积的效能。
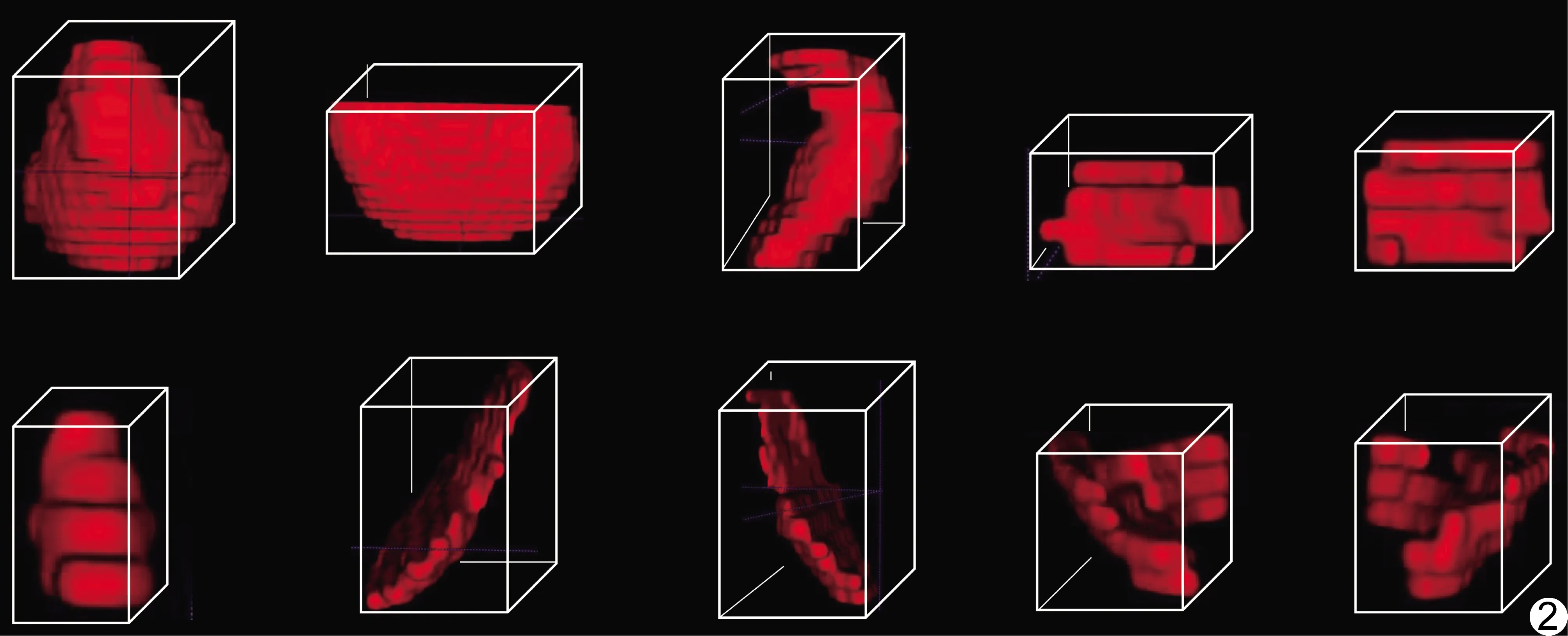
图2 采用最小体积包围盒算法测量盆腔各结构体积的示意图 上排从左至右依次为前列腺、膀胱、直肠、左侧精囊腺及右侧精囊腺;下排自左至右依次为尿道、左侧闭孔内肌、右侧闭孔内肌、左侧耻骨直肠肌及右侧耻骨直肠肌
1.6 统计学分析 采用SPSS 18.0和MedCalc 14.8统计分析软件。计数资料以频数表示。采用Kolmogorov-Smirnov法对计量资料进行正态性检验,符合正态分布者以±s表示,行方差分析或独立样本t检验;非正态分布以中位数(上下四分位数)表示,行Kruskal-WallisH检验。P<0.05为差异有统计学意义。
2 结果
2.1 组间一般资料比较 训练集113例中,86例前列腺增生、27例前列腺癌;调优集17例中,11例前列腺增生、6例前列腺癌;测试集17例中,11例前列腺增生、6例前列腺癌;其间患者年龄及血清前列腺特异性抗原(prostate specific antigen, PSA)水平差异均无统计学意义(P均>0.05),见表1。
表1 训练集、调优集及测试集患者一般资料比较
2.2 3D U-Net模型的分割效能 以手动标注盆腔组织结构标签为参考标准,3D U-Net DL模型对于测试集盆腔各组织结构的分割结果与手动标注结果的重合度较好,其分割前列腺癌及前列腺增生患者盆腔结构的DSC及JSC均>0.90,ACC、PRE及REC亦均>90%,见表2、3及图3。

表3 3D U-Net DL 模型分割测试集前列腺增生患者盆腔各结构的效能
2.3 体积比较 以手工标注体积为参考标准,3D U-Net模型分割测试集前列腺癌和前列腺增生患者盆腔各结构体积与手动标注差异均无统计学意义(P均>0.05),见图4。

图4 3D U-Net DL模型分割测试集患者盆腔各结构体积与手动标注分割体积比较 A.前列腺癌患者; B.前列腺增生患者
3 讨论
目前已有学者[8]基于mpMRI对前列腺及前列腺癌灶进行自动分割和定位。ZHU等[8]通过建立级联卷积神经网络,实现了于T2WI上分割前列腺腺体及外周带和中央腺体结构;朱丽娜等[9]采用U-Net网络基于表观弥散系数图自动分割和定位前列腺癌;这些研究多针对前列腺或前列腺癌灶本身,未对其周围组织结构进行研究。本研究训练的3D U-Net DL模型可分割盆腔T2WI所示前列腺及其周围正常组织,达到了定位周围组织结构的目的,为后续研究前列腺癌周围组织侵犯奠定了基础。
精准放疗是根治性治疗前列腺癌的主要方式之一[10]。放疗中不仅要准确治疗肿瘤,亦须保护盆腔正常脏器,以减少不良反应。CT是目前放疗过程中勾画靶区的主要成像途径,能提供电子密度信息,用于计算放疗剂量[11],并可较好地显示盆腔结构,但显示前列腺肿瘤病灶效果不佳。MRI对软组织分辨率高,对判断肿瘤病灶与正常组织的关系更具优势[12]。研究[13]表明,针对MRI勾画的前列腺体积与根治术中切除的实际前列腺体积基本吻合。
3D U-Net用于分割多种医学图像时均表现出良好性能,尤其样本量较少时亦能取得较好效果。DSC和JSC是衡量2个体素集合之间的相似性的度量指标,取值范围为0~1,其值越大,2个集合之间的相似度越高。本研究采用3D U-Net DL模型分割147例前列腺疾病患者的175组盆腔T2WI所示盆腔软组织结构,测试集结果显示,该模型对前列腺癌及前列腺增生患者盆腔各组织结构的分割结果与手动标注结果均具有较好的重合度,DSC及JSC均>0.90,以单个体素是否正确进行分类分析,模型分割各组织结构的ACC、PRE、REC均>90%,且分割测试集中前列腺癌和前列腺增生患者盆腔各结构的体积与手动标注差异均无统计学意义,表明其分割前列腺癌及盆腔组织的效能良好。本研究中双侧耻骨直肠肌的DSC及JSC略低于其他组织,分析原因,可能在于相比其他组织,耻骨直肠肌在图像中所占体素较少,且其形态不甚规则。另外,本研究采用两种不同MR设备获取MRI数据,多设备来源的训练数据可能有利于强化模型的适应能力。
本研究的主要局限性:①为单中心回顾性研究,且无外部验证;②纳入前列腺癌患者肿瘤分期均≤T2期,故该模型是否适用于前列腺癌侵犯周围组织患者有待进一步验证。
总之,采用3D U-Net DL模型自动分割盆腔T2WI所示盆腔软组织结构,有望用于制定前列腺癌放疗计划。
