质粒连接克隆法用于提高单细胞全基因组扩增产物覆盖率及质量的初探
2022-02-23陈伟珊黄一芳叶昕怡郑磊
陈伟珊,黄一芳,叶昕怡,郑磊*
(1.南方医科大学 南方医院 检验科,广东 广州 510515;2.广西医科大学 第一附属医院 检验科,广西 南宁 530000)
近年来稀有单细胞基因组学的研究得到了广泛的关注[1],这些稀有细胞往往具有重要的生物学意义或临床价值,如宫颈脱落滋养层细胞、母体循环胎儿细胞或循环肿瘤细胞(circulating tumor cells,CTCs)等,在产前无创筛查和肿瘤转移等检测中发挥着重要的作用。但这些珍贵的稀有细胞含有的DNA量极少,仅约5.6 pg,难以满足测序的要求。因此,稀有细胞往往需要先进行全基因组扩增(whole-genome amplification,WGA)以获取足够量的DNA再进行下游分析。然而,目前单细胞WGA技术由于存在碱基偏好性和背景污染等问题,导致全基因组的扩增存在一定的区域偏向性,无法达到预期的WGA覆盖率和均一性[2-4]。近年来提出的液滴-多重置换扩增(multiple displacement amplification,MDA)和离心驱动液滴生成方法,可减轻DNA背景污染和扩增偏倚,但都存在通量低、处理过程繁琐、样本浪费等缺点[5-6]。多次退火环状循环扩增(multiple annealing and looping-based amplification cycles,MALBAC)是目前应用最广泛的单细胞全基因组扩增方法,该方法联合了聚合酶链式反应(polymerase chain reaction,PCR)和MDA技术,同时兼顾了单细胞全基因组扩增的保真度和均一性,极大提高了扩增产物的全基因组覆盖率。即便如此,在实际操作中,临床标本中如宫颈脱落滋养层细胞、母体循环胎儿细胞或CTCs等稀有细胞因为数量及质量受限、环境影响等因素的存在,其MALBAC扩增产物同样存在覆盖率低、质量差等困境。如何提高临床标本中稀有细胞的全基因组扩增产物质量是目前亟需解决的问题。而随着基因工程技术和分子生物学研究的飞速发展,用大肠杆菌感受态细胞进行转化的质粒连接克隆技术得到越来越广泛的应用[7]。已有大量研究表明质粒连接克隆技术在提高靶标DNA片段中发挥了重要作用,被广泛用于DNA片段的单克隆分析。通过将单细胞扩增产物的DNA片段与质粒片段进行连接克隆,提高低丰度扩增产物DNA片段的含量,可能是解决稀有细胞的全基因组扩增产物覆盖率低、质量差的重要手段。然而,目前关于质粒克隆技术在单细胞WGA产物的应用尚缺乏相关报道,本研究在MALBAC扩增的基础上,联合质粒连接克隆技术,旨在探究一种更有效地提高单细胞WGA产物覆盖率和质量的联合方法。
1 材料与方法
1.1 样本收集
本研究收集南方医科大学南方医院54例临床样本,其中包括19例宫颈脱落滋养层细胞样本及35例CTCs样本,所有样本均已通过单细胞挑选并使用MALBAC扩增技术(亿康基因公司,上海)完成单细胞WGA。样本收集及后续研究均已通过南方医科大学南方医院医学伦理委员会审查批准(批件号:NFEC-2019-211、NFEC-2019-188),且在患者充分知情的情况下进行。
1.2 试剂与仪器
MALBAC单细胞全基因组扩增试剂盒,Endo-Free Plasmid Mini KITⅡ购于广州三询生物科技有限公司;pMDTM19-T Vector Cloning kit载体,2×Accurate Taq Master Mix,GLDNAMarker2000购于广州瑞真生物技术有限公司;E.coli DH5α感受态细胞购于广州天骏生物科技有限公司;琼脂糖粉购于Biowest公司;STR检测试剂盒购于普洛麦格(北京)生物技术有限公司;引物均于上海生工生物工程股份有限公司合成,聚丙烯酰胺凝胶电泳(polyacrylamide gel electrophoresis,PAGE)纯化。
恒温水浴锅购于上海一恒科技有限公司;各式离心机购于Eppendorf公司;Nanodrop 2000分光光度计购于Thermo Scientific公司;凝胶成像分析系统购于SYNGENE公司;电泳仪购于北京六一仪器厂;普通PCR扩增仪购于Eppendorf公司;3500DX基因测序仪购于APPLIED BIOSYSTEMS公司。
1.3 质粒连接克隆
将1μL载体pMDTM19-T与5μLWGA产物进行连接,形成重组载体pMDTM19-T-WGA,转入E.coli DH5α感受态细胞中,经过冰浴、热激、冰上冷却和抗性基因表达等步骤进行质粒连接克隆。
1.4 质粒DNA提取
将增菌后的样本3000 r/min离心10 min,使用Endo-Free Plasmid Mini KITⅡ对离心后的沉淀物进行质粒DNA提取,具体步骤按试剂盒操作说明进行。提取的DNA用NanDrop 2000进行质粒DNA浓度及A260/A280、A260/A230检测,评估质粒DNA的纯度。
1.5 PCR扩增与琼脂糖凝胶电泳
合成针对人类基因组22条常染色体特定片段的22对扩增引物[1](表1),使用2×Accurate Taq Master Mix扩增目标片段,以总量为100 ng的克隆前或克隆后单细胞扩增产物DNA作为PCR反应模板,配置反应体系。具体PCR反应程序如下,预变性:95℃,5 min;变性:95℃,30 s,退火:Tm℃,30 s,延伸:72℃,45 s,进行35个循环;最终延伸72℃,5 min。克隆前与克隆后扩增产物经过琼脂糖凝胶电泳进行分离,利用电泳仪和凝胶成像分析系统检测阳性扩增位点的数量及扩增产物覆盖率的改变。

表1 单细胞全基因组扩增产物质量评估引物序列Table 1 Primers for quality assessment of single-cellwhole genome am plification products
1.6 Sanger测序
随机挑选样本,将克隆前后产物针对人类基因组22条染色体相应的特定位点进行Sanger测序,测序结果使用Chromas 2.3软件进行分析。
1.7 短串联重复序列(short tandem repeats,STR)
以定量约0.1~0.5 ng/μL的DNA作为PCR反应模板,配制反应体系。具体PCR反应程序如下,初始变性:95℃,2 min;热循环:94℃,30 s;60℃,1 min;72℃,1 min;进行10个循环;90℃,30 s;58℃,1min;72℃,1min;进行20个循环;最终延伸72℃,10 min。利用3500DX遗传分析仪检测,结果采用SoftGenetics GeneMarker 2.2.0软件进行分析。
1.8 统计学分析
采用SPSS 20.0软件进行统计分析,计数资料以百分率(%)表示,组间比较采用配对t检验,以P<0.05为差异有统计学意义。
2 结果
2.1 验证分子克隆转化过程
使用针对人类基因组22条染色体特定片段的22对扩增引物对克隆前后产物进行PCR反应,PCR产物经1.6%琼脂糖凝胶电泳分离检测。结果显示在100~250 bp合适区域内,质粒连接克隆前后PCR产物在22条染色体特定位点相应的引物片段大小处均出现了大略一致的条带(图1),说明载体pMDTM19-T可成功将单细胞WGA产物转入大肠杆菌DH5α中进行克隆增殖,初步证明质粒连接克隆法可用于单细胞WGA产物的转化。

图1 单细胞全基因组扩增产物质量评估Figure 1 Quality assessment of single cell whole genome amplification products
2.2 克隆前后全基因组覆盖率的比对
根据既往文献报道[8-10],本研究通过计算54例单细胞样本克隆前后WGA产物中22条染色体上特定22个位点的扩增阳性率来代表其各自WGA产物覆盖率(表2),即WGA产物覆盖率=阳性扩增位点数量/总扩增位点数量×100%。采用配对t检验(表3)对纳入的54例稀有单细胞WGA产物进行克隆前后扩增覆盖率的比较(图2),结果表明稀有细胞全基因组覆盖率(78.67%)克隆后显著高于克隆前(73.37%),差异有统计学意义(P<0.001)。分组分析显示宫颈脱落滋养层细胞全基因组覆盖率克隆后也显著高于克隆前(P<0.001),CTCs全基因组覆盖率克隆后也显著高于克隆前(P<0.05)。

图2 克隆前后基因组覆盖率分析Figure 2 Analysis of genome coverage before and after cloning

表2 54例稀有细胞样本克隆前后22对引物的PCR结果及覆盖率比较Table 2 PCR results of 22 pairs of primers and coverage before and after cloning of 54 rare cell sam ples

表3 全基因组扩增产物克隆前后配对t检验结果Table 3 Results of paired t-test of whole genome amp lification products before and after cloning
2.3 Sanger测序结果验证
随机选取12例样本针对人类基因组22条染色体相应的特定位点进行Sanger测序。结果显示:样本45的位点13(45-13)、样本49的位点22(49-22)和样本50的位点12(50-12)克隆前为杂峰,而克隆后Sanger测序成功,峰图正常。而样本5的位点21(5-21)、样本6的位点21(6-21)、样本7的位点21(7-21)、样本16的位点12(16-12)、样本17的位点21(17-21)、样本23的位点13(23-13)、样本31的位点15(31-15)、样本51的位点12(51-12)和样本54的位点16(54-16)均为克隆前琼脂糖电泳无条带或条带淡,Sanger测序失败;克隆后条带清晰明亮,Sanger测序成功,峰图正常(图3)。
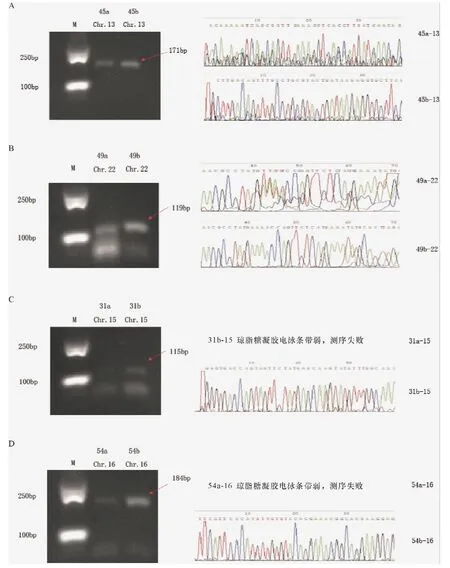
图3 稀有细胞全基因组扩增产物克隆前后的Sanger测序结果Figure 3 Sanger sequencing results of whole-genome amplified products in rare cells before and after cloning
2.4 STR分析
随机选取2例样本进行STR分析以验证克隆后单细胞WGA产物质量是否有提升。结果2例样本克隆后STR分析显示的基因座均多于克隆前,其中样本23克隆前STR仅检测出3个基因座:D19S433、D18S51及AMEL;通过质粒连接克隆后,STR 检测到了15个基因座:D8S1179、D21S11、D7S820、CSF1PO、D3S1358、TH01、D13S317、D16S539、D19S433、vWA、TPOX、D18S51、AMEL、D5S818及FGA(图4)。

图4 稀有细胞全基因组扩增产物克隆前后的STR结果Figure 4 STR results of whole-genome amplification products in rare cells before and after cloning
3 讨论
目前以稀有胎儿细胞和CTCs为基础的诊断技术得到了广泛关注[11-12]。但这些珍贵的稀有单细胞含有的DNA量极少,需要依赖于WGA扩增技术才可达到测序要求。目前关于改善单细胞WGA扩增质量的技术方法绝大多数是基于MDA技术[5-6,13],但由于MDA本身存在指数扩增、随机引物偏好以及不可避免的试剂残留DNA污染等局限性,并没有很好地改善单细胞WGA覆盖不均匀等问题。而MALBAC则是联合了PCR和MDA技术,采用拟线性而非指数扩增的方式来实现高覆盖率,成为目前应用最广泛的WGA技术。此外,质粒连接克隆法作为一项成熟的技术,已被广泛应用于基因工程和分子生物学研究[14-15],但尚无质粒连接克隆法在单细胞WGA产物应用的相关报道。因此,本研究提出以宫颈脱落滋养层细胞和CTCs的MALBAC扩增产物作为研究对象,利用质粒连接克隆技术,以提高稀有细胞WGA扩增产物覆盖率和质量的技术路线。目前使用多对引物对不同染色体上的位点进行PCR的组合法已广泛应用于WGA的质量控制[16]。Wang等[10]的研究针对22条常染色体设计了22对引物,对22个位点进行PCR反应,并用琼脂糖凝胶电泳法分离产物,以确定单细胞WGA产物的扩增效率。在Leung等[8]的研究中,也同样采用了22个位点组合法来评估WGA的扩增效率。评估扩增效率时,通常会用到覆盖率这一指标。覆盖率是度量扩增完整性的一个手段,在基因组测序中表示测序获得的序列占整个靶基因如全基因组序列的比例。Chen等[9]在研究中发现,在采用22个位点组合法时,当阳性扩增位点数量在20个(20/22)以内时,“阳性扩增位点数量”与在50X平均深度下的1X覆盖率呈线性相关性。因此,本研究通过使用22个位点组合法,初步验证了质粒连接克隆法用于单细胞WGA产物转化的可行性。同时通过计算WGA扩增产物阳性位点数量占其总扩增位点数量的百分比,作为克隆前后WGA产物的扩增覆盖率,即WGA产物覆盖率=阳性扩增位点数量/总扩增位点数量×100%,并通过配对t检验比较克隆前后WGA产物覆盖率的变化是否存在统计学差异,评估质粒连接克隆法能否提高WGA产物的覆盖率。结果显示,克隆后的产物全基因组覆盖率显著增加,部分原本由于扩增偏倚导致扩增产物量低于检测值的基因组依靠克隆增殖而达到分子技术检测限,提高了单细胞WGA覆盖率和均一性,为深入解析这些稀有细胞的生物学意义及临床价值提供了极大的便利,为无创产前诊断和癌症治疗等生物医学的应用提供了基础。
通常单细胞基因组DNA的含量约在pg到ng水平,因此WGA扩增时如何减少扩增偏倚、提高扩增纯度及全基因组覆盖率是目前单细胞分析面临的首要问题。由于MALBAC方法的局限性,WGA产物含有大量的离子dNTP,降低了DNA的纯度。而联合质粒连接克隆技术后,大量的离子dNTP得不到复制,克隆后的单细胞WGA产物DNA纯度可能会提高。Lu等[17]研究显示88个临床相关癌基因和抑癌基因评估MALBAC扩增的覆盖率为(73.6±9.6)%;Liu等[18]使 用MALBAC成功扩增了4例晚期实验室Dd2寄生虫样本,其基因组的覆盖率平均达到67.9%;而Chen等[19]在42例MALBAC扩增的样本中得出平均覆盖率为60.44%。本研究通过22个位点组合法来计算单细胞克隆前后的WGA覆盖率,结果显示稀有细胞经MALBAC扩增后全基因组覆盖率为73%,与既往的研究结果相似。基于MALBAC联合质粒连接克隆技术后,稀有细胞克隆后的全基因组覆盖率(79%)显著高于克隆前(73%)。对于宫颈脱落滋养层细胞而言,提升效果更为显著(克隆前72%、克隆后81%)。对于WGA方法的改善,有研究者提出离心驱动液滴生成方法可以最大限度减少WGA的背景污染和扩增偏倚[5-6],然而离心时会有大量样本残留在连接管和芯片通道中,造成样本的浪费,这对于稀有细胞扩增研究来说是不可接受的。虽然Li等[20]通过简化装置,改善了这一问题,但该技术取得的单细胞WGA覆盖率仅为(46.9±3.0)%,远低于本研究克隆后单细胞WGA覆盖率(79%)。本研究所用的质粒连接克隆法不仅在覆盖率上占优势,且由于一个重组质粒对应一个DH5α,最大程度地利用了稀有细胞,避免样本的浪费。Tian等[16]还提出了通过微柱阵列(GAMA)进行基因组扩增的方法,可使WGA覆盖率达到81.08%,与本研究中宫颈脱落滋养层细胞克隆后的WGA覆盖率(81%)相当,但GAMA方法还不成熟,尚无优化微流控设备的流速及其对最终放大性能的影响,且设备昂贵。相比之下,质粒克隆技术经过了半个世纪的发展与验证,成为一项成熟稳定的技术,且价格低廉,可行性高。
迄今为止,关于WGA方法用于检测单基因病或目标区域突变的效率评估并不多见[21]。本研究随机分析的12个样本中,有3个样本相应位点克隆前Sanger测序为杂峰,克隆后测序成功;另外9个样本克隆前Sanger测序失败,克隆后测序成功,提示质粒连接克隆法在一定程度上提高了MALBAC扩增产物的Sanger测序峰图质量及成功率,提高了稀有细胞在检测特定点突变等单基因性疾病的效率,对胎儿遗传性疾病诊断和肿瘤突变位点检测提供了重要的价值。此外,微量DNA的使用对法医学的研究至关重要,目前许多用以提高微量DNA质量和数量的技术会导致更多重叠峰以及等位基因的丢失[22-23]。有研究者评估了MALBAC扩增后DNA的产量和STR分型的成功率,提示MALBAC用于人体微量DNA分析具有可行性[24-25]。本研究的代表性样本23于克隆前STR仅检测出3个基因座;通过质粒连接克隆后,STR检测到了15个基因座,比克隆前增加了12个基因座,初步表明MALBAC结合质粒克隆法在微量DNA分析中的巨大潜力,在法医学和遗传学领域具有广泛的应用前景。
本研究的质粒连接克隆法在一定程度上提高了单细胞WGA扩增的覆盖率及质量,但仍存在一定的不足与缺陷。研究中某些样本在克隆前后出现极少量非特异性扩增,这可能与样本的引物偏好、扩增随机效应以及MALBAC所用的Tap聚合酶保真性低相关[26]。MALBAC扩增是先使用Bst DNA聚合酶形成环状的DNA片段后再行线性的PCR扩增[27],而Bst DNA聚合酶是缺乏校对性的,因此可采用具有校对功能的高持续合成能力和高保真性DNA聚合酶来替代原有的Bst DNA聚合酶,形成更低错误率和更长的扩增子,从而减少扩增误差,避免非特异性扩增,从而优化MALBAC技术。今后可扩大样本量以进一步分析与验证。
本研究提出在MALBAC的基础上联合质粒连接克隆技术用以提高单细胞WGA产物覆盖率及质量的技术路线。经评价验证,克隆后的单细胞WGA产物的覆盖率和质量都显著提高,且该联合方法成本低廉、操作简单。因此,本研究提出的技术切实可行、效果良好,可以使原本扩增效果不佳的稀有单细胞WGA产物质量得到改善,从而更好地应用于生物医学,为更多学者开展单细胞水平的研究提供了思路。
作者贡献声明
陈伟珊:设计实验流程、开展实验、统计分析数据、撰写论文初稿;黄一芳:构思研究思路和框架、实验数据评估、审阅修改论文;叶昕怡:协助实验开展,辅助样本收集;郑磊:实验课题的资金支持和平台建设、实验流程的指导和监督、终稿的确认。
利益冲突声明
本研究未受到企业、公司等第三方资助,不存在潜在利益冲突。
