基于深度学习的脑肿瘤分级预测方法研究
2022-02-10梁国标李贞妮
闻 亮,于 跃,梁国标*,李贞妮
(1.北部战区总医院神经外科,沈阳 110016;2.中国医科大学,沈阳 110122;3.东北大学信息科学与工程学院,沈阳 110819)
0 引言
大脑是人类身体中最重要的器官,而脑部肿瘤是在颅腔内生长的异常组织,又称颅内肿瘤、脑癌。脑部肿瘤相比于其他器官的肿瘤来说小得多,其中胶质瘤占所有脑部肿瘤发病率的一半以上,死亡比例超过2.4%[1]。根据胶质瘤的性质,可以将其分为高级别胶质瘤(high-grade glioma,HGG)和低级别胶质瘤(low-grade glioma,LGG)。由于HGG 容易变异,常常会被称为恶性肿瘤,其死亡率较高,患者痊愈后恢复效果也比较差。而LGG 则不一定为恶性肿瘤,但其在其他因素影响下即使治疗后仍然可以演变为恶性肿瘤。因此,胶质瘤是脑部肿瘤治疗领域的研究重点。在临床中越早发现脑部肿瘤,患者就越有希望被成功治愈。Jiang 等[2]论述了世界卫生组织中枢神经系统(central nervous system,CNS)肿瘤分类标准对于临床预后判断的价值与优势,这说明如果能够更早发现患者的脑部肿瘤并对其级别进行预测,对患者的后续诊疗以及恢复有很大帮助。
目前确定脑胶质瘤的等级仍需要对患者脑部肿瘤区域进行活体生理切片,脑组织大切片的制作在神经病理诊断中有着重要应用,可用切片检测结果来诊断HGG 或LGG,但是对于HGG 患者,组织生理切片对其生命存在一定威胁。因此,基于MRI图像对脑肿瘤的级别进行预测具有重要意义。然而,MRI 图像边界模糊,脑肿瘤区域占比较少,对肿瘤进行手动分割进而预测其级别难度很大且耗时耗力。
在临床诊断中,多模态MRI 对脑胶质瘤有着较高的诊断符合率,因此可以采用多模态MRI 序列(FLAIR、T1、T2、及T1CE等)对脑胶质瘤进行多参数成像。常规MRI 平扫和增强检查难以实现脑胶质瘤的诊断和分级,而基于其病理学基础的功能MRI 检查方法能够提供包括微环境、微血管、细胞增殖等更加丰富的信息,可以有效提高脑胶质瘤的临床诊断效能[3]。不同MRI 序列针对的病理变化区域也不同[4],T2模态图像可突出显示脑胶质瘤的水肿部分,FLAIR模态图像有助于区分脑胶质瘤水肿和脑脊液,而T1和T1CE模态图像则能明显观察到高信号脑胶质瘤的核心。
随着“互联网+”概念的兴起,人工智能已经渗透到人类生活的方方面面。深度学习算法因其特有的属性,在很多领域突破了传统机器学习算法的瓶颈,特别是在医疗影像识别领域展现了其强大的识别能力[5-6]。随着深度学习算法的不断发展,其在医疗领域的应用也越来越多,例如精确切割病灶、预测心脑血管疾病等[7]。国内外也有许多专家学者开始研究脑肿瘤患者MRI 图像的分割,例如卷积神经网络(convolutional neural networks,CNN)和支持向量机(support vector machine,SVM)结合的脑肿瘤分割方法[8-9]、基于U-Net 的分割方法[10]、基于双流解码网络(dualsteam decoding U-net,DDU-net)的分割方法[11]、多任务深度监督注意力R2U 分割方法[12]以及基于多重注意力机制的分割方法[13]等。这些方法都是对MRI图像进行分割,标记感兴趣的肿瘤区域,但不具备完成肿瘤检测识别的功能。随着网络深度的增加,近年来图像分类算法也层出不穷,例如K 最邻近算法(K-nearest neighbors,KNN)、反向传播神经网络(back propagation neural network,BPNN)、视觉几何群网络(visual geometry group network,VGG)等,但是基于三维MRI 图像进行分类的难度很大。
针对以上问题,本研究基于深度学习算法提出一种脑肿瘤分级预测模型(brain tumor groding model,BTGM),既可以对输入的MRI 图像进行脑肿瘤的分割处理,为医生提供一个直观的病态分析,又可以对分割后的肿瘤图像进行分级预测,进一步判断脑胶质瘤的等级,从而为医师对患者的后续治疗提供辅助诊断。因为MRI 具有无创性、多模式的优点,人们期望用医学成像来探知人体内是否存在隐藏疾病[14],若可以在病情萌芽阶段及时发现,则能够大大提升患者的生存率。因此,本研究提出的BTGM 具有重要的理论价值和应用前景。
1 BTGM 建模
基于深度学习的BTGM 结构如图1 所示。本模型由脑肿瘤分割模型、脑肿瘤分类模型及患者术后生存周期模型3 个部分组成,可以基于对MRI 图像中脑肿瘤的分割与提取来完成脑肿瘤等级的分级预测,同时通过多项式拟合法实现对患者术后生存周期的预测。
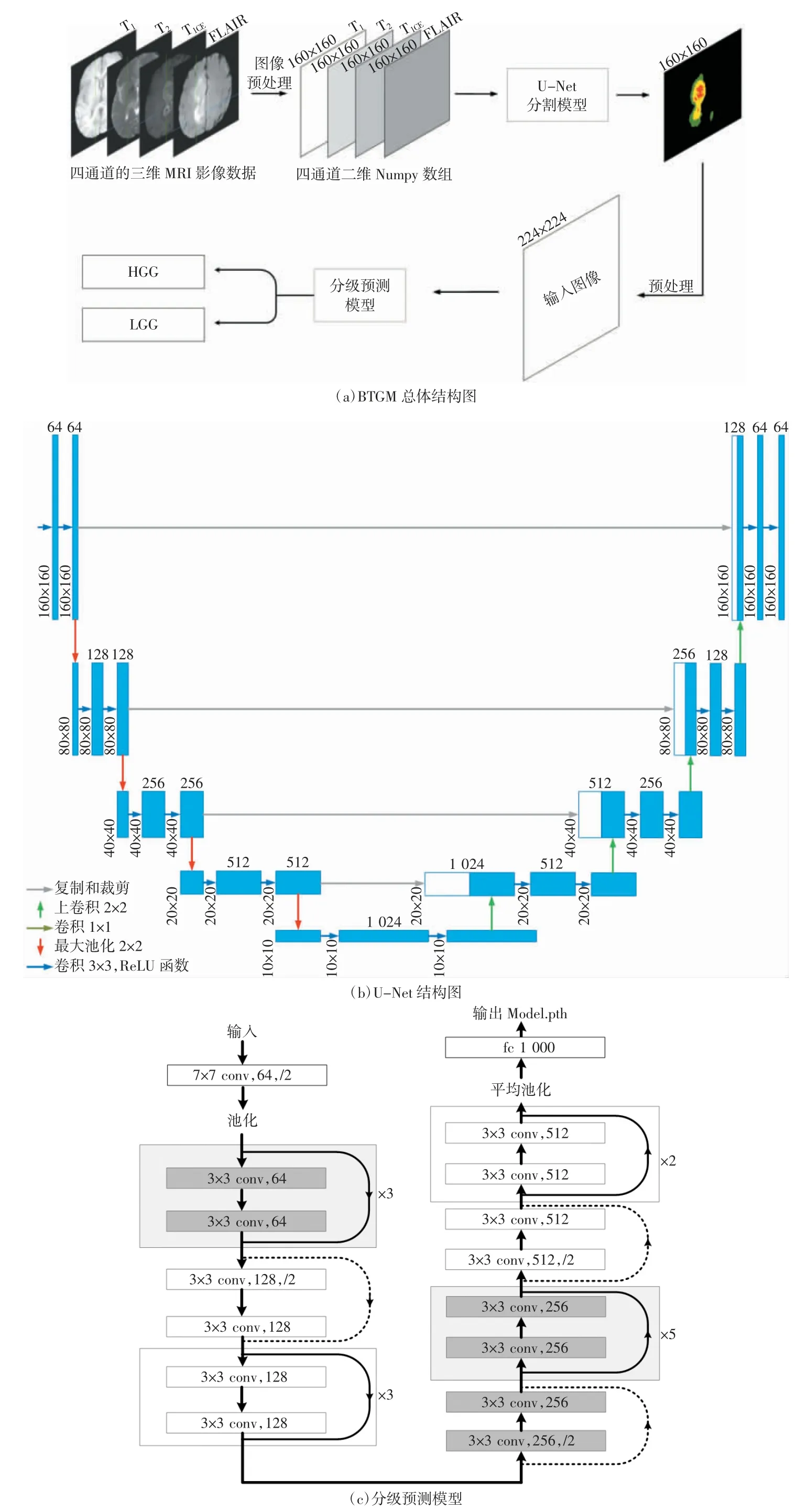
图1 基于深度学习的BTGM 结构图
本模型基于BraTS 2019 数据集,通过设计、改进损失函数来训练分割网络并获得最优解,通过数据集提供的Dice 函数、PPV 函数及Sensitivity 函数来评价模型的分割效果。以分割模型结果为基础,基于残差网络设计BTGM,并基于患者的脑胶质瘤级别、术后生存周期和患者年龄等信息,利用多项式拟合方法对患者预后的效果进行预测,最后对预测结果进行综合评价。
1.1 脑肿瘤分割模型的设计
1.1.1 数据集选择
本研究选用MICCAI BraTS 2019[15]多模态脑胶质瘤分割挑战赛数据库提供的临床数据进行训练和验证。其中,BraTS 2019 数据集由259 例HGG 病例和76例LGG 病例构成,所有脑影像均带有专家手动分割的真值标签,每个病例都包括FLAIR、T1、T2、T1CE4 个模态,影像的三维尺寸是155×240×240 像素。多模态MRI脑肿瘤图像如图2 所示。

图2 多模态MRI 脑肿瘤图像
根据实验需要,本研究将对3 个区域的分割算法进行探讨。BraTS 2019 数据集规定,绿色为浮肿(peritumoral edema,ED)区域(标签2)、黄色为增强肿瘤(enhancing tumor,ET)区域(标签4)、红色为坏疽(non-enhancingtumor,NET)区域(标签1)、黑色为背景区域(标签0),多模态MRI 图像分割后的输出图像如图3 所示。ED、ET、NET 3 个区域经过嵌套就是最终提取的感兴趣区域,即全部肿瘤(whole tumor,WT)区域(WT=ED+ET+NET)、肿瘤核心(tumor core,TC)区域(TC=ET+ENT)和增强肿瘤区域ET。

图3 多模态MRI 图像分割后的输出图像
1.1.2 数据预处理
本研究提出的BTGM基于U-Net 二维网络。由于直接基于三维MRI 图像进行分级预测对算力需求较高,本研究为了降低计算量并提高处理速度,首先将三维MRI 数据进行图像预处理转变为二维数据,同时在对图像预处理的过程中通过几何变换(随机角度旋转、水平翻转、垂直翻转)实现数据增强,提高模型的泛化性。因此,在训练模型前需要对三维MRI 图像进行预处理从而提高图像的分割效果[16],具体分为以下3 个步骤:
(1)对各个模态进行标准化;
(2)切割各模态数据集和真实值(ground truth,GT)数据集;
(3)将数据切割成切片,合并每个模态的部分并保存为“Numpy 数组”。
由于FLAIR、T1、T2、T1CE4 个序列属于不同模态的图像,具有明显的特征对比度,所以采用Z-score方式对每个模态图像进行标准化,即将每个模态的数据标准化为零均值和单位标准差。经过处理的数据均值为0,标准差为1,其换算公式为

式中,x 为原始数据;x¯为原始数据的均值;σ 为原始数据的标准差;X*为标准化的数据,高于平均水平的数据获得标准化的正分数,低于平均水平的数据获得标准化的负分数。在此基础上,利用normalize 函数进行数据归一化,去掉最高分和最低分数据,得到标准差较低的公平数据集。
此外,BraTS 数据集中边缘区域大多是黑色无效区域图像,因此剪裁掉周边非感兴趣区域对提高整体识别度大有帮助。本研究基于最小矩阵法将MRI 图像中周边的黑色区域剪裁掉,只留下脑部区域,这样最终分割出来的图像中大部分都是脑部区域及肿瘤区域,观察更直观、更清晰,数据预处理前后的效果对比如图4 所示。

图4 数据预处理前后的效果
由于MRI 图像数据是三维的,所以需要切成二维数据才能输入U-Net。以50%概率将切片中无病灶区域舍弃,缓解数据类别不平衡的问题,最后将4 个模态保存至4 个通道,并剪裁成160×160 像素的大小,最终Numpy 数组保存的“shape”是(160,160,4)。
1.1.3 网络结构设计
BTGM 中的脑肿瘤分割网络包括4 层下采样和4 层上采样,每层下采样部分包括2 个卷积核为3×3的卷积操作,并通过ReLU 激活函数激活有效信号,再进行一次卷积核为2×2 的最大池化操作。经过4层下采样操作可以有效提取脑肿瘤的特征信息,而上采样部分与下采样相反,可以还原下采样得到的有效信息,并且上采样过后进行了跳跃链接,这样的叠操作使得信息提取更加充分。本研究输入通道为4,每一通道对应原始MRI 图像的T1、T2、T1CE和FLAIR 图像,相关的3 个感兴趣特征区域ED、ET 和NET 分别用绿色、黄色和红色标记。
1.1.4 损失函数设计
为了避免数据不均衡,本研究应用Dice 损失函数[17]。Dice 损失函数是通过计算Dice 系数得出的,二者之和为1。Dice 系数是一种集合相似度度量函数,通常用于计算2 个样本的相似度,其取值范围为[0,1],计算公式为

式中,X 表示GT 分割图像;Y 表示预测分割图像;|X∩Y|表示X 和Y 交集中元素的个数;|X|和|Y|分别表示X 集合和Y 集合中各自元素的个数。分子的系数是2,是因为分母中重复计算了X 和Y 的公共元素。
Dice 损失函数的计算公式如下[17]:

考虑到Dice 损失函数比较适合高度不均匀的样本,因此在设计本研究损失函数时,为了提高训练的稳定性,引入二分类损失函数(binary cross entropy loss,BCELoss),并使用Sigmoid 函数将数据映射到(0,1)。最终设计的损失函数Loss 计算公式如下:
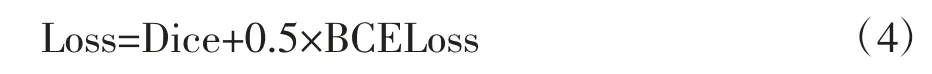
二分类损失函数BCELoss 公式如下:

式中,y 为GT 分割图像;x 为模型输出值;n 为样本的总数。
1.1.5 训练结果及模型评估标准
本研究提出的BTGM 基于RTX2080ti 显卡,使用Linux 系统,配合深度学习框架PyTorch 和conda环境管理系统进行训练。训练数据为BraTS 2019 提供的官方数据集,共335 例病例(259 例HGG+76 例LGG)。本研究按照70%训练、20%验证、10%测试的比例划分数据集。首先对数据集进行预处理,对三维MRI 图像进行降维,并将其保存为数组中的Numpy数据。然后进入conda 环境,设置历元大小、学习速率,并使用Adam 优化算法[18]减少内存占用。
根据BraTS 官网给出的3 个判断依据(分别为Dice、Sensitivity、PPV 3 个函数)对算法模型进行评估。其中3 个函数是由专业医师绘制的3 个肿瘤区域预测结果P{0,1}的二值图和真值标号T{0,1}的二值图。Dice 函数计算2 个分割区域的真实正样本数量标准化值,即预测病变区域中有多少像素区域是正确预测的,其计算公式如下:

式中,Pi表示预测分割图像的像素集合;Ti表示GT分割图像的像素集合;i 表示第i 种肿瘤区域。
Sensitivity 函数代表实际阳性样本率,即将预测为肿瘤区域的阳性样本数据与实际分割结果为阳性样本的数据进行比较,其计算公式如下:
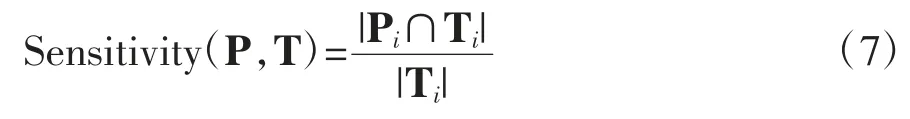
PPV 函数用于将肿瘤区域的实际样本、全样本数据与实际分割结果进行比较,其计算公式如下:

式中,TP 为真阳性的数据,即预测结果与GT 均为正样本(肿瘤区域)的数据;FP 为假阳性的数据,即预测结果为负样本(背景)而GT 为正样本的数据;PPV表示预测为正的样本中预测正确的数据所占比例。
基于上述判断依据,通过对图像分割后自动分割的肿瘤面积与真实值手动分割面积的占比重叠大小来评价模型的好坏。
1.2 BTGM 设计
基于分割后得到的RGB 图像,设计以Resnet34[19]残差网络为基础的分类模型学习HGG 和LGG 的特征,并对脑肿瘤的级别进行预测。由于训练中存在过度拟合的问题,本研究在BraTS 2019 数据集的基础上增加了BraTS 2018 数据集中的部分数据。
本研究设计的分类模型分为2 个部分,一个是向下采样模块,一个是正常卷积模块。正常卷积模型包含2 个卷积层、2 个正则层和2 个连接前向传播函数的ReLU 激活函数。由于Resnet 残差块的输出表示为y=F(x)+x,因此存在一个单独的res 变量来存储初始输入张量x。
下采样模块的结构包括2 个连续的卷积层,其中第一层步长为2,残差连接部分需要使用步长为2 的1×1 卷积进行下采样。同时,模型设计通过增加通道数以匹配输出特征映射、相应的正则化函数和ReLU 函数激活特征值。由于分割后得到的是RGB图像,所以通道数为3,输出为一维张量,卷积层后连接池化层的池化核为3×3,基于前向传播函数将2 个模块连接起来构成分类模型的网络结构。
经过脑肿瘤分割网络后的RGB 图像大小为160×160 像素,需要用transpose 进行图像扩展和tensor等预处理,预处理后的图像大小为224×224 像素。训练过程中,数据集遍历10 次,依次输入32 张图片。初始学习率为0.001,每次学习后学习率提高10 倍。使用交叉熵损失函数和Adam 优化器,衰减策略设定为固定步长衰减,每2 轮学习速率衰减到原来的一半。由于设计的网络模型中Resnet 残差部分中的深度卷积会引起梯度爆炸问题,因此采用optimizer函数消除梯度。
由于分割图像中脑肿瘤大小不一致,并且不是所有图像都有3 个特征区域,如果只训练具有所有区域特征的图像,则会出现过拟合现象,因此本研究使用了数据增强。首先将含有脑肿瘤的图像放入数据集,然后将具有3 个完整特征区域的肿瘤图像导入数据集。通过上述方式,脑肿瘤分割网络可以学习到与识别肿瘤接近区域的特征,使模型更加成熟,分类预测效果更好。由于本研究处理的脑肿瘤分为HGG 和LGG 2 个级别,因此模型识别只有2 种情况,所以本研究通过大量的训练图像数据进行验证,将识别出肿瘤分级正确的图像数量和所有参与验证的图像数量的比值定义为分类模型的准确率,并以此作为评价模型的标准。准确率越高,该分类模型对肿瘤分级的预测效果越好。
2 结果
2.1 分割模型的分割效果评价
从Dice、Sensitivity 和PPV 3 个方面来评价分割效果,具体结果详见表1。
表1 分割效果
从分割效果可以看出,全部肿瘤区域的分割准确率可以达到0.891,所有脑肿瘤图像都得到了较好的分割。但由于对MRI 图像进行了降维处理,损失了一部分图像信息,导致BTGM 模型对脑肿瘤分割的准确率下降,与数据库中的较优算法间存在差距。尤其对于存在脑肿瘤损伤和强化的区域,分割的准确率较低,其主要原因是脑肿瘤坏死和强化区域较为分散,使得全脑图像的提取效果不佳。但是本研究提出的模型在增加脑肿瘤分割环节后,可以有效改善对脑肿瘤进行分级预测的效果,且可以为医师提供一个直观的病态分析,帮助其对患者的后续治疗提供辅助诊断,具有一定的应用价值。
2.2 分类模型的预测能力评价
本研究提出的分类模型增加了网络深度,可以使低维特征更好地映射到高维特征。对于BraTS 2019数据集中的HGG 和LGG 病例,本研究对预测模型进行分组测试。首先对分割后的脑肿瘤图像进行了筛选,保留了1 000 张WT 区域、TC 区域、ET 区域图像,并将图像分成10 组来评价模型的脑肿瘤分级预测能力。各组图像中HGG 和LGG 的病例随机选取,即每组包含不同人和不同等级脑肿瘤的图像。实验结果表明,其中有8 组分级预测准确率为100%,其余2 组准确率均在95%以上,平均准确率可以达到98%,与直接使用残差网络的分类效果相比,准确率提高了约10%,分级预测效果良好。分析可知,由于大部分HGG 和LGG 的差异主要在ET 区域,所以学习权值较高。而另外一小部分的LGG 和HGG 病例在ET 区域差异不大,但在TC 区域差异明显,这种情况可能会导致脑肿瘤分级的结果出现一些误差。
2.3 患者术后生存周期预测模型的预测能力评价
基于多项式分析法对患者术后生存周期进行预测,利用和方差(sum of squared error,SSE)、R 平方系数对拟合结果进行评价。初次拟合的线性模型多项式如下:

式中,Age 代表患者年龄。R 平方系数=0.190 7,SSE=1.302×107。
为了进一步减小拟合误差,本研究对同龄数据进行均值处理。实验结果表明,均值处理后得到的SSE约为初次拟合的1/10,R 平方系数更接近于1,术后生存周期预测模型拟合效果更好,模型能力更稳健,具有更好的泛化能力。
3 讨论
本研究针对脑肿瘤分割和脑肿瘤分级预测问题,提出了一种基于深度学习的BTGM。试验结果表明,脑肿瘤分割模型的肿瘤区域平均预测准确率达到0.827。该模型对TC 区域的分割精度达到0.848,未来可在提高其准确率方面作出进一步的改进。脑肿瘤分类模型的分类效果较好,多组测量准确率稳定在98%左右,可以给予医生更准确的指导和辅助判断。
在脑肿瘤分割模型中,需要对三维MRI 数据进行降维处理,因此在三维到二维图像的变换过程中可能存在信息丢失,导致模型分割精度下降。在下一阶段研究中,考虑将MRI 三维数据直接输入神经网络进行训练,以减少信息丢失,进一步提高训练效果。另外,虽然本研究提出的分割模型具有较高的识别度,但数据集的增强需要人工完成。下一步的主要研究方向是使分割模型网络更有针对性地找到脑内最大肿瘤区域的残差位置,从而进一步提高脑肿瘤分级的识别精度。
本文提出的模型能够在术前对患者进行脑肿瘤分割和分级预测,但是脑肿瘤患者术后容易出现并发症,对患者术后进行生存周期的预测能够为针对性的护理提供参考,从而能够有效地预防脑水肿、呼吸道感染、泌尿系统感染等的发生。因此,通过进一步对脑肿瘤患者术后的生存周期进行预测,可以进一步指导患者家属进行及时有效的护理配合,预防并发症的出现,延缓患者术后的生存周期。
4 结语
针对基于MRI 图像对脑胶质瘤分级困难的问题,本研究设计并实现了基于深度学习的BTGM。该模型由脑肿瘤分割模型、脑肿瘤分类模型及患者术后生存周期预测模型3 个部分组成。脑肿瘤分割模型基于医学MRI 图像,提取深度学习特征并完成脑肿瘤的分割提取。然后,基于监督学习方法对分割后的肿瘤图像进行分级预测,并结合患者年龄等生理数据,预测术后脑肿瘤患者的生存周期。结果表明,本研究提出的脑肿瘤分类模型对HGG 和LGG 的预测准确率可以达到98%,能够很好地辅助医师在患者发病早期作出脑肿瘤级别的判断,对患者的肿瘤进行早期干预,延长患者的生命。
本研究提出的BTGM 基于U-Net 二维网络,需要将三维MRI 数据预处理为二维数据,这个过程中不可避免会有一定的图像信息丢失,导致BTGM 中的分割部分准确率下降。且目前的研究中,将脑肿瘤只分成了HGG 和LGG 2 个级别,不同切片间可能存在高相关性,模型泛化性能在不同数据集间可能会存在较大差异。未来的研究中,将直接构建基于三维MRI 数据的脑肿瘤分割模型,并在此基础上改进残差网络构建BTGM,提高脑肿瘤分级预测的精度,同时考虑将本院实际病例增加到数据集当中进行训练,进一步提高模型的泛化能力,并将脑肿瘤的级别扩展到世界卫生组织提出的四级分类[20]。
