“鸡尾酒”式混合抗生素对细菌耐药性的影响
2022-01-21韩宝苍陈志彬张跃恒林志芬孙昊宇
韩宝苍,陈志彬,张跃恒,林志芬,孙昊宇
“鸡尾酒”式混合抗生素对细菌耐药性的影响
韩宝苍1,陈志彬1,张跃恒1,林志芬1,孙昊宇2*
(1.同济大学环境科学与工程学院,上海 200092;2.上海大学环境与化学工程学院,有机复合污染控制工程教育部重点实验室,上海 200444)
选择四环素、磺胺、磺胺增效剂3类常用抗生素,以其对大肠杆菌()的单一兴奋效应(hormesis)最大促进效应对应浓度(HCmax)配制等HCmax比的二元及三元混合体系,探究混合体系对细菌生长和耐药性(突变、质粒接合转移)的联合效应.结果表明,二元混合抗生素对生长和接合转移频率起到协同抑制,对的突变频率起协同刺激;三元混合抗菌剂对这三种效应均协同抑制.因此,推测二元混合体系会导致毒性和耐药性风险同时增加;三元混合体系会导致毒性风险增加、耐药性风险降低.
混合抗生素;细菌耐药性;hormesis;突变;接合转移
抗生素被广泛应用于医疗卫生、畜禽养殖等行业[1].然而,目前抗生素大规模生产与使用导致大量抗生素经由废水排放、畜禽排泄等途径进入环境中[2],增加细菌耐药性发生频率,加速固有抗性微生物和抗性基因的扩散[3].细菌耐药性污染的传播导致临床治疗中常规抗生素剂量无法抑制细菌而需要加大抗生素用量,甚至诱导了超级细菌的出现,这对人类健康构成极大危害.因此,如何控制细菌耐药性的发生与传播是目前亟待解决的问题.环境中细菌耐药性污染主要以抗性基因作为媒介[4].已有文献表明,抗性基因的产生和传播往往与环境中的抗生素残留密切相关[5].环境中抗生素浓度越大,抗性基因往往污染越严重.因此,有学者认为,可以通过减少抗生素使用量从源头降低抗性基因的产生[6].
“鸡尾酒疗法”作为一种药物使用策略,指联合使用3种或3种以上的药物进行治疗,其可以减少单一用药造成生物体对药物的依赖并加强疗效,在源头上减少耐药性的产生[7].因此,依照“鸡尾酒疗法”将低剂量抗生素混合使用,可能是临床上保持药效、减少细菌耐药性的抗生素联用手段.然而,按照“鸡尾酒疗法”使用抗生素后,会导致进入环境中的抗菌剂会以低剂量、混合的形式存在.在以往的研究中,低剂量抗生素对细菌往往会产生低浓度促进、高浓度抑制的双向剂量-效应,即hormesis效应[8].已有文献表明,hormesis效应在抗生素作用于大肠杆菌()、费氏弧菌等多种细菌时普遍存在[5,9].
四环素类(TCs)、磺胺增效剂类(SAPs)与磺胺类(SAs)抗生素是常见的临床使用抗生素,对革兰氏阳性菌与革兰氏阴性菌均表现出很好的抑菌效果.因此,本文以细菌耐药突变、RP4质粒接合转移表征细菌耐药性,选择可能作为“鸡尾酒疗法”配方的四环素类(3种)、磺胺增效剂类(3种)和磺胺类(1种)抗生素作为受试化合物,以经典模式生物为受试生物,探究单一抗生素对生长与突变、接合转移的影响;将单一抗生素的hormesis促进效应最高的浓度点(HCmax)按等HCmax比进行二元或三元混合,探究混合抗生素对生长与突变、接合转移的影响和机制;并与单一抗生素毒性与细菌耐药性进行对比,借鉴独立作用(IA)模型,判别单一抗生素在等HCmax比进行混合时对生长及耐药性的联合作用方式.本文旨在探究抗生素“鸡尾酒”暴露体系对细菌毒性与耐药性的影响,为实际环境中低浓度抗生素的联合作用和风险评估提供参考.
1 材料与方法
1.1 试剂与生物
实验所用抗生素购自 Sigma-Aldrich 化学制品有限公司(上海,中国),纯度为95%以上.受试化合物的基本信息见表1.购于 Biovetor 生物科技有限公司(北京,中国).毒性及突变实验所用生物为野生型MG1655.接合转移实验所用供体菌是(RP4),其RP4质粒上携带有卡那霉素、氨苄青霉素和四环素的抗性基因,受体菌是具有能够稳定遗传的萘啶酮酸(Nal)耐药标记的(Nal).
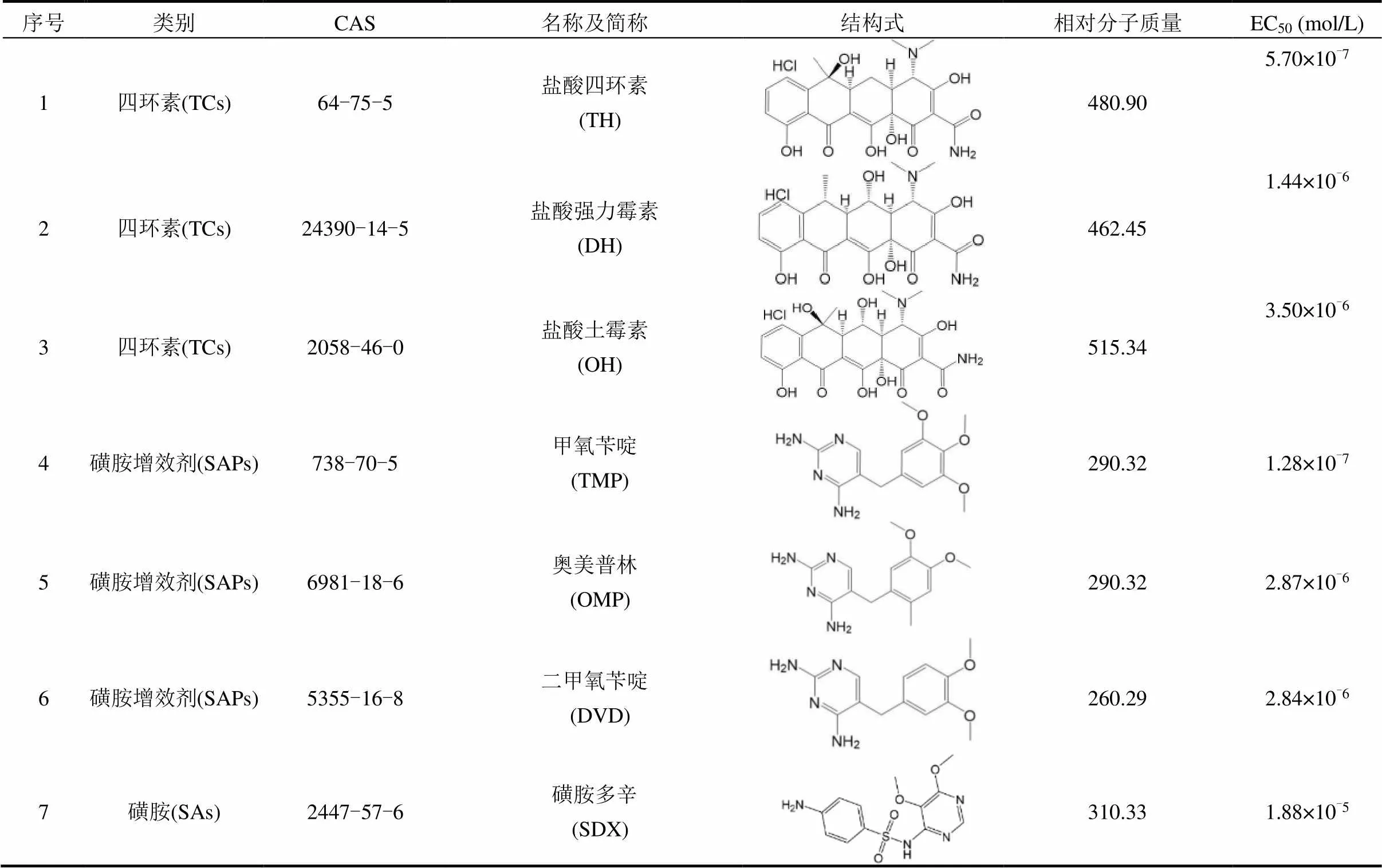
表1 抗生素信息及单一毒性
1.2 毒性实验
精确称取测试抗生素,使用二甲基亚砜(DMSO)助溶,随后用质量分数为1%的氯化钠溶液稀释成等对数浓度梯度的系列溶液.取80μL稀释后的化合物溶液加入到100孔板中,同时加入80μL培养基及40μL稀释后的菌液,每个浓度设置3个平行,设置的浓度组数及浓度范围见表2.混合抗生素各抗生素的比例和浓度值见表3.每组实验设置3个空白对照,操作同上,仅以80μL氯化钠溶液代替稀释后的抗生素溶液.于Bioscreen全自动微生物生长曲线分析仪(Multiskan GO,美国赛默飞世尔科技公司)中37℃震荡培养,第22h测试并记录600nm处的光密度值(OD600)的读数.
抗生素对的生长促进率由式(1)计算得出:
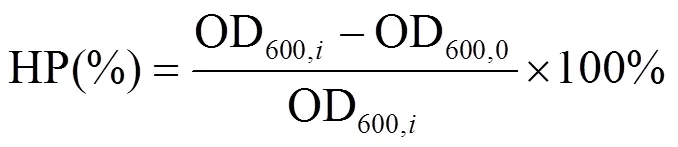
式中:HP表示抗生素对的生长促进率,OD600,0表示空白对照组的OD600值,OD600,表示各个实验组的OD600值.
1.3 突变实验
突变实验的培养条件与毒性实验相同.将含有溶液的100孔板在Bioscreen分析仪中于37℃培养22h后,取20μL稀释106倍的菌液加至不含任何抗生素的固体培养基上,37℃恒温静置培养12h后,进行菌落形成单位(CFU)计数,计算总菌数.另取20μL浓缩3倍的菌液加至含有40mg/L利福平的固体培养基上,37℃恒温静置培养12h后进行突变子CFU计数.对照组与实验组每个浓度点均设置3个平行.设置的浓度组数及浓度范围见表2.混合抗生素各抗生素的比例和浓度值见表3.
抗生素对的突变频率促进率的由式(2)、式(3)计算得出:

式中:为突变频率;m为突变子个数,CFU/mL;t为总菌数,CFU/mL;
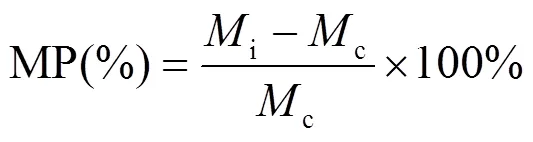
式中:MP为抗生素对的突变频率促进率;i和c分别为实验组和对照组突变频率.
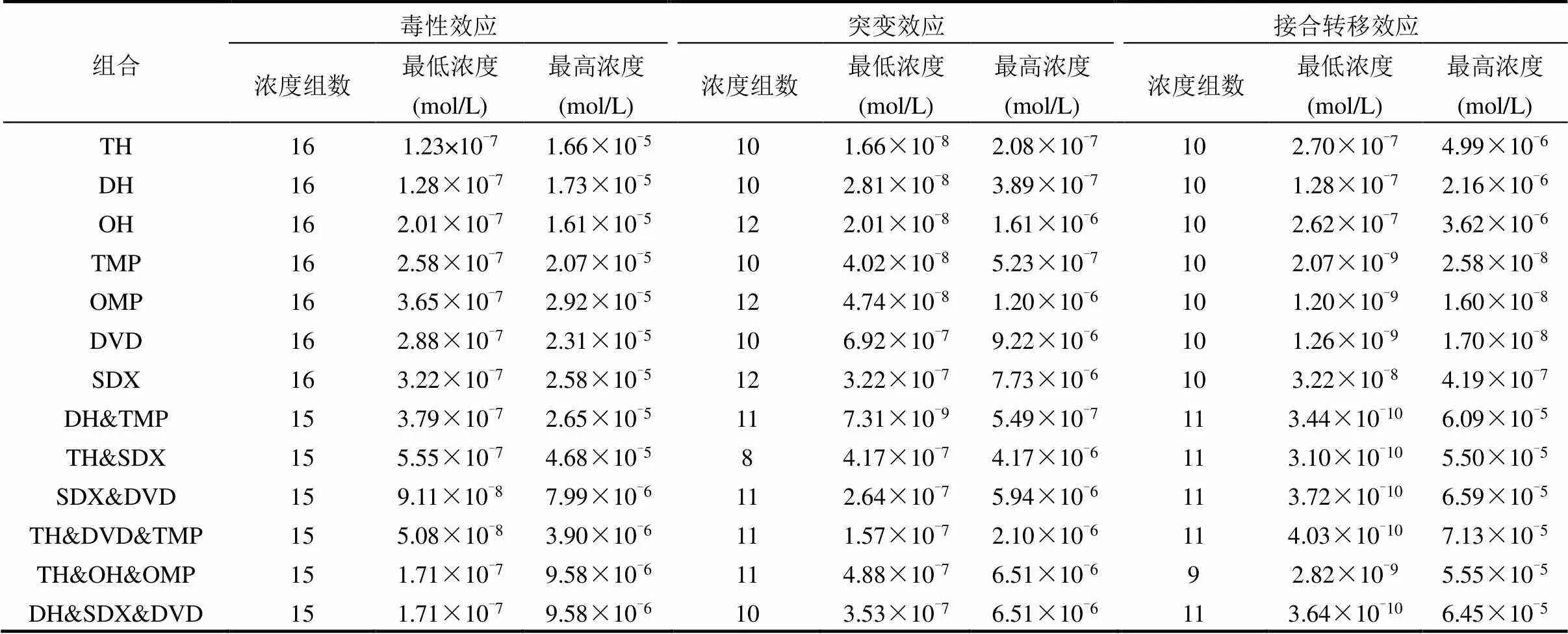
表2 实验设置浓度组数及范围
1.4 接合转移实验
将RP4质粒供体菌与受体菌分别接种至含有相应抗生素的单倍LB培养基中,在37℃条件下以180r/min振荡培养9h,用同样体积的生理盐水进行2次洗菌,去除多余的培养基及抗生素,调节其OD600值为0.5左右,再以供体菌:受体菌=1:2的比例混合制成工作菌液.
配置200μL的培养体系,其中包含40μL的单一或混合抗生素溶液、100μL的培养基和60μL的工作菌液,混匀后静置培养8h.之后进行梯度稀释,分别取稀释后的菌液30μL添加至含有萘啶酮酸和卡那霉素的筛选板上,37℃静置培养12h后分别进行接合子CFU计数与受体菌CFU计数.对照组与实验组均设置3个平行.设置的浓度组数及浓度范围见表2.混合抗生素各抗生素的比例和浓度值见表3.
抗生素对E. coli的质粒接合转移频率促进率由式(4)、式(5)计算得出:
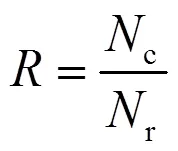
式中:为RP4质粒接合转移频率;c为接合子个数, CFU/mL;r为受体菌个数,CFU/mL;

式中:RP为抗生素对E. coli的RP4质粒接合转移频率促进率;i和c分别为实验组和对照组的RP4质粒接合转移频率.
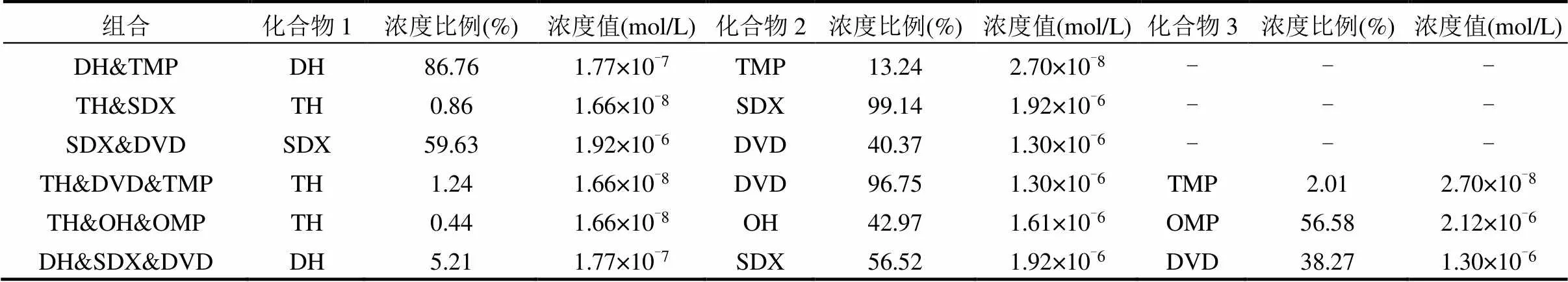
表3 化合物混合液中各抗生素的比例和浓度值
注:“-”表示无数据.
1.5 特征参数表征
抗生素对的毒性效应曲线如图1a所示.根据单一抗生素的毒性实验结果,当生长抑制率达到50%时,在图1a上对应浓度为EC50; HPmax在图1a上对应的浓度为HCmax.混合抗生素浓度为HCmix在图1a上对应的生长促进率为HPmix.
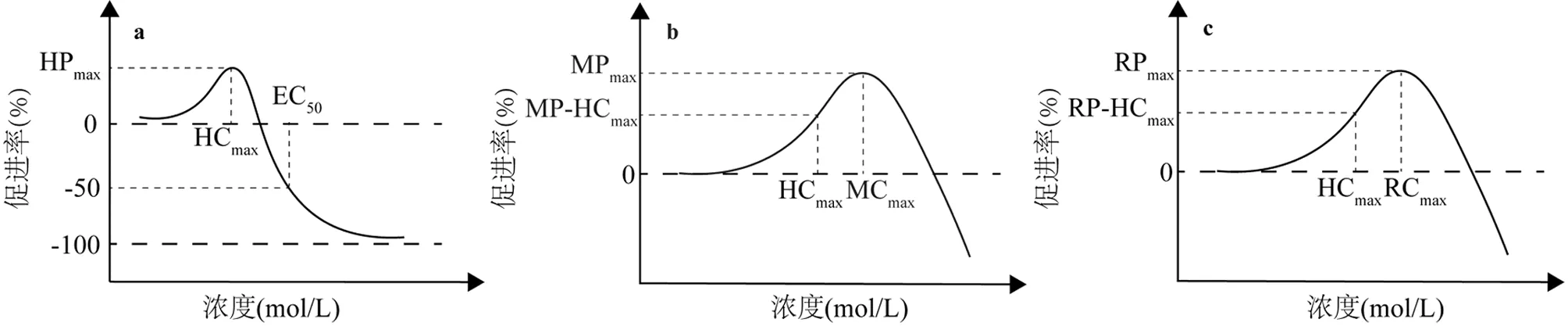
图1 抗生素对E. coli的毒性、突变与接合转移效应曲线
抗生素对的突变效应曲线如图1b所示.单一抗生素MPmax在图1b上对应浓度为MCmax.单一抗生素处于HCmax浓度时在图1b上对应的突变促进率为MP-HCmax.混合抗生素处于HCmix时在图1b上对应的突变促进率为MP-HCmix.
抗生素对的质粒接合转移效应曲线如图1c所示.单一抗生素RPmax图1c上对应的浓度为RCmax.单一抗生素处于HCmax浓度时在图1c对应的质粒接合转移促进率为RP-HCmax.混合抗生素处于HCmix时在图1c上对应的质粒接合转移促进率为RP-HCmix.
本文涉及到的生长、突变与接合转移指标参数及其具体释义在表4中列出.
1.6 联合作用判别
混合暴露实验中,由于TCs与SAs、SAPs作用通路不同,基于IA模型对混合抗生素的毒性、突变与接合转移效应的联合作用方式进行判别.
基于IA模型的判别方法,构造式(6)计算理论毒性效应HPmix-IA.

式中:HPmax(i)是第种抗生素单一暴露时产生的hormesis最大促进效应.将混合抗生素实际的HPmix与计算得到的HPmix-IA进行比较,当实际的HPmix小于HPmix-IA时,认为混合抗生素对的生长抑制起到协同作用;当实际的HPmix等于HPmix-IA时,认为混合抗生素对的生长抑制起到相加作用;当实际的HPmix大于HPmix-IA时,认为混合抗生素对的生长抑制起到拮抗作用.
相应地,构造式(7)计算理论突变效应MP- HCmix-IA.

式中:MP-HCmax(i)是第种抗生素单一暴露时在HCmax浓度处的突变频率促进率.将混合抗生素实际的MP-HCmix与计算得到的MP-HCmix-IA进行比较,当实际的MP-HCmix小于MP-HCmix-IA时,认为混合抗生素对的突变促进效应起到拮抗作用;当实际的MP-HCmix等于MP-HCmix-IA时,认为混合抗生素对的突变促进效应起到相加作用;当实际的MP-HCmix大于MP-HCmix-IA时,认为混合抗生素对的突变促进效应起到协同作用.
此外,构造式(8)计算理论接合转移效应RP- HCmix-IA.
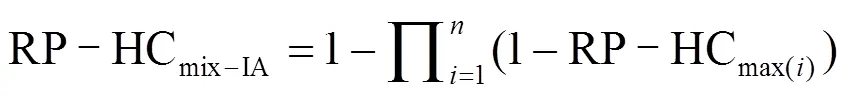
式中:RP-HCmax(i)是第种抗生素单一暴露时在HCmax浓度处的接合转移频率促进率.将混合抗生素实际的RP-HCmix与计算得到的RP-HCmix-IA进行比较,当实际的RP-HCmix小于RP-HCmix-IA时,认为混合抗生素对的质粒接合转移促进效应起到拮抗作用;当实际的RP-HCmix等于RP-HCmix-IA时,认为混合抗生素对的质粒接合转移促进效应起到相加作用;当实际的RP-HCmix大于RP-HCmix-IA时,认为混合抗生素对的质粒接合转移促进效应起到协同作用.

表4 E. coli生长与突变、接合转移指标参数释义
2 结果与讨论
2.1 单一抗生素对E. coli的生长及耐药性的影响
2.1.1 单一抗生素对生长的影响 单一抗生素对生长的EC50见表1,各抗生素毒性大小顺序为:SDX < OH < OMP < DVD < DH < TH < TMP.在实验浓度范围内,所有单一抗生素均对的生长表现出hormesis效应, hormesis效应的HPmax与HCmax数据见表5,其中HPmax的大小顺序为TH < DVD < TMP < OH < DH < OMP < SDX;HCmax的大小顺序为TH < TMP < DH < DVD < OH < SDX < OMP.依据现有文献报道,TCs、SAs、SAPs抑制生长的机制如下(图2a):TCs经由细胞外膜的亲水性孔,通过内膜转移系统进入细胞,与核糖体30S亚基16s RNA上的A位点结合,阻止氨基酰-tRNA进入核糖体,从而抑制肽链延长与蛋白质合成[10];SAs与对氨基苯甲酸(PABA)竞争结合二氢蝶酸合成酶(DHPS),阻碍二氢叶酸(DHF)的合成,进而抑制二氢叶酸还原酶(DHFR)与DHF的结合[11-12],阻碍四氢叶酸(THFA)的合成,最终抑制的生长[13];SAPs作用通路与SAs相似,SAPs通过与DHF竞争结合DHFR从而阻碍THFA合成,抑制生长[14-15].以往的研究表明,活性氧簇(ROS)在抗生素抑制细菌生长的过程中也起重要作用.ROS在低浓度抗生素下对细菌起到保护作用,而在高浓度抗生素下则协同抗生素抑菌[16].因此推测,当暴露于抗生素时,低剂量抗生素会引起ROS水平轻微升高,使ROS扮演信号分子的角色,调控的相关通路从而引起生长促进作用[17],最终表现出hormesis剂量-效应关系.
2.1.2 单一抗生素对突变的影响 单一抗生素均能促进的突变效应,其对应的MPmax与MCmax见表5.其中各抗生素MPmax的大小顺序为SDX < DH < TH < OH < TMP < OMP < DVD;MCmax的大小顺序为OH < TH < TMP < DH < OMP < SDX < DVD.基于以往的研究,推测TCs、SAs、SAPs促进突变频率的可能机制如下(图2b):对TCs的耐药性是由16S rRNA基因对应位点上AGA926-928发生单或多碱基突变引起,该区域在核糖体螺旋环31(helix31)处,是TCs与16S rRNA主要的结合位点[18]; helix31区域的核苷酸突变会影响16S rRNA与TCs的亲和力,从而使对TCs产生耐药性[19].SAs、SAPs阻碍THFA生物合成,从而减少嘌呤与嘧啶的合成,在DNA复制转录时,可能会导致碱基的错配,并最终促进的突变频率[20].此外,当TCs、SAPs、SAs浓度超过阈值时,抗生素对产生明显的生长毒性,致使细菌总数与突变体数量大幅下降,进而导致各抗生素对突变频率促进作用的下降[21].
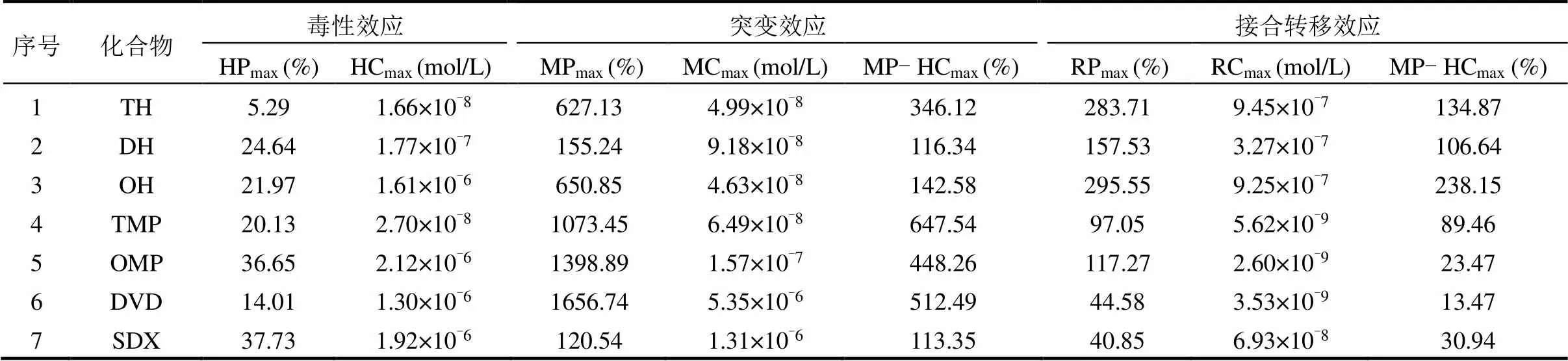
表5 单一抗生素毒性、突变与接合转移效应相关参数汇总
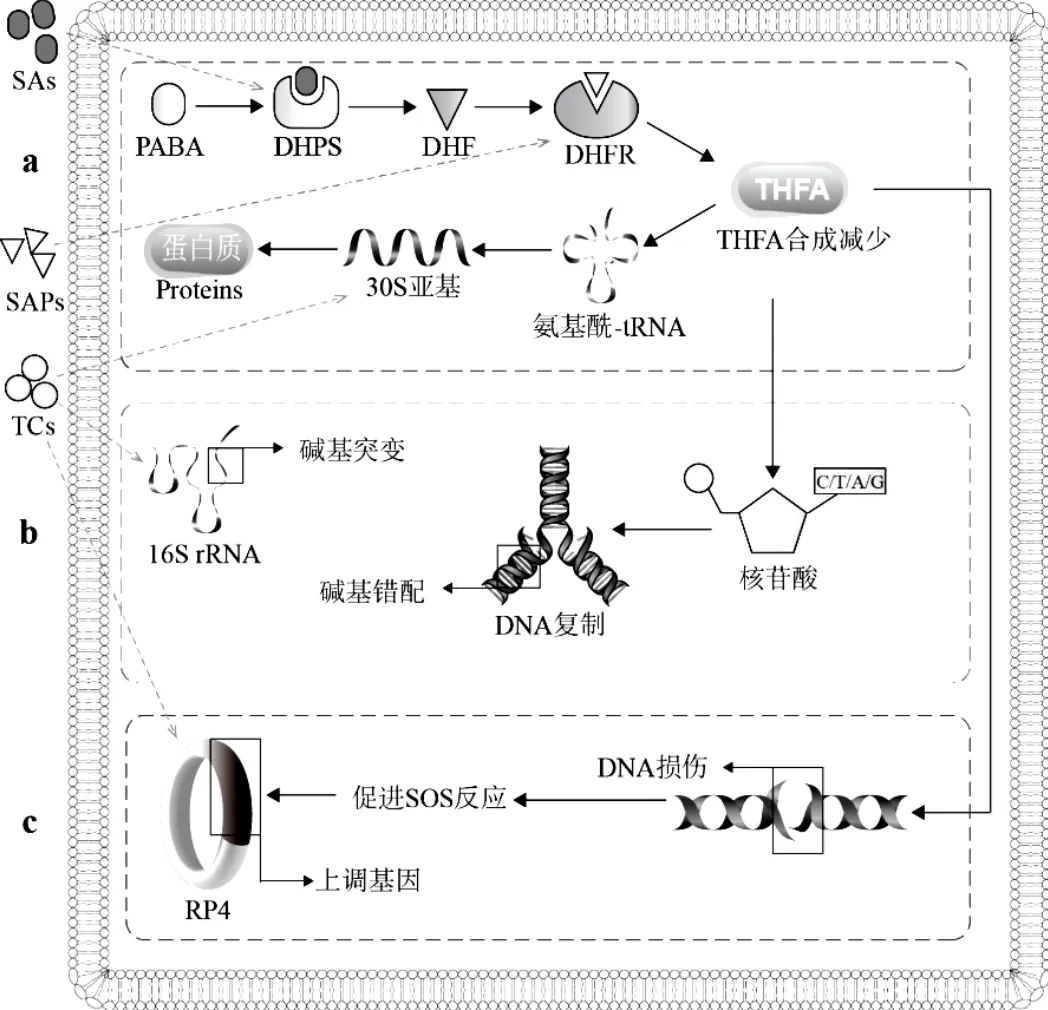
图2 SAs、SAPs、TCs的毒性、突变与接合转移作用通路 [15,20,24]
PABA,对氨基苯甲酸;DHPS,二氢蝶酸合成酶;DHF,二氢叶酸;DHFR,二氢叶酸还原酶;THFA,四氢叶酸
2.1.3 单一抗生素对的质粒接合转移的影响 单一抗生素均能促进的质粒接合转移效应,其对应的RPmax与RCmax见表5.其中,各抗生素RPmax的大小顺序为SDX < DVD < TMP < OMP < DH < TH < OH; RCmax的大小顺序为OMP < DVD < TMP < SDX < DH < OH < TH.TCs、SAPs、SAs促进的质粒接合转移频率的可能机制如下(图2c):暴露在TCs药物中含有RP4质粒的供体菌,可以通过特异性识别RP4质粒上的序列[22],促使闭合环状DNA双链进行解螺旋[23],质粒上的R,A,1和2等基因得以表达,从而促进质粒的接合转移.当TCs浓度达到一定阈值时,抗生素对供体菌与受体菌产生明显的生长毒性,致使接合子数量大幅下降,进而导致TCs对的质粒接合转移频率促进作用的下降.SAPs、SAs的暴露可引起体内DNA与蛋白质损伤,触发SOS反应[24-25]. SOS反应可能上调RP4质粒编码基因,以及控制供体和受体细菌细胞膜通透性的基因,从而促进RP4质粒的接合转移[26-27].当SAPs、SAs胁迫逐渐增大,SOS反应不能完全修复DNA与蛋白质损伤,可能影响供体与受体菌活性,甚至是接合子活性,导致SAPs、SAs对接合转移频率的促进作用逐渐降低[28-30].
2.2 二元及多元抗生素混合对E. coli的生长及耐药性的影响
2.2.1 混合抗生素对生长的影响 根据单一抗生素毒性实验结果,将3类抗生素按照等HCmax浓度比进行混合,毒性效应结果如图3所示.在图3中利用虚线表征当单一抗生素以浓度HCmax混合时的混合抗菌剂浓度HCmix,找到其对应的生长促进率HPmix,可以看出:TH&SDX组合的HCmix虚线落在无效应区间内,表示TH&SDX二元混合在HCmix对无毒性效应;其余5种组合的HCmix垂线均落在促进率为负的区间范围(即抑制区间范围)内,表示该5种组合在HCmix对的生长有抑制效应,且抑制率均达到80%以上,说明混合抗生素能够明显抑制的生长.为进一步明确混合抗生素对生长的联合作用方式,计算得到IA模型下混合抗生素的HPmix-IA,并与实际HPmix进行比较,相关数据见表6.二元混合抗生素和三元混合抗生素的HPmix均小于HPmix-IA,说明混合抗生素对的生长抑制起到协同作用.由单一抗生素对的毒性作用机制可以看出(图2),SAs、SAPs分别与DHPS、DHFR结合后,会抑制THFA的合成[31].当阻断THFA的合成时,氨基酸甲酰化受到阻碍,从而使核蛋白的合成受阻.同时,TCs与核糖体30S亚基16s RNA上A位点结合,阻止氨基酰-tRNA进入核糖体A位置,抑制肽链延长和蛋白质合成[14].因此,TCs与SAs、SAPs的联用能够共同抑制蛋白质合成,对的生长抑制起到协同作用.

表6 混合抗生素毒性、突变与接合转移效应的实际与计算促进率

图3 混合抗生素对E. coli生长的毒性效应
2.2.2 混合抗生素对突变的影响 如图4所示, 利用虚线表征当单一抗生素以浓度HCmax混合时的混合抗生素浓度HCmix,找到其对应的突变频率促进率MP-HCmix,可以看出:混合抗生素依然能够促进突变的产生.为进一步明确混合抗生素对突变的联合作用方式,计算得到IA模型下混合抗生素的MP-HCmix-IA,并与实际MP-HCmix进行比较,相关数据见表6.二元混合抗生素的MP- HCmix均大于MP-HCmix-IA,说明二元混合抗生素对促进突变频率起到协同作用;三元混合抗生素的MP-HCmix均小于MP-HCmix-IA,说明三元混合抗生素对促进的突变频率起到拮抗作用.由单一抗生素对的突变作用机制可以看出(图2),TCs造成16S rRNA上helix31区域的核苷酸突变;SAs、SAPs阻碍THFA生物合成,减少嘌呤与嘧啶合成,导致碱基错配.TCs&SAs及TCs&SAPs的二元协同是核苷酸突变与碱基错配共同作用的结果;SAs&SAPs的二元协同则是在于其均可阻碍THFA合成并最终导致碱基错配从而起到协同作用.然而,当加入第3种抗生素时,三元联合对的生长起到明显抑制作用,影响细菌核酸和氨基酸的生成,导致突变所需要的蛋白供给减少,进而抑制的突变频率上升.
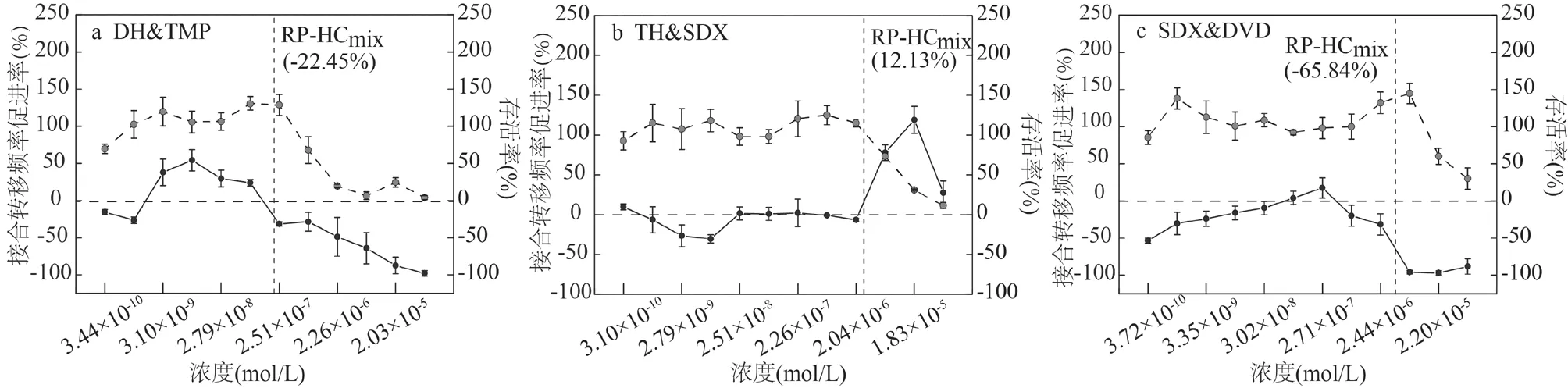
2.2.3 混合抗生素对的质粒接合转移的影响 如图5所示, 利用虚线表征当单一抗生素以浓度HCmax混合时的混合抗生素浓度HCmix,找到其对应的接合转移频率促进率RP-HCmix,可以看出: TH&SDX组合的HCmix虚线落在接合转移频率促进率促进区间,表示TH&SDX二元混合在此浓度下促进的质粒接合转移的发生;其余5种组合的HCmix垂线均落在接合转移频率促进率抑制区间,表示该5种组合此浓度下可抑制的质粒接合转移的发生.为进一步明确混合抗生素对的质粒接合转移的联合作用方式,计算得到IA模型下混合抗生素的RP-HCmix-IA,并与实际RP-HCmix进行比较,相关数据见表6.二元抗生素与三元抗生素的RP-HCmix均小于RP-HCmix-IA,说明以上组合对促进的质粒接合转移频率均起到拮抗作用.由单一抗生素对的质粒接合转移作用机制可以看出(图2):TCs使供体菌RP4质粒解螺旋,R,A,1和2基因表达,促进质粒接合转移;SAs、SAPs触发SOS反应,上调胞内基因,促进RP4质粒接合转移.当混合抗生素浓度处于HCmix时,低浓度抗生素联用使细菌总数减少,从而影响供体和受体细菌的活性,甚至是结合子的活性,使TCs解螺旋的RP4质粒减少,SOS上调胞内基因减少,从而使RP4质粒接合转移频率降低[32],表现为混合抗生素对的质粒接合转移促进的拮抗作用.
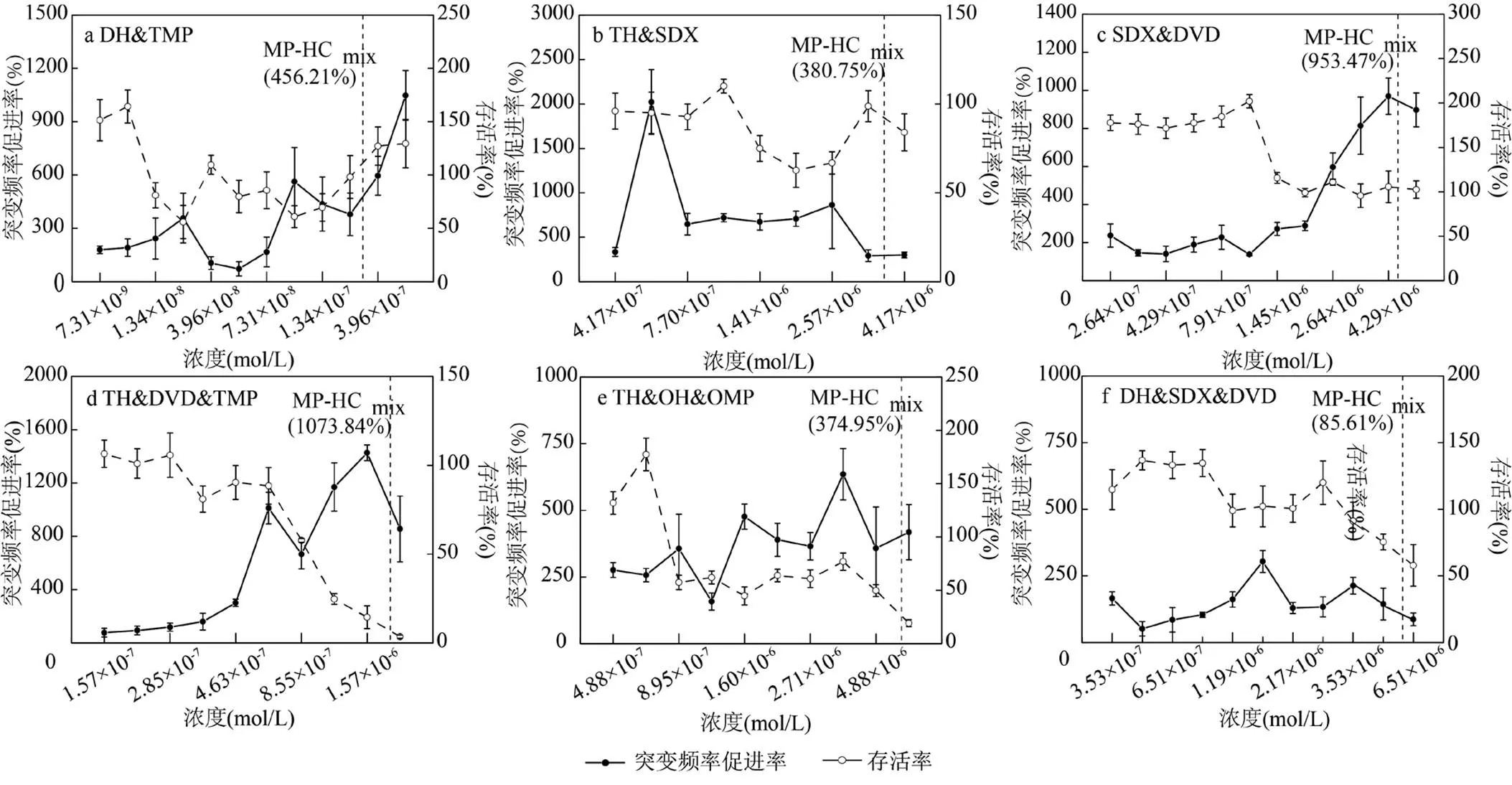
图4 混合抗生素对E. coli的突变效应
2.3 基于“鸡尾酒”暴露体系的混合抗生素的可能环境风险
二元混合抗生素处于HCmix浓度时,实际生长促进率HPmix小于计算生长促进率HPmix-IA,实际突变促进率MP-HCmix大于计算突变促进率MP- HCmix-IA,表示对的生长抑制与突变频率上升起到协同作用;实际质粒接合转移促进率RP-HCmix小于计算质粒接合转移促进率RP-HCmix-IA,表示对接合转移频率上升起到拮抗作用.三元混合抗生素处于HCmix浓度时,实际生长促进率HPmix小于计算生长促进率HPmix-IA,表示对的生长抑制起到协同作用;实际突变促进率MP-HCmix小于计算突变促进率MP-HCmix-IA,实际质粒接合转移促进率RP-HCmix小于计算质粒接合转移促进率RP- HCmix-IA,表示对的突变频率上升与接合转移频率上升起到拮抗作用.上述结果表明,二元与三元混合抗生素对细菌的毒性都增加,但是抗生素种类的增加能够抑制细菌耐药突变与质粒接合转移的发生.因此,由上述混合抗生素实际与计算的毒性、突变、RP4质粒接合转移效应大小比较,推测环境中基于“鸡尾酒”暴露体系的混合抗生素(3种或3种以上),会增加生物的毒性风险,但却能够降低耐药性风险.发现同时包含TC与SAP的三元混合方式较其他三元混合方式具有更明显的“毒性风险增加、细菌耐药性风险降低”的环境影响.
本文研究结果表明,一定程度上增加抗生素混合种类,会增加毒性风险,但却能够降低细菌耐药性风险.可能原因在于混合抗生素中抗生素种类的增加,使抗生素作用于更多靶蛋白,这些靶蛋白可能处于同一条信号通路,使得对这一通路的抑制性更好,共同抑制细菌生长,放大抑制效果,增加毒性协同,从而增加毒性风险.由于混合抗生素毒性增强,影响细菌核酸和氨基酸的生成,导致突变所需要的蛋白供给减少,进而抑制的突变频率上升.混合抗生素使细菌总数减少,影响供体和受体细菌的活性,甚至是结合子的活性,从而使RP4质粒接合转移数减少,抑制的质粒接合转移频率上升,减少细菌耐药性风险[33].
此外,本研究还说明混合抗生素的毒性风险与细菌耐药性风险似乎无法同时降低,毒性风险与细菌耐药性风险同时降低的目的可能需要通过新型抗生素的替代使用来达到.另一方面,本文发现即使是在抗生素多重混合使用的情况下,其对于生长、耐药突变与RP4质粒接合转移的促进作用区间始终存在,这表明环境中多种抗生素的残留带来的生态风险仍然不可忽视.在今后的研究中,需要关注环境中抗生素暴露的浓度和种类,以及抗生素与新型抗生素混合对细菌生长与耐药性的影响.
3 结论
3.1 在HCmix时二元混合虽能够有效抑制的生长,但却促进细菌耐药性的发生.
3.2 三元混合对生长抑制起到协同作用,对耐药突变频率上升与接合转移频率上升起到拮抗作用,其中TCs&SAPs&其他测试抗生素的三元混合方式具有更明显的“毒性风险增加、细菌耐药性风险降低”的环境效应.
3.3 基于“鸡尾酒疗法”使用的抗生素在进入环境后,其“鸡尾酒”暴露特征可能增加毒性风险,降低细菌耐药性风险,但整体上造成的生态风险仍不容忽视.
[1] Kovalakova P, Cizmas L, Mcdonald T J, et al. Occurrence and toxicity of antibiotics in the aquatic environment: A review [J]. Chemosphere, 2020,251:126351.
[2] 刘晓晖,卢少勇.大通湖表层水体中抗生素赋存特征与风险[J]. 中国环境科学, 2018,38(1):320-329.
Liu X H, Lu S Y. Occurrence and ecological risk of typical antibiotics in surface water of the Datong Lake, China [J]. China Environmental Science, 2018,38(1):320-329.
[3] Qiao M, Ying G G, Singer A C, et al. Review of antibiotic resistance in China and its environment [J]. Environment International, 2018,110: 160-172.
[4] 罗 晓,张文丽,袁立霞,等.纳污河流抗性基因和微生物群落相关性[J]. 中国环境科学, 2019,39(6):2606-2613.
Luo X, Zhang W L, Yuan L X, et al. Correlation between resistance genes and microbial community in polluted rivers [J]. China Environmental Science, 2019,39(6):2606-2613.
[5] Sun H, Calabrese E J, Zheng M, et al. A swinging seesaw as a novel model mechanism for time-dependent hormesis under dose-dependent stimulatory and inhibitory effects: A case study on the toxicity of antibacterial chemicals to Aliivibrio fischeri [J]. Chemosphere, 2018,205:15-23.
[6] Van Buul L W, Van Der Steen J T, Veenhuizen R B, et al.Antibiotic use and resistance in long-term care facilities [J]. Journal of the American Medical Directors Association, 2012,13(6):568.e1-568.e13.
[7] Xu K J, Song J, Zhao X M. The drug cocktail network [J]. Bmc Systems Biology, 2012,6:(Suppl 1):S5.
[8] Calabrese E J, Baldwin L A.Toxicology rethinks its central belief - Hormesis demands a reappraisal of the way risks are assessed [J]. Nature, 2003,421(6924):691-692.
[9] Cheng Y, Zeng H, Yu Y, et al. Study on the hormesis of sulfonamides on different gram-negative bacteria [J]. Asian Journal of Ecotoxicology, 2018,13(2):57-65.
[10] Gossen M, Freundlieb S, Bender G, et al. Transcriptional activation by tetracyclines in mammalian cells [J]. Science, 1995,268(5218): 1766-1769.
[11] Seydel J K. Sulfonamides, structure-activity relationship, and mode of action. Structural problems of the antibacterial action of 4- aminobenzoic acid (PABA) antagonists [J]. Journal of Pharmaceutical Sciences, 1968,57(9):1455-1478.
[12] Brown G M. The biosynthesis of folic acid. II. Inhibition by sulfonamides [J]. The Journal of biological chemistry, 1962,237:536-40.
[13] Skold O. Sulfonamide resistance: mechanisms and trends [J]. Drug Resistance Updates, 2000,3(3):155-160.
[14] Long X, Wang D, Lin Z, et al. The mixture toxicity of environmental contaminants containing sulfonamides and other antibiotics in Escherichia coli: Differences in both the special target proteins of individual chemicals and their effective combined concentration [J]. Chemosphere, 2016,158:193-203.
[15] 卢文强,孙昊宇,王雅娟,等.抗生素的胁迫与抗生素抗性基因产生与传播关系的研究[J]. 生态毒理学报, 2020,15(4):129-138.
Lu W Q, Sun H Y, Wang Y J, et al. The relationship of antibiotic stress with emergence and dissemination of antibiotic resistance genes [J]. Asian Journal of Ecotoxicology, 2020,15(4):129-138.
[16] Zhao X, Drlica K. Reactive oxygen species and the bacterial response to lethal stress [J]. Current Opinion in Microbiology, 2014,21:1-6.
[17] Tasho R P, Shin W T, Cho J Y. Acclimatization of Pisum sativum L., grown in soil contaminated with veterinary antibiotics, an attribute of dose hormetic response of root metabolites [J]. Science of the Total Environment, 2018,635:364-374.
[18] 文学琴,吴芳草,刘 芳,等.幽门螺杆菌四环素耐药与16SrDNA突变的关系[J]. 中国病原生物学杂志, 2019,14(4):433-438.
Wen X Q, Wu F C, Liu F, et al. Relationship betweenresistance to tetracycline and 16SrDNA mutation [J]. Journal of Pathogen Biology, 2019,14(4):433-438.
[19] Gerrits M M, De Zoete M R, Arents N L A, et al.16S rRNA mutation-mediated tetracycline resistance in Helicobacter pylori [J]. Antimicrobial Agents and Chemotherapy, 2002,46(9):2996-3000.
[20] Wang T, Liu Y, Wang D, et al. The joint effects of sulfonamides and quorum sensing inhibitors on Vibrio fischeri: Differences between the acute and chronic mixed toxicity mechanisms [J]. Journal of Hazardous Materials, 2016,310:56-67.
[21] Vedantam G, Guay G G, Austria N E, et al. Characterization of mutations contributing to sulfathiazole resistance in Escherichia coli [J]. Antimicrobial Agents and Chemotherapy, 1998,42(1):88-93.
[22] Parke D. Construction of mobilizable vectors derived from plasmids rp4, puc18 and puc19 [J]. Gene, 1990,93(1):135-137.
[23] Pansegrau W, Balzer D, Kruft V, et al. Invitro assembly of relaxosomes at the transfer origin of plasmid-RP4 [J]. Proceedings of the National Academy of Sciences of the United States of America, 1990,87(17):6555-6559.
[24] Sultan I, Rahman S, Jan A T, et al. Antibiotics, resistome and resistance mechanisms: A bacterial perspective [J]. Frontiers in Microbiology, 2018,9:2066.
[25] Andersson D I, Hughes D. Microbiological effects of sublethal levels of antibiotics [J]. Nature Reviews Microbiology, 2014,12(7):465-478.
[26] Wang Y, Lu J, Mao L, et al. Antiepileptic drug carbamazepine promotes horizontal transfer of plasmid-borne multi-antibiotic resistance genes within and across bacterial genera [J]. Isme Journal, 2019,13(2):509-522.
[27] Zhang Y, Gu A Z, Cen T, et al. Sub-inhibitory concentrations of heavy metals facilitate the horizontal transfer of plasmid-mediated antibiotic resistance genes in water environment [J]. Environmental Pollution, 2018,237:74-82.
[28] Bae Y S, Oh H, Rhee S G, et al. Regulation of reactive oxygen species generation in cell signaling [J]. Molecules and Cells, 2011,32(6): 491-509.
[29] Schieber M, Chandel N S. ROS function in redox signaling and oxidative stress [J]. Current Biology, 2014,24(10):453-462.
[30] 张崇淼,牛治瑶,王 真,等.氯消毒对产ESBLs菌β-内酰胺酶类抗性基因接合转移的抑制及作用机制研究[J]. 中国环境科学, 2021,41(10):4763-4769.
Zhang C M, Niu Z Y, Wang Z, et al. Inhibition and associated mechanism of conjugative transfer of β-lactamase resistance genes in ESBLs producing bacteria by chlorine disinfection [J]. China Environmental Science, 2021,41(10):4763-4769.
[31] Li X F, Shi J Y, Sun H Y, et al. Hormetic dose-dependent response about typical antibiotics and their mixtures on plasmid conjugative transfer of Escherichia coli and its relationship with toxic effects on growth [J]. Ecotoxicology and Environmental Safety, 2020,205:111300.
[32] 李旭飞,巫晓丹,孙昊宇,等.抗生素和纳米银对大肠杆菌耐药性的联合效应[J]. 中国环境科学, 2020,40(11):5045-5054.
Li X F, Wu X D, Sun H Y, et al. Joint effects of antibiotics and silver nanoparticles on resistance of Escherichia coli [J]. China Environmental Science, 2020,40(11):5045-5054.
[33] Li X F, Liu Y Y, Wang Y J, et al. Resistance risk induced by quorum sensing inhibitors and their combined use with antibiotics: Mechanism and its relationship with toxicity [J]. Chemosphere, 2021,265:129153.
The effects of antibiotic mixtures on bacterial resistance based on “cocktail” exposure.
HAN Bao-cang1, CHEN Zhi-bin1, ZHANG Yue-heng1, LIN Zhi-fen1, SUN Hao-yu2*
(1.College of Environmental Science and Engineering, Tongji University, Shanghai 200092, China;2.Key Laboratory of Organic Compound Pollution Control Engineering (MOE), School of Environmental and Chemical Engineering, Shanghai University, Shanghai 200444, China)., 2022,42(1):434~443
Three kinds of commonly used antibiotics, i.e., tetracyclines, sulfonamides, and sulfonamide potentiators, were selected as representatives to obtain their concentration for the hormetic maximum promoting effect on() (HCmax). The binary and ternary mixtures of test antibiotics were designed based on equal HCmaxrations, and their combined effects on bacterial growth and resistance (mutation and plasmid conjugation) ofwere explored. The results showed binary mixtures synergistically inhibited the growth and conjugation frequency, but synergistically stimulated the mutation frequency. The combined effects of ternary mixtures on toxicity and resistance were all synergistic inhibition. Therefore, it could be speculated that binary mixtures might increase the risk of toxicity and resistance, while ternary mixtures might result in an increased toxicity risk but a decreased resistance risk.
antibiotic mixtures;bacterial resistance;hormesis;mutation;conjugation transfer
X171.5
A
1000-6923(2022)01-0434-10
韩宝苍(1997-),男,山东临沂人,同济大学博士研究生,主要从事抗生素环境毒理研究.发表论文2篇.
2021-06-04
国家自然科学基金资助项目(22006116,21777123);博士后创新人才支持计划(BX20190247);中国博士后科学基金(2019M661624);上海“超级博士后”激励计划(2019194)
* 责任作者, 副研究员, sunhaoyu2021@shu.edu.cn
