基于eDNA技术的长江上游珍稀特有鱼类国家级自然保护区重庆段鱼类多样性研究
2022-01-20刘智皓陈启亮李英文沈彦君
王 梦 杨 鑫 王 维 段 聪 刘智皓 陈启亮 李英文* 沈彦君*
(1. 重庆师范大学重庆市动物生物学重点实验室, 重庆 401331; 2. 重庆市珍稀特有鱼类国家级自然保护区管理处, 重庆 402260)
环境DNA(Environmental DNA, eDNA)是指从生物体的皮肤、唾液和分泌物等释放到环境中的混合DNA分子[1,2], 其广泛存在于水体、土壤、沉积物和空气等环境介质中。eDNA metabarcoding通常指直接提取环境样本中的总DNA, 并使用特异或通用引物进行聚合酶链式反应(Polymerase chain reaction, PCR), 通过测序、生物信息学分析来识别环境中目标物种序列的一种新兴的生物多样性检测手段[3,4]。eDNA技术最早起源于环境微生物学领域[5], 在2000年之后得到广泛认可和应用[6]。Ficetola等[7]在2008年首次利用eDNA技术监测出池塘中存在入侵物种美国牛蛙(Rana catesbeiana), 余玥[8]基于eDNA技术成功检测出长江中下游干流中的15种鱼类, Zhang等[9]利用eDNA技术研究了长江口及其邻近水域鱼类群落结构的季节变化特征。总之, eDNA技术的出现为水生生态系统生物多样性的研究开辟了一条新道路, 整个过程无需采集目标生物个体, 弥补了传统方法调查成本大、缺乏标准化流程和对生态系统干扰高等的不足, 在水生生物多样性评估方面具有极大的应用潜能[10]。
长江上游珍稀特有鱼类国家级自然保护区(下简称“保护区”)是长江上游最大的水生生物自然保护区, 涉及三省一市(云南、贵州、四川和重庆), 其流域复杂的地形地貌和发达的河网水系等孕育了丰富的鱼类种类[11—14], 是我国重要的淡水鱼类资源宝库[15]。其中重庆段(28°55′—29°20′N, 105°53′—106°24′E)起于重庆市江津区石蟆镇羊石街道, 止于珞璜镇地维大桥, 全长118.8 km, 分为核心区、缓冲区和实验区3个部分, 其主要的保护对象为达氏鲟(Acipenser dabryanus)和胭脂鱼(Myxocyprinus asiaticus)等珍稀特有鱼类[16]。重庆段是保护区最下游的江段, 是关系到上游保护区内珍稀特有鱼类生存和三峡库区渔业资源增殖的重要通道, 对维系我国淡水鱼类种质资源、鱼类生物多样性及三峡库区水域生态系统稳定性具有重要意义[17]。然而,近年来, 由于过度捕捞、水体污染、鱼类繁殖场所的破坏和航道整治工程建设等的影响[18], 长江上游的鱼类资源呈现出明显下降的趋势, 在一定程度上打破了原有的水生态系统平衡。
当前, 在长江大保护的背景下, 为了修复长江流域生态, 保护鱼类自然资源, 迫切需要了解长江流域鱼类资源的现状及存在的问题, 为渔业管理以及生态保护政策的制定与实施提供科学依据。但传统捕捞调查方法费时费力, 不仅对鱼类具有伤害性, 还必须依赖鱼类分类学家对采集样本进行鉴定[19],且目前正是“十年禁渔”时期, 传统调查方法工作的开展受到很大程度的限制。因此, 本文首次运用eDNA技术对保护区重庆段鱼类多样性进行初步评估, 探索适用于长江鱼类资源监测和保护的新方法[20],并为后期长江“十年禁渔”效果评估提供一定的基础资料。
1 材料与方法
1.1 采样时间及采样点的设置
本次调查于2021年3月28日进行, 为提高环境DNA检测成功率, 本研究结合地理环境和有关鱼类“三场”分布的历史资料, 在该江段共布设了三抛河(SPH)、松溉(SJ)、高占滩(GZT)、丁家沱(DJT)、綦江口(QJK)和马夫沱(MFT)6个采样点, 各采样点具体分布见图 1。
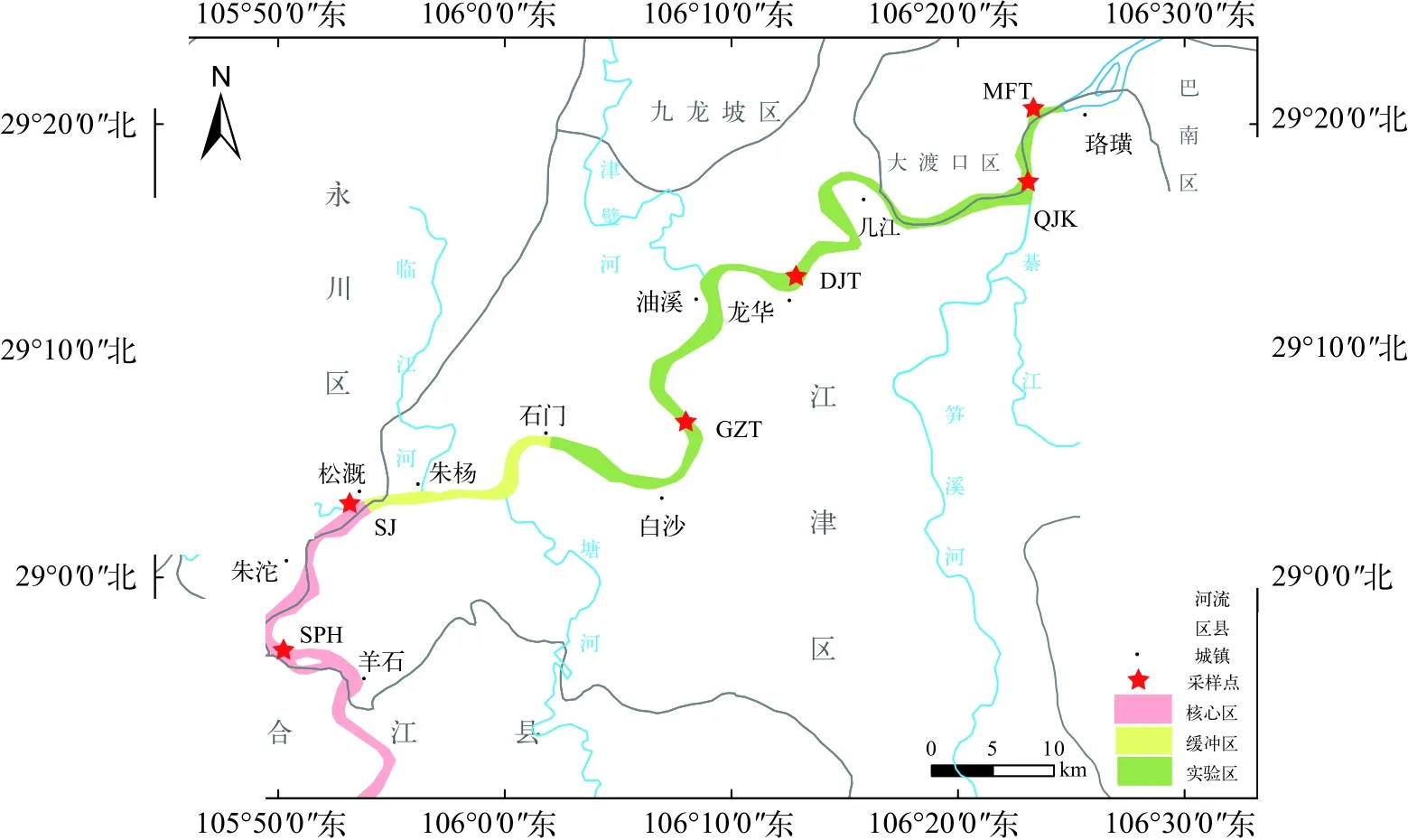
图 1 样品采样点分布图Fig. 1 Information of sampling locations
1.2 水样的采集与处理
当前正处于长江“十年禁渔”期, 本次调查经过相关渔业主管部门的批准后进行。到达指定采样点后, 在每个取样位置记录水体温度、深度和地理坐标, 待调查船关闭发动机15min后, 用采水器从表层水体采集2 L水样放置于聚乙烯瓶中[21]。各采样点采集3瓶平行水样。采水器及采样瓶在使用前均用10%漂白粉溶液进行消毒, 且在采集过程中, 采集人员每次取样后需更换一次性手套[22]。收集到的水样储存于冷藏条件下, 运送回实验室, 于24h内使用真空泵抽滤到孔径为0.45 μm的混合纤维素滤膜(Whatman, UK)上。若水样较浑浊, 则先将水样用无菌的医用纱布进行粗过滤后再进行抽滤[19]。每份样品抽滤前后, 均对器材进行消毒以清除装置中残余的DNA, 避免样品间的交叉污染。且在每次抽滤时, 还需设置1个阴性对照来评估是否存在外源DNA污染。最后将滤膜置于4.5 mL无酶冻存管(Thermo)中于-80℃冷冻保存, 直至DNA提取。
1.3 水样总DNA提取
使用PowerWater DNA Isolation Kits(Qiagen公司)试剂盒提取滤膜中捕获的水样总DNA, 为了减少DNA降解, 用无菌TE溶液代替PW6(无菌洗脱液)进行最后一步洗脱。最后用1%的琼脂糖凝胶电泳检测提取到的基因组DNA质量[23]。每份样品独立提取, 为评估提取过程是否存在污染, 需设置空白滤膜作为阴性对照。最后将DNA样品置于-20℃下冰箱保存, 直至PCR扩增。
1.4 PCR扩增及高通量测序
本研究以鱼类多样性监测中使用最广泛的分子标记—线粒体基因12S rRNA为靶点, 选取Valentini 等[24]开发的鱼类特异引物“Teleo” (teleo-F: 5′-ACACCGCCCGTCACTCT-3′; teleo-R: 5′-CT TCCGGTACACTTACCATG-3′)进行扩增, 并添加样本特异性的 Barcode 序列。PCR采用TransStart Fastpfu DNA Polymerase, 扩增为20 μL反应体系: 4 μL 5×FastPfu Buffer、2 μL dNTPs、0.4 μL FastPfu Polymerase、2—5 μL模板DNA(10 ng/μL)和上下游引物各0.8 μL(10 μmol/L), 最后用ddH2O将体系补至20 μL。PCR反应程序: 预变性95℃ 5min, 变性95℃ 30s, 退火55℃ 30s, 延伸72℃ 45s, 终延伸72℃10min, 于10℃保存(变性-延伸-退火为27个循环)。为评估P C R扩增中是否出现污染, 需要使用ddH2O为模板进行PCR阴性对照。进行PCR扩增时, 每个样本进行3次重复, 并将PCR产物混合。经2%琼脂糖凝胶电泳检测PCR产物。在本研究中,18个样品均获得了可检测的PCR产物。PCR产物胶回收后送至上海凌恩生物科技有限公司通过Illumina NovaSeq 6000测序平台进行高通量测序。
1.5 数据处理
数据质量控制 Illumina PE250测序序列首先根据barcode得到所有样品的有效序列; 然后对reads的质量进行质控过滤; 再根据PE reads之间的overlap关系, 将成对的reads拼接(merge)成一条序列; 最后按照barcode和引物序列拆分得到每个样本的优质序列, 并在过程中根据正反barcode和引物方向校正序列方向。
OTU聚类与注释 按序列相似性≥97%进行OTU聚类分析后将OTU代表序列与MitoFish(http://mitofish.aori.u-tokyo.ac.jp/)和NCBI(https://www.ncbi.nlm.nih.gov/)数据库进行比对、分类注释, 并得到相应的OTU丰度表。在各采样点的平行样品中未匹配到的物种序列被剔除, 匹配到物种的序列数按均值处理。
统计分析 本研究以OTU聚类分析结果为基础,进行了物种组成分析、Alpha多样性和Beta多样性分析(包括未鉴定到种水平的3属)。(1)物种组成分析。去除比对到非鱼类(如细菌、鸟类、两栖类和哺乳类等)的数据信息后, 筛选出比对至鱼类且identity值≥97%,E-value≤10-5的OTU, 再将比对至同一物种的OTU进行合并。若有OTU无法比对至种水平, 则向上一级如属、科等进行统计。在Excel中统计各个样本中每种鱼的有效序列数占比并参考Fishbase数据库和《中国内陆鱼类物种与分布》注释、完善鱼类分类学信息。最后通过R语言绘制各个采样点鱼类组成柱状图等。(2)Alpha多样性分析。本文选取Chao1指数(Chao1 index)、香农指数(Shannon index)、辛普森指数(Gini-Simpsonindex)及覆盖度(Coverage)指数, 分别反映群落丰富度(Community richness)、群落多样性(Community diversity)及群落覆盖度(Community coverage)。(3)Beta多样性。本研究基于Bray-Curtis距离矩阵进行PCoA分析, 探索不同分组样本间的群落组成差异性或相似性。
2 结果
2.1 鱼类物种组成
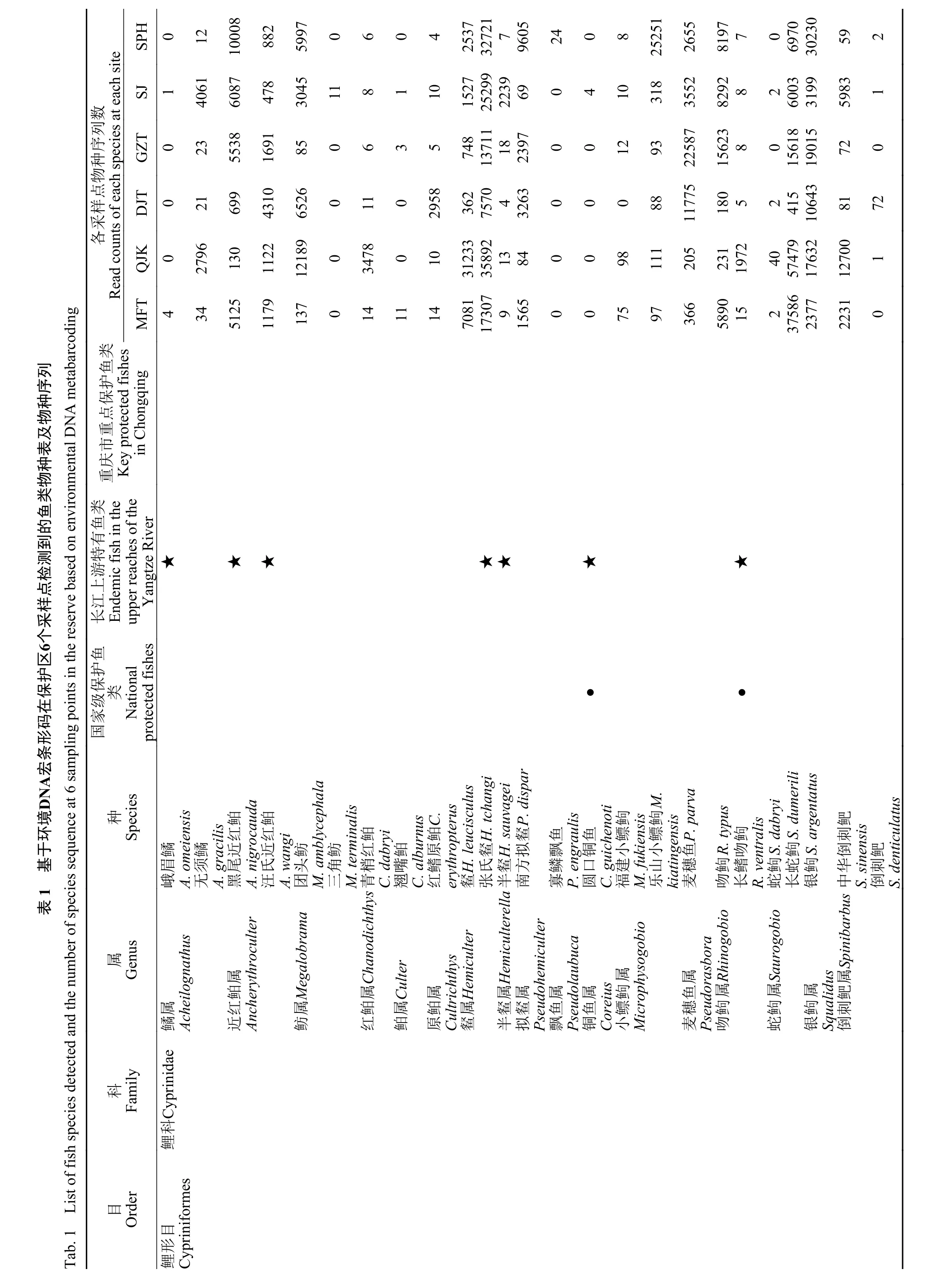
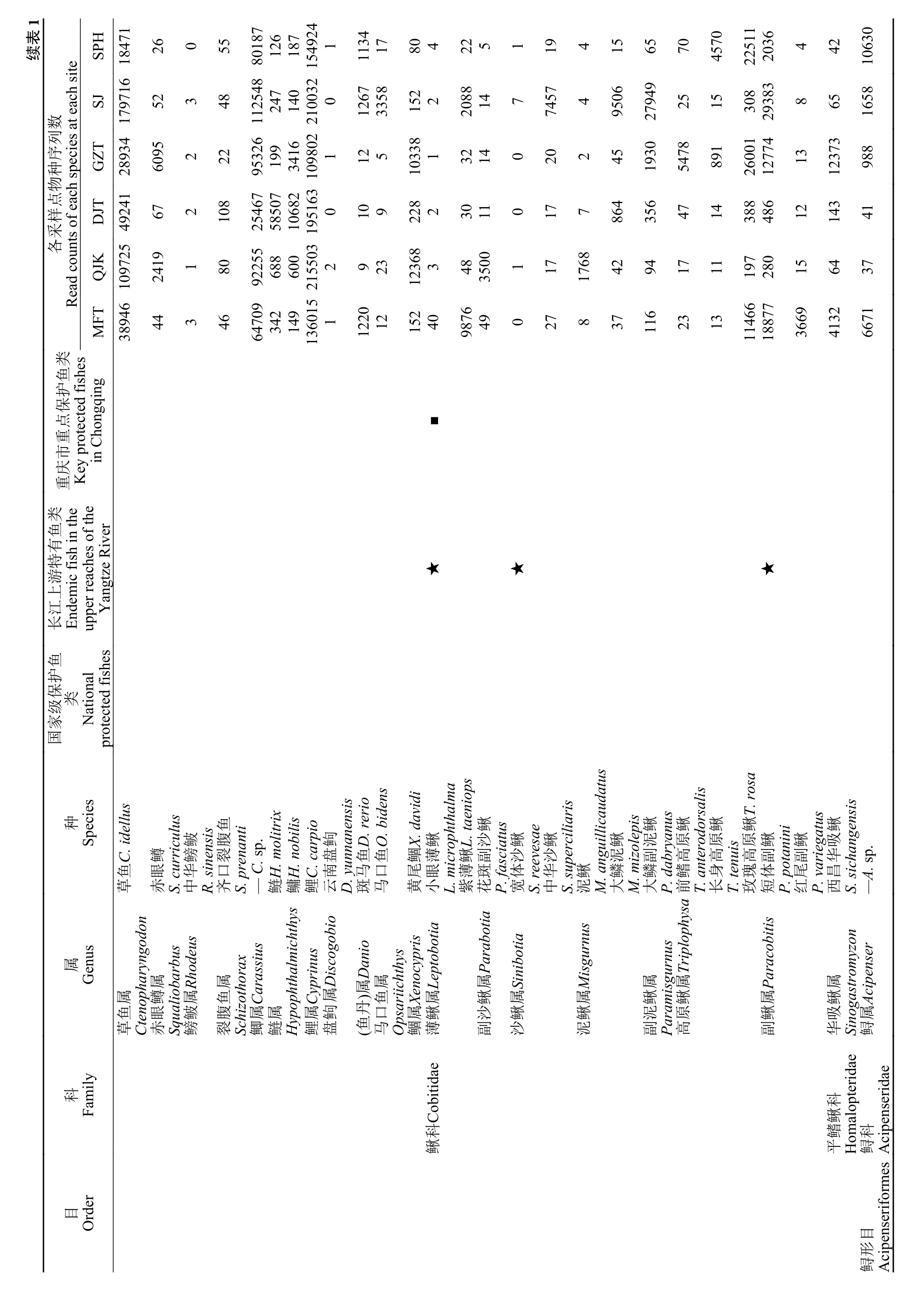
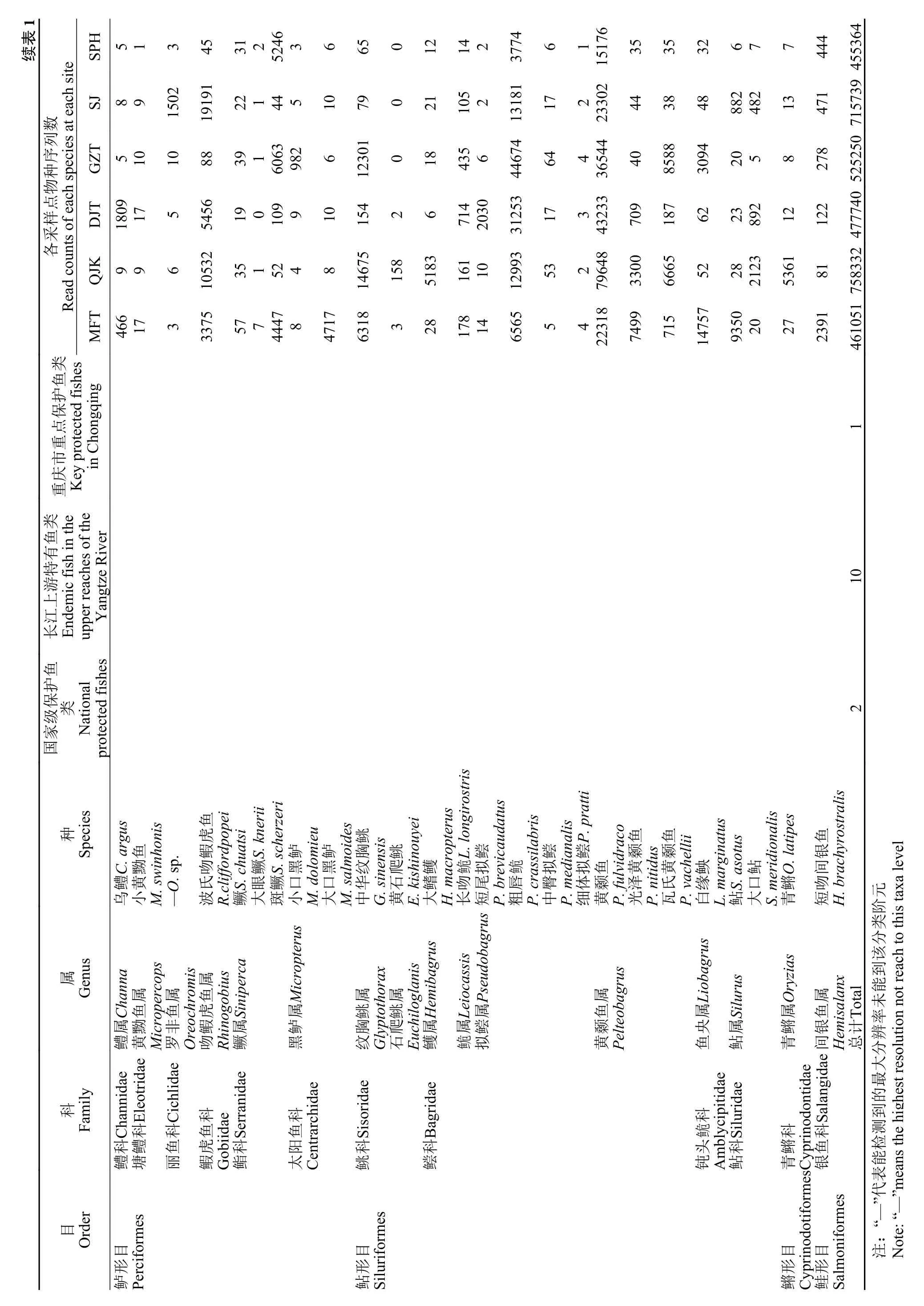
从6个采样点共获得1565条OTU, 不同采样点OTU数量差异见图 2。由图 2可看出, 6个采样点有724个共有的OTU。其中QJK的OTU数量最多, 共1252条; DJT最少, 仅有1054条。对OTU进行注释后, 本次调查共检测出鱼类74种(不包括未鉴定到种水平的3属; 表 1), 隶属6目16科52属, 含有圆口铜鱼(Coreiusguichenoti)和长鳍吻(Rhinogobioventralis)两种国家级保护鱼类; 长江上游特有鱼类10种;重庆市重点保护鱼类1种。鲤形目(Cypriniformes)的鲤科鱼类最多, 总计38种, 占鱼类物种总数的48.72%; 而鲟形目(Acipenseriformes)、鳉形目(Cyprinodotiformes)和鲑形目(Salmoniformes)的种类最少, 均仅含1种。根据序列比对结果, 本次调查中的鲫属(Carassius)、罗非鱼属(Oreochromi)及鲟属(Acipenser)未鉴定到种水平。
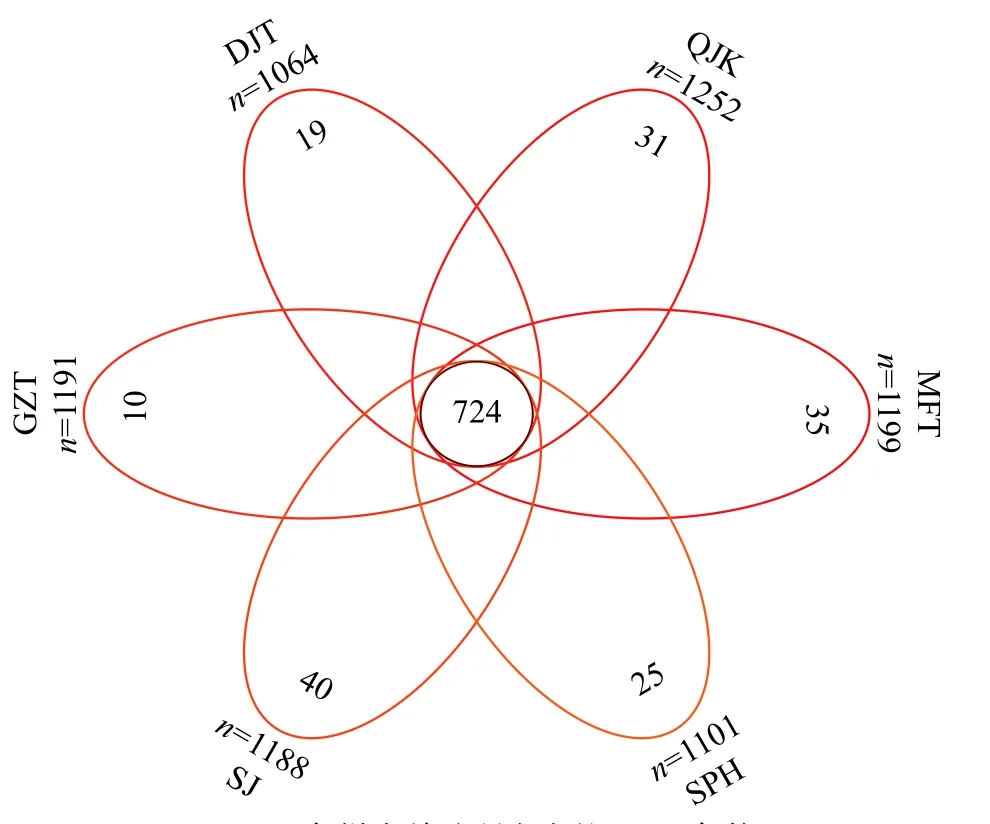
图 2 各采样点的韦恩图展示Fig. 2 Venn diagram display of each sampling point
?
?
?
基于各采样点的序列丰度, 在属水平的物种组成情况见图 3。由图 3可知, 鲤属(Cyprinus)、鲫属(Carassius)、草鱼属(Ctenopharyngodon)和黄颡鱼属(Tachysurus)等在各采样点均被监测到并显示出相对较高的序列丰度。此外, 由表 1的物种序列数可知, 除了峨眉鱊(Acheilognathus omeiensis)、倒刺鲃(Spinibarbu denticulatus)、圆口铜鱼(C. guichenoti)和宽体沙鳅(Sinibotia reevesae)等12种鱼类仅在个别采样点中检测到, 其余鱼类在6个采样点中均被检测到, 但在鱼类序列数上呈现出了明显的差异,例如大口黑鲈(Micropterus salmoides)、赤眼鳟(Squaliobarbus curriculus)和半䱗(Hemiculterella sauvagei)等在各采样点的序列数相差较大。
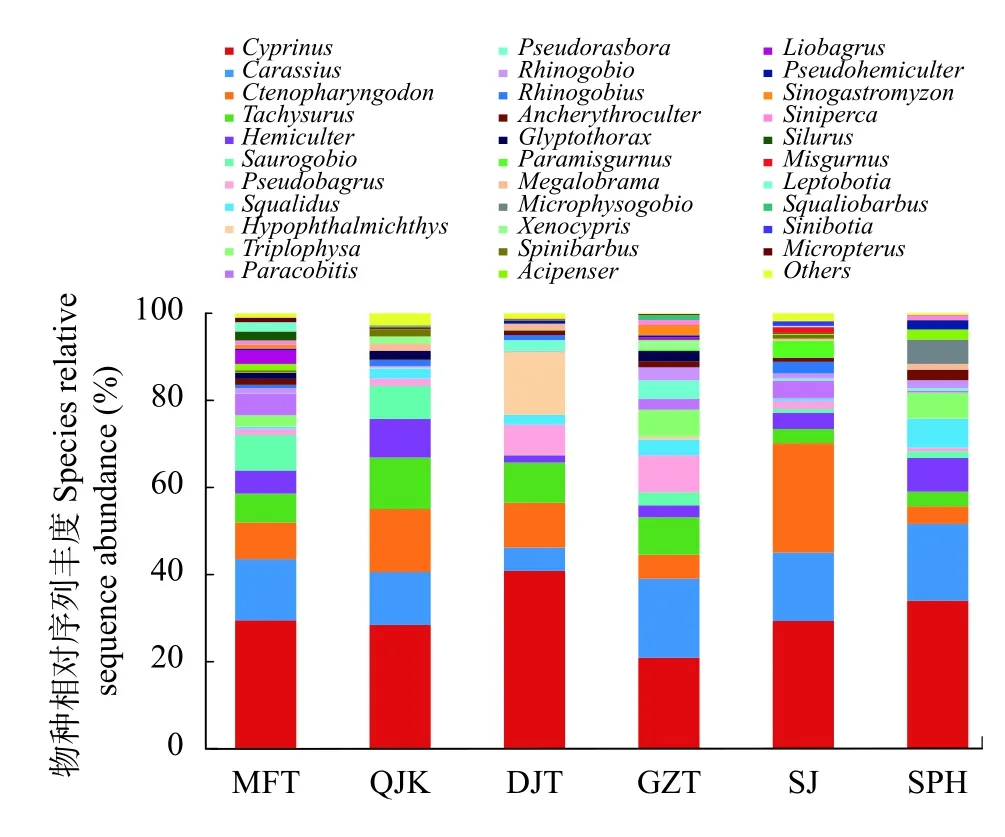
图 3 各采样点的鱼类物种组成Fig. 3 The composition of fish species at each sampling site
2.2 Alpha多样性分析
本研究通过计算Chao1、Shannon和Simpson多样性指数及覆盖度Coverage来对该保护区鱼类群落进行Alpha多样性分析, 各样本的Alpha多样性指数见表 2。由表 2可知, 各采样点Alpha多样性的各项指数较为均匀。各个样本的Coverage值范围为0.9996—0.9998, 表明测序基本覆盖到了全部的OTU数据, 能反映样本的真实情况。
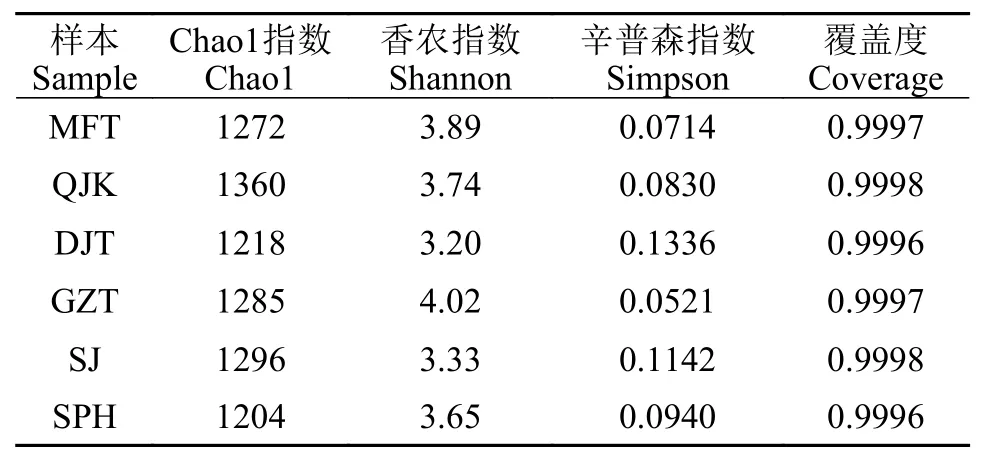
表 2 各样本的Alpha多样性指数统计Tab. 2 Alpha diversity index of each sample
2.3 Beta多样性分析
本研究基于各采样点的序列丰度, 采用Bray-Curtis距离矩阵进行PCoA分析( 图 4), 对比样本间鱼类种类组成的相似性或差异性。由图 4可知, 采样点MFT与SPH及QJK与SJ具有相似的鱼类多样性, 而DJT和GZT具有不同于其他采样点的鱼类组成。
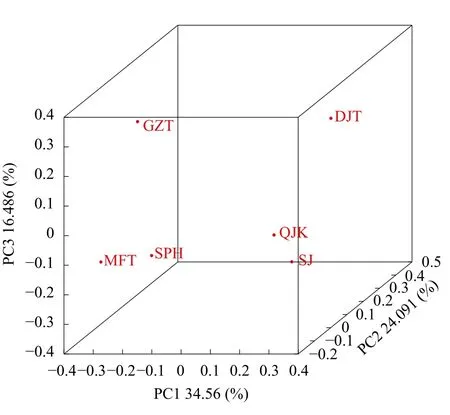
图 4 基于Bray-Curtis距离矩阵的鱼类的主坐标分析(PCoA)Fig. 4 Analysis of fish principal coordinates based on Bray-Curtis distance matrix
3 讨论
3.1 基于环境DNA的长江鱼类组成
本研究首次运用eDNA技术分析了长江上游珍稀特有鱼类国家级自然保护区重庆段鱼类多样性,共检测出鱼类74种(不包括未鉴定到种水平的3属),其中鲫属(Carassius)、罗非鱼属(Oreochromi)及鲟属(Acipenser)未鉴定到种水平。罗非鱼属(Oreochromi)未鉴定到种是因为数据库中没有相应的参考序列; 而鲫属(Carassius)及鲟属(Acipenser)只能鉴定到属, 其原因是比对到不同的物种但有相同的比对质量。这可能与扩增片段较短及数据库所含物种信息不完整有一定的关系。
根据相关资料记载, 近年来通过刺网和撒网等传统方法监测到有103种渔获物(表 3), 其中吻鮈属(Rhinogobio)、铜鱼属(Coreius)、黄颡鱼属(Pelteobagrus)及蛇鮈属(Saurogobio)等为主要优势种群[25—30]。在本次调查中, 鲤属(Cyprinus)、鲫属(Carassius)和草鱼属(Ctenopharyngodon)鱼类的丰度和占到50%以上, 这可能与采样时间和地点有较大关系。为提高环境DNA检测成功率, 本研究布设的6个采样点均为历史资料记载的产卵场。其次, 本次调查于2021年3月28日进行, 这正处于鲤鱼等鱼类的繁殖季节。因此造成鲤属(Cyprinus)、鲫属(Carassius)和草鱼属(Ctenopharyngodon)鱼类的丰度较高。除上述3个属以外的鱼类与传统方法监测的结果相比,鱼类结构组成较为一致, 蛇鮈属(Saurogobio)、黄颡鱼属(Pelteobagrus) 、䱗属(Hemiculter)及吻鮈属(Rhinogobio)等鱼类为当前优势种。这也凸显了eDNA技术作为一种新兴的生物多样性调查方法,在水生生物监测和保护的应用中具有检测灵敏度高等特点[31,32]。

表 3 基于传统方法在保护区监测到的渔获物种类Tab. 3 Types of catches monitored in protected areas based on traditional methods

续表 3

续表 3
此外, 本研究还检测出短吻间银鱼(H. brachyrostralis)、罗非鱼(O.sp.)、大口黑鲈(M. dolomieu)和团头鲂(M. amblycephala)等7个外来物种, 这可能是该保护区流域周边的渔业养殖场缺乏必要的物种隔离设施及防逃手段, 造成上述外来物种的逃逸而进入长江[33]。严太明等[34]的相关研究表明, 外来物种的引入可能会给土著鱼类和生态系统带来巨大的灾难。因此, 对于保护区上游周边的养殖场应当建造物种隔离及防逃设施, 防止外来物种的入侵对保护区土著鱼类资源造成一定的生态胁迫。
3.2 基于环境DNA的长江鱼类多样性分析
表 3反映了鱼类群落丰度的Alpha多样性指数[35]。本研究中的Chao1指数范围为1204—1360, 其中QJK采样点值最大, 表明该采样点的群落丰富度最高; 而SPH采样点指数最小, 表明群落丰富度最低。这可能与各采样点的地理位置和环境有密切关系。QJK位于支流(綦江)与干流(长江)的交汇口,其水域的复杂性可能使该处的群落丰富度最高; 而SPH采样点位于赵家中坝内浩附近, 内浩进、出口水流紊乱, 激流回漩, 可能在一定程度上导致了鱼类分布量相对较少, 从而使群落丰富度较低。其他样点的Chao1指数较为均匀, 表明该保护区鱼类生态结构较为稳定[36]。Shannon和Simpson指数均揭示了鱼类的多样性水平[37]。在本研究中, Shannon和Simpson指数表明GZT鱼类多样性最高, 这可能与小口黑鲈(M. dolomieu)、西昌华吸鳅(S. sichangensis)和赤眼鳟(S. curriculus)等物种的序列丰度较高有关。而DJT鱼类多样性最低, 这可能与该样点的DNA提取浓度偏低有关。不同指数评估鱼类多样性的侧重点具有一定差异, 群落丰富度指数并不能完全衡量群落多样性高低[38]。如SJ位点的Simpson多样性指数较低但群落丰富度较高, 推测可能与样品中少数鱼类物种序列丰度过高有关。
基于Bray-Curtis距离矩阵的Beta多样性分析显示DJT和GZT两个采样点与其他样点的鱼类组成相似度较低, 共有物种较少(图 4)。这可能是受到了自然因素和人为因素的共同影响。DJT和GZT分别位于保护区的高占滩和丁家沱附近, 其周围人为活动频繁。如耕种养殖及浣洗衣物等活动会改变河流中氮磷含量, 从而影响到鱼类营养物质的获取及鱼类多样性[39,40]。从自然因素来看, 刘军等[41]探究了长江上游主要河流的鱼类分布与流域面积、含沙量和平均径流量等自然地理、水文变量有着密切关系。其中以含沙量为代表的悬浮物浓度会对鱼类的生存和生长产生影响, 从而改变鱼类多样性[42,43]。DJT和GZT两个采样点较其他采样点水样更为浑浊, 所以与其他样点的鱼类组成相比, 在一定程度上表现出了差异性。而其他各样点共有物种较为集中, 这可能与采样点之间距离较近, 鱼类可以进行频繁的交流有关。
3.3 影响eDNA检测的因素
由于长江流域环境的复杂性, eDNA的产生及降解速率易受水温、pH、流速、紫外线和水域底质等诸多环境因素的影响[44,45]。本次调查共设置6个采样点, 18个样品。其中仅DJT采样点的PCR扩增结果不理想, 在3个平行样品中, 一个样品PCR产物浓度偏低, 其余2个样品PCR产物目的条带太弱或未检测到。而其余5个采样点PCR扩增结果达到A, 即产物目的条带大小正确, 浓度合适, 符合测序要求。本研究认为造成这种差异性可能与该采样点的环境有密切关系。eDNA得率除了水样本身所包含的生物物质多少之外, 还受到单张滤膜抽滤体积、水样泥沙含量和抽提操作等多种因素影响[46]。DJT采样点位于保护区重庆段丁家沱附近, 该处水样浑浊, 泥沙含量较高, 在进行eDNA抽提时, 这些泥沙导致抽滤速度慢、耗时长。这就有可能使部分DNA片段在抽滤时发生了降解, 从而影响了DNA的提取结果和浓度。此外, 泥沙可能会吸附一些脱落游离的DNA, 这也可能会对DNA抽提效率产生一定的影响[23]。因此, 在进行采样时应尽量选择泥沙较少的区域, 或者在过滤时用一次性的医用纱布进行粗过滤后再对水样进行过滤[19]。此外, 还可以选用孔径较大的滤膜进行预过滤后再将滤液重新过滤到孔径较小的滤膜上。
3.4 长江流域鱼类资源保护建议
在本次调查中, 6个采样点共监测到的鱼类种类为74种(不包括未鉴定到种水平的3属), 其中包含小眼薄鳅(L. microphthalma)、圆口铜鱼(C. guichenoti)、汪氏近红鲌(A. wangi)和短体副鳅(P.potanini)等长江上游特有鱼类。然而, 环境DNA未检测到有历史资料记载的达氏鲟(A. dabryanus)和岩原鲤(P. rabaudi)等珍稀特有鱼类, 可能是因为上述长江特有鱼类在该江段的资源量较为稀少, 从而在环境中缺少对应的物种DNA。如不采取有效措施, 可能会导致这些长江鱼类的资源量进一步下降。而鱼类作为水生态系统的重要组成部分, 其物种多样性及组成结构的变化会影响水生态系统的健康状态[47]。因而, 保护鱼类多样性对于水生态系统的稳定具有重大的意义。为此, 针对长江流域鱼类资源的保护提出了以下两点建议: (1)严格监督、合理解决保护区段流域生态安全问题。比如合理搭建人工鱼巢和人工鱼礁; 及时关注流域内生物入侵情况, 筑好生态安全屏障; 增殖放流应合法合理,征求相关科研人员专业建议; 密切关注沿江城镇和企业排污问题, 从源头控制污染负荷; 严格监管两岸消落带及中坝上作物种植和畜禽养殖, 避免面源污染对鱼类生境产生影响[11,48]。(2)加强科学研究,建立适应性管理机制。尽管长江流域鱼类资源丰富, 但缺乏绝大部分鱼类的基本生物学信息, 使得我们无法对其种群状态进行准确评估, 同时也不利于人工繁殖等相关保护工作的开展[49]。因此, 建议全面调查长江流域鱼类的物种数量、种群状况和受威胁程度等, 系统开展鱼类基础生物学和生态学研究, 重点研究珍稀特有鱼类的人工繁殖技术; 同时加强水生态系统的健康监测[50], 建立适应性管理机制。
4 结论
传统的鱼类资源调查耗时费力, 而eDNA技术在灵敏度、标准化和种类鉴别等方面均优于传统方法, 且操作简单, 在鱼类多样性的监测与保护工作中具有巨大的应用前景。本研究首次运用eDNA技术调查长江上游珍稀特有鱼类国家级自然保护区重庆段鱼类多样性, 共检测出鱼类74种(不包括未鉴定到种水平的3属), 成功验证了eDNA技术检测长江鱼类物种组成的有效性和可行性。各样点的鱼类Alpha和Beta多样性分析结果差异较小, 表明该保护区鱼类的生态结构较为稳定。在现阶段的长江鱼类资源监测中, 虽然eDNA技术无法完全取代传统的鱼类监测方法, 但可根据监测任务的需要, 与传统监测方法结合使用, 用于快速调查长江鱼类的多样性组成及分布等, 为后期长江“十年禁渔”效果评估提供一定的基础资料。
致谢:
感谢陶晔、查富蓉和刘静(上海凌恩生物公司)在生物信息学分析工作中提供的帮助; 感谢重庆市珍稀特有鱼类国家级自然保护区管理处工作人员在本次采样中所提供的帮助。
