基于SSR分子标记的紫薇遗传多样性分析
2022-01-07冷嘉文乔中全唐丽王晓明陈前欣邵雯雯
冷嘉文,乔中全,唐丽*,王晓明,陈前欣,邵雯雯
(1.中南林业科技大学林学院,湖南 长沙 410004;2.湖南省林业科学研究院,湖南 长沙 410004;3.长沙市木本花卉工程技术研究中心,湖南 长沙 410004)
紫薇(拉丁名:Lagerstroemia indicaL.)[1]又名百日红,痒痒树。紫薇是千屈菜科紫薇属的落叶乔木或灌木,树皮光滑,嫩枝具四棱,淡褐色;叶对生,花红色、紫色等;原产于亚洲南部及东部温带,中国各地均有种植,是夏季著名观赏植物。紫薇的枝条具有柔软、易愈合、萌芽力强的特点,因此紫薇也是一种优秀的观花乔木,在园林绿化中,被广泛用于公园、庭院、道路、街区城市等绿化,也是做盆景的好材料[2-5]。由于紫薇的品种多样,来源复杂,导致紫薇在品种分类鉴定中产生困难。遗传多样性是种质资源研究和评价的主要内容之一,也是种质资源保护、开发和利用工作的基础和前提;种质资源的遗传多样性为改良品种、选育新品种及开展遗传生物学研究提供了丰富的遗传变异和基因资源。随着分子生物学的发展,利用分子标记开展紫薇遗传多样性、亲缘关系分析、遗传图谱构建等方面的研究已成为紫薇研究中的重点和热点。王献等[6]在2004年建立并优化了20个紫薇和南紫薇的AFLP银染色反应系统,构建了紫薇品种和其他几种的AFLP的指纹图谱;此外,该技术用于分析30种紫薇品种和2个近缘种的亲缘关系,为紫薇的良种选育、基因连锁图谱的构建以及遗传关系等一系列研究奠定了理论基础。顾翠花[7]在王献的基础上优化并建立了适合紫薇的AFLP实验反应体系,筛选出了适合紫薇种质资源评价分析的引物,研究紫薇中13个群体的亲缘关系。Pounders等[8]以紫薇的栽培品种“Tonto”和‘White VI’DNA为模板,以GA、AAG、ATG和CAG为基序,筛选了15个SSR位点。蔡明等[9]从紫薇基因组中分离出39个SSR,其中有12个多态的SSR结合现有的14个SSR,合计26个SSR可以明确识别所有品种,并分成两个不同的组。新开发的SSR对于遗传多样性评估、遗传图谱构建和分子育种是有效的。徐静静[10]等使用ISSR标记技术对4种不同花色紫薇群体进行分析,研究证明4种紫薇群体具有丰富的遗传多样性,聚类分析结果表明白色、紫色及粉色系的单株基本聚在一起,但红色系的聚类结果比较分散,且红色和白色亲缘关系较近,与粉色的亲缘关系较远。Wang等[11]使用78个SSR标记来评价51个紫薇品种、5个屋久岛紫薇品种和37个种间杂种的遗传多样性。SSR基因座在不同的品种之间有很大差异,每个位点平均检测到6.6个等位基因。聚类分析确定了紫薇栽培品种和屋久岛紫薇单个品种及其种间杂种的三个主要组。谢宪[12]通过AFLP技术分析了紫薇3个杂交组合的亲代和子代43个材料的聚类分析表明,紫薇紫色和红色单株能聚成一支,粉色单株比较分散。乔中全等[13]在2019年基于ISSR分子标记,分析了紫薇栽培品种(其中引自美国30个品种,中国8个品种)的亲缘关系。分析结果表明,38个品种共聚为3支。第一个分支全部是美国引进品种,第二个分支包括10个美国引进的品种和7个中国种植的品种,第三个分支引进品种和中国栽培品种是同一个。这些研究在分子水平对紫薇品种的分类、鉴定方面取得一定的进展,但对从其他国家进口的紫薇品种研究比较少,在遗传多样性和种质资源评价方面不能进行全面的概括。SSR分子标记由于其在基因组中覆盖率高、实验操作步骤简单、多态性丰富、稳定性好等优点,能够检测出品种亲缘关系之间的细小差异,现在已经广泛应用于品种种质资源亲缘关系、遗传多样性的研究,本试验对30种来源于美国的紫薇品种利用SSR分子标记的方法探讨不同紫薇品种的亲缘关系。本研究对来自30个不同品种名的紫薇进行简单重复序列扩增多态性标记研究,避免表型分类有同名异物及同物异名的差异,希望深入探究来源不同的紫薇品种的亲缘关系。
1 材料与方法:
1.1 植物材料
30份试验材料均取自湖南省林业科学实验林场紫薇种质资源圃,在选定的植株上随机选取生长健康且无病虫污染的植物叶片,用密封袋半封口并放置于冰袋上带回实验室,用双蒸水清洗干净于-80℃冰箱保存备用。试验材料编号见(表格1)。

表1 实验材料Table 1 Experiment Material
1.2 紫薇基因组DNA提取
紫薇叶片基因组DNA采用天根生化科技(北京)有限公司生产的新植物基因组DNA提取试剂盒,提取和改良CTAB法[14],具体操作步骤参考新植物基因组DNA提取试剂盒使用说明书。根据紫薇属植物的特性和实际情况进行改进,使用1%的琼脂糖凝胶电泳和蛋白测定仪检测DNA的完整性及浓度。
1.3 引物筛选和PCR扩增
采用优化好的SSR-PCR反应体系,即25μL反应体系,模板DNA 1μL(50 ng/L),上下游引物各1μL(10µmol/L),2×Rapid Taq PCR Master Mix(含有Taq酶、dNTP和缓冲液)12.5μL,双蒸水补足至25μL。在95℃预变性3 min,95℃变性15 s,退火温度为60℃15 s,在72℃延伸15 s,总共34个循环,在4℃保存。通过5%的琼脂糖凝胶电泳和凝胶成像系统BIO-RAD中成像,从100对引物中选择背景清楚、扩增条带清楚且亮度较高的10对引物用于紫薇样品的SSR-PCR反应。
1.4 数据处理与分析
统计扩增条带,选择清晰、可辨认的条带记为“1”,不清晰或模糊的记为“0”形成原始矩阵,使用POPGENE 32软件计算出观测等位基因数(Na)、有效等位基因数(Ne)、Shannon’s信息指数(I)、遗传距离、遗传一致度;利用MAGE 7.0进行紫薇品种间的聚类分析。
2 结果与分析
2.1 紫薇叶片基因组DNA提取结果
通过1%的琼脂糖凝胶电泳检测,所得到的紫薇基因组DNA条带清晰,检测结果表明紫薇基因组DNA完整性好,无降解,浓度高,基本没有蛋白质及RNA的污染。

图1 DNA检测电泳图Fig.1 Electrophoresi of DNA detection
2.2 品种间遗传多样性分析
本实验从100对SSR引物中筛选出10对背景清晰、亮度高、多态性好的引物对30种紫薇品种进行SSR-PCR扩增,均能扩增出清晰的条带,总共扩增出10条带,多态性比率为100%。30份样品的观测等位基因(Na)平均含有2.7个,在SSR09位点有等位基因数量最多为4个、有效等位基因数(Ne)为1.6839、Shannon’s信息指数(I)分布范围在0.3251~0.8434,平均值为0.650 2(见表格2),在SSR13位点最高为0.843 4,表明在此位点具有较为丰富的遗传信息。以上遗传多样性分析均表明30个供试样品间的遗传多样性较高、具有丰富的遗传变异。

图2 引物SSR1对30个品种的扩增产物电泳图Fig.2 Electrophoresis of the amplified products of primer SSR1 on 30 varieties
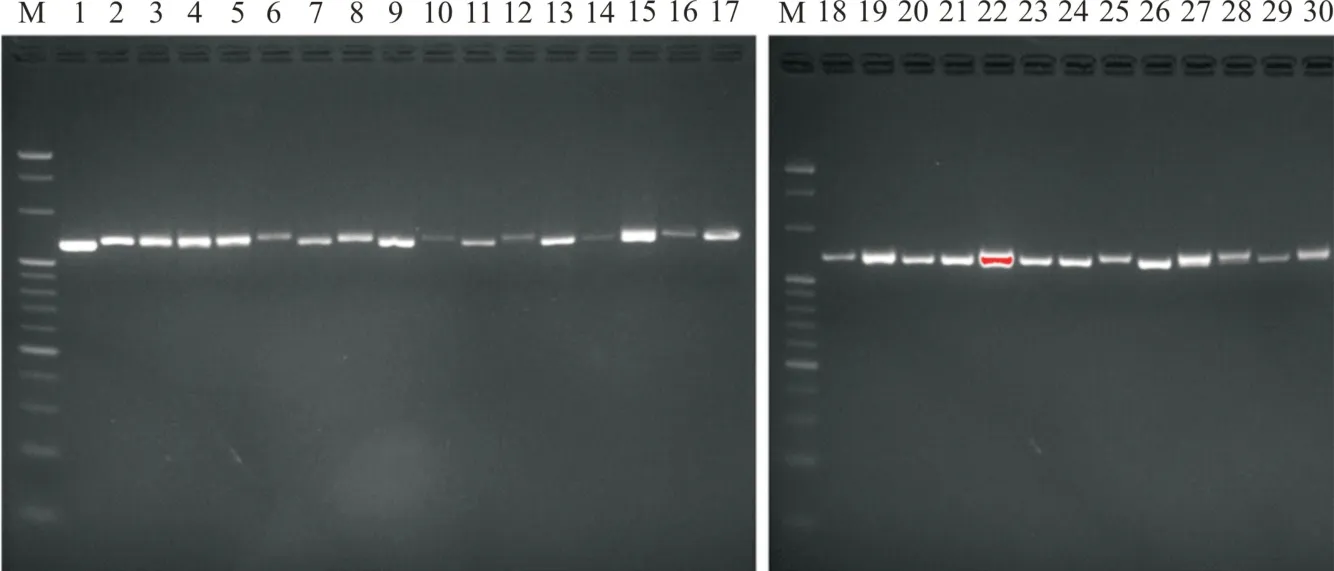
图3 引物SSR7对30个品种的扩增产物电泳图Fig.3 Electrophoresis of the amplified products of primer SSR7 on 30 varieties

图4 引物SSR32对30个品种的扩增产物电泳图Fig.4 Electrophoresis of the amplified products of primer SSR32 on 30 varieties

表2 30个紫薇品种的遗传多样性Table 2 Genetic diversity of 30 crape myrtle varieties
2.3 遗传一致度及遗传距离分析
遗传距离和遗传一致度可以评价种品种之间遗传分化程度和亲缘关系远近,遗传距离越小,越接近于“0”,遗传一致度越高,越接近于“1”,表明种群之间的遗传分化程度越小,亲缘关系越近;反之,种群之间的遗传分化程度越大,亲缘关系越远。通过PopGen 32软件进行对30个紫薇样品间遗传相似性系数及遗传距离的计算。30个紫薇品种中,遗传一致度的范围介于0.111 2~0.911 8,平均值为0.719 7,“Bradberry Wine”和“Catawba”遗传一致度最高为0.911 8,表明两者亲缘关系相近;“Victor”和“Blush”遗传一致度最低,为0.111 2,表明两者存在遗传差异较大。Nei’s遗传距离的范围介于0.027 0~0.896 5,平均值为0.404 1,“Bradberry Wine”和“Catawba”遗传距离最近,为0.027 0,表明两者遗传分化程度小,亲缘关系相近;“Victor”和“Blush”遗传距离最远,为0.896 5,表明两者存在遗传分化程度大,亲缘关系较远,与遗传相关性数据一致。
2.4 紫薇品种的聚类分析
根据UPGMA法对30种紫薇品种进行聚类分析,得到不同品种间的遗传关系树状图(见图5)。30个紫薇品种可以大致分为4个类群。类群Ⅰ由12个品种组成:“Double Feature”、“Bradberry Wine”、“Catawba”、“Siren Red、Red Filli”、“Red Roost”、“Shell Pink”、“Purple Majic”、“Sacramento”、“Sacramento”、“Dynamite”、“Victor”、“Berry Dazzle”;类群Ⅱ由15个品种组成:“Rhapsody in Pink”、“Best Red”、“Midnight Majic”、“Peppermint”、“Cherry Dazzle”、“Crimson Red”、“Coral Malic”、“Purely Purple”、“Red Hot”、“Shawberry Dazzle”、“Pure velvet”、“Pure White”、“Tonto”、“Moonlight Majic”、“Natche”;类群Ⅲ由2个品种组成:“Chisam Fire”、“Mystic Magenta”;类群Ⅳ由单独1个品种组成:“Blush”。

图5 30个紫薇品种的UPGMA聚类图Fig.5 UPGMA cluster map of 30 crape myrtle varieties
3 讨论
利用改良CTAB法从紫薇叶片中提取基因组DNA,通过检测结果可知DNA无明显降解,浓度较高但纯度一般;使用试剂盒提取紫薇基因组DNA,通过检测结果可知DNA无明显降解,纯度较高,但浓度相比较改良CTAB法较低,使用试剂盒提取紫薇DNA基因组操作相对于改良CTAB法更方便快捷。实验所提取的DNA可进一步用于后续分子生物学试验。
SSR分子标记由于其在基因组中覆盖率高,同时具有实验操作步骤简单、多态性丰富、稳定性好等优点,是研究作物种质资源遗传多样性及群体结构分析的重要工具[15],广泛应用于大部分植物遗传多样性研究。从100对SSR引物中筛选出10对背景清晰、亮度高、多态性好的引物。用筛选出的10对引物通过PCR扩增30个紫薇品种,可以扩增出10个清晰的多态位点,多态位点百分率为100%,表明SSR分子标记可以用于紫薇品种的遗传多样性和亲缘关系鉴定。刘阳等[16]利用41对SSR引物对48个紫薇品种遗传多样性分析中总共获得317个等位基因位点,其中单对引物最多获得15个等位基因位点,最少有3个。相比之下,此次试验结果均低于该研究。
聚类分析是根据试供材料的多个数据指标进行分类,是探究种质资源遗传多样性和遗传背景的有效手段[17],是植物保护和利用的前提,分析后聚集在同一类别的材料具有很大程度的相似性,不同类别的则差异性较大。聚类分析可以直观的表示品种或群类之间的遗传关系及遗传相似性。SSR聚类分析依据分子水平上的遗传一致度和遗传距离关系分类,可以直观的了解紫薇品种亲缘关系的远近。群类Ⅰ和群类Ⅱ中包含30个紫薇品种的大部分,其中“Dynamite”、单独为一类的品种“Blush”分类意见与乔中全等[13]研究结果意见不一致。群类Ⅱ中“Crimson Red”和“Coral Malic”两者亲缘关系较为接近却出现不同的花色,确与遗传距离更远的品种花色相同,这与王献等[6]提出的不能单独以花色这一形态特征鉴别紫薇品种的意见一致。本实验未能扩增出特异性片段与紫薇品种的花色或其他形态特征相关,因此还需大量开发特异性引物,更深入探究紫薇品种的遗传背景。
本次试验中,由于所采用的紫薇品种不够丰富,不能完全概括紫薇的遗传关系,应该增加试验材料品种的丰富度,选用不同引物,结合株型、花萼特征、花色等表型形状,表型形状是受基因型和环境因素共同影响的[18],由于紫薇的品种多样,来源复杂,只凭传统的形态学标记不能对品种进行有效鉴定,易致紫薇在品种分类,遗传多样性的鉴定中产生困难,同时以表型形状为选育标准可能导致紫薇新品种育种失败。往后会继续结合紫薇的表型形状、生物学特性、细胞学分析、分子水平上的鉴定综合分析紫薇品种的亲缘关系。本研究采用SSR分子标记技术对来自美国的30个紫薇品种遗传多样性分析及亲缘关系进行初步探讨。目前我国紫薇属植物有18种,但尚处于研究初期,起步较晚,与美国、日本等国家存在一定差距[19]。因此,我们要大力加强对紫薇属植物的开发利用,积极开展相关试验工作,进一步深入研究紫薇属植物遗传特性,为紫薇属植物种质资源多样性提供重要信息,以期对紫薇新品种开发选育、繁殖栽培等方面提供技术支撑。
