微型生物群落eDNA-PFU监测
2021-12-24姜传奇冯伟松龚迎春
姜传奇 熊 杰 黄 洁 冯伟松 龚迎春 缪 炜
(中国科学院水生生物研究所, 武汉 430072)
主要由藻类和原生动物等组成的微型生物群落在水生态系统中起着至关重要的作用, 是水生态系统食物链的关键环节。微型生物个体小, 对环境变化响应灵敏, 非常适合用于环境的生物监测。大量研究表明微型生物群落的结构和功能可以很好地反映不同生境的水质特征。
1991年发布的《水质 微型生物群落监测 PFU法》(GB/T 12990-91)[1]是我国自行制订的首个生物监测国家标准。该标准应用聚氨酯泡沫塑料块(PFU)作为人工基质采集微型生物, 并根据该生物群落的结构与功能参数评价水质。相比于直接采水样, PFU法可采集到水体中85%以上的微型生物,同时还可避免河流等流水生境中采集的瞬时性和随机性等问题[2]。标准颁布后, PFU法在全国范围内对不同河流(包括长江、汉江、乌江、清江和沅江等)的水质进行评价, 被证明是一种快速、经济、准确的监测方法[3]。
PFU法需要专业人员利用显微镜对微型生物进行种类鉴定, 对专业知识要求高、费时耗力且不易标准化。环境DNA(eDNA)技术的发展则突破了以往监测人员无法准确鉴定微型生物的瓶颈, 能更为高效和准确地鉴定出水环境中包括微型生物在内的各种水生生物[4—6]。eDNA方法在进行物种鉴定时依赖的参考序列库具有通用性和标准性, 所获鉴定数据更客观且可比性强。尤其在万种原生生物基因组计划[7]支撑下, 微型生物基因组和条形码数据库不断丰富和完善, 使得微型生物种类的鉴定更为准确; 同时由于DNA测序成本的降低和设备的国产化, 检测快速、经济, 易于推广。
基于此提出微型生物群落(藻类和原生动物)eDNA-PFU法, 即基于eDNA技术改进的PFU法, 对微型生物群落的结构和功能进行监测, 并将藻类和原生动物种类数、指示种类和功能类群等微型生物群落的结构和功能参数作为湖泊河流水库等水体的生态考核指标。为了对上述提议进行验证, 我们针对武汉东湖水体中的原生动物群落开展了预实验, 全面评价了eDNA-PFU法监测微型生物群落的准确性和有效性。
1 材料与方法
1.1 样品采集与制备
2020年8月12日至9月12日, 本研究在湖北武汉东湖中设置了一个采样点(30°53′95″N, 114°37′51″E;图 1)。实验方法参照《水质 微型生物群落监测PFU法》(GB/T 12990-91), 将PFU(孔径为100—150 μm)制作成5 cm×6.5 cm×8 cm的小块, 采用沉式挂放法将PFU块挂放于距离岸边20 m处, 挂放深度为距离水面30—50 cm。分别在放置3d、5d、7d、9d、11d、13d、15d、17d、19d、23d和30d后随机取6块PFU样品, 采集的PFU块放到密封袋中带回实验室, 获取各PFU块中的水样, 同时在PFU周围利用采水器采集3 L原位水环境样品带回实验室。取PFU样品和原位水环境样品分别进行微型生物活体鉴定, 另取等量的PFU样品和水环境样品进行eDNA样品制备(每组样品设置3个平行)。
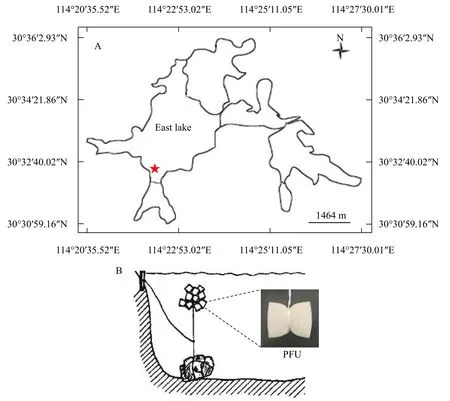
图1 PFU在武汉东湖的样点位置及PFU的放置方式Fig. 1 Location and PFU systems applied in East Lake, Wuhan
1.2 原生动物的形态学鉴定与丰度测定
吸取一定体积的PFU挤出液或原位水在倒置显微镜下进行物种鉴定和计数。
原生动物的鉴定和计数都是在显微镜(OLYMPUS BX53, 日本)下进行[3]。镜检时用微吸管从含PFU挤出液或原位水的烧杯底部吸3滴水样进行观察。第一片在高倍镜下观察, 第二、三片分别用中倍镜和低倍镜观察。计数时先把烧杯中的水样摇匀, 用微吸管吸0.1 mL水样于0.1 mL的技术框内,全片进行活体计数。原生动物的物种鉴定主要参考《微型生物监测新技术》[8]和《原生动物学》[9]。
1.3 eDNA的提取、测序与数据分析
采用十六烷基三甲基溴化铵(Cetyltrimethylammonium Bromide, CTAB )法对样本的DNA进行提取, 即将适量的样品加入到CTAB裂解液中, 然后经酚-氯仿-异戊醇抽提除去蛋白质、多糖等杂质, 再加入乙醇沉淀分离出核酸[10]。用2%的琼脂糖凝胶对DNA的纯度和浓度进行检测, 对于质量合格的DNA, 使用M220聚焦超声仪(Covaris Inc., Woburn,MA, USA)将DNA破碎成大约300 bp的片段, 以建立文库。在检验文库质量后, 所有样品均基于MGISEQ-T1进行测序。
对测序所得的原始序列用fastQC(v0.11.9)进行质量评估, 并移除低质量碱基, 得到测序质量较好的Reads(Clean data), 将经过质控的Reads, 通过BLASTN比对至SILVA数据库[11], 设置e值小于1e-5, 筛选出符合要求的Reads, 使用Megahit (v1.1.3)将这些Reads组装到一起得到全部样本的 rDNA contigs。用nhmmer判定组装得到的 rDNA contigs中属于18S rDNA的部分并进行截取, 经97%相似度聚类后得到最终18S contigs用于物种鉴定。使用Bwa(0.7.17-r1188)将每个样品的 Reads分别比对至18S contigs上, 得到比对结果文件, 并利用Featurecounts(v1.6.0)进行统计, 然后采用RPKM(Reads Per Kilobase per Million mapped reads)的方式进行标准化得到OTU(Operational Taxonomic Units)丰度表。
1.4 数据分析
本研究主要对eDNA-PFU法和传统PFU法在物种鉴定的全面性和准确性等方面进行了比较, 包括两种方法在不同天数所监测到的种数和具体物种组成等数据的比较。
2 结果与讨论
2.1 eDNA-PFU法在物种鉴定多样性方面的特征
经过持续30d共11次的样品采集, 通过显微镜活体观察分别从PFU块和原位水样检测到153种(11门)和79种(9门)原生动物; 通过eDNA宏基因组测序法分别从PFU块和原位水样中获得473个OTU(14门)和440个OTU(14门)的原生动物序列。eDNA-PFU法鉴定到原生动物的物种数远远高于传统PFU法及原位水环境样品中所检出的数目, 监测到的多样性更高。eDNA鉴定出的物种比传统方法高这么多倍的原因主要是因为前者可以鉴定出许多丰度相对较低的物种及在形态上很难辨别的物种。
基于累积所检出的原生动物进行比较分析, 发现eDNA-PFU法逐次累加检测物种数目均显著高于PFU法和水样eDNA检测方法, 通过可重复双因素方差分析发现, eDNA-PFU法所检测到的原生动物数目与PFU法和单次eDNA-水样检测到的物种数目均有显著差异, 并且eDNA-PFU法检测稳定性更高, 仅在前两次采样时存在部分偏差, 之后数次采样偏差均较小(图 2)。此外, eDNA-PFU法较传统PFU法达到检测饱和的速度更快, 第7天即检测到原生动物总物种数的98.19%, 而传统的PFU法仅可达到60.78%(图 3)。
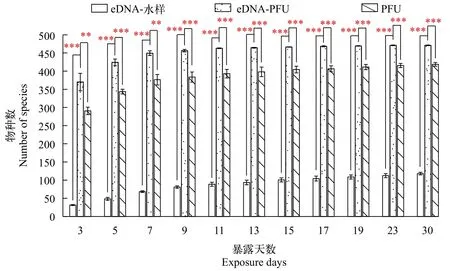
图2 eDNA-PFU法与水样eDNA法和传统PFU法累积采集原生动物物种数比较分析Fig. 2 Comparative analysis of the number of protozoan species collected by eDNA-PFU and water eDNA and traditional PFU methods
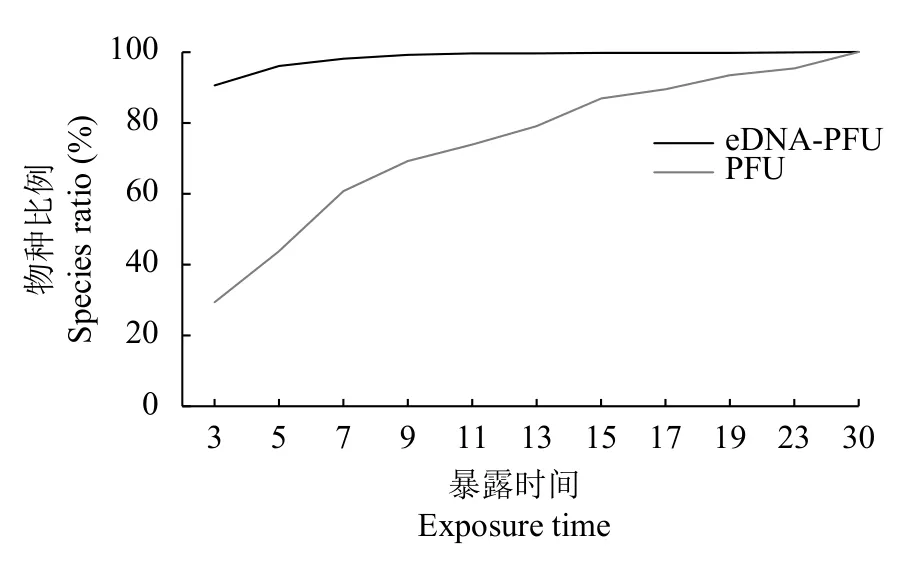
图3 eDNA-PFU法和传统PFU法累积采集原生动物物种数所占比例Fig. 3 The proportion of the number of protozoa species collected by eDNA-PFU and traditional PFU method
2.2 eDNA-PFU法在物种鉴定准确性方面的特征
将eDNA-PFU法和传统PFU法所检出的原生动物进行比较(表 1), 发现eDNA-PFU法鉴定到种级阶元的有64.4%, 到属级阶元的有20.0%, 鉴定到科及以上阶元的有15.6%; PFU法鉴定到种级阶元的有62.4%, 到属级阶元的有34.8%, 鉴定到科及以上阶元的有2.8%。随后对两种方法所鉴定的原生动物在各分类阶元进行比较(图 4), 发现在门水平100%覆盖, 纲、目和科水平的覆盖程度较好, 均大于70%,传统PFU法所检出的原生动物大部分包含在eDNAPFU法检出的原生动物中, 且在属水平的覆盖度也较高, 可达到45%。但值得注意的是两种方法在种类水平上匹配度很低, 镜检确定的70个种类, 仅有5种被eDNA监测到, 仅6%鉴定为同一物种(图 4)。我们分析一方面可能是由于传统PFU法是根据活体特征对原生动物进行物种鉴定, 而原生动物个体微小, 运动较快, 因此很难鉴定到种, 其种水平上的鉴定结果很难保证准确性, 从而造成两种方法在种水平的鉴定结果偏差较大; 另一方面由于原生动物的多样性极为丰富, 很多物种还没有可以使用的分子序列信息, 从而影响了eDNA-PFU法在物种鉴定中的准确性。此外, 本研究加入内参(嗜热四膜虫细胞)作为鉴定参照, 发现eDNA-PFU法可以鉴定到种, 而传统PFU法仅可鉴定到四膜虫属。因此, 整体而言, eDNA-PFU法与传统PFU法比, 覆盖度和灵敏度均更高。
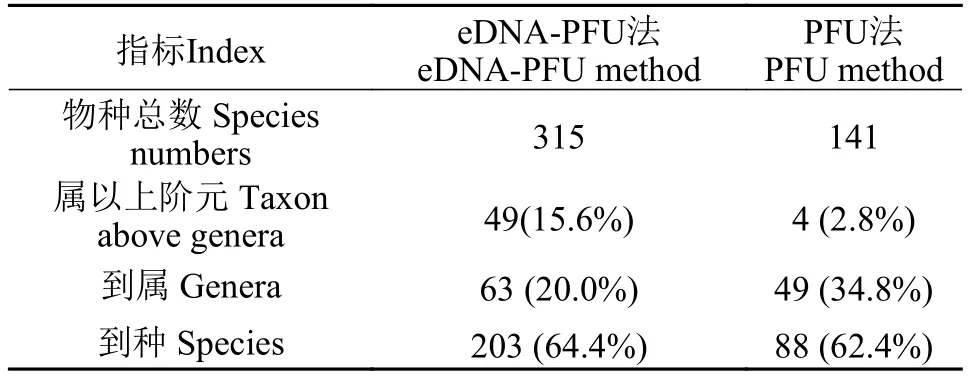
表1 eDNA-PFU法和传统PFU法鉴定原生动物的精准度比较Tab. 1 Comparison of accuracy in identifying protozoa by eDNA-PFU and traditional PFU methods
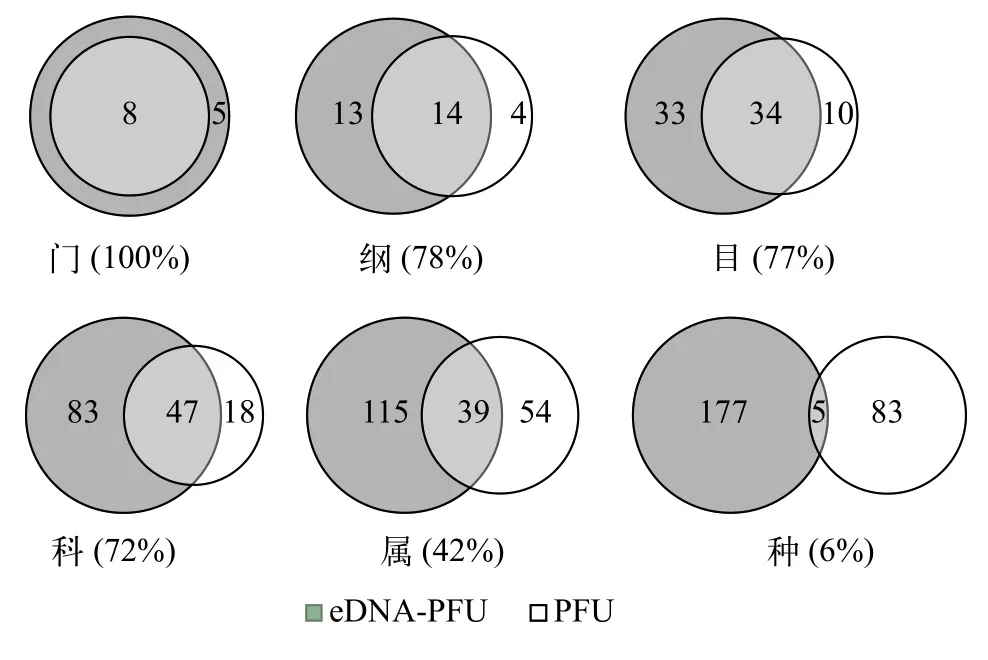
图4 eDNA-PFU法和传统PFU法所检出的原生动物各阶元比较图Fig. 4 Comparison of each category of protozoa detected by eDNA-PFU and conventional PFU methods
3 结论与展望
本研究提出利用eDNA-PFU法对水体中的微型生物群落(藻类和原生动物)进行监测, 在东湖水体中对该方法监测微型生物群落的有效性进行了评估。结果表明eDNA-PFU法较传统PFU法覆盖度更高, 可以全面地揭示水体中微型生物群落结构特征。此外, 本文发现eDNA-PFU法鉴定到原生动物的物种数远远高于传统PFU法以及原位水环境样品中所检出的数目, 检测到的物种多样性更高, 且与传统PFU法相比, 基于eDNA-PFU法的检测在科属水平上具有较高的覆盖度和准确性。因此, 我们的结果表明eDNA-PFU法可以全面、真实、准确地揭示水体中微型生物群落结构特征。
然而, eDNA-PFU法在水质监测的应用中仍有许多亟待解决和完善的地方。首先, 由于原生动物和藻类等微型生物的多样性极为丰富, 其分子序列信息也十分多样, 为了更好地对eDNA-PFU加以应用, 后续需要进一步丰富和完善本土原生动物等物种的DNA条形码数据库。再者, 基于eDNA-PFU法获得的物种和丰度信息较之传统PFU法在数据的呈现形式上不一样, 例如传统PFU法可以提供不同类群的绝对丰度(由于依据活体样品对原生动物的定量难度非常大, 因此在实际应用中常常很难获得这方面的数据), 而eDNA-PFU法只能提供相对丰度, 因此后续需要开展系统研究以制定和规范依据eDNA-PFU法的水质监测参数。最后, 由于原生动物和藻类等微型生物的分布与季节、地理位置, 及局部小环境特征都有非常紧密的关系, 本文仅用了1个调查点的数据, 虽然重复较多, 但仍不具备代表性。后续将增加调查样点数, 涵盖多个浮游生物多样性高、中、低梯度的样点。通过比较不同生物多样性背景的水体监测结果的异同, 以对eDNAPFU法和传统PFU方法做进一步的比较和评价, 并全面分析基于eDNA-PFU获得的微型浮游生物种类数、指示种类和功能类群等微型生物群落的结构和功能参数对生态考核的支撑作用, 从而为湖泊河流水库等水体的生态考核提供新一代的生物监测标准。总之, 通过进一步完善, eDNA-PFU法有望成为水体微型生物群落监测的新一代监测方法。
致谢:
感谢中国科学院水生生物研究所何舜平研究员对于eDNA评估水质方法的提出给予的指导, 感谢刘国祥研究员在藻类的eDNA监测评估中给予的建议, 感谢徐军研究员在长江中下游水生态系统结构和功能方面提供的信息, 感谢水生生物数据分析管理平台的曾宏辉高级工程师在水生生物DNA条形码库构建中给予的支持。
