基于SNP 和InDel 标记的巴西木薯遗传多样性与群体遗传结构分析
2021-12-17邹枚伶张辰笈江思容EderJorgedeOliveira张圣奎夏志强王文泉李有志
孙 倩 邹枚伶 张辰笈,4 江思容,5 Eder Jorge de Oliveira张圣奎 夏志强,3,4,* 王文泉,3,4,5,* 李有志
1 广西大学生命科学与技术学院 / 亚热带农业生物资源保护与利用国家重点实验室, 广西南宁 530004; 2 中国热带农业科学院热带生物技术研究所, 海南海口 571101; 3 中国热带农业科学院热带生物组学大数据中心, 海南海口 571101; 4 海南大学, 海南海口570203; 5 南京农业大学, 江苏南京 210095; 6 Embrapa Mandioca e Fruticultura, Cruz das Almas, Bahia 44380-000, Brazil; 7 齐鲁工业大学, 山东济南 250306
木薯(Manihot esculentaCrantz) 是大戟科(Euphorbiaceae)木薯属(ManihotP. Mill.)的多年生灌木植物, 具有高生物量、抗贫瘠、抗病虫能力强等特点, 被广泛种植于亚、非、美三洲等多个国家或地区[1]。木薯起源于亚马逊河流域, 于19 世纪20 年代传入中国, 最初种植于广东省, 之后逐渐在海南、广西、贵州、云南等地大量种植。它是世界三大薯类作物之一, 同时也是世界上第六大粮食作物, 仅次于小麦、水稻、玉米、马铃薯和大麦[2]。其用途广泛, 除可食用、饲用外, 还可用于生产加工, 如造纸、纺织、生物燃料等[3]。木薯块根还可用于提取淀粉, 加工成薯条、面包, 以及生产燃料乙醇等; 茎秆可用来进行木薯繁殖、粉碎还田、做燃料等; 叶片可作蔬菜食用或喂鱼、养蚕等[4]。
木薯基因组具有高度杂合的特性, 原因是其异花授粉, 且长期进行无性繁殖。由于基因组的高度杂合, 从而增加了木薯遗传变异的多样性, 这些多样性可为木薯育种人员提供更多可选择的良好亲本,但同时由于木薯的基因组高度杂合、亲缘关系不清晰、遗传改良周期长等特点也增加了育种的工作难度[5]。目前已有一些利用相关序列扩增多态性(sequence-related amplified polymorphism, SRAP)、简单重复序列(simple sequence repeat, SSR)、扩增片段长度多态性 (amplified fragment length polymorphism, AFLP)、单核苷酸多态性(single nucleotide polymorphisms, SNP)等分子标记进行木薯遗传多样性的研究。Fregene 等[6]利用SSR 标记对来源于哥伦比亚、巴西和秘鲁等地的木薯地方品种的种质资源多样性评价发现, 不同国家来源的木薯种质的遗传多样性水平都很高, 其中来自巴西和哥伦比亚材料的基因多样性水平最高。Alex 等[7]利用13 对SSR
标记对巴西多地的传统甜木薯品种的群体结构和遗传多样性评估结果显示, 该群体的遗传多样性平均值为0.5407, 范围为0.3138 (GA21)~0.6502(GA140), 表明该群体的遗传变异性宽泛。Carvalho等[8]采用SSR 标记和RAPD 标记的研究中发现, 巴西的木薯种质资源的地理来源和遗传聚类具有显著正相关关系。
丰富木薯种质资源的遗传多样性, 并对其遗传背景和性状进行综合评价, 发掘控制优良性状的优异等位基因, 对今后木薯育种具有重大意义。全基因组关联分析(genome-wide association study,GWAS)能够鉴定目的表型性状与遗传标记或基因间的关系, 并检测出控制相关性状的优良等位基因位点[9]。而进行关联分析需要先评估实验群体的遗传多样性、遗传结构及亲缘关系[10]。但目前利用 SNP 和 InDel 标记对木薯进行遗传多样性、亲缘关系及群体结构分析等的相关研究还鲜为报道。
本研究拟利用SNP 和InDel 分子标记, 对由巴西Embrapa 机构提供的来源于巴西多地的192 份木薯种质资源进行遗传多样性和群体结构分析。本研究将为以后木薯育种亲本选配提供材料和理论指导, 也可为下一步通过关联分析发掘控制木薯种质中优良性状的优异等位基因提供理论依据,从而促进利用分子标记辅助选择技术培育木薯新品种。
1 材料与方法
1.1 试验材料
供试木薯材料共192 份, 均为巴西栽培种木薯(表1)。

表1 192 份木薯栽培种Table 1 List of 192 cassava cultivars
1.2 木薯基因组DNA 提取及建库测序
采用改良的CTAB 法[11]提取木薯叶片的基因组DNA, 经1%的琼脂糖凝胶电泳检测及浓度测定后,将工作液浓度稀释到100 ng μL-1, -20℃保存。然后利用AFSM[12]技术对192 份木薯DNA 样品使用96孔PCR 板分别构建EcoR I-MspI 和EcoR I-HpaII文库, 单克隆检测达到要求后, 将相应的EcoR IMspI 和EcoR I-HpaII 文库按1∶1 比例混合成1 个文库, 总共192 个样本, 构建了2 个AFSM 文库, 并利用Hiseq 2500 对构建好的测序文库进行双端150 bp 测序。
1.3 SNP 和InDels 分子标记检测
利用Perl 脚本(http://afsmseq.sourceforge.net/)对原始测序数据进行过滤, 同时统计测序得到的总reads 数, 再将reads 根据AFSM 技术设计的Barcodes分配到每个个体中, 并统计每个个体的reads 数[12]。使用Bowtie 2 软件[13]将优化后的测序reads 比对到木薯AM560 参考基因组[14], 再使用SAMtools[15]和VCFtools (http://vcftools.sourceforge.net/)检测 SNP和InDels 位点。基于木薯AM560 参考基因组V6.1,利用snpEff 软件[16]识别其变异位置(间隔区、非翻译区/UTR、基因上游区或基因下游区)、变异类型(同义突变和错义突变、移码突变和非移码突变), 同时对其进行注释。
1.4 巴西木薯群体结构、遗传多样性及群体分化分析
先利用 PHYLIP (http://evolution.genetics.washington.edu/phylip.html)计算样品的遗传距离矩阵,然后用Notepad++软件将遗传距离矩阵的文件调整成合适的格式, 采用邻接法构建系统进化树结构,生成tree 文件后, 再使用iTOL (https://itol.embl.de/)绘制进化树图。通过GCTA 软件[17]利用检测出的SNP 对参试木薯群体材料进行主成分分析(PCA)。再使用R 软件计算各个主成分的向量, 绘制PCA散点图。
此外, 使用ADMIXTURE 软件[18]进行群体结构分析, 估算出最佳群体亚群数。先用PLINK 软件[19]调整ADMIXTURE 软件的输入文件格式, 并输入文件, 然后将亚群数K值范围设置为1~12, 根据得到的cross-validation error 值选择合适的亚群数K值,利用各个材料占各亚群的遗传成分系数(Q)构成群体遗传结构矩阵。
利用VCFtools 软件(https://vcftools.github.io/index.html)计算群体遗传多样性指数(π)和群体分化指数(Fst)[20]。根据Wright 的研究, 当群体分化指数(Fst)等于0 或1 时, 分别表明亚群间没有分化, 或亚群间完全分化。而当0 < Fst < 0.05、0.05 ≤ FST <0.15、0.15 ≤ FST < 0.25, 或0.25 ≤ FST < 1 时,则分别表明亚群间具有较弱、中等、比较强或非常强的遗传分化[21]。
2 结果与分析
2.1 巴西木薯群体基因型分析
通过对192 份巴西栽培种木薯基因组DNA 进行AFSM 建库及测序, 总共得到了155 G 数据, 过滤后得到134 G 数据, 893,020,018 条reads。再利用木薯参考基因组 AM560 V6.1, 通过 SAMtools 和VCFtools 软件对192 份木薯样品基因组进行扫描,得到796,006 个SNPs 和116,821 个InDels。
通过哈迪温伯格检测(HWE)>0.001、次等位基因频率(MAF)≥0.05 过滤, 并舍去低质量的变异位点后, 仅保留了9443 个高质量的变异位点(7946 个SNPs 和1997 个InDels)用于后续分析。其中, 3287个SNPs 和InDels 位于基因间隔区, 4005 个SNPs 和InDels 位于基因上游区, 471 个SNPs 和InDels 位于基因下游区, 2 个SNPs 和InDels 位于5′端UTR。845个SNPs 和InDels 属于错义突变, 745 个SNPs 和InDels 属于同义突变, 417 个SNPs 和InDels 属于移码突变, 另有171 个SNPs 和InDels 属于其他类型突变(表2)。
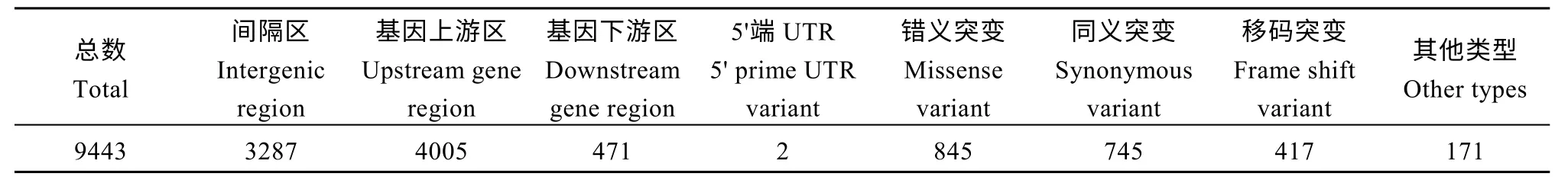
表2 SNPs 和InDels 的统计Table 2 Summary of SNPs and InDels
2.2 巴西木薯群体结构分析
通过ADMIXTURE 软件利用9943 个高质量的SNPs 和InDels 分子标记对192 份巴西栽培种木薯进行群体遗传结构分析。将亚群数K值范围设置为1 ~1 2, 计算不同K值下的交叉验证错误率(cross-validation error, CV error)。当K从1 到2 时, CV error 值迅速减小;K从2 到4 时, CV error 值又逐渐增加; 当K从4 到9 时, CV error 值逐渐减小并趋于平缓; 当K大于9 时, CV error 值又出现一定的增幅(图1-a)。说明在K等于9 时, CV error 值最小, 因此巴西栽培种木薯群体可分为 9 个亚群(Subgroup 1~Subgroup 9)。
192 份巴西木薯可以被分为9 个亚群, 再根据每个个体在这9 个亚群的Q 值, 将每个个体归类到Q值最大所在的亚群(图1-b)。9 个亚群中分别含有3份、22 份、27 份、6 份、20 份、25 份、24 份、12份和53 份材料。
主成分分析以所有的高质量SNPs 和InDels 为基础, 通过R 软件分析绘图, 得到如下结果: 该木薯群体的9 个亚群在PC1 轴上可以看出一定的分布差距, 大部分亚群可以聚类在一起, 该结果说明聚类结果与群体结构的划分具有一致性(图1-c)。
由图2 可知, 聚类结果与群体结构的划分相一致, 亚群1、亚群2、亚群4、亚群6 和亚群8 能较好地分别聚在一起, 而其他亚群样品大致能聚在一起, 且样品间有一定的交叉。巴西木薯各栽培种之间并未聚类到一起, 可能是由于木薯栽培历史比较短, 来源于巴西多地的木薯栽培种还未产生明显的分化。
2.3 巴西栽培种木薯遗传多样性分析
利用9943 个高质量的SNPs 和InDels, 通过计算遗传多样性指数(π), 评估巴西栽培种木薯群体和各个亚群的遗传多样性。通过vcftools 计算发现, 巴西栽培种木薯群体的遗传多样性指数为0.274, 亚群1~9 的遗传多样性指数在0.192~0.289 之间, 其中亚群1 具有最低的遗传多样性指数(0.192), 亚群7、亚群2、亚群6 和亚群3 具有相对较高的遗传多样性指数, 分别达到0.284、0.281、0.264 和0.261, 而亚群5 具有9 个亚群中最高的遗传多样性指数(0.289)(表3)。说明巴西栽培种木薯群体具有相对较高的遗传多样性水平。
利用群体分化指数(Fst)评估巴西栽培木薯亚群间的差异程度(表4)发现, 除亚群1 和亚群4 之间有较强遗传分化外, 其他亚群之间均为中等或较弱遗传分化, 亚群间的遗传分化指数在0.031~0.152 之间。其中, 亚群8 与其他各亚群之间均为中等分化;而除亚群4 与亚群1 外, 亚群4 和亚群1 分别与其他各亚群之间也均为中等分化。表明, 除亚群1 与亚群4 间遗传分化较强、亲缘关系较远外, 其余各亚群间的为中等或较弱遗传分化程度, 即亚群间的亲缘关系相对均较近。
另外, 本研究对试验所用的巴西栽培木薯的遗传距离分析发现, 这些木薯种质间的遗传距离为0.084~0.297, 平均遗传距离为 0.228。其中, BGM 1883 与Valencia 遗传距离最近(0.084); BGM0640 与BRSJari 遗传距离最远(0.297)。
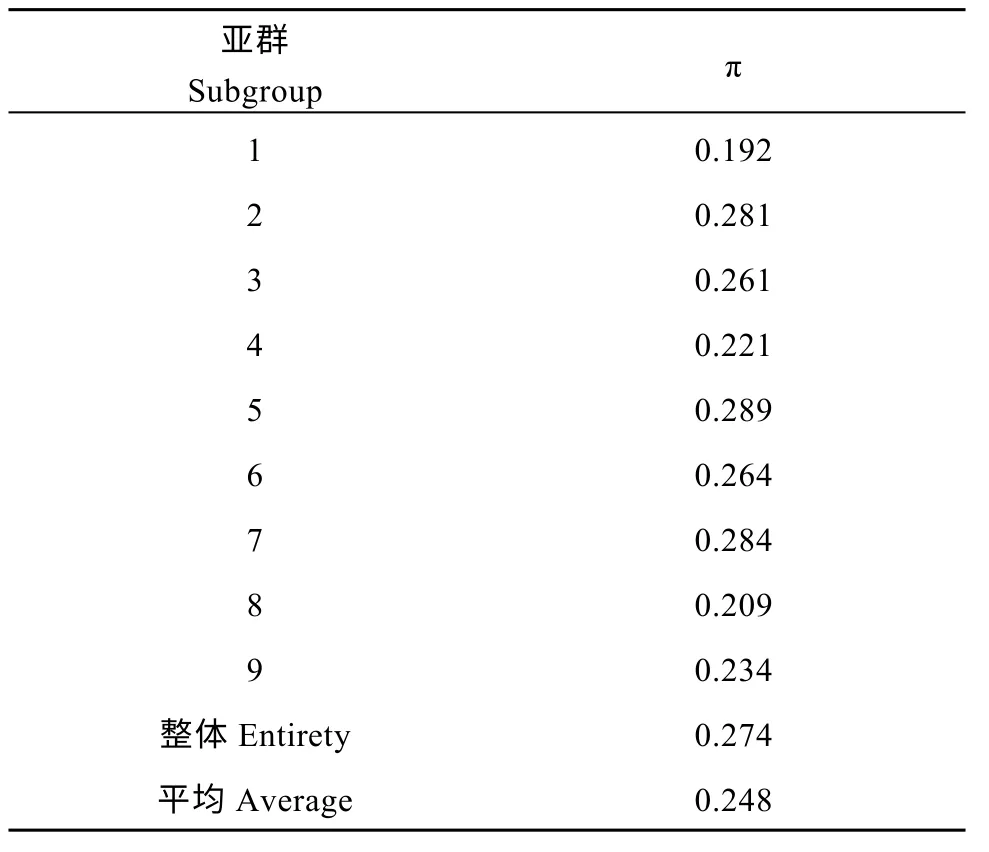
表3 遗传多样性指数(π)的统计Table 3 Statistics of genetic diversity index (π)
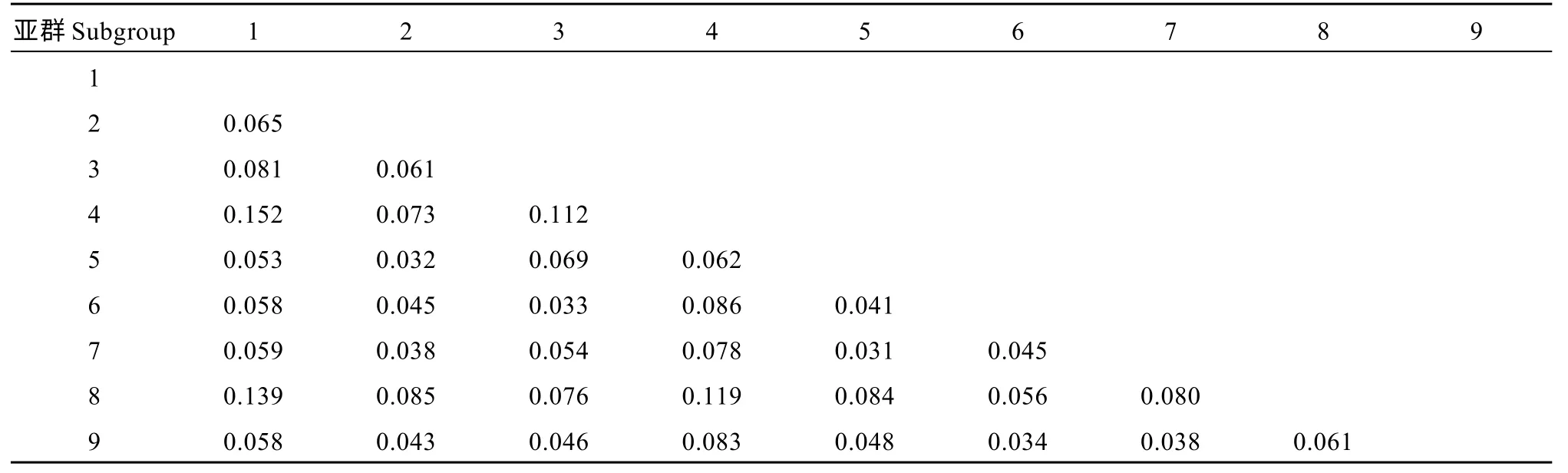
表4 群体分化指数(Fst)的统计Table 4 Statistics of population differentiation index (Fst)
3 讨论
3.1 木薯群体结构分析
研究表明, 基因型与性状之间会产生假关联,其原因可能是群体结构分层、等位基因分布不均等[22]。为了消除造成关联分析不准确的因素, 我们需要先对试验群体进行群体结构分析。本研究利用ADMIXTURE 软件对巴西木薯自然群体的群体结构分析表明, 当K=9 时, CV error 值最小, 由此将192份木薯种质划分为9 个亚群, 该结果与聚类分析、主成分分析的结果大概相符, 它们之间相互补充及印证, 说明该木薯群体的遗传结构较为可靠。在这9个亚群中, 群体分化指数在0.03~0.15 之间, 且大部分亚群间的群体分化指数均小于0.09, 表明该木薯群体存在一个中等偏弱的遗传分化。前人的研究结果显示, 中国热带农业科学院热带品种资源种质圃收集的158 份木薯种质的群体分化指数在0.03~0.07之间[23]; 在其他地区的栽培木薯中, 群体分化指数在0.01~0.05 之间[14], 表明国内收集的木薯遗传分化程度较低。比较看来, 本研究中的巴西木薯种质的群体分化指数高于国内收集的木薯种质, 可挑选优质巴西木薯品种并引进中国, 从而丰富已有的木薯种质资源。
3.2 木薯遗传多样性分析
本研究对遗传多样性指数(π)进行了计算, 从而评估该木薯群体的遗传多样性。Ramu 等[14]的研究表明, 来源于不同地区(尼日利亚、哥伦比亚、巴西等地)的国外栽培木薯的遗传多样性指数为0.0036,低于其祖先(M. esculentassp.flabellifolia, π =0.0051); Fregene 等[6]对来源于哥伦比亚、巴西和秘鲁等地的木薯地方品种的种质资源多样性评价发现,巴西和哥伦比亚的木薯种质具有最高的遗传多样性水平; 在张圣奎对中国热带农业科学院热带品种资源种质圃收集的158 份木薯种质的遗传多样性研究中发现, 该群体的遗传多样性指数为1.21×10-4, 表明该群体的遗传多样性较低[23], 同时也表明目前国内的木薯种质资源丰富度较为缺乏。本研究发现,该巴西木薯群体中各亚群的遗传多样性指数在0.19~0.29 之间, 平均遗传多样性指数为0.248, 说明巴西木薯群体的遗传多样性较为丰富, 可引进部分优良巴西种质以丰富国内的木薯种质资源。除此之外, 样本之间的亲缘关系也会对关联分析的结果造成一定的影响。本研究对192 份木薯种质间的遗传距离进行分析, 从而评估不同材料之间的亲缘关系,结果发现这些木薯种质的平均遗传距离为0.228。
4 结论
本研究利用9943 个高质量的SNPs 和InDels 对192 份巴西Embrapa 机构提供的木薯种质进行了和群体遗传结构分析。遗传多样性分析结果显示, 巴西木薯群体的遗传多样性水平较为丰富, 高于中国和哥伦比亚等地区; 群体遗传结构分析结果显示,该群体被划分为9 个亚群, 此结果与主成分分析及聚类分析结果基本一致。另外, 该木薯群体的分化程度较低, 但高于国内的木薯种质资源。遗传距离分析显示, BGM1883 与 Valencia 遗传距离最近,BGM0640 与BRSJari 遗传距离最远。该研究将为之后关联分析挖掘优良基因及引进优良巴西木薯种质提供依据。
