胶原蛋白作为胃癌潜在生物标志物的生物信息学分析
2021-12-04杨婵君陈雯恬
杨婵君,陈雯恬,刘 静
(1.汕头大学医学院第二附属医院输血科,广东汕头 515041;2.汕头大学医学院长江学者实验室/广东省乳腺癌诊疗重点实验室/生理学教研室,广东 汕头 515041)
根据《2020年癌症统计》的报告,恶性肿瘤是全球主要的公共卫生问题之一,已成为仅次于心脑血管疾病的非传染性疾病死亡原因。在各种恶性肿瘤中,胃癌是全球范围内癌症死亡的第3大原因。每年约有72万人死于胃癌,70%的确诊病例发生在发展中国家,尤其是在东亚。目前,胃癌的诊断技术包括内镜检查、影像学检查、病理组织学检查及肿瘤标志物检测等,手术联合化疗和放疗是胃癌患者的主要治疗方法。然而,由于缺乏特异性标志物,胃癌的早期诊断率非常低,2/3的胃癌患者确诊时已是晚期。更糟糕的是,有转移的胃癌患者的5年生存率只有20%~30%。因此,迫切需要寻找特异性高的生物标志物,用于胃癌的筛查、早期诊断和治疗。本研究拟利用GEO数据库筛选胃癌组织与正常组织的差异表达基因(differentially expressed genes,DEGs),并使用相关生物信息学软件和方法识别出潜在的关键基因,以期发现可用于胃癌的诊断、预后判断和治疗的新的生物标志物。
1 材料与方法
1.1 GEO数据库和R语言
在GEO数据库搜索关键词“Gastric cancer”,选取人体组织标本检测结果,并从中筛选样本量较多的3组GEO数据库,分别为GSE65801、GSE79973和GSE118916胃癌微阵列基因表达数据。3个数据集中共含114例数据,其中GSE65801共32对64个样本,GSE79973共10对20个样本,GSE118916共15对30个样本。用R语言中的Limma软件包对这些数据集进行归一化分析,以获得胃癌中与正常组织相比表达水平发生变化的基因。表达变化基因筛选的阈值设置为|logFC|≥2,P
≤0.05。使用在线韦恩图工具(http://bioinformatics.psb.ugent.be/webtools/Venn/)对从每个数据集中获得的结果进行计算和图形重叠,得出胃癌中的DEGs。1.2 DEGs的功能注释
DAVID 6.8(https://david.ncifcrf.gov/)是一套全面的功能注释工具,包括从分子到生物体水平的基因本体。京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)是基于基因组测序和其他高通量实验技术分子水平信息的综合知识库。为了验证DEGs在胃癌中的生物学意义,本研究应用DAVID 6.8进行功能富集分析和KEGG通路分析,并将其标准设为P
<0.05。1.3 蛋白相互作用网络和核心基因的确定
为了探索胃癌的潜在分子机制,本研究利用STRING数据库(http://string-db.org/)预测DEGs的蛋白质-蛋白质相互作用(protein-protein interactions,PPIs),并用Cytoscape软件及其cytoHubba插件计算了蛋白质间的相互作用系数得分(hub score)。Hub score>1 000的基因被鉴定为胃癌中的核心基因,在PPIs网络中具有高连通性。
1.4 构建miRNA-mRNA网络
miRNAs在胃癌中的功能因其调节通路的不同而不同。使用miRBD(http://mirdb.org)在线分析工具预测核心基因相关的miRNA,并用Cytoscape软件绘制miRNA靶向调控网络图。
1.5 核心基因在胃癌中的表达情况
为了评价和验证核心基因在胃癌中的生物学意义,本研究使用The Human Protein Atlas工具(https://www.proteinatlas.org)分析核心蛋白在胃癌组织和正常组织中的表达及定位情况;使用Oncomine数据库(https://www.oncomine.org/resource/main.html)在 线 分 析工具对核心蛋白mRNA表达水平在正常组织和胃癌组织中的表达差异进行分析;使用UALCAN数据库(http://ualcan.path.uab.edu)分析核心基因在不同种族胃癌患者中的表达情况。
1.6 核心基因在胃癌患者中的预后价值
为了评估核心基因在胃癌患者中的预后价值,使用Kaplan-Meier数据库(http://kmplot.com/analysis)分析核心基因对胃癌患者生存的影响。
2 结果
2.1 数据集的基因表达差异
在GEO数据库获得GSE65801、GSE79973和GSE118916等3个胃癌基因表达数据集,火山图(图1A~C)显示,大多数基因的表达无明显改变,少数基因表达水平发生了显著变化,这可能是胃癌发生发展的关键因素。图1中D~F的聚类热图显示出了核心基因表达的显著变化,有助于区分胃癌组织和正常组织。
2.2 胃癌组织中差异表达基因的筛选
为了探索胃癌组与正常组差异基因的表达,本研究设置阈值P
≤0.05且差异倍数|logFC|≥2进行筛选;分别筛选GSE65801、GSE79973和GSE118916的上调和下调基因,在线绘制韦恩图,获得了25个上调差异表达基因和85个下调差异表达基因(图1G~I)。
图1 数据集的基因表达模式及胃癌组织中3个DEGs
2.3 功能富集分析
生物过程功能富集分析发现DEGs主要富集于消化、骨骼系统发育和胶原纤维组织(图2A);细胞组分功能富集分析主要涉及胶原蛋白、纤维胶原蛋白、胞外区、细胞外基质部分,以及蛋白质类细胞外基质(图2B);分子功能分析显示其与细胞外基质结构成分显著相关(图2C);根据KEGG分析,DEGs主要富集于细胞外基质受体相互作用和局部黏附(图2D)。

图2 通过GO和KEGG通路对DEGs的功能富集分析
2.4 PPIs网络蛋白-蛋白间相互作用分析
为了进一步分析胃癌中DEGs之间的相关性,构建了PPIs网络并计算了蛋白与其他蛋白间相互作用分数,揭示了所识别DEGs之间的密切关系。蛋白间相互作用分数高(hub score>1 000)的DEGs与其他DEGs具有较强的相关性,被认为是胃癌中的核心基因,即COL1A1、COL1A2、COL12A1、COL6A3、COL2A1和COL10A1。所有的核心基因都属于胶原家族成员,与细胞外基质的调控密切相关(图3)。

图3 蛋白-蛋白互作网络(PPIs)与hub评分
2.5 miRNA-mRNA网络分析
在miRDB数据库预测miRNAs调控核心基因的基础上,进一步筛选出得分大于80的miRNA。通过Cytoscape软件构建了miRNA-mRNA网络,表明属于miR-29s家族的hsa-miR-29a-3p、hsa-miR-29b-3p和hsa-miR-29c-3p可以同时调控多个核心基因(图4)。
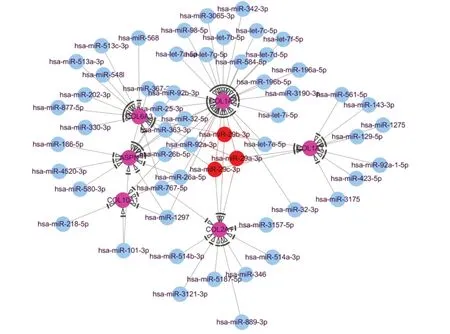
图4 核心基因的miRNA-mRNA网络
2.6 核心基因mRNA表达水平的差异分析
为了验证核心基因在胃癌中的潜在功能,利用Oncomine数据库评估了胃癌和正常胃组织之间核心基因mRNA表达水平的差异,发现除了COL2A1无明显差异外,其他核心基因在胃癌组织中的表达与正常胃组织相比均上调(均为P
<0.05,见图5)。
图5 核心基因在正常组织和胃癌组织中的表达差异
由于胃癌在不同种族人群中的发病率不同,我们进一步利用TCGA数据库分析核心基因mRNA在胃癌组织中的表达情况,发现不同种族的胃癌患者中COL1A1、COL1A2和COL10A1的表达均高于正常胃组织(均为P
<0.05,图6A、B和F)。除非洲裔美国人外,与正常胃组织相比,白种人和亚洲人胃癌患者COL12A1和COL6A3的表达增加(P
<0.05,图6C和D)。在不同种族背景的胃癌患者中未发现COL2A1的异常表达(图6E)。
图6 TCGA数据库中不同民族胃癌患者hub基因表达的差异
除了核心基因的mRNA水平外,人类蛋白质图谱Human Atlas也提供了不同组织中相关蛋白的表达情况。典型图片提示,与正常胃组织相比,胃癌组织中COL1A1和COL12A1的蛋白表达水平升高(图7),而其他核心基因在胃癌和正常胃组织之间蛋白表达未见显著差异。

图7 胃癌和正常胃组织中核心基因的蛋白表达水平
2.7 核心基因的生存期分析
在Kaplan-Meier绘图仪数据库的所有胃癌患者中,核心基因的mRNA水平的高表达与生存期短显著相关,可以预测这些患者的不良预后(P
<0.05)。相反,核心基因mRNA低表达的胃癌患者生存期较长(图8)。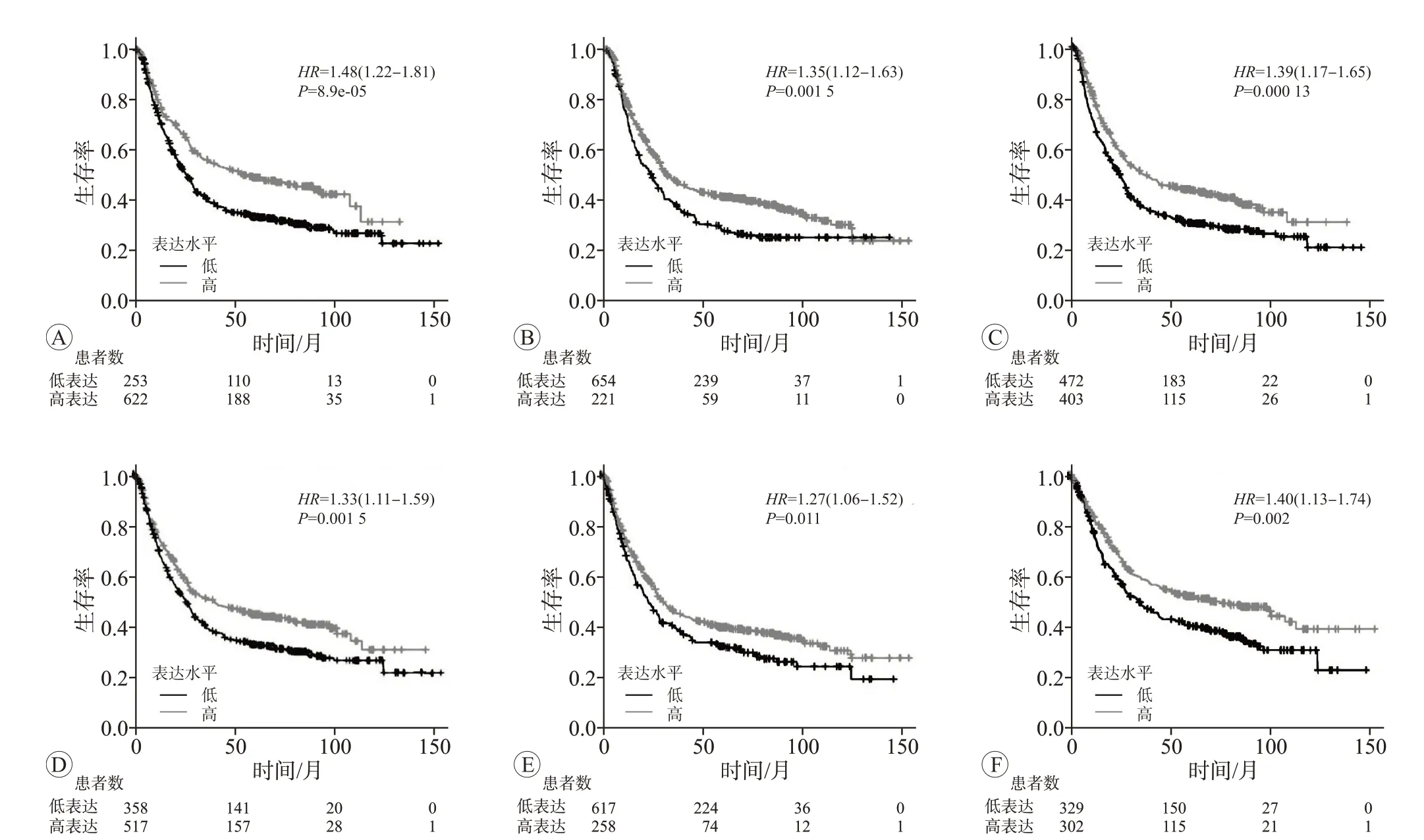
图8 核心基因表达情况对胃癌患者生存期的影响
3 讨论
胃癌作为临床常见的恶性肿瘤,其发病具有明显的地域差异性,我国东部沿海和西北部地区的发病率明显高于南方地区。尽管在过去的一个世纪里发现胃癌的发病率有所下降,但在我国它仍居男性常见癌症第2位,女性常见癌症第5位。因此,揭示胃癌发生发展的机制,挖掘可用于胃癌诊断、治疗和预后判断的特异性生物标志物尤为重要。随着高通量技术的发展和普及,以及多种生物信息学工具的开发,极大地促进了基因图谱的研究,为胃癌患者的早期筛查和临床治疗提供了显著的优势。
为了探索胃癌患者的潜在生物标志物,本研究利用3个基因芯片数据集,建立了胃癌组织和正常组织的基因表达谱。GO功能富集分析和KEGG通路分析表明,COL1A1、COL1A2、COL12A1、COL6A3、COL2A1和COL10A1等胶原蛋白基因在细胞外基质相关功能或通路中富集。人们普遍认为,细胞外基质(extracellular matrix,ECM)重构的稳态,即受损分子或旧分子在降解的同时被新分子替换,是维持组织稳态的关键过程。ECM的组成和数量的改变对肿瘤和基质细胞的性质有很大的影响,这些改变会激活下游信号,从而调节细胞行为,促进肿瘤的进展。
据报道,相关研究发现ECM在恶性肿瘤中通常是失调的,并参与恶性细胞增殖、黏附、迁移、血管生成和药物代谢。胶原蛋白基因参与肿瘤ECM与受体的相互作用和局灶性黏附途径,在肿瘤的侵袭、转移过程中担任着重要的角色。大多数的研究表明,胶原蛋白基因的高表达促进了癌细胞的增殖和肿瘤的发生。Liu等发现,COL1A1在宫颈癌组织中的表达水平显著增加,并抑制了辐射诱导的宫颈癌细胞凋亡,而COL5A1的过度表达诱导了肺癌的转移。COL1A1在结直肠癌中高表达,是一种潜在的诊断性生物标志物和治疗靶点。COL3A1和POSTN在食管癌中高表达,与病理分期呈正相关,可能在食管癌的发生和发展中起重要作用。而COL12A1在结肠直肠癌中高表达,提示预后不良,可作为其预后的生物标志物。COL1A2
、COL6A3
和THBS2
基因沉默可通过PI3K-Akt信号通路抑制胃癌细胞增殖、迁移和侵袭,同时促进细胞凋亡。I型胶原还破坏细胞间钙黏蛋白,增加胰腺癌细胞的迁移,破坏PTHrP、IL-6和IL-8的表达。本研究发现COL1A1、COL1A2和COL6A3在胃癌组织中的表达显著高于正常胃组织,而生存结果分析显示COL1A1和COL6A3高表达与预后不良相关。对不同种族的基因表达分析和生存分析显示,COL1A1、COL1A2和COL6A3在不同种族胃癌中表达均上调。尽管COL2A1在GEO数据集中显著上调,但在肿瘤数据库中,COL2A1在胃癌和正常组织之间的表达无显著差异。功能富集分析表明,COL2A1可能在肿瘤细胞外基质与受体的相互作用和局灶性黏附过程中起作用。提示COL2A1可能是一个新的潜在诊断胃癌的生物标志物和治疗靶点。
如上文所述,胶原蛋白是肿瘤细胞外基质的主要成分,因此在胃癌组织中发现了多种不同胶原蛋白类型。Sun证实了与正常组织相比,COL1A1在胃癌中表达上调,表明其在胃癌细胞侵袭和转移中发挥了重要作用。Guo等进一步发现COL1A1过表达可诱导TGF-β信号通路激活,促进胃癌细胞增殖和迁移。为了探讨胃癌发生、发展和多样性的潜在机制,Hippo等在22例原发性人类晚期胃癌组织和8例正常组织中进行了高密度寡核苷酸芯片研究,揭示了一些基因在癌组织中高表达,即与细胞周期、生长因子、细胞运动、细胞黏附和基质重塑等相关的基因。在异常表达的基因中,发现胃癌组织中胶原降解增加,而胶原降解是肿瘤细胞侵袭周围组织的重要步骤。Yasui等报道COL1A1和COL1A2是胃癌的新生标记物,因为它们在胃癌中普遍过表达,与侵袭和转移相关。此外,一项基于Oncomine数据库的分析发现,在mRNA转录水平上,与正常胃组织相比,COL1A2
基因在胃癌组织中高表达;进行在线生存分析发现,与COL1A2
基因低表达组相比,高表达组的胃癌患者总体生存期降低;进一步分析发现,COL1A2
基因高表达的肠型、弥漫型胃癌患者总体生存期降低。胶原蛋白家族的另一成员COL10A1受miR-26a-5p调控,并在胃癌细胞中高表达,以促进癌细胞的增殖、迁移和侵袭。Jiang等通过定量PCR和免疫组织化学检测,发现COL12A1在胃癌组织中表达增加,与肿瘤侵袭、转移和临床晚期显著相关。重要的是,COL12A1表达增加导致总生存率降低,多变量Cox分析表明COL12A1高表达是胃癌患者一个强有力的独立预后指标。敲低胃癌细胞SGC-7901中COL12A1的表达水平后,细胞的生长和增殖能力被显著抑制,细胞的侵袭能力也显著降低,表明COL12A1具有促进细胞生长和侵袭能力的效应,提示其可能在胃癌的生长和侵袭转移中发挥了重要作用。另外,miR-29被认为是多种恶性肿瘤的抑 癌基因。Qi等通过Meta分析评估了miR-49在多种人类恶性肿瘤中的预后价值,并指出miR-29的低表达与恶性肿瘤的侵袭性和预后不良有关,可能成为预测人类恶性肿瘤复发和进展的关键生物标志物。Cushing等报道了miR-29通过抑制ECM,包括胶原蛋白,以及调节胶原合成、交联和降解的酶发挥作用。为了证实miR-29a在癌细胞增殖和侵袭中的功能作用,Muniyappa等对人工改变miR-29a表达的细胞进行了2D-DIGE蛋白质组学分析,发现了几种候选蛋白,包括RAS癌基因家族成员RAN
,显著降低了癌细胞的侵袭能力。核糖体蛋白S15a(RPS15A)被证实通过激活Akt/IKK-β/NF-κB信号通路促进胃癌的进展,并且通过沉默RPS15A抑制胃癌细胞的增殖和迁移。Zhang等发现miR-29s直接结合到RPS15A的3′-UTR,并通过抑制肝细胞中RPS15A的表达抑制细胞生长和细胞周期。An等发现miR-29a-3p可以结合到COL1A1并抑制其蛋白表达,并证实了circKRT7通过EMT途径吸收miR-29a-3p,可促进卵巢癌细胞的增殖和转移。在本研究中,miR-29s被认为是多种胶原基因的调节因子,可能在胃癌的发生发展中起到癌基因的作用。miR-29-3p在鼻咽癌细胞中直接结合和调节COL1A1,被证实介导癌细胞的放射敏感性。在结直肠癌中,干扰素-γ通过将干扰素调节因子1(interferon regulatory factor 1,IRF-1)吸引到miR-29b启动子区域的结合位点来增加miR-29b的表达水平,并且miR-29b增加了IRF-1的表达,形成了一个正的IRF1/miR-29b反馈环。此外,IGF1被证实为miR-29b的靶点,而IGF1是PI3K/Akt信号通路的激活因子,miR-29b可通过阻断PI3K/Akt信号通路从而抑制结直肠癌细胞生长和侵袭。Wang等证实了miR-29b对肺动脉平滑肌细胞胶原Ⅰ合成的调节作用,预测了miR-29s在ECM胶原沉积中的重要作用。此外,Yu等研究发现,miR-29c在胃癌组织和细胞系中下调,miR-29c过表达可抑制细胞增殖,促进细胞凋亡,阻滞G1/G0期细胞周期。van Rooij等发现miR-29是一群编码纤维化相关蛋白(如胶原蛋白、纤维蛋白和弹性蛋白)的miRNAs,上调miR-29的表达可以降低体内外胶原蛋白的表达。在肿瘤微环境中,miR-29可能通过不同的分子生物学过程参与胶原蛋白的调控。然而,目前miR-29家族的研究尚在初级阶段,其在肿瘤学研究中的意义有待挖掘,miR-29家族与胶原蛋白家族的相互调控机制尚需进一步阐明。
综上所述,本研究在胃癌组织中发现了一组胶原基因,其在胃癌组织中高表达,与胃癌患者的预后密切相关,在胃癌中具有潜在诊断作用,可作为新的治疗靶点,且其表达和相关生物学过程可能受miR-29s的调控。
