转录因子OsMADS25提高水稻对低温的耐受性
2021-11-27闫凌月张豪健郑雨晴丛韫起刘次桃樊帆郑铖袁贵龙潘根袁定阳段美娟
闫凌月,张豪健,郑雨晴,丛韫起,刘次桃,樊帆,郑铖,袁贵龙,潘根,袁定阳,段美娟
研究报告
转录因子OsMADS25提高水稻对低温的耐受性
闫凌月1,张豪健1,郑雨晴1,丛韫起1,刘次桃1,樊帆1,郑铖1,袁贵龙2,潘根3,袁定阳2,段美娟1
1. 湖南农业大学农学院,水稻逆境生物学湖南省重点实验室,长沙 410128 2. 湖南杂交水稻研究中心,杂交水稻国家重点实验室,长沙 410125 3. 中国农业科学院麻类研究所,长沙 410205
低温冷害是影响水稻高产的关键环境因素,鉴定和克隆具有重要应用价值的耐低温基因并培育耐低温新品种对于保障粮食安全具有重要意义。MADS转录因子在植物逆境信号途径中扮演着重要的角色。本研究利用qRT-PCR检测,发现受低温和脱落酸(abscisic acid, ABA)诱导表达上调,预示可能参与ABA依赖的逆境信号途径。进一步构建了水稻的过表达载体pCambia1300-221--Flag,利用根癌农杆菌介导的遗传转化法将其导入水稻品种中花11 (ZH11),选取两个表达量高的纯合株系进行表型鉴定。结果表明,过表达株系显著提高了水稻苗期对低温的耐受性以及对ABA的敏感性。利用3,3-二氨基联苯胺(diaminobezidine, DAB)和氯化硝基四氮唑蓝(nitrotetrazolium blue chloride, NBT)组织化学染色结果表明:低温处理后,过表达株系比野生型ZH11染色浅,表明过表达株系在低温胁迫下积累的活性氧(reactive oxygen species, ROS)相对较少,增强了对低温的耐受性。综合结果表明,低温逆境下响应ABA信号,通过提高水稻对ROS的清除能力,避免水稻受到低温伤害。
水稻;;脱落酸;活性氧;耐低温
水稻(L.)是最主要粮食作物,世界上有一半以上的人口以水稻为主食[1]。相比其他主粮作物如小麦(L.)、大麦(L.)等,水稻对低温更加敏感[2]。世界上约有24个国家如中国、日本、朝鲜等遭遇过低温冷害问题[3]。低温对水稻的影响主要发生在幼苗期和生殖生长期。例如在我国的华南和长江中下游双季稻区早稻苗期遇到“倒春寒”,晚稻抽穗扬花期遇到“寒露风”低温灾害气候,导致水稻大幅减产[4~7]。低温灾害使我国每年粮食减产30~50亿公斤[8]。低温严重制约水稻的产量,因此挖掘耐低温新基因及培育耐低温新品种是水稻育种的重要研究方向。
植物耐受低温胁迫是一个复杂的遗传性状,受多个基因/数量性状基因座(quantitative trait locus, QTL)控制。与其他农艺性状相比,水稻低温耐受性的遗传研究进展缓慢,在过去几十年内,通过正向遗传学方法鉴定出的耐低温基因相对有限,包括8个QTL定位的基因和,2个由全基因组关联分析(genome-wide association study, GWAS)定位的基因和。编码一个富含甘氨酸(GRP)的保守结构域,第17位氨基酸为赖氨酸(Lys)基因型表现为低温下种子萌发率增强;而第17位氨基酸为组氨酸(His)基因型则表现为穗期耐低温[9,10]。编码G蛋白信号调节因子,其与G蛋白α亚基RGA1互作,促进Ca2+流入细胞内,并增强G蛋白的GTP酶活性,进而增强水稻对低温的耐受性[11]。过表达提高水稻对低温的耐受性[12]。第99位氨基酸为异亮氨酸(Ile)时,对提高水稻苗期耐低温起重要作用[13]。编码氧化酶,催化生物活性的茉莉酰基-L-异亮氨酸(JA-Ile)转化为无活性形式的12-羟基-JA-Ile (12OH- JA-Ile),并通过调节茉莉酸(jasmonic acid, JA)含量来调控水稻对低温的耐受性[14]。编码酪蛋白激酶I (casein kinase I),提高低温下水稻的生长速率及产量[15]。编码一个含有F-box结构域的蛋白,其与E3泛素连接酶Skp1互作,参与泛素-蛋白酶体的低温信号传导途径[16]。编码一个保守的富亮氨酸受体激酶LRR-RLK (leucine-rich repeat receptor- like kinase),其与ATP合成酶的β亚基AtpB互作,并在低温下复合体通过影响ATP合成酶的活性,从而影响水稻灌浆期的能量供应,进而提高水稻抽穗期对低温的耐受性[3]。bZIP转录因子,与另一个bZIP蛋白bZIP71互作来调节水稻体内植物激素脱落酸(abscisic acid, ABA)、活性氧(reactive oxygen species, ROS)和可溶性糖水平,从而提高水稻苗期和抽穗期对低温的耐受性[10,17]。在343位的核苷酸为G时,表现出在苗期和抽穗期都耐低温[18]。
植物响应低温逆境信号的基因可分为有两大类:第一类是功能基因,其编码产物在植物受逆境胁迫时直接起保护作用,例如渗透保护剂合成酶基因、抗氧化酶类等;第二类是调节基因,其编码产物通过调节功能基因的协同表达,从而使植物避免受到逆境的伤害,例如蛋白激酶类和转录因子[19]。一个转录因子可以调控一类相关性状的很多基因,从而有效改变植物的相关特性。因此,转录因子往往是植物耐受逆境(包括低温)等的主效基因。因此,从一些关键调节因子入手,可促使多个胁迫应答基因发挥作用。
MADS转录因子广泛参与激素和非生物逆境胁迫应答。例如拟南芥()开花时间基因的突变提高植物对低温的耐受性[20];拟南芥通过调节ABA信号基因的表达来调节植物对渗透胁迫的耐受性[21]。小麦(L.)受条锈菌感染后表达上调[22]。水稻负调节水稻对病原体的抗性和对干旱的耐受性[23],而参与调节水稻对高温的敏感性[24]。最近研究表明,通过影响硝酸盐的积累以及调节生长素的方式调节水稻根系发育,进而提高水稻对盐的耐受性[25,27]。然而是否响应低温逆境胁迫仍不清楚。为了研究是否响应低温逆境信号途径,本文探究了过表达转基因株系对逆境关键激素ABA的响应,以及转基因株系在低温下的存活率、下游调控基因和清除ROS的能力,揭示响应水稻低温胁迫的可能分子机制。
1 材料与方法
1.1 材料
水稻粳稻品种ZH11和日本晴(Nipponbare),根癌农杆菌()菌株AGL0、大肠杆菌()菌株DH5α由湖南农业大学农学院湖南省逆境生物学重点实验和湖南杂交水稻研究中心提供。pCambia1300-221-Flag质粒由中国科学院遗传与发育生物学研究所储成才研究员惠赠。高保真DNA聚合酶(KOD FX酶)购于TOYOBO(日本)公司;酶、限制性内切酶、T4 连接酶等购于TaKaRa (日本)公司;DEPC以及组培试剂购于Sigma (美国)公司;RNA提取Trizol购于Invitrogen (美国)公司;反转录试剂盒购于TOYOBO (日本)公司;质粒提取、胶回收试剂盒购于TIANGEN (中国)公司;3,3-二氨基联苯胺(diaminobezidine, DAB)和氯化硝基四氮唑蓝(nitrotetrazolium blue chloride, NBT)购于Solarbio (中国)生物公司。引物合成和测序由擎科生物技术有限公司(中国)完成。
1.2 OsMADS25过表达载体的构建
提取日本晴两周龄幼苗总RNA,经反转录获得cDNA。以cDNA为模板,以引物F:5ʹ- CTAGATGGGGAGAGGGAAGATTGC-3ʹ和R:5ʹ-ACGCTTCATCTTCAACTTCTTTTTGACTCA-3ʹ (下划线为酶切位点),高保真酶KOD FX (TOYOBO公司,日本)进行PCR扩增,获得() CDS序列。用I和I酶切PCR纯化产物和双元表达载体pCambia1300-221-Flag质粒(构建过程如图1所示),利用T4连接酶连接,转入大肠杆菌DH5α体内,提取质粒。经酶切鉴定正确的质粒由美国Invitrogen公司测序。将测序正确的质粒,转化于根癌农杆菌,成功转化到农杆菌的菌落再浸染ZH11的愈伤组织,经潮霉素筛选获得过表达的阳性转基因植株。
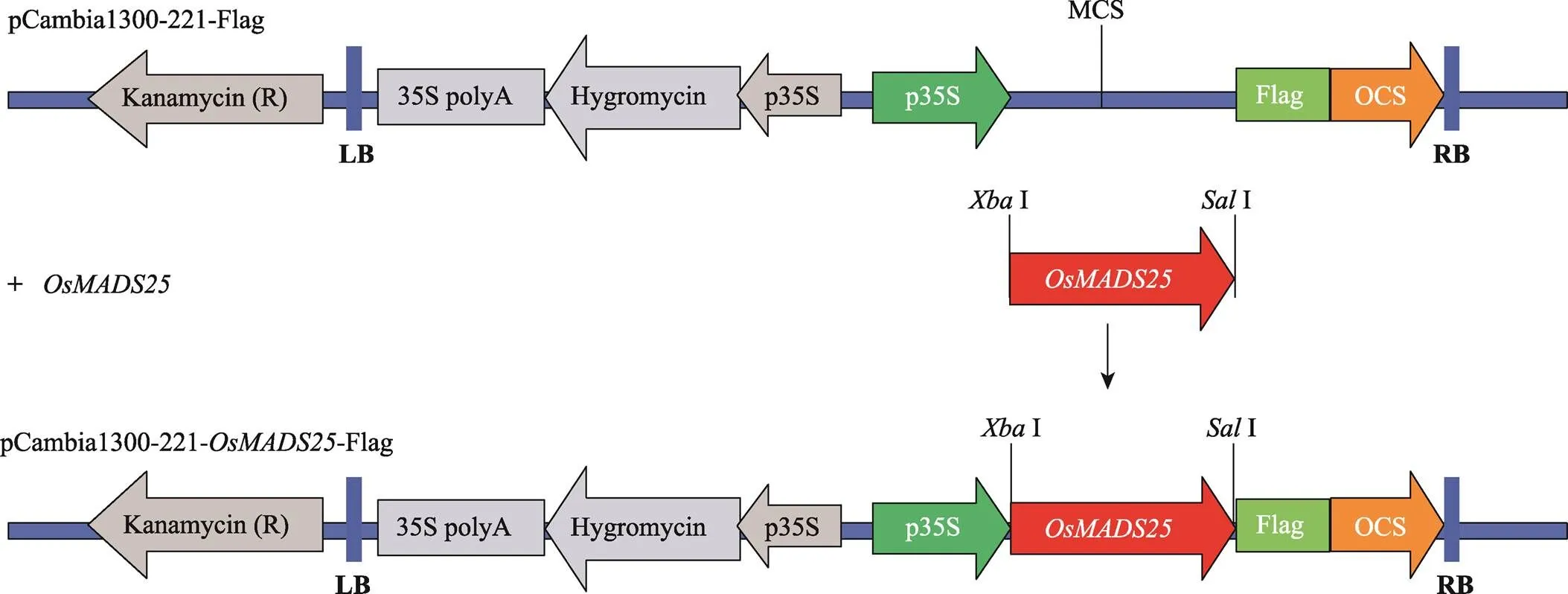
图1 OsMADS25过表达载体构建示意图
1.3 逆境诱导表达谱分析
对两周龄的ZH11分别进行低温逆境(4℃)和ABA (100 μmol/L)处理,以正常条件为对照,分别于0、0.5、3、6、12、24 h收集根和苗,按照美国Invitrogen公司RNA提取Trizol说明书提取总RNA,按照日本TOYOBO公司反转录试剂盒操作说明进行反转录,获得cDNA,以()为内参,qRT-PCR检测不同时间点和不同部位在RNA水平的表达量。引物序列见表1。
1.4 转基因株系低温耐受性检测
转基因株系及野生型ZH11种子放入鼓风干燥箱箱(42℃,7 d)以破除休眠,放入37℃恒温箱浸种2 d,露白后的种子移至剪孔型的PCR板中,用IRRI 营养液(International Rice Research Institute Protocol, http://irri.org)培养至三叶一心时期,在光照培养箱(Percival公司,美国)进行低温(4℃)处理,70%相对湿度、14 h白天/10 h夜晚。低温处理3 d后,恢复至正常温度(28±2℃),统计成活率。每个处理120棵苗,3个平行重复。利用SAS软件(http://www.sas.com/)进行统计分析。
1.5 DAB和NBT染色
DAB和NBT染色分别用于检测过表达株系及野生型ZH11对照在低温处理后植株中双氧水(H2O2)和超氧阴离子(O2–)含量。
DAB染色采用Solarbio生物公司DAB浓缩型试剂盒。NBT染色:称取100 mg NBT加入到100 mL 10 mmol/L的磷酸缓冲液(0.2 mol/L KH2PO45 mL、0.2 mol/L NaOH 4.52 mL、pH=7.8)中,充分搅拌至NBT完全溶解。两者均过夜染色,染色过程中避光,95%酒精清洗叶片,并在沸水中煮10 min,以至叶片褪色,拍照。
1.6 ABA敏感性检测
过表达株系及野生型ZH11种子于37℃浸种2 d,将露白的种子放入铺有滤纸的培养皿中,分别用含0 μmol/L、5 μmol/L、10 μmol/L ABA的水溶液处理10 d,测量苗的长度。实验在人工气候室进行(28℃、70%相对湿度,14 h白天/10 h夜间光周期)。每个平皿30棵苗,每个处理3个平行重复,用SAS软件进行统计分析。
1.7 OsMADS25下游基因的检测
提取两周龄过表达株系及野生型ZH11幼苗的总RNA,经反转录合成cDNA第一条链,qRT-PCR检测过表达株系和野生型ZH11中低温响应基因()、()和()的相对表达量,以()为内参。引物序列见表1。

表1 qRT-PCR检测引物
2 结果与分析
2.1 OsMADS25受低温和ABA诱导
对三叶一心时期(约两周龄)的野生型ZH11进行低温(4℃)逆境和逆境激素ABA处理,为区别是否响应光周期,本研究设置了正常培养条件作为对照。结果显示:经ABA处理,野生型ZH11根和地上部分的表达量上调;在根中仅0.5 h就表现出上调,在6 h表达量达到峰值(图2);在地上部分,在6 h表达上调,在12 h表达量达峰值。经低温处理,的表达量仅在根中12~24 h上调(图2)。根是植物最先感知和响应逆境的部位,经ABA处理后,表达量在根中增加的速度比地上部分要快(图2)。结果表明,受低温和ABA诱导表达上调,预示着可能参与响应ABA依赖的低温信号途径。
2.2 OsMADS25提高水稻对低温的耐受性
受低温诱导表明它可能参与低温逆境信号。为了鉴定在水稻中是否参与对低温耐受性的调节,本研究通过对过量表达的T3纯合株系中相对表达量的检测,选取相对表达量在转录水平上表达量高且差异显著的两个纯合株系OE-1和OE-3进行表型鉴定(图3 B)。结果表明,在低温胁迫下,过表达株系OE-1和OE-3的成活率分别比野生型ZH11高53%和38% (图3:A,C)。过表达株系极大的提高了水稻在低温下的成活率;且相对表达量高的株系OE-1的成活率显著高于OE-3 (图3:B,C),表明对低温的耐受性与相对表达量呈正相关。
2.3 OsMADS25提高水稻对ABA的敏感性
正调控水稻对低温的耐受性,且受ABA诱导表达上调,表明可能参与ABA依赖的低温信号途径。为探究是否参与ABA信号,本研究对过表达株系(OE-1和OE-3)及野生型ZH11进行ABA敏感性测试。结果表明:在5 µmol/L和10 µmol/L的ABA处理后,过表达株系都表现为苗长都显著比ZH11矮14%和6%以上(图4:C~F);而过表达株系与野生型ZH11在正常条件下无显著差异(图4:A,B)。过表达株系幼苗的生长受ABA抑制比野生型ZH11更强,这表明提高水稻对ABA的敏感性,预示了可能参与ABA依赖的低温逆境信号。
2.4 OsMADS25上调低温响应相关基因
低温逆境下,植物通过提高抵御渗透胁迫和清除ROS的能力来应对逆境胁迫[28]。本研究利用qRT-PCR检测了在低温逆境中参与渗透胁迫、清除ROS的相关基因在过表达转基因株系的表达情况。结果表明,和表达上调(图5)。影响生长素运输、合成和信号传导,正调控水稻在营养生长期的低温耐受能力[15]。影响渗透物质和ROS的积累,正向调控水稻对低温胁迫的耐受性[29]。过表达株系,提高水稻海藻糖的含量,调节渗透压,进而使水稻对多种胁迫产生抗性[30~32]。这些结果表明:可能通过上调和的表达,进而提高水稻耐低温的能力。
2.5 OsMADS25提高水稻对活性氧的清除能力
上调的表达(图5),而在逆境中发挥清除ROS的功能。另外,Xu等[26]报道通过激活下游基因谷胱甘肽转移酶4 (glutathione S–transferase 4,)和的表达,进而提高水稻在盐处理下清除ROS以及提高应对渗透胁迫的能力。那么参与低温逆境是否与耐盐有相似的生理机制?为了进一步探讨参与低温逆境的生理机制,本研究对转基因株系及野生型ZH11对照在低温(4℃)处理6 h后,进行DAB和NBT染色,检测叶片中H2O2及O2–的含量。结果表明:野生型ZH11的叶片染色深,而转基因株系OE-1、OE-3的叶片染色浅(图6),说明转基因株系中积累的活性氧H2O2及O2–的含量比ZH11少,由此说明可能通过提高水稻在低温下对ROS的清除能力进而提高对低温的耐受性。
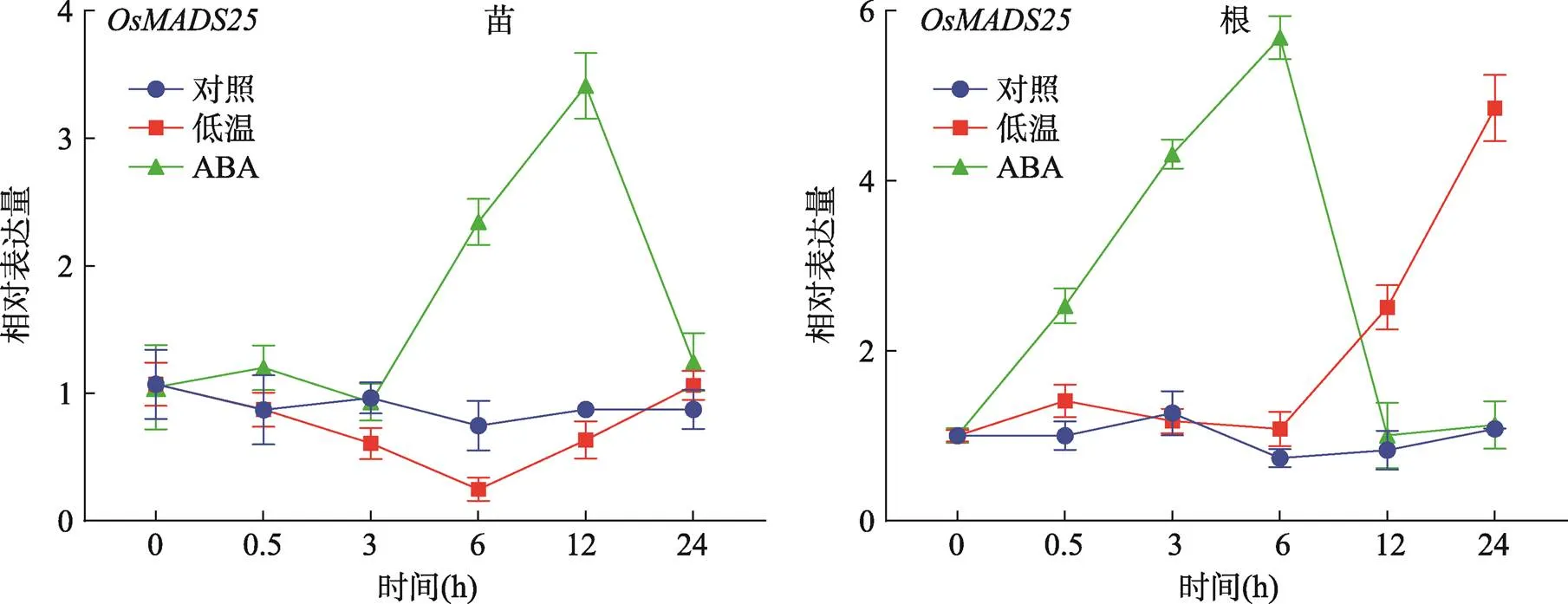
图2 野生型ZH11中OsMADS25受低温和ABA诱导表达上调
对照:两周龄的野生型ZH11在温室正常条件下培养;低温:两周龄的野生型ZH11在4℃光照培养箱处理;ABA:两周龄的野生型ZH11用含100 µmol/L ABA的IRRI营养液处理。

图3 OsMADS25提高水稻对低温的耐受性
A:低温(4℃)处理过表达株系及野生型对照ZH11。OE-1、OE-3为过表达T3代的两个纯合株系;比例尺:5 cm。B:过表达株系中在转录水平上的相对表达量。C:转基因株系成活率统计。统计分析采用SNK-q单因素多重比较分析,不同的字母表示具有显著差异(<0.01)。
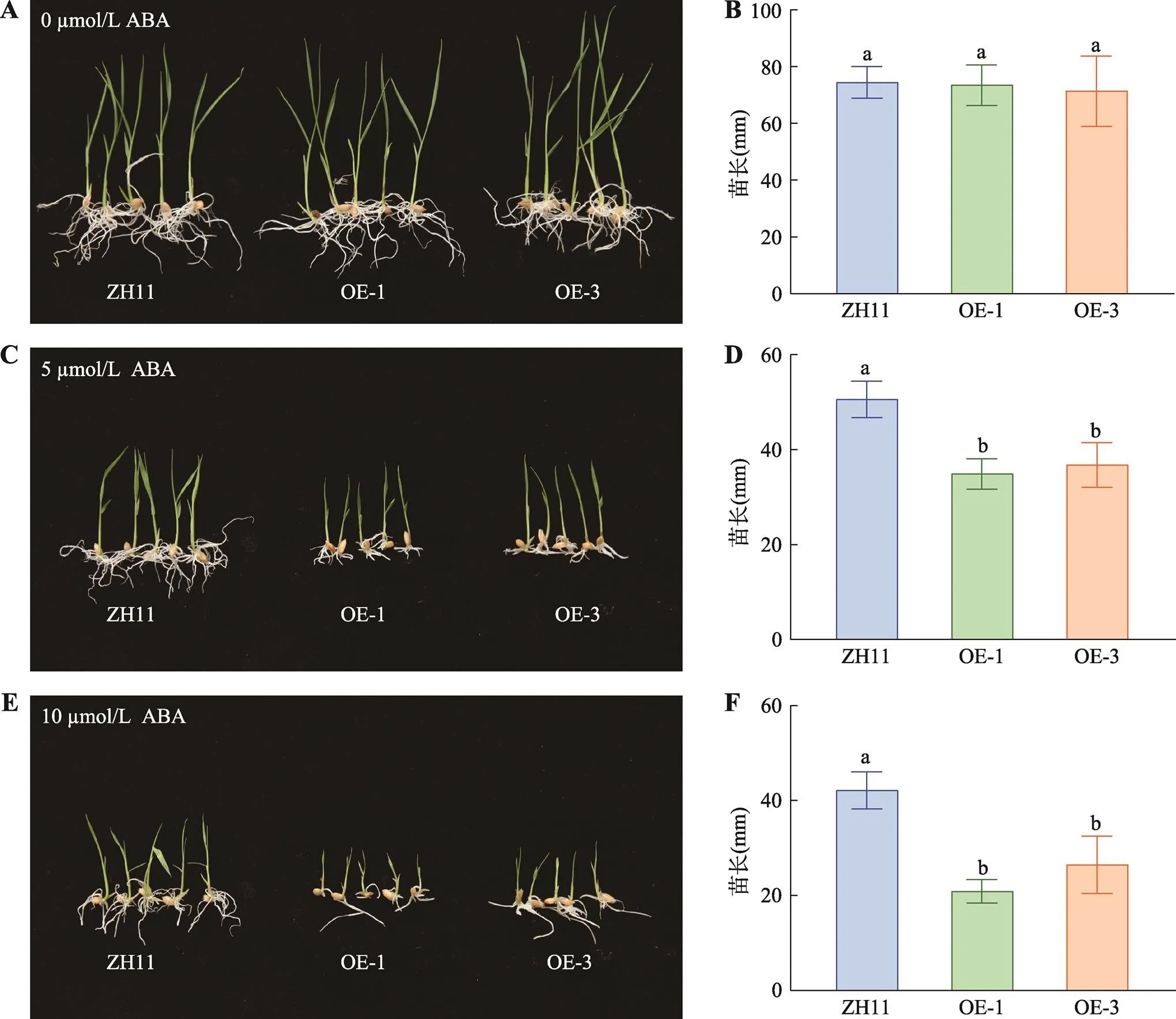
图4 OsMADS25过表达株系幼苗的生长受ABA抑制比野生型ZH11更强
A,B:0 µmol/L ABA处理过表达株系与野生型ZH11并在10 d后统计苗长;C,D:5 µmol/L ABA处理过表达株系与野生型ZH11并在 10 d后统计苗长;E,F:10 µmol/L ABA处理过表达株系与野生型ZH11并在 10 d后统计苗长。OE-1、OE-3为过表达T3代的两个纯合株系;统计分析采用SNK-q单因素多重比较分析,不同的字母表示具有显著差异(<0.01)。
3 讨论
()在水稻4号染色体13.6 Mb位置上,与Schläppi等[5]利用全基因组关联分析(genome-wide association study, GWAS)定位的水稻微核心种质资源群体(Minicore)苗期耐低温QTL的位置重叠。过表达提高了水稻对低温的耐受性,说明可能是耐低温QTL的主效基因。
过表达转基因的T3代纯合株系OE-1的表达量高于OE-3,对低温的耐受性也是OE-1高于株系OE-3,这表明正调控水稻对低温的耐受性。受低温和ABA诱导表达上调(图2),转基因株系提高了水稻对ABA的敏感性(图4),下游调控基因、受ABA诱导表达上调[29,31]。这些结果表明:参与ABA依赖的低温逆境信号。低温处理后,转基因株系NBT和DAB染色浅,ROS积累少,对低温耐受性强。Xu等[26]报道通过提高水稻在盐逆境下对ROS的清除能力,从而提高水稻的耐盐性。另外,的另一下游调控基因,在逆境具有调节渗透压的作用[30~32]。这说明在低温逆境下可能通过调控的表达,进而调节渗透压,提高水稻对低温的耐受性。这表明在耐盐和耐低温有相似的生理机制。综上所述,低温逆境时,转录因子响应ABA信号,通过调节的表达,进面提高水稻对ROS的清除能力;同时,通过调节的表达,进而调节渗透压,降低低温对水稻的伤害(图7)。是一个耐低温新的基因,其功能的鉴定对于丰富水稻耐低温基因资源,以及培育耐低温水稻新品种具有一定的意义。

图5 OsMADS25过表达株系提高耐低温相关基因的表达
ZH11为野生型对照;OE-1、OE-3为过表达T3代的两个纯合株系;统计方法为-test;**<0.01,差异极显著。

图6 OsMADS25提高水稻对ROS的清除能力
DAB染色表示植物在低温处理后H2O2的积累情况,NBT染色表示植物在低温处理后O2–的积累情况。ZH11为野生型对照;OE-1,OE-3为过表达T3代的两个纯合株系;比例尺:5 mm。

图7 OsMADS25提高水稻耐低温的可能机制
[1] Maclean JL, Dawe DC, Hardy B, Hettel GP. Rice almanac: source book for the most important economic activity on earth (3rd edition). Oxon, U.K.: CABI Pub., 2002.
[2] Gross BL, Zhao ZJ. Archaeological and genetic insights into the origins of domesticated rice.,2014, 111(17): 6190–6197.
[3] Zhang ZY, Li JJ, Pan YH, Li JL, Zhou L, Shi HL, Zeng YW, Guo HF, Yang SM, Zheng WW, Yu JP, Sun XM, Li GL, Ding YL, Ma L, Shen SQ, Dai LY, Zhang HL, Yang SH, Guo Y, Li ZC. Natural variation inenhances rice adaptation to cold habitats.,2017, 8: 14788.
[4] Wang D, Liu JL, Li CG, Kang HX, Wang Y, Tan XQ, Liu MH, Deng YF, Wang ZL, Liu Y, Zhang DY, Xiao YH, Wang GL. Genome-wide association mapping of cold tolerance genes at the seedling stage in rice.,2016, 9(1): 61.
[5] Schläppi MR, Jackson AK, Eizenga GC, Wang AJ, Chu CC, Shi Y, Shimoyama N, Boykin DL. Assessment of five chilling tolerance traits and GWAS mapping in rice using the USDA Mini-Core collection.,2017, 8: 957.
[6] Zhao JL, Zhang SH, Yang TF, Zeng ZC, Huang ZH, Liu Q, Wang XF, Leach J, Leung H, Liu B. Global transcriptional profiling of a cold-tolerant rice variety under moderate cold stress reveals different cold stress response mechanisms.,2015, 154(3): 381–394.
[7] Pan YH, Zhang HL, Zhang DL, Li JJ, Xiong HY, Yu JP, Li JL, Rashid MAR, Li GL, Ma XD, Cao GL, Han LZ, Li ZC. Genetic analysis of cold tolerance at the germination and booting stages in rice by association mapping.,2015, 10(3): e0120590.
[8] Zhu YJ, Chen K, Mi XF, Chen TX, Ali J, Ye GY, Xu JL, Li ZK. Identification and fine mapping of a stably expressed QTL for cold tolerance at the booting stage using an interconnected breeding population in rice.,2015, 10(12): e0145704.
[9] Fujino K, Sekiguchi H, Matsuda Y, Sugimoto K, Ono K, Yano M. Molecular identification of a major quantitative trait locus,, controlling low-temperature germinability in rice.,2008, 105(34): 12623–12628.
[10] Liu CT, Schläppi MR, Mao BG, Wang W, Wang AJ, Chu CC. The bZIP73 transcription factor controls rice cold tolerance at the reproductive stage.,2019, 17(9): 1834–1849.
[11] Ma Y, Dai XY, Xu YY, Luo W, Zheng XM, Zeng DL, Pan YJ, Lin XL, Liu HH, Zhang DJ, Xiao J, Guo XY, Xu SJ, Niu YD, Jin JB, Zhang H, Xu X, Li LG, Wang W, Qian Q, Ge S, Chong K.confers chilling tolerance in rice.,2015, 160(6): 1209–1221.
[12] Zhao JL, Zhang SH, Dong JF, Yang TF, Mao XX, Liu Q, Wang XF, Liu B. A novel functional gene associated with cold tolerance at the seedling stage in rice.,2017, 15(9): 1141–1148.
[13] Kim SI, Andaya VC, Tai TH. Cold sensitivity in rice (L.) is strongly correlated with a naturally occurring I99V mutation in the multifunctional glutathione transferase isoenzyme GSTZ2.,2011, 435(2): 373–380.
[14] Mao DH, Xin YY, Tan YJ, Hu XJ, Bai JJ, Liu ZY, Yu YL, Li LY, Peng C, Fan T, Zhu YX, Guo YL, Wang SH, Lu DP, Xing YZ, Yuan LP, Chen CY. Natural variation in thegene confers chilling tolerance in rice and allowed adaptation to a temperate climate.,2019, 116(9): 3494–3501.
[15] Lu GW, Wu FQ, Wu WX, Wang HJ, Zheng XM, Zhang YH, Chen XL, Zhou KN, Jin MN, Cheng ZJ, Li XY, Jiang L, Wang HY, Wan JM. Riceis involved in adaptive growth and fitness under low ambient temperature.,2014, 78(3): 468–480.
[16] Saito K, Hayano-Saito Y, Kuroki M, Sato Y. Map-based cloning of the rice cold tolerance gene.,2010, 179(1-2): 97–102.
[17] Liu CT, Ou SJ, Mao BG, Tang JY, Wang W, Wang HR, Cao SY, Schläppi MR, Zhao BR, Xiao GY, Wang XP, Chu CC. Early selection offacilitated adaptation ofrice to cold climates.,2018, 9(1): 3302.
[18] Xiao N, Gao Y, Qian HJ, Gao Q, Wu YY, Zhang DP, Wang Z, Zhang XX, Yu L, Li YH, Pan CH, Liu GQ, Zhou CH, Jiang M, Huang NS, Dai ZY, Liang CZ, Chen Z, Chen JM, Li AH. Identification of genes related to cold tolerance and a functional allele that confers cold tolerance.,2018, 177(3): 1108–1123.
[19] Yang SJ, Vanderbeld B, Wan JX, Huang YF. Narrowing down the targets: towards successful genetic engineering of drought-tolerant crops.,2010, 3(3): 469–490.
[20] Seo E, Lee H, Jeon J, Park H, Kim J, Noh YS, Lee I. Crosstalk between cold response and flowering inis mediated through the flowering-time geneand its upstream negative regulator.,2009, 21(10): 3185–3197.
[21] Yu LH, Wu J, Zhang ZS, Miao ZQ, Zhao PX, Wang Z, Xiang CB.MADS-box transcription factor AGL21 acts as environmental surveillance of seed germination by regulatingexpression.,2017, 10(6): 834–845.
[22] Wei B, Cai T, Zhang RZ, Li AL, Huo NX, Li S, Gu YQ, Vogel J, Jia JZ, Qi YJ, Mao L. Novel microRNAs uncovered by deep sequencing of small RNA transcriptomes in bread wheat (L.) and(L.) Beauv.,2009, 9(4): 499–511.
[23] Khong GN, Pati PK, Richaud F, Parizot B, Bidzinski P, Mai CD, Bès M, Bourrié I, Meynard D, Beeckman T, Selvaraj MG, Manabu I, Genga AM, Brugidou C, Nang Do V, Guiderdoni E, Morel JB, Gantet P.negatively regulates resistance to pathogens and drought tolerance in rice.,2015, 169(4): 2935–2949.
[24] Chen C, Begcy K, Liu K, Folsom JJ, Wang Z, Zhang C, Walia H. Heat stress yields a unique MADS box transcription factor in determining seed size and thermal sensitivity.,2016, 171(1): 606–622.
[25] Zhang GP, Xu N, Chen HL, Wang GX, Huang JL.regulates root system development via auxin signalling in rice.,2018, 95(6): 1004–1022.
[26] Xu N, Chu YL, Chen HL, Li XX, Wu Q, Jin L, Wang GX, Huang JL. Rice transcription factor OsMADS25 modulates root growth and confers salinity tolerance via the ABA-mediated regulatory pathway and ROS scavenging.,2018, 14(10): e1007662.
[27] Yu CY, Liu YH, Zhang AD, Su S, Yan A, Huang LL, Ali I, Liu Y, Forde BG, Gan YB. MADS-box transcription factor OsMADS25 regulates root development through affection of nitrate accumulation in rice.,2015, 10(8): e0135196.
[28] Liu CT, Wang W, Mao BG, Chu CC. Cold stress tolerance in rice: physiological changes, molecular mechanism, and future prospects., 2018, 40(3): 171–185.刘次桃, 王威, 毛毕刚, 储成才. 水稻耐低温逆境研究: 分子生理机制及育种展望. 遗传, 2018, 40(3): 171–185.
[29] Ke YG, Yang ZJ, Yu SW, Li TF, Wu JH, Gao H, Fu YP, Luo LJ. Characterization ofresponsive to osmotic and cold stresses in rice.,2014, 57(3): 150–161.
[30] Habibur Rahman Pramanik M, Imai R. Functional identification of a trehalose 6-phosphate phosphatase gene that is involved in transient induction of trehalose biosynthesis during chilling stress in rice.,2005, 58(6): 751–762.
[31] Ge LF, Chao DY, Shi M, Zhu MZ, Gao JP, Lin HX. Overexpression of the trehalose-6-phosphate phosphatase geneconfers stress tolerance in rice and results in the activation of stress responsive genes.,2008, 228(1): 191–201.
[32] Zhang ZY, Li JH, Li F, Liu HH, Yang WS, Chong K, Xu YY. OsMAPK3 phosphorylates OsbHLH002/OsICE1 and inhibits its ubiquitination to activateand enhances rice chilling tolerance.,2017, 43(6): 731–743.e5.
Transcription factor OsMADS25 improves rice tolerance to cold stress
Lingyue Yan1, Haojian Zhang1, Yuqing Zheng1, Yunqi Cong1, Citao Liu1, Fan Fan1, Cheng Zheng1, Guilong Yuan2, Gen Pan3, Dingyang Yuan2, Meijuan Duan1
Cold stress is the limiting factor of rice growth and production, and it is important to clone cold stress tolerant genes and cultivate cold tolerance rice varieties. The MADS transcription factors play an important role in abiotic stress signaling in rice. This study showed thatwas up-regulated by low temperature and abscisic acid (ABA), suggesting thatmay be involved in ABA-dependent signaling. Theoverexpression vector, pCambia1300-221--Flag, was constructed and introduced into the rice variety Zhonghua 11 (ZH11) through-mediated genetic transformation. Two homozygous lines with high expression levels were selected for phenotypic identification.overexpression lines show significantly improved cold stress tolerance and the sensitivity to ABA at the seedling stage of rice.Reactive oxygen species (ROS) was detected by diaminobenzidine (DAB) staining and nitroblue tetrazolium (NBT) staining. After treatment with cold stress, little ROS accumulation was observed inoverexpression lines compared to wild-type ZH11. In conclusion,plays a role in scavenging reactive oxygen species (ROS) and could improve rice tolerance to cold stress involved in ABA-dependent pathway.
rice (L.);; abscisic acid (ABA); reactive oxygen species(ROS); cold tolerance
2021-06-20;
2021-08-21
湖南省科技重大专项(编号:2018NK1010),湖南省科技人才支持项目(编号:2019TJ-Q08),湖南农业大学人才引进启动基金(编号:20154/5407419002),杂交水稻国家重点实验室(湖南杂交水稻研究中心)开放课题(编号:2020KF05)和湖南省自然科学基金青年基金项目(编号:2019JJ50714)资助[Supported by the Hunan Science and Technology Major Project (No.2018NK1010), the Hunan Science and Technology Talents Support Project (No.2019TJ-Q08), the Research Initiation Fund of Hunan Agricultural University (No.20154/5407419002), the Open Research Fund of the State Key Laboratory of Hybrid Rice, Hybrid Rice Research Center (No.2020KF05) and the Hunan Province Natural Science Fund (No. 2019JJ50714)]
闫凌月,在读硕士研究生,专业方向:作物遗传育种。E-mail: yanlingyue0203@163.com
袁定阳,博士,研究员,研究方向:作物遗传育种。E-mail: yuandingyang@hhrrc.ac.cn
段美娟,博士,研究员,研究方向:作物遗传育种。E-mail: duanmeijuan@163.com
10.16288/j.yczz.21-217
2021/10/13 11:34:37
URI: https://kns.cnki.net/kcms/detail/11.1913.R.20211012.2042.002.html
(责任编委: 储成才)
