基于COⅠ基因的滆湖鲌类国家级水产种质资源保护区3种鲌类的遗传多样性分析
2021-11-19李大命刘洋唐晟凯刘燕山蒋琦辰何浩然沈冬冬张彤晴
李大命 刘洋 唐晟凯 刘燕山 蒋琦辰 何浩然 沈冬冬 张彤晴

摘要:为探究滆湖鲌类国家级水产种质资源保护区主要保护对象3种鲌类遗传资源状况,利用线粒体DNA COⅠ基因序列评价3种鲌类的遗传多样性及种群历史动态。通过PCR技术和测序技术,获得长度为630 bp COⅠ基因片段。序列分析结果表明,3种鲌类COⅠ基因序列碱基组成相似,且具有明显碱基组成偏向性,A+T的含量高于G+C的含量。翘嘴鲌的46条COⅠ基因片段发现4个变异位点,定义了4种单倍型(Hq1-Hq4),翘嘴鲌的单倍型和核苷酸多样性指数分别为0.422和0.00 085;蒙古鲌的20条COⅠ基因片段发现4个变异位点,定义4种单倍型(Hm1-Hm4),蒙古鲌的单倍型和核苷酸多样性指数分别为0.695和0.001 90;达氏鲌的41条COⅠ基因片段发现4个变异位点,定义5种单倍型(Hd1-Hd5),达氏鲌的单倍型和核苷酸多样性指数分别为0.230和0.000 53。中性检验和歧点分布分析显示,3种鲌类在历史进化过程中经历了不明显的种群扩张。整体来看,滆湖鲌类国家级水产种质资源保护区3种鲌类的遗传多样性水平较低,种群遗传组成趋于单一化,需要采取措施增加鲌类种群数量,提高其遗传多样性水平。
关键词:国家级水产种质资源保护区;遗传多样性;COⅠ基因;翘嘴鲌;蒙古鲌;达氏鲌
中图分类号:S917.4;S931 文献标志码: A
文章编号:1002-1302(2021)20-0177-05
收稿日期:2021-04-08
基金项目:江苏省水生生物资源重大专项暨首次水生野生动物资源普查(编号:ZYHB16-3);2019年淡水渔业资源监测(编号:2019-SJ-018-2);2020年渔业生态监测(编号:2020-SJ-016-2)。
作者简介:李大命(1981—),男,河南省汝南人,博士,副研究员,主要从事大水面渔业资源监测和评估研究。E-mail:ldm8212@126.com。
国家级水产种质资源保护区是依法予以特殊保护和管理的水产种质资源主要生长繁殖区域,以实现水产种质资源及其生存环境的保护和合理利用,水产种质资源保护区的建立对于保护水产种质资源起到重要作用[1]。多年来,受气候变化、过度捕捞、环境污染、工程建设的影响,水产种质资源保護区的生态环境及主要保护对象受到巨大威胁,亟待开展水产种质资源遗传状况的研究和保护[2]。滆湖鲌类国家级水产种质资源保护区位于江苏省常州市武进区西南处滆湖水域的北端,保护区面积为1 520 hm2,其中核心区面积509 hm2,实验区面积1 011 hm2。保护区特别保护期为全年,主要保护对象为翘嘴鲌(Culter alburnus)、蒙古鲌(Culter mongolicus)及达氏鲌(Culter dabryi)。迄今,尚无有关于保护区保护对象3种鲌类遗传状况的研究。
遗传多样性是评价物种资源状况的一个重要指标,是物种适应周围环境变化、维持生存和进化的物质基础[3]。因此,掌握物种的遗传多样性和遗传结构是开展生物种质资源保护和利用的前提和基础。鱼类的线粒体基因组是一种双链DNA分子,具有分子小、结构简单、编码效率高、母系遗传、重组率低、进化速率快等特点,已成为群体遗传学和系统学研究的重要分子标记[4-5]。COⅠ基因是位于线粒体DNA的蛋白质编码基因,其长度适宜,进化速度适中又相对保守,被广泛应用于鱼类物种鉴定及群体遗传学领域,研究结果为认识鱼类种质遗传资源提供了重要依据[6-9]。
本研究利用线粒体DNA的COⅠ基因序列,首次探讨滆湖鲌类国家级水产种质资源保护区主要保护对象的遗传多样性及遗传结构,研究结果可为掌握3种鲌类的种质资源遗传状况、科学保护及合理利用水产种质资源提供重要依据。
1 材料与方法
1.1 样品采集和处理
2019年7—11月和2020年5月、9月,在滆湖鲌类国家级自然保护区开展渔业资源调查。采用三层刺网采集鱼类样本,鱼类鉴定依据《江苏鱼类志》[10]。共采集翘嘴鲌46尾,蒙古鲌20尾,达氏鲌41尾,测定样本体长与体质量。现场剪取样本的肌肉组织,置于无水乙醇中保存,带回实验室用于提取基因组DNA。
1.2 DNA提取、PCR和测序
采用广谱性基因组提取试剂盒提取肌肉组织基因组DNA。利用琼脂糖凝胶电泳检验DNA完整性及紫外分光光度计测定DNA浓度。
COⅠ基因扩增引物为F1:5′-TCAACCAACCACAAAGACATTGGCAC-3′和R1:5′-TAGACTTCTGGGTGGCCAAAGAATCA-3′[11]。COⅠ基因的PCR体系为50.0 μL,其中,含有10×PCR Buffer (含 20 mmol/L Mg2+)5.0 μL,Taq DNA polymerase(5 U/μL)0.4 μL,dNTP(各2.5 mmol/L)4.0 μL,上、下游引物(10 μmol/L)各2.0 μL,DNA 模板1.0 μL(80 ng/μL),剩余体积用超纯水补足。PCR程序为:94 ℃预变性4 min;94 ℃变性40 s,55 ℃退火40 s,72 ℃延伸50 s,进行30个循环;最后72 ℃延伸5 min,4 ℃保存。
利用琼脂糖凝胶电泳对PCR产物进行检验,将合格的PCR产物送生工生物工程(上海)股份有限公司进行双向测序。
1.3 序列分析
使用BioEdit7.05.3软件[12]读取序列并辅以人工校对,利用Clustal X1.83软件[13]进行多序列比对。采用MEGA 7.0软件[14]计算碱基组成、变异位点及保守位点,计算种内及种间的遗传距离,采用邻接法(Neighbor-joining,NJ)构建3种鲌类单倍型的分子系统发育树。利用DnaSP 6.0软件[15]确定单倍型种类和个数,计算单倍型多样性指数(h)和核苷酸多样性指数(π),并进行Tajimas D和Fus Fs中性检验。
2 结果
2.1 COⅠ基因序列碱基组成
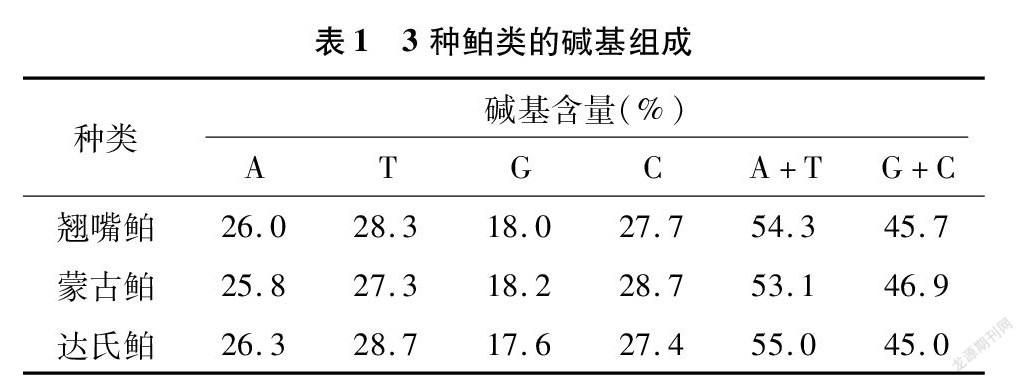
经比对和分析,获得3种鲌类COⅠ基因片段,长度为630 bp,无碱基插入或缺失。翘嘴鲌4种碱基A、T、G、C的平均含量分别为26.0%、28.3%、18.0%和27.7%。同样,蒙古鲌4种碱基的平均含量分别为25.8%、27.3%、18.2%和28.7%,达氏鲌4种碱基的平均含量分别为26.3%、28.7%、17.6%和27.4%。由表1可知,3种鲌类的碱基组成相近,碱基G的含量最低,碱基A+T含量高于 G+C 含量,表现出明显碱基偏向性,其中,达氏鲌的A+T含量(55.0%)最高,蒙古鲌的A+T含量(53.1%)最低。
2.2 群体遗传多样性
由表2可知,翘嘴鲌46条COⅠ基因序列有4个变异位点,其中,1个是单一信息位点,3个是简约信息位点,定义4种单倍型,单倍型多样性和核苷酸多样性分别为0.422和0.000 85;蒙古鲌20条 COⅠ 基因序列有4个变异位点,全部是简约信息位点,定义4种单倍型,单倍型多样性和核苷酸多样性分别为0.695和0.001 90;达氏鲌41条COⅠ基因序列有4个变异位点,其中,3个是单一信息位点,1个是简约信息位点,定义5種单倍型,单倍型多样性和核苷酸多样性分别为0.230和0.005 3。整体来看,3种鲌类的遗传多样性较低,其中蒙古鲌遗传多样性最高,达氏鲌遗传多样性最低。
翘嘴鲌群体有4种单倍型(Hq1~Hq4)组成,其中,单倍型Hq2数量最多,有34个,占群体比例为73.9%,单倍型Hq4有2个,单倍型Hq1和Hq3各有1个;蒙古鲌群体有4种单倍型(Hm1~Hm4)组成,单倍型Hm2数量最多,有10个,占群体的比例为50.0%,单倍型Hm1和Hm4各有4个,单倍型Hm3有2个;达氏鲌群体有5种单倍型(Hd1~Hd5)组成, 单倍型Hd1数量最多,有36个,占群体的比达87.8%,单倍型Hd4有2个,单倍型Hd2、Hd3和Hd5各有1个。
2.3 种内和种间遗传距离
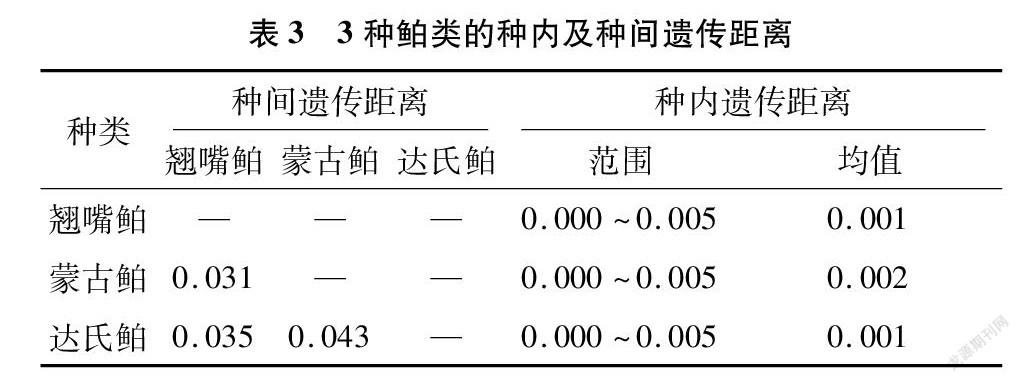
3种鲌类的种内遗传距离均为0.000~0.005,翘嘴鲌和达氏鲌的种内遗传距离均值为0.001,蒙古鲌的种内遗传距离均值为0.002。3种鲌类的种间遗传距离为0.031~0.043,其中,蒙古鲌和达氏鲌间的遗传距离最大,蒙古鲌和翘嘴鲌之间的遗传距离最小。种内遗传距离均小于2%,种间遗传距离为种内遗传距离的15.5~43.0倍(表3)。
2.4 分子系统发育
以鲤(Cyprinus carpio,MK843657.1)为外类群,基于NJ法构建鲌类的分子系统发育树,并应用Bootstrap(重复次数1 000)检验聚类树各分支置信度。由图1可知,3种鲌类均单独聚为独立分支,且节点的置信度水平较高,翘嘴鲌分支的支持率为99%,蒙古鲌和达氏鲌的分支支持率为100%,表明COⅠ基因可作为鲌类鉴定的有效分子标记[8]。同时可看出,达氏鲌和翘嘴鲌最先聚集在一起,然后与蒙古鲌聚集在一起,表明达氏鲌和翘嘴鲌的亲缘关系较近,与蒙古鲌的亲缘关系较远,与王伟的研究结果[16]一致。
2.5 群体进化历史
由表4可知,利用DnaSP 6.0对3种鲌类进行Tajiams D和FuFs中性检验,结果表明,3种鲌类的中性检验值均为负值,但统计结果不显著(P>0.1)。由图2可知,3种鲌类的歧点分布图中,歧点分布曲线呈明显的单峰型。
3 讨论
3.1 3种鲌类的遗传多样性
通常采用单倍型多样性指数(h)和核苷酸多样性指数(π)2个常用参数衡量物种遗传多样性的高低,h和π值越大,表明群体的遗传多样性越丰富[17]。本研究采用COⅠ基因片段,首次分析滆湖鲌类国家级水产种质资源保护区主要保护对象的遗传多样性。结果显示,翘嘴鲌、蒙古鲌和达氏鲌的单倍型多样性分别为0.422、0.695和0.230,核苷酸多样性分别为0.000 85、0.001 90和0.000 53。Grant和Bowen通过研究,将鱼类的单倍型多样性指数和核苷酸多样性指数大小的阈值分别设定为0.5和0.005[18]。本研究结果表明,3种鲌类中只有蒙古鲌的单倍型多样性大于0.5,表明保护区3种鲌类的遗传资源较为贫乏。与三峡库区翘嘴鲌(h:0.972 2~0.990 5,π:0.002 4~0.014 1)、蒙古鲌(h:0.866 7~1,π:0.002 2~0.002 4)、达氏鲌(h:0.969 0~1.000 0,π:0.033 0~0.071 5)[19]、长江中下游5个达氏鲌地理群体(h:0.412 3,π:0.001 1)[20]及长江流域湖北段和江苏段5个蒙古鲌群体(h:0.664,π:0.002)[21]的遗传多样性相比,同样表明保护区3种鲌类的遗传多样性指数较小。多年来,滆湖环境污染严重,蓝藻水华频繁发生,水生植物消失,涉水工程项目建设较多,加之过度捕捞等多种不利因素的影响,导致保护区鱼类的栖息
环境被破坏,鱼类的小型化、低龄化趋势加剧,种群资源量下降,遗传多样性丧失[22-25]。另外,受水利工程建设的影响,滆湖处于相对封闭状态,与周边水体的鱼类群体基因交流受阻,进一步导致滆湖鱼类种质资源遗传多样性下降[26]。
一般来说,鱼类的遗传多样性特征与进化历史密切相关。Grant和Bowen根据单倍型多样性和核苷酸多样性的关系,将群体遗传多样性分为4种模式:第一类模式为低h和低π(h<0.5,π<0.005),表明种群近期经历了瓶颈效应;第二类模式为高h和低π(h≥ 0.5,π<0.005),表示是受瓶颈效应后群体数量的迅速扩张导致;第三类模式为低h和高π(h<0.5,π ≥ 0.005),表示群体经历了轻微的瓶颈效应,但没有影响到核苷酸变异;第四类模式为高h和高π(h≥ 0.5,π≥0.005),表示群体稳定且具有比较悠久的进化历史[18]。本研究结果显示,翘嘴鲌和达氏鲌的单倍型多样性<0.5,核苷酸多样性<0.005,属于第一种类型;蒙古鲌单倍型多样性>0.5,核苷酸多样性<0.005,属于第二种类型,这表明3种鲌类近期均经历了瓶颈效应,群体尚未有足够长的时间积累遗传变异。3种鲌类的中性检验结果显示,Tajimas D 值和Fus Fs值均为负值,但统计结果不显著,表明进化过程中偏离了中性选择,发生轻微的有害突变或有害突变被清除[27-28]。3种鲌鱼的歧点分布图呈现单峰型[29],综合来看,表明3种鲌鱼进化历史上经历了不明显的种群扩张过程。
3.2 3种鲌类的遗传结构
Hebert等基于COⅠ序列,对动物界11个门、13 320 个物种进行了研究,结果显示,种内遗传距离多在2%以下[30]。本研究结果显示,翘嘴鲌、蒙古鲌和达氏鲌的种内遗传距离均为0.000~0.005,明显<2%,这说明在保护区内3种鲌类群体是一个自由交配的群体,群体内个体间存在广泛的基因交流。从单倍型组成来看,特定类型的单倍型在群体中占据明显优势,翘嘴鲌的优势单倍型Hq2有34个,占群体的比例为73.9%,蒙古鲌的优势单倍型Hm2有10个,占群体的比例为50%,达氏鲌的优势单倍型Hd1有36个,占群体的比例为87.8%,说明3种鲌类群体的遗传组成趋于单一化,这可能由于鲌类的群体数量较小,受遗传漂变的影响所致[31]。从保护生物学的角度来看,数量较少的单倍型容易丢失,对种群的遗传多样性产生较大影响,更需要加大保护力度。
3.3 鲌类种质资源保护
本研究显示,种质资源保护区的3种鲌类遗传多样性水平较低,种群遗传组成单一化,需要采取措施保护和修复鲌类资源,提高遗传多样性水平:(1)控制环境污染,治理蓝藻水华,改善鱼类的栖息环境;(2)构建水域生态牧场和建设人工鱼巢,为繁殖和孵化提供基质和空间;(3)开展增殖放流,补充鱼类资源量,优化鱼类遗传结构;(4)强化执法,严厉打击非法捕捞,保护鱼类繁殖和生长。
参考文献:
[1]郭子良,张曼胤,崔丽娟,等. 中国国家级水产种质资源保护区建设及其发展趋势分析[J]. 水生态学杂志,2019,40(5):112-118.
[2]盛 强,茹辉军,李云峰,等. 中国国家级水产种质资源保护区分布格局现状与分析[J]. 水产学报,2019,43(1):62-80.
[3]钱迎倩,马克平.生物多样性研究的原理与方法:生物多样性研究系列专著 1[M]. 北京:中国科学技术出版社,1994:13-36.
[4]肖武汉,张亚平.鱼类线粒体DNA的遗传与进化[J]. 水生生物學报,2000,24(4):384-391.
[5]袁 娟,张其中,罗 芬.鱼类线粒体DNA及其在分子群体遗传研究中的应用[J]. 生态科学,2008,27(4):272-276.
[6]彭居俐,王绪祯,王 丁,等. 基于线粒体COⅠ基因序列的DNA条形码在鲤科鲌属鱼类物种鉴定中的应用[J]. 水生生物学报,2009,33(2):271-276.
[7]张龙岗,杨 玲,李 娴,等. 利用mtDNA COⅠ基因序列分析引进的澳洲虫纹鳕鲈群体遗传多样性[J]. 水产学杂志,2013,26(2):14-18.
[8]王利华,罗相忠,王 丹,等. 基于COⅠ和Cytb DNA条形码在鲌属鱼类物种鉴定中的应用[J]. 淡水渔业,2019,49(4):22-28.
[9]李大命,孙文祥,许 飞,等. 高邮湖大银鱼、太湖新银鱼Cytb和COⅠ基因序列多态性分析[J]. 水产科学,2020,39(2):258-264.
[10]倪 勇,伍汉霖.江苏鱼类志[M]. 北京:中国农业出版社,2006:283-288.
[11]Ward R D,Zemlak T S,Innes B H,et al.DNA barcoding Australias fish species[J]. Philosophical Transactions of the Royal Society of London(Series B,Biological Sciences),2005,360(1462):1847-1857.
[12]Hall T A. BioEdit:A user-friendly biological sequence alignment editor and analysis program for Windows 95 /98 /NT[J]. Nucleic Acids Symp Ser,1999,41:95-98.
[13]Thompson J D,Gibson T J,Plewniak F,et al.The CLUSTAL_X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research,1997,25(24):4876-4882.
[14]Kumar S,Stecher G,Tamura K.MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution,2016,33(7):1870-1874.
[15]Rozas J,Ferrer-Mata A,Sánchez-Delbarrio J C,et al.DnaSP 6:DNA sequence polymorphism analysis of large data sets[J]. Molecular Biology and Evolution,2017,34(12):3299-3302.
[16]王 伟. 翘嘴鲌(Culter alburnus)群体遗传多样性及鲌亚科鱼类系统发生的研究[D]. 上海:华东师范大学,2007.
[17]沙 航,罗相忠,李 忠,等. 基于COⅠ序列的长江中上游鲢6个地理群体遗传多样性分析[J]. 中国水产科学,2018,25(4):783-792.
[18]Grant W,Bowen B.Shallow population histories in deep evolutionary lineages of marine fishes:insights from sardines and anchovies and lessons for conservation[J]. Journal of Heredity,1998,89(5):415-426.
[19]王 丹,程慶武,杨镇宇,等. 三峡库区鲌属鱼类线粒体COⅠ基因遗传多样性的初步分析[J]. 水生生物学报,2015,39(5):1054-1058.
[20]谢佳燕,颜 渊,杨钰慧.达氏鲌不同地理种群线粒体COⅠ基因遗传多态性的研究[J]. 江苏农业科学,2019,47(3):37-40.
[21]谢佳燕,颜 渊,杨钰慧,等. 基于线粒体COⅠ基因序列的蒙古鲌群体遗传结构分析[J]. 淡水渔业,2019,49(3):3-7.
[22]彭自然,陈立婧,江 敏,等. 滆湖水质调查与富营养状态评价[J]. 上海水产大学学报,2007,16(3):252-258.
[23]周 刚.滆湖水生植物生物量、演替规律及合理利用[J]. 湖泊科学,1997,9(2):175-182.
[24]童合一,刘其根,陈马康,等. 滆湖天然鱼类小型化及其对策[J]. 上海水产大学学报,1992,1(增刊2):124-135.
[25]唐晟凯,张彤晴,孔优佳,等. 滆湖鱼类学调查及渔获物分析[J]. 水生态学杂志,2009,30(6):20-24.
[26]张 迪,雷光春,龚 成,等. 基于COⅠ基因序列的太湖新银鱼遗传多样性[J]. 湖泊科学,2012,24(2):299-306.
[27]Fu Y X. Statistical tests of neutrality of mutations against population growth,hitchhiking and background selection[J]. Genetics,1997,147(2):915-925.
[28]Hickerson M J,Meyer C P. Testing comparative phylogeographic models of marine vicariance and dispersal using a hierarchical Bayesian approach[J]. BMC Evolutionary Biology,2007,8(3):322-340.
[29]Barbosa A M,Real R,Munoz A R,et al. New measures for assessing model equilibrium and prediction mismatch in species distribution models[J]. Diversity and Distributions,2013,19(10):1333-1338.
[30]Hebert P D N,Ratnasingham S,deWaard U R,et al. Barcoding animal life:cytochrome c oxidsse subunit 1 divergences among closely related species[J]. Proceeding of the Royal Society of London B:Biological Science,2003,270(suppl. 1):96-99.
[31]沈银柱,黄占景. 进化生物学[M]. 北京:高等教育出版社,2018:145-146.
