甘薯、番茄、拟南芥中SPL转录因子的生物信息学分析
2021-11-19荣誉磊周志林赵冬兰唐君彭湘君赵玉琪宗凯吕亚宁姚改芳胡康棣张华
荣誉磊 周志林 赵冬兰 唐君 彭湘君 赵玉琪 宗凯 吕亚宁 姚改芳 胡康棣 张华


摘要:对甘薯、番茄、拟南芥中63个SPL基因家族进行了系统进化树分析、保守蛋白基序(Motif)分析,筛选归纳出同源性较高的2个分支的12个SPL基因进行理化性质分析、核定位预测等,氨基酸序列比对结果表明这些基因的功能可能较为保守。通过对番茄中的SPL基因Solyc05g015510.2、Solyc10g078700.1进行表达量分析,发现这2个基因可能参与调控果实成熟衰老进程。另外,通过对非生物胁迫下的转录水平进行分析得知,拟南芥中的AT5G43270可能参与对盐胁迫、热胁迫条件下的响应,AT1G27360、AT1G27370可能参与热胁迫条件下的响应,AT2G42200可能参与冷胁迫条件下的响应,而AT3G57920在非生物胁迫条件下表达量没有特别明显的变化,表明AT3G57920可能不参与非生物胁迫下的响应。
关键词:SPL转录因子家族;番茄;甘薯;拟南芥;生物信息学
中图分类号: S188 文献标志码: A
文章编号:1002-1302(2021)20-0074-10
收稿日期:2021-03-08
基金项目:国家重点研发计划(编号:2019YFD100070、2019YFD100071、2019YFD1001300、2019YFD1001303);现代农业产业技术体系项目(编号:CARS-10-B1);国家自然科学基金(编号:31670278、31970200、31970312、31901993、31872078);安徽省科技重大项目(编号:16030701073);中央高校基础研究基金(编号:JZ2018HGTB0241、JZ 20181)。
作者简介:荣誉磊(1992—),男,安徽阜阳人,硕士,研究方向为采后生物学。E-mail:990953597@qq.com。
通信作者:胡康棣,博士,副教授,研究方向为采后生物学,E-mail:kangdihu@hfut.edu.cn;张 华,博士,教授,研究方向为植物分子生物学,E-mail:hzhanglab@hfut.edu.cn。
植物基因转录水平的调控发生在基因表达的初期,是许多基因表达调控的主要方式。在此调控过程中,被称为转录因子的蛋白质特异结合到靶基因启动子,控制着一系列相关基因的表达[1-2]。squamosa promoter binding protein box(简称SBP-box)基因别称 squamosa promoter binding protein like(简称SPL)基因,可编码一类绿色植物特有的转录因子,调节多个不同并且重要的生物过程,如叶片发育[3]、调节细胞数量和大小[4]、花和果实的发育[5-9]、拟南芥花粉孢子的形成[10]和响应赤霉素(GA)信号[11]等。SPL基因最初是從金鱼草(Antirrhinum majus)中分离出来,人们把它命名为SBP1和SBP2,研究发现SPL蛋白通过结合MADS-box基因启动子的squamosa元件,控制植物花的早期发育。已知的SPL家族成员都具有高度保守的DNA结合结构域(SBP),大约含有76个氨基酸残基,具有2个典型的C3H(C—C—CH)和C2HC(C—C—H—C)锌指结构特征,其 C端区域具有核定位信号[12],能介导SPL蛋白进入细胞核。SPL基因由不同数量的外显子组成,但所有陆地植物的SBP结构域都是由第一和第二外显子编码的,并且这2个外显子之间的内含子位置也是保守的[13]。SBP锌指遵循 CC—x—C—x—C—x—[HC]—x—C—x—C—x—H—x—C(x表示氨基酸)的一般模式,其他3种组氨酸都很保守,但不参与锌的结合[6]。此外,核磁共振技术(NMR)被用来研究拟南芥中SBP转录因子SPL4 和SPL7的DNA结合结构域[14]。体外试验表明,SBP锌指结构域优先结合序列-TNCGTACAA-[15],然而,除了它们与DNA结合的能力之外,人们对这些潜在的转录调节因子的生理功能知之甚少。
自发现以来,SBP-box基因家族在拟南芥(Arabidopsis thaliana)[16]、水稻(Oryza sativa)[17]、玉米(Zea mays)[18]、小麦(Triticum aestivum)[19]和番茄(Solanum lycopersicum)[20]等植物中均有报道。在拟南芥中,有11个SPL基因受 miR156 调控,已有文献报道miR156 通过转录切割以及翻译抑制来调节SPL3表达[21],拟南芥AtSPL1基因能够参与植物发育和对伏马菌素B1的敏感性[22]。拟南芥SBP-box基因家族转录因子参与花期的转变,有报道阐明了它们在拟南芥发育中的作用,分析了可能的同源基因SPL9和SPL15的单突变体和双突变体表型,发现这些基因在控制生殖生长的转变过程中起着冗余作用。此外,在营养生长过程中,它们的功能缺失导致花序结构改变和分枝增强[23]。对LeSPL-CNR的研究表明,SBP-box基因在调控番茄果实成熟过程中起着重要作用,同时也证明了番茄的大量自然变异受表观遗传过程控制的论点[24]。部分SPL可通过调节其他转录因子来发挥功能,并且也能参与葡萄糖、无机盐和腺嘌呤核苷三磷酸(ATP)生成相关的代谢过程[25]。但是SPL基因家族在甘薯发育中的功能相关研究鲜有文献报道,在番茄和拟南芥中的基因功能研究也不是很全面,本研究运用生物信息学方法对甘薯、拟南芥、番茄中SPL家族成员理化性质、亚细胞定位、蛋白质基因序列分析、氨基酸序列比对、蛋白结构域及转录水平进行分析,并对可能参与的功能进行探讨,以期为进一步研究SPL基因在甘薯、番茄、拟南芥中的功能提供借鉴。
1 试验方法
1.1 SPL基因家族系统进化树分析与保守蛋白基序(motif)分析
从Phytozome(https://phytozome.jgi.doe.gov/pz/portal.html)网站下载拟南芥和番茄中已鉴定的33条SPL蛋白序列作为参考,其中拟南芥中16条,番茄中17条,利用甘薯网站(http://sweetpotato.plantbiology.msu.edu)中蛋白序列数据库blastp 检索甘薯蛋白数据库获得30条候选序列。利用MEGA 7.0软件使用Neighbor-Joining法构建SPL家族蛋白的系统发育树,利用iTOL工具网站(http://itol.embl.de/)对系统发育进化树进行优化。利用MEME网站(https://meme-suite.org/meme/tools/meme)对SPL蛋白基序(motif)進行分析。将得到的系统发育树与motif分析结果进行对比,找到同源性较高且系统发育树结果和motif分析结果吻合的SPL分支进行深入分析。
1.2 SPL基因家族理化性质分析与亚细胞定位预测
利用ExPASy网站(https://web.expasy.org/protparam/)对2个同源性较高的分支中的12个SPL基因的序列长度、蛋白质等电点、分子量等理化参数进行分析,并利用核定位信号NLS Mapper工具(http://nls-mapper.iab.keio.ac.jp/cgi-bin/NLS_Mapper_form.cgi)预测蛋白质的核定位信号,随后通过 Cell-PLoc 2.0 预测了这12个蛋白质的亚细胞定位。
1.3 同源SPL蛋白结构域分析及氨基酸序列比对
将上述筛选得到的2个分支12个SPL基因利用SMART(http://smart.embl.de/)网站分别进行蛋白结构域分析,并利用ESPript 3.0(https://espript.ibcp.fr/ESPript/ESPript/)网站进行氨基酸序列比对分析。
1.4 番茄中SPL基因表达量分析
将筛选得到的2个分支中番茄的SPL基因利用TomExpress(http://tomexpress.toulouse.inra.fr/)网站,挖掘基因表达数据,并进行绘图和分析。
1.5 非生物胁迫条件下的拟南芥中SPL基因表达量分析
将上述筛选得到的2个分支中拟南芥中的SPL基因利用ePlant工具(http://bar.utoronto.ca/eplant/)获取拟南芥中非生物胁迫条件下SPL基因的表达数据,并进行绘图和分析。
2 结果与分析
2.1 甘薯、番茄、拟南芥中SPL家族蛋白进化树与motif分析
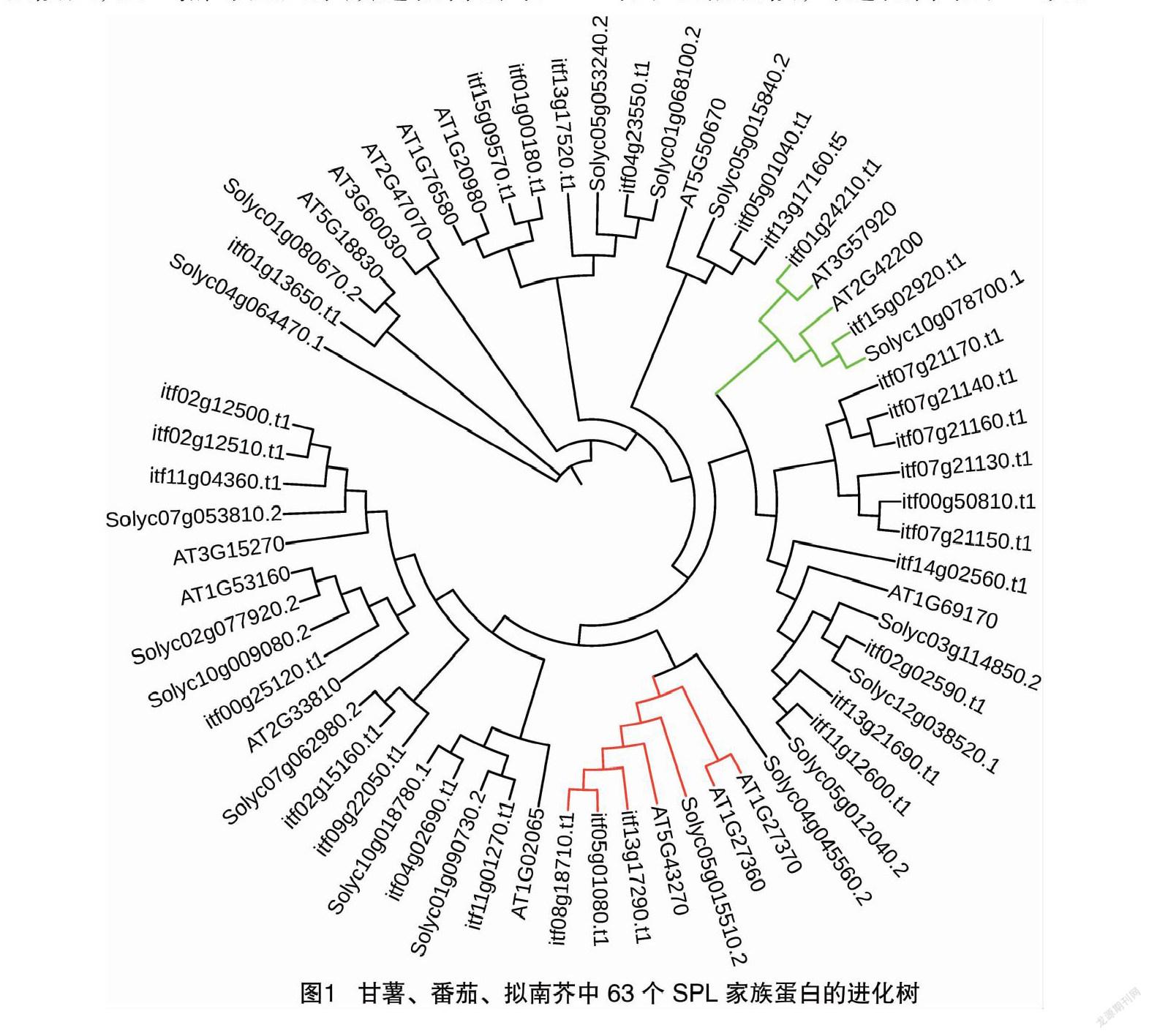
将BLAST同源比对获得的63条SPL家族蛋白利用MEGA 7.0 软件构建系统发育树。如图1所示,3个分支中,Solyc04g064470.1独自在一个分支中,表明较其他家族成员亲缘性较远,第2个分支中包括AT5G18830、Solyc01g080670.2、itf01g13650.t1,第3个分支包括其余甘薯、番茄、拟南芥中59个SPL基因。其中甘薯itf05g01080.t1、itf08g18710.t1、itf13g017290.t1与番茄Solyc05g015510.2以及拟南芥AT5G43270、AT1G27370、AT1G27360在同一个分支,说明它们亲缘性较高,可能具有相似的基因功能。甘薯itf15g02920.t1、itf01g24210.t1与拟南芥AT2G42200、AT3G57920以及番茄Solyc10g078700.1在同一个分支,说明它们亲缘性较高,可能具有相似的基因功能。
随后,利用MEME网站分析甘薯、番茄、拟南芥中63个SPL家族蛋白的保守蛋白基序(motif)。由图2可以看到SBP-box 蛋白基序分布,不同颜色的方块代表不同基序。其中,itf05g01080t1、itf08g18710.t1、itf13g017290.t1与Solyc05g015510.2以及AT5G43270、AT1G27370、AT1G27360的motif具有很大的相似性,这一点与基因家族进化树的结果相符合,另外,itf15g02920.t1、 itf01g24210.t1与AT2G42200、AT3G57920、Solyc10g078700.1的motif结果也很相似,与进化树结果一致。
2.2 甘薯、番茄、拟南芥中SPL蛋白家族理化性质与亚细胞定位预测
通过对图2中保守性较高的2个分支中12个SPL基因核酸序列和氨基酸序列进行理化分析,结果(表1)表明,蛋白质编码区(CDS)序列长度为 1 038~1 392 bp,蛋白质编码序列为345~463个氨基酸残基,蛋白分子量为37 017.17~50 418.90 g/mol,等电点(pI)介于7.94~9.11之间。在蛋白质序列的整个区域内,对SPL蛋白的核定位信号序列(NLS)进行分析,通过 NLS Mapper 网站进行预测。如表1所示,5个拟南芥SPL基因中有3个含有潜在的 NLS,2个番茄SPL基因都没有含有潜在的NLS,5个甘薯SPL基因中只有1个含有潜在的 NLS,说明部分SPL蛋白可能在细胞核中发挥作用。
同时,利用Cell-PLoc 2.0 预测了这12个SPL家族蛋白的亚细胞定位。如表2所示,12个SPL家族蛋白均定位在细胞核内,值得注意的是其中有3个SPL家族蛋白也存在细胞质内。
2.3 SPL家族蛋白的结构域分析及氨基酸序列比对
对上述12个同源性较高的SPL基因进行结构域分析(图3),发现这12个基因都具有典型的SBP结构域,且第1个分支的SBP结构域的位置在第170~250个氨基酸,第2个分支的SBP结构域的位置在第50~160个氨基酸,说明这2个分支都具有保守的SBP转录因子结构域,但SBP结构域的位置明显不同。
通过对上述SPL基因进行氨基酸序列对比分析发现,itf05g01080.t1、itf08g18710.t1、itf13g017290.t1、Solyc05g015510.2、AT5G43270、AT1G27370、AT1G27360的序列之间具有差异性,然而均具有高度保守的SBP结构域(图4-a),保守的SBP结构域包括2个锌指结构和NLS,第2个锌指结构与NLS有重叠,itf15g02920.t1、itf01g24210.t1、AT2G42200、AT3G57920、Solyc10g078700.1也具有高度保守的SBP结构域(图4-b),也具有2个锌指结构和1个NLS。该结构域富含半胱氨酸和组氨酸的基序,表明在分子层面这些基因也具有高度同源性。
2.4 番茄中SPL基因表达水平分析
分析番茄中2个的SPL基因表达量(图5)可知,Solyc10g078700.1在番茄组织中大部分时间表达量是处于一个较稳定的状态,然而在开花后4 d表达量急速增加,在开花后41 d及以后,表达量减少,这说明这个基因可能参与果实成熟过程中相关通路的途径。Solyc05g015510.2在其他组织中的表达量在0.04左右,但是在果实破色期到红熟期之间,基因表达量快速上升,这表明该基因可能参与果实成熟衰老的过程。
2.5 拟南芥SPL基因在非生物胁迫条件下的转录水平分析
筛选出来的12个SPL基因中,有5个拟南芥基因,在网站里检索这5个SPL基因在冷、热、干旱等胁迫下基因的表达量。如图6-A所示,AT5G43270在遭受盐胁迫、热胁迫0.5 h后幼苗中SPL基因表达量急速下降,在这之后的24h内呈上升趋势,有的甚至超过对照组,说明AT5G43270可能參与拟南芥对盐胁迫、热胁迫条件下的反应。图6-B 中AT1G27360在遭受热胁迫12 h后幼苗中SPL基因表达量急速上升,超过对照组,说明AT1G27360可能参与热胁迫条件下的响应。图6-C中AT1G27370在遭受热胁迫6 h后幼苗中SPL基因表达量急速上升,在8 h左右时超过对照组,说明AT1G27370可能参与热胁迫条件下的响应。而根中基因表达量与对照组相比,没有明显差异。由图6-D可知, 另外一个分支中的AT2G42200在冷胁迫条件下 12 h 后幼苗中SPL基因表达量急剧上升,说明AT2G42200可能参与冷胁迫条件下的响应,根中的SPL基因表达量在受到非生物胁迫时几乎没有变化。由图6-E可知,AT3G57920在非生物胁迫条件下表达量没有特别明显的变化,说明AT3G57920可能不参与非生物胁迫条件下的响应,根中的基因表达量在受到非生物胁迫时也几乎没有变化。
3 讨论
本研究利用BLAST同源比对,获得甘薯、番茄、拟南芥中的63个SPL家族蛋白,并进行进化树分析,结果表明甘薯itf05g01080.t1、itf08g18710.t1、itf13g017290.t1与番茄Solyc05g015510.2以及拟南芥AT5G43270、AT1G27370、AT1G27360在同一个分支,这7个SPL基因虽然不在同一物种中,但是亲缘性较高。甘薯itf15g02920.t1、itf01g24210.t1与拟南芥AT2G42200、AT3G57920以及番茄Solyc10g078700.1在同一个分支,说明它们亲缘性较高。motif分析结果与基因家族进化树的结果相符合,进而归纳出2个SPL基因家族的分支,基因结构域分析发现这2个分支的12个SPL基因都具有典型的SBP结构域。
通过对上述12个SPL基因进行氨基酸序列的对比分析发现,其中含有itf05g01080.t1、itf08g18710.t1、itf13g017290.t1、Solyc05g015510.2、AT5G43270、AT1G27370、AT1G27360的分支序列之间虽然具有差异性,但是均具有高度保守的SBP结构域,保守的SBP结构域包括2个锌指结构和NLS,第2个锌指结构与NLS有重叠,itf15g02920.t1、itf01g24210.t1、AT2G42200、AT3G57920、Solyc10g078700.1也具有2个锌指结构和1个NLS。该结构域的相同结构表明在分子层面这些基因也具有结构同源性。
最后进行基因功能验证,表明Solyc10g078700.1这个基因可能参与果实成熟过程中相关通路的途径,Solyc05g015510.2这个基因可能参与果实成熟衰老的过程,拟南芥中AT5G43270可能参与拟南芥对盐胁迫、热胁迫条件下的反应,AT1G27360、AT1G27370可能参与热胁迫条件下的响应。而另外一个分支中可以看到,AT2G42200在冷胁迫条件下12 h后幼苗中SPL基因表达量上升,说明AT2G42200可能参与冷胁迫条件下的响应,AT3G57920在非生物胁迫条件下表达量没有特别明显的变化。本研究初步筛选了甘薯、番茄、拟南芥中的SPL基因,并利用生物信息学方法对理化性质、亚细胞定位预测、蛋白基序、氨基酸序列比对、蛋白结构域及转录水平进行了分析,并对可能参与的功能进行了探讨,为进一步研究SPL基因在番茄、拟南芥、甘薯中的功能提供了一定借鉴作用。
参考文献:
[1]Cawley S,Bekiranov S,Ng H H,et al. Unbiased mapping of transcription factor binding sites along human chromosomes 21 and 22 points to widespread regulation of noncoding RNAs[J]. Cell,2004,116(4):499-509.
[2]杨致荣,王兴春,李西朋,等. 高等植物转录因子的研究进展[J]. 遗传,2004,26(3):403-408.
[3]Wu G,Poethig R S,Wu G,et al. Temporal regulation of shoot development in Arabidopsis thaliana by miR156 and its target SPL3[J]. Development,2006,133(18):3539-3547.
[4]Usami T,Horiguchi G,Yano S,et al. The more and smaller cells mutants of Arabidopsis thaliana identify novel roles for SQUAMOSA PROMOTER BINDING PROTEIN-LIKE genes in the control of heteroblasty[J]. Development,2009,136(6):955-964.
[5]Cardon G H,Hhmann S,Nettesheim K,et al. Functional analysis of the Arabidopsis thaliana SBP-box gene SPL3:a novel gene involved in the floral transition[J]. Plant Journal,1997,12(2):367-377.
[6]Klein J,Saedler H,Huijser P,et al. A new family of DNA binding proteins includes putative transcriptional regulators of the Antirrhinum majus floral meristem identity gene SQUAMOSA[J]. Molecular and General Genetics,1996,250(1):7-16.
[7]Tr M,Poole M,Hong Y G,et al. A naturally occurring epigenetic mutation in a gene encoding an SBP-box transcription factor inhibits tomato fruit ripening[J]. Nature Genetics,2006,38(8):948-952.
[8]Jiao Y Q,Wang Y H,Xue D W,et al. Regulation of OsSPL14 by OsmiR156 defines ideal plant architecture in rice[J]. Nature Genetics,2010,42(6):541-544.
[9]Wang H,Nussbaum-Wagler T,Li B,et al. The origin of the naked grains of maize[J]. Nature,2005,436(7051):714-719.
[10]Unte U S,Sorensen A,Pesaresi P,et al. SPL8,an SBP-box gene that affects pollen sac development in Arabidopsis[J]. Plant Cell,2003,15(4):1009-1019.
[11]Zhang Y,Schwarz S,Saedler H,et al. SPL8,a local regulator in a subset of gibberellin-mediated developmental processes in Arabidopsis[J]. Plant Molecular Biology,2007,63(3):429-439.
[12]Birkenbihl R P,Jach G,Saedler H,et al. Functional dissection of the plant-specific SBP-domain:overlap of the DNA binding and nuclear localization domains[J]. Journal of Molecular Biology,2005,352(3):585-596.
[13]Cuo J,Song J,Wang F,et al. Genome-wide identification and expression analysis of rice cell cycle genes[J]. Plant Molecular Biology,2007,64(4):349-360.
[14]Yamasaki K,Kigawa T,Inoue M,et al. A novel zinc-binding motif revealed by solution structures of DNA-binding domains of Arabidopsis SBP-family transcription factors[J]. Journal of Molecular Biology,2004,337(1):49-63.
[15]Cardon G,Hhmann S,Klein J,et al. Molecular characterisation of the Arabidopsis SBP-box genes[J]. Gene,1999,237(1):91-104.
[16]Cardon G H,Hhmann S,Nettesheim K,et al. Functional analysis of the Arabidopsis thaliana SBP-box gene SPL3:a novel gene involved in the floral transition[J]. Plant Journal,1997,12(2):367-377.
[17]Miura K,Ikeda M,Matsubara A,et al. OsSPL14 promotes panicle branching and higher grain productivity in rice[J]. Nature Genetics,2010,42(6):545-549.
[18]Hultquist J F,Dorweiler J E. Feminized tassels of maize mop1 and ts1 mutants exhibit altered levels of miR156 and specific SBP-box genes[J]. Planta,2008,229(1):99-113.
[19]劉 霞,张 斌,毛新国,等. 小麦tae-miR156前体基因的克隆及基靶基因TaSPL17多态性分析[J]. 遗传,2014,36(6):592-602.
[20]Salinas M,Xing S P,Hhmann S,et al. Genomic organization,phylogenetic comparison and differential expression of the SBP-box family of transcription factors in tomato[J]. Planta,2012,235(6):1171-1184.
[21]Gandikota M,Birkenbihl R P,Hhmann S,et al. The miRNA156/157 recognition element in the 3′ UTR of the Arabidopsis SBP box gene SPL3 prevents early flowering by translational inhibition in seedlings[J]. Plant Journal,2010,49(4):683-693.
[22]Stone J M,Liang X W,Nekl E R,et al. Arabidopsis AtSPL14,a plant-specific SBP-domain transcription factor,participates in plant development and sensitivity to fumonisin B1[J]. The Plant Journal,2005,41(5):744-754.
[23]Schwarz S,Grande A V,Bujdoso N,et al. The microRNA regulated SBP-box genes SPL9 and SPL15 control shoot maturation in Arabidopsis[J]. Plant Molecular Biology,2008,67(1/2):183-195.
[24]Manning K,Tr M,Poole M,et al. A naturally occurring epigenetic mutation in a gene encoding an SBP-box transcription factor inhibits tomato fruit ripening[J]. Nature Genetics,2006,38:948-952.
[25]Wang Y,Hu Z L,Yang Y X,et al. Function annotation of an SBP-box gene in Arabidopsis based on analysis of co-expression networks and promoters[J]. International Journal of Molecular Sciences,2009,10(1):116-132.
