猪基因组选择“两步走”策略的计算机模拟评估
2021-11-19唐振双殷东尹立林马云龙项韬朱猛进余梅刘小磊李新云邱小田赵书红
唐振双, 殷东, 尹立林, 马云龙, 项韬, 朱猛进, 余梅, 刘小磊, 李新云, 邱小田, 赵书红
猪基因组选择“两步走”策略的计算机模拟评估
唐振双1, 殷东1, 尹立林1, 马云龙1, 项韬1, 朱猛进1, 余梅1, 刘小磊1, 李新云1, 邱小田2, 赵书红1
1华中农业大学动物科技学院/农业动物遗传育种与繁殖教育部重点实验室/农业农村部猪遗传育种重点实验室/国家家畜工程技术研究中心,武汉 430070;2全国畜牧总站 北京 100107
基因组选择育种自2001年被MEUWISSEN等提出以来,已广泛应用在奶牛、猪等重要家畜的育种中,并显著加快了其重要经济性状的遗传改良速度。2017年,在全国畜牧总站的组织协调下,在全国生猪遗传改良计划框架内,猪全基因组选择育种平台项目正式启动。【】尽管基因组选择在种猪选育中取得了良好的效果,基因分型技术的不断升级也带来了成本的持续下降,但对于我国多数核心育种场依然面临着基因芯片分型个体数量不足、基因组选择实施流程不完善等问题,限制了该技术的大规模推广应用。结合我国生猪育种的实际情况,研究提出了一种“终测选择-早期选择”的“两步走”基因组选择策略。“终测选择”指在终测结束后利用一步法基因组BLUP对后备猪进行遗传评估,当群体中芯片分型个体数量达到一定规模后进行“早期选择”。以杜洛克、长白和大白三个种猪品种真实的50K基因芯片数据作为基础群体对不同品种分别进行大群模拟,共模拟4个世代,前3个世代作为基础群体,第4个世代作为测试群体,每个个体模拟两个性状(中等遗传力性状和低遗传力性状),利用猪基因组选择育种平台基于HIBLUP软件计算不同品种、不同性状的育种值,比较一步法基因组BLUP和常规BLUP两种方法的预测准确性。根据测试群个体有无终测成绩对其基因组育种值影响大小来评估早期选择效果。分析表明在3个品种内中等遗传力性状的终测选择效果和早期选择效果均好于低遗传力性状。一步法基因组BLUP的选择准确性均优于常规BLUP的选择准确性,并且随着测试群中芯片分型个体数量的增加、群体规模的扩大,预测准确性越来越高。一步法基因组BLUP的早期选择效果好于常规BLUP,当群体中芯片数量达到2 000张时就可以开展早期选择,阉割排名后30%的个体,可以保证前1%的优秀个体不会被错误淘汰,并且随着芯片数量的增加、早期选择的效果会越来越好。基因组选择“两步走”的策略符合我国国情、容易在生猪育种中推广实施。当芯片数量较少时,可以开展“终测选择”,一定程度上提高选择的准确性,提高育种效率;当芯片数量较多时,可以开展“早期选择”,对排名靠后的猪只个体进行早期阉割,增加优秀个体的测定量,增大选择强度、加快遗传进展。“两步走”策略符合我国生猪产业基因组选择育种的实际需求,该策略的实施将有利于推动我国猪基因组选择的应用、加快种猪改良进程。
基因组选择;猪;两步走;终测选择;早期选择
0 引言
【研究意义】种猪质量是影响生猪产业效益的核心要素,育种则是提升种猪质量的关键措施[1]。基因组选择育种是当前效率最高的育种技术,已广泛应用在奶牛、猪、鸡等多个物种中[2-4]。2017年,在全国畜牧总站的组织协调下,我国猪全基因组选择育种平台项目正式启动[5]。【前人研究进展】20世纪四十年代,HAZEL通过建立选择指数的方法来选择个体,标志着现代育种正式开始[6]。七十年代,HENDERSON开创基于表型和系谱信息快速求解BLUP(best linear unbiased perdition)的方法,该方法极大地提高了选择的准确性,随后的几十年间BLUP成为遗传评估的主要方法[7-9]。九十年代初,标记辅助选择(marker- assisted selection, MAS)方法兴起,但较低的标记密度使估计的准确性较差[10-11]。随着测序技术和计算机的快速发展,2001年MEUWISSEN等提出基因组选择(genomic selection, GS),该方法能对一些低遗传力的性状、无法直接度量的性状进行选择[12],并且能显著提高育种值估计的准确性、缩短世代间隔、降低群体近交水平的增长速度等[13-15]。2010年,AGUILAR和CHRISTENSEN等提出“一步法”选择(single-step genomic BLUP, SSGBLUP)用于育种值估计,该方法能同时利用基因组信息和系谱信息对有基因型个体和无基因型个体进行育种值的估计,具有较高的预测准确性[16-18]。【本研究切入点】虽然基因组选择在多个物种上都取得了良好的效果,国内部分生猪龙头企业也已尝试利用该方法进行种猪选育,但核心育种群体的芯片分型个体数量不足、基因组选择实施流程不完善等因素仍然限制着该技术的大规模推广应用[2,19]。【拟解决的关键问题】本研究提出了一种“终测选择-早期选择”的“两步走”基因组选择策略,并以杜洛克、长白和大白3个品种的真实基因芯片数据作为基础群体,通过大群模拟对终测选择和早期选择效果进行评估。结果表明“两步走”策略有利于公司尽早开展基因组选择育种、提高种猪效率和降低育种成本,也有利于我国猪基因组选择育种技术快速推广。
1 材料与方法
1.1 基因组选择“两步走”的策略
基于目前基因组选择在我国猪育种领域应用的现状,本研究提出猪基因组选择“两步走”的策略,第一步:终测选择,第二步:早期选择。
开展终测选择,即终测结束后利用SSGBLUP对后备猪进行遗传评估。终测选择需要在终测前一个月收集生长、体型外貌正常猪只的组织样品进行基因分型,终测时获得基因型数据和表型数据,在选留前一天利用全部的表型、系谱和基因型数据,使用SSGBLUP计算目标性状的基因组育种值(genomic estimated breeding values,GEBV),进一步构建综合选择指数,根据指数的排名进行选留和分流。终测选择能够在芯片数量较少时尽早地开展基因组选择育种,提高选择准确性和育种效率。
当群体中芯片分型个体数量达到一定规模后进行早期选择。早期选择是指在仔猪出生初期,利用基础群数据及仔猪个体基因芯片数据,对仔猪进行育种值评估和综合选择指数计算,根据指数排名进行选留和分流。早期选择重点针对小公猪开展,具体做法是在仔猪出生当天,收集待评估的初生仔猪的组织样本,尽快进行基因型检测,仔猪阉割前一天,将最新的基因型数据、系谱数据和表型数据上传至猪基因组选择育种平台,利用SSGBLUP模型计算目标性状基因组育种值和综合选择指数,根据指数排名高低对小公猪进行选留、对淘汰个体进行阉割。对母猪也可以进行早期选择,基因型检测做法同公猪,选留可以在断奶前一天进行,综合指数高的选留、低的个体淘汰分流到扩繁群或商品群。所有早期选择的种猪,只要正常生长就进入测定站,终测后再进行一次终测选择,最终选定后备猪进入核心群。
基因组选择“两步走”的策略符合我国国情,其优势主要有两点:一是当芯片数量较少时可以开展“终测选择”,一定程度上提高选择的准确性,提高育种效率;二是当芯片数量比较多时,可以开展“早期选择”,对排名靠后的猪只个体进行早期阉割,增加优秀个体的测定量,增大选择强度、加快遗传进展。
1.2 基因型数据模拟
本研究采用Simer软件(https://github.com/xiaolei- lab/SIMER),基于1 030头杜洛克、530头长白和2 030头大白的50K(50 697 SNPs)基因芯片数据进行计算机模拟[20],利用基因芯片数据的染色体、物理位置和基因分型等信息,模拟群体的繁殖过程,在群体生成过程中同时模拟系谱数据、基因型数据和表型数据。
每个品种模拟4个世代,其中N1—N3世代的核心群作为基础群体,N4世代的核心群作为测试群体。各群体的参数设置如下:每个世代核心群规模固定,杜洛克基础母猪为1 000头,长白基础母猪为500头,大白基础母猪为2 000头,各品种核心群公猪为30头。杜洛克产仔数为8头/窝,长白为13头/窝,大白为14头/窝,每窝产公猪、母猪的比例为1﹕1。杜洛克母猪的留种率为15%,长白和大白母猪留种率为10%,各品种公猪的留种率为1%。每个世代各品种公猪更新率为100%,母猪更新率为60%。根据上述设置,杜洛克核心群每个世代能产8 000头仔猪,从中挑选600头母猪和30头公猪作为核心群。长白核心群每个世代能产6 500头仔猪,从中挑选300头母猪和30头公猪作为核心群。大白核心群每个世代能产28 000头仔猪,从中挑选1 200头母猪和30头公猪作为核心群。在测试群有表型的个体中随机抽取10%或30%的个体进行基因型数据模拟,群体内所有个体都有完整的系谱记录。
1.3 表型数据模拟
在基础群体内随机抽取50%的个体进行表型模拟,测试群内所有个体都模拟表型,每个个体模拟2个性状,性状1的遗传力设为0.3、性状2的遗传力设为0.1,每个性状受100个QTNs(Quantitative Trait Nucleotides)控制,且QTN效应分布服从正态分布。模拟使用的参数见表1。
1.4 基因组选择“两步走”策略的评估
群体内芯片数量较少时实施终测选择。在测试群(N4世代)内,分别采用基于系谱信息的BLUP和SSGBLUP两种方法估计育种值,预测的准确性用EBV(estimated breeding values)与TBV(true breeding values)之间的皮尔森相关系数表示,相关系数越大,表明育种值估计的准确性越高。
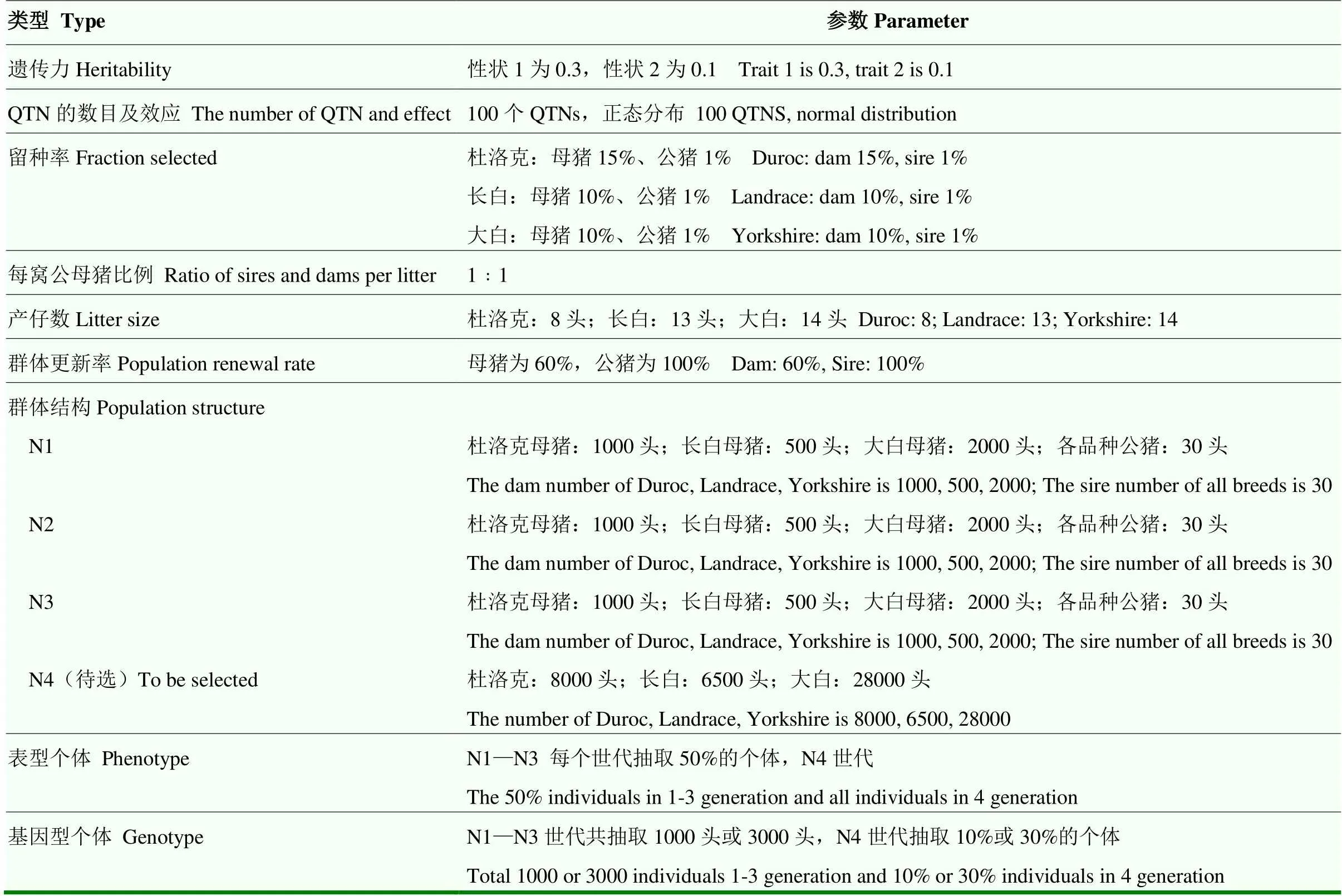
表1 模拟参数
当群体中芯片分型个体数量积累到一定规模后实施早期选择,根据测试群个体有无终测成绩对其基因组育种值影响大小来评估早期选择效果。在每个品种的基础群体(N1—N3世代)内随机抽取1 000或3 000个个体进行基因型数据的模拟作为积累的芯片数据,利用基础群体内积累的芯片数据、表型数据、系谱数据及测试群(N4世代)内新检测的芯片数据和系谱数据,分别采用BLUP和SSGBLUP两种方法计算测试群内不同性状的育种值。在测试群内,计算有终测表型时GEBV排名前1%或5%的个体被无表型时GEBV排名前30%、50%或70%覆盖的比例,个体相同条件下覆盖比例越高,表明早期选择效果越好。
1.5 育种值的估计和综合选择指数的计算
育种值估计和综合选择指数计算均在猪基因组选择育种平台(http://ubreed.pro:90/ GSPlatform/webroot/ user/login.html)完成,该平台使用HIBLUP软件(https://www.hiblup.com/)进行育种值估计。
2 结果
2.1 数据模拟
3个品种的数据概况见表2,基因型数据模拟基于真实的50K芯片数据,标记密度图见电子附图1。在每个品种内随机挑选1 000个个体,利用rMVP软件进行主成分分析(https://cran.r-project.org/web/packages/ rMVP/index.html),结果如图1所示,3个品种的模拟数据质量良好、满足分析要求[21]。
2.2 终测选择效果评估
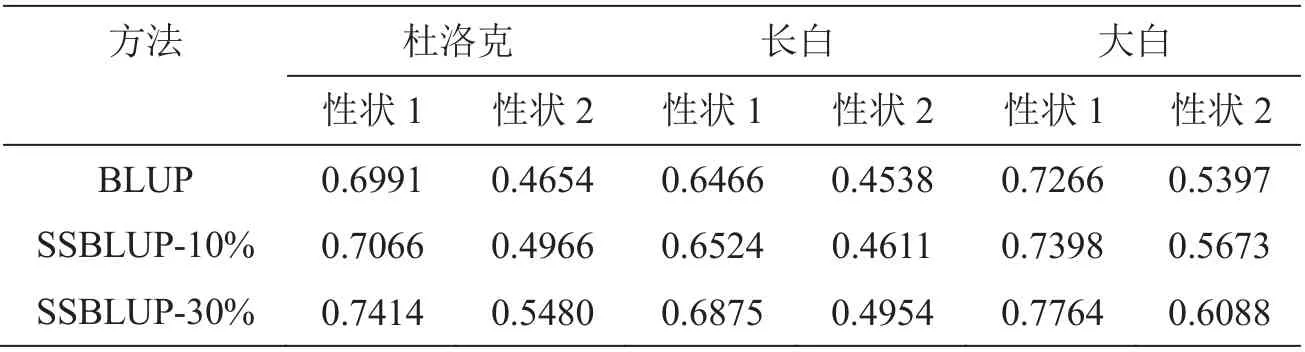
芯片数量较少时实施终测选择,即使用GEBV代替EBV进行选种。在3个品种内对比分析BLUP和SSGBLUP两种方法育种值估计的准确性,分析时数据使用情况见表2。在这里由于想对基因芯片较少的情况进行分析,所以只用了测试群体的基因型数据,不包括基础群体的基因型数据,分析结果见图2和电子附表1。结果表明:(1)随着测试群中芯片分型个体数量的增加、群体规模的扩大,预测准确性越来越高;(2)性状1的预测准确性高于性状2,表明中等遗传力的性状育种值预测的准确性比低遗传力的性状要高;而且不论是性状1还是性状2,SSGBLUP的准确性始终高于BLUP,表明尽管芯片数量不多,但是终测选择使用SSGBLUP效果更好。因此,建议在芯片数量较少时开展终测选择,这有利于提高选择的准确性和育种效率。
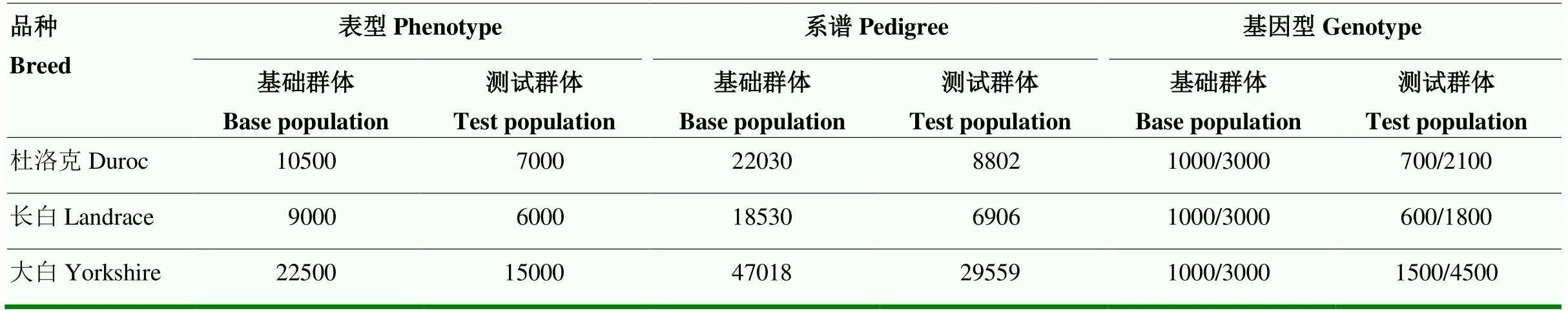
表2 3个品种数据的概况
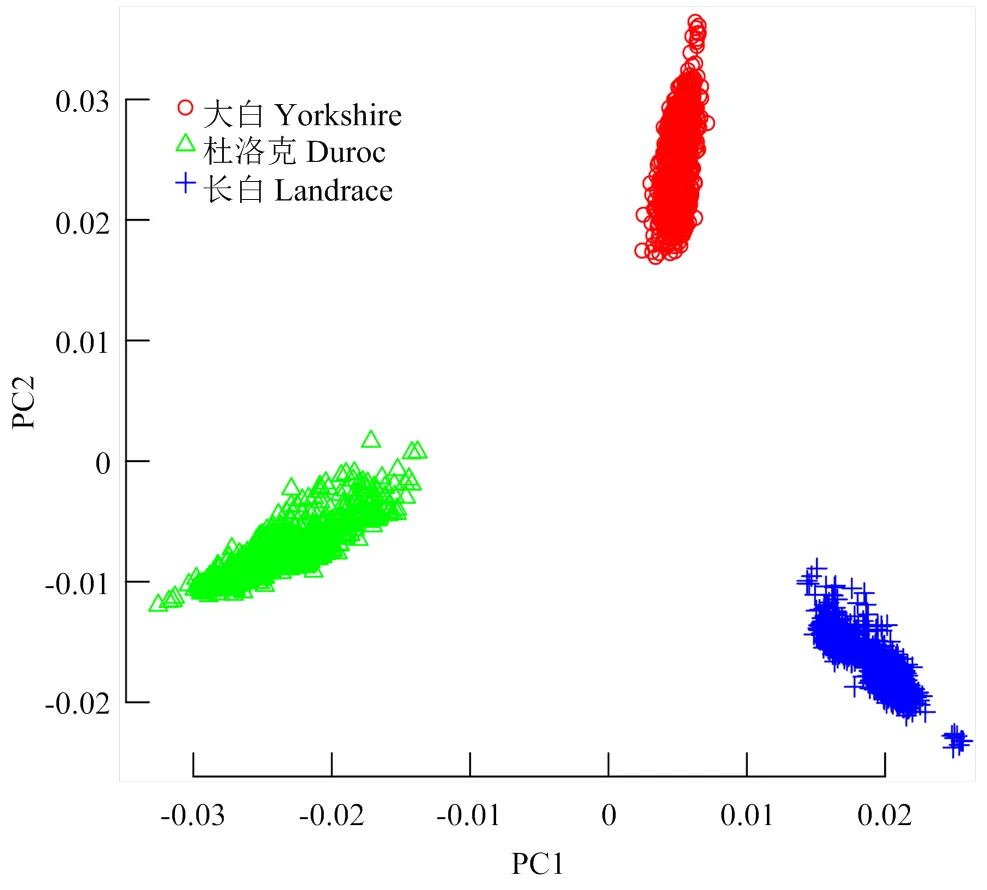
图1 3个品种的主成分分析图
2.3 早期选择效果评估
评估早期选择效果时,假设群体已经积累1 000张或3 000张芯片,在此基础上分别根据GEBV和EBV在测试群内进行早期选择,通过对比最优秀个体保留比例来分析早期选择效果,不同阉割比例下早期选择的效果见图3和电子附表2—4。结果发现SSGBLUP早期选择效果好于BLUP,并且随着芯片数量的增加、早期选择的效果越来越好;性状1的早期选择效果略好于性状2;不同品种、不同性状的选择效果趋势类似,随着群体规模的扩大,早期选择的效果也愈明显。以杜洛克群体为例,在性状1中,当阉割排名后70% 时,测试群中最优秀的1%(有终测表型时的GEBV排名),根据常规BLUP预测,有85.71%的个体不会被错误阉割,而根据SSGBLUP预测(1 700张芯片),有95.71%的个体不会被错误阉割;在性状2中,比例分别是75.71%和97.14%。当阉割比例为30%时,测试群中最优秀的1%(有终测表型时的GEBV排名)都不会被错误阉割。这表明早期选择是可行的,而且SSGBLUP比常规BLUP更好。
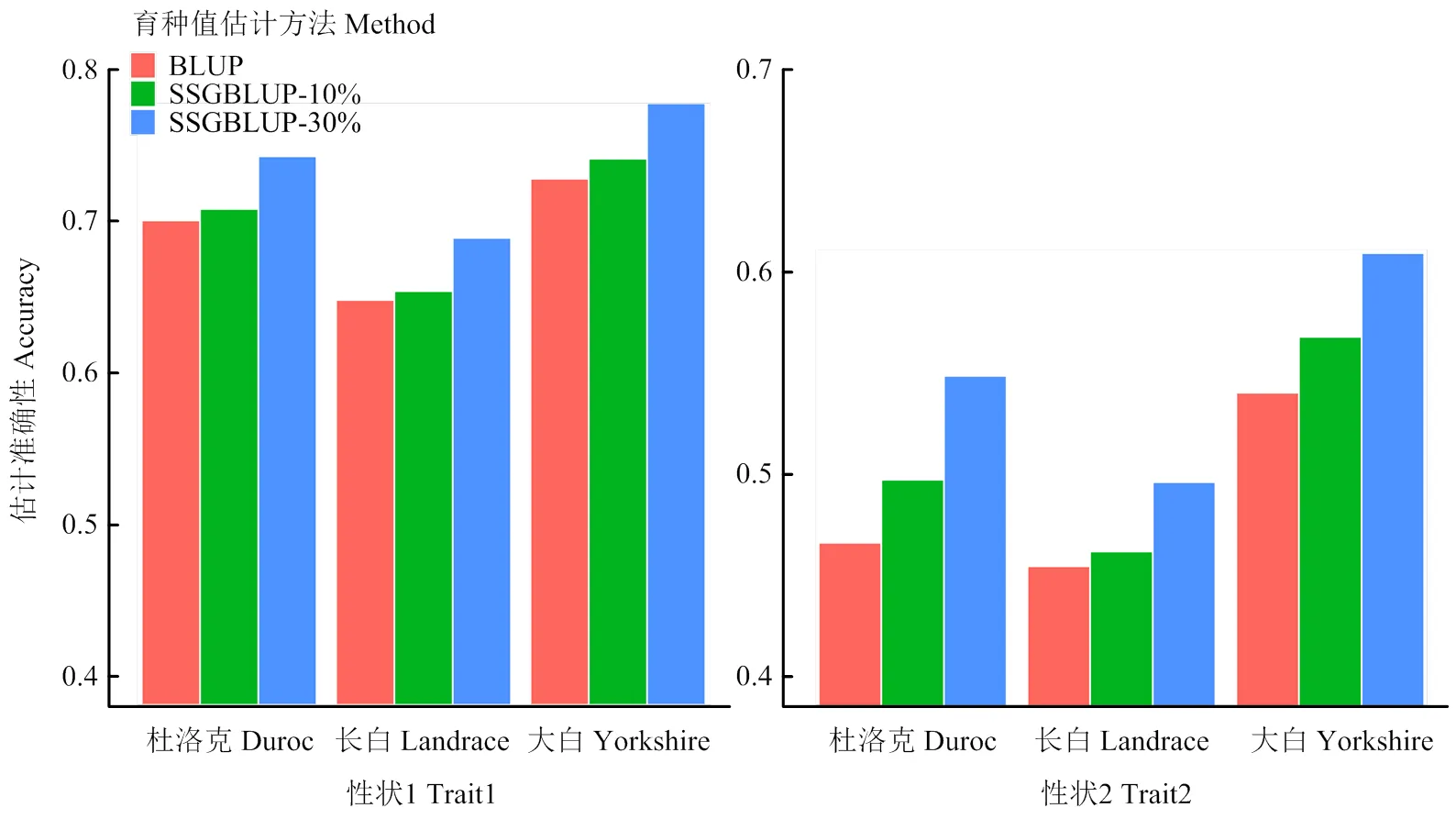
图2 育种值估计的准确性
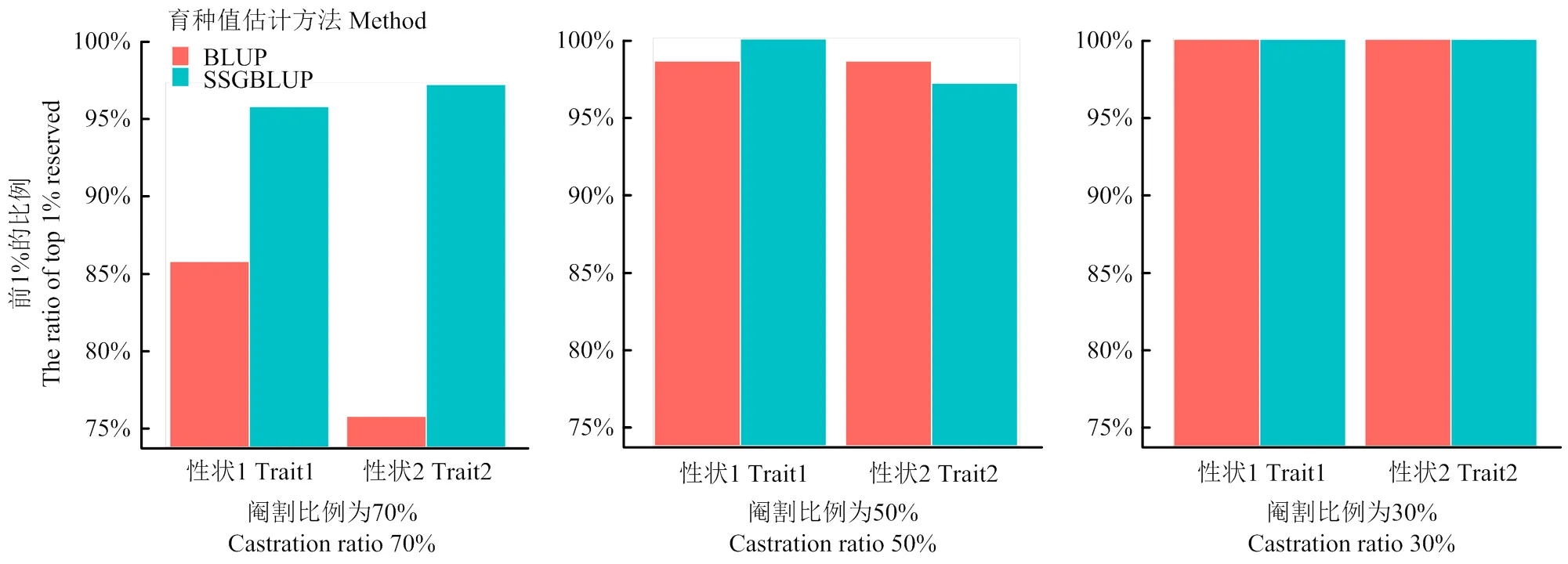
该图展示杜洛克品种早期选择的部分结果。SSGBLUP方法估计育种值时,基础群体有1000张芯片、测试群有700张芯片;基于最优秀的前1%(有终测表型时的GEBV排名)个体保留比例进行早期选择效果评估
3 讨论
通过数据模拟分析,分别对杜洛克、长白、大白3个主流瘦肉猪品种进行了分析,结果表明基因组选择“两步走”策略是可行的,也提示了“两步走”的阶段。育种公司可以根据基因芯片累积的数量,以2 000张为界限将基因组选择分成终测选择和早期选择两个阶段实施,这种策略有利于育种公司提高选择准确性和育种效率、降低育种成本,同时也有利于在我国推广猪基因组选择育种技术。
3.1 终测选择效果评估
比较了BLUP和SSGBLUP两种方法育种值估计的准确性,结果表明即使在芯片分型个体数量较少的情况下SSGBLUP也能提高估计的准确性,本研究与前人的研究结果一致[22]。SSGBLUP可以同时利用系谱信息和基因组信息构建亲缘关系矩阵(H矩阵),由于使用了基因组信息,使个体间亲缘关系估计得更准,所以育种值估计的准确性比常规BLUP更高[23-24]。另外,有研究表明在低遗传力性状的评估中,间接法与直接法效果类似,但以Bayes为理论基础的间接法在估计遗传参数时需要多次迭代才能收敛,因此需要更多的计算时间和内存[22, 25-26]。与其相比,SSGBLUP在保持一定计算准确性的前提下,能够显著缩短计算时间、降低计算负担,而且,SSGBLUP无需构建参考群就能实施基因组选择[8, 26]。基于此,建议在芯片较少的情况下,终测结束后利用SSGBLUP开展遗传评估、进行选种,这样可尽早开展基因组选择、提高育种效率。
3.2 早期选择效果评估
本研究的结果表明当芯片数量达到2 000张左右时可以开展早期选择,在这种情况下,阉割后30%—50%的个体,最优秀的前1%个体是比较安全的;而阉割后70%的个体时,前1%的个体会有一定风险。另外,本研究是基于模拟数据进行早期选择效果的评估,在真实情况下,由于系谱记录错误或缺失、表型测定数量和质量等诸多因素的影响,预测的准确性会打折扣。阉割比例为50%和30%的结果类似,SSGBLUP在两类性状上都取得了较好的早期选择效果,虽然在低遗传力性状的评估中会出现个别BLUP的选择效果好于SSGBLUP选择效果的情况,主要原因是早期选择效果评估过程复杂,单次模拟就需要进行144次的育种值估计和比较分析,但单次模拟的结果基本符合预期。通过观察阉割比例为70%的结果,可知SSGBLUP的早期选择效果要明显好于BLUP的效果。
基于此,建议在2 000张芯片时阉割后30%的个体,随着芯片数量的增多,阉割比例可以进一步上升。早期选择可以对无种用价值的公猪实施早期阉割,有利于减少肉公猪比例,降低育种成本[27]。同时,早期选择还能够通过扩大初选范围,增加选择强度,加快遗传进展。因此,随着育种公司基因组选择育种的逐渐开展,建议择机实施早期选择。
4 结论
本研究提出基因组选择“两步走”的策略,第一步,当芯片分型个体数量较少的时候开展终测选择;第二步,当芯片分型个体数量达到一定规模后,开展早期选择。研究结果表明即便在芯片数量较少时,SSGBLUP的育种值估计的准确性也会高于常规BLUP,终测选择有效;当芯片数量达到2 000张左右时开展早期选择,阉割后30%的个体是比较安全的,随着芯片数量增加,阉割比例可以上升,早期选择不但能够降低育种成本,还能提升选择准确性和选择强度,实施早期选择对提高育种公司效益是有帮助的。本研究提出的基因组选择“两步走”的策略容易实施推广,符合我国当前的国情,也有利于推动猪基因组选择育种技术在养猪业中的广泛应用。
[1] 彭中镇. 《全国生猪遗传改良计划》解读及实施中有关问题的讨论(待续). 猪业科学, 2011, 28(8): 100-103. doi:10.3969/j.issn.1673- 5358.2011.08.037.
PENG Z Z. Discussion on the relevant issues in the interpretation and implementation of the “National Swine Improvement Program”. Swine Industry Science, 2011, 28(8): 100-103. doi:10.3969/j.issn. 1673-5358.2011.08.037. (in Chinese)
[2] KNOL E. ASAS-EAAP Exchange Speaker Talk: Is genomic selection beneficial for the pig industry? Journal of Animal Science, 2019, 97l(1): 1-7.
[3] CESARANI A, GASPA G, CORREDDU F, CELLESI M, DIMAURO C, MACCIOTTA N P P. Genomic selection of milk fatty acid composition in Sarda dairy sheep: Effect of different phenotypes and relationship matrices on heritability and breeding value accuracyJournal of Dairy Science, 2019, 102l(4): 3189-3203.
[4] MRODE R, OJANGO J M K, OKEYO A M, MWACHARO J M. Genomic selection and use of molecular tools in breeding programs for indigenous and crossbred cattle in developing countries: Current status and future prospects. Frontiers in Genetics, 2018, 9: 694. doi:10.3389/fgene.2018.00694.
[5] 丁向东, 邱小田, 王志刚, 杨红杰, 陈瑶生, 张勤. 全国生猪遗传改良计划(2009—2020)给我国生猪种业带来的变化和未来工作建议. 中国畜牧杂志, 2020(6): 169-174.
DING X D, QIU X T, WANG Z G, YANG H J, CHEN Y S, ZHANG Q. The changes and suggestions brought by the National Pig Genetic Improvement Program (2009-2020) for pig breeding industry in China. Chinese Journal of Animal Science, 2020(6): 169-174. (in Chinese)
[6] HAZEL L N, LUSH J L. The efficiency of three methods of selectionJournal of Heredity, 1942, 33l(11): 393-399.
[7] HENDERSON. Inverse of a matrix of relationships due to sires and maternal grandsiresElsevier, 1975, 58l(12): 1917-1921.
[8] 尹立林, 马云龙, 项韬, 朱猛进, 余梅, 李新云, 刘小磊,赵书红.全基因组选择模型研究进展及展望畜牧兽医学报, 2019, 50(2): 233-242.
YIN L L, MA Y L, XIANG T, ZHU M J, YU M, LI X Y, LIU X L, ZHAO S H. The progress and prospect of genomic selection models. Acta Veterinaria et Zootechnica Sinica, 2019, 50(2): 233-242. (in Chinese)
[9] VANRADEN P M. Efficient methods to compute genomic predictions. Journal of Dairy Science, 2008, 91(11): 4414-4423. doi:10.3168/ jds.2007-0980.
[10] FERNANDO R L, GROSSMAN M. Marker assisted selection using best linear unbiased predictionGenetics Selection Evolution, 1989, 21l(4): 467.
[11] GODDARD M E, HAYES B J. Mapping genes for complex traits in domestic animals and their use in breeding programmes. Nature Reviews Genetics, 2009, 10(6): 381-391. doi:10.1038/nrg2575.
[12] MEUWISSEN T H E, HAYES B J,GODDARD M E. Prediction of total genetic value using genome-wide dense marker mapsGenetics, 2001, 1571(4): 1819-1829.
[13] WRAY N R, YANG J, HAYES B J, PRICE A L, GODDARD M E, VISSCHER P M. Pitfalls of predicting complex traits from SNPs. Nature Reviews Genetics, 2013, 14(7): 507-515. doi:10.1038/nrg3457.
[14] HICKEY J M, CHIURUGWI T, MACKAY I, POWELL W, HENDRE P. Genomic prediction unifies animal and plant breeding programs to form platforms for biological discoveryNature Genetics, 2017, 491(9): 1297-1303.
[15] HILL W G. Applications of population genetics to animal breeding, from wright, fisher and lush to genomic prediction. Genetics, 2014, 196(1): 1-16. doi:10.1534/genetics.112.147850.
[16] CHRISTENSEN O F, LUND M S. Genomic prediction when some animals are not genotypedGenetics Selection Evolution, 2010, 42l(1): 2.
[17] AGUILAR I, MISZTAL I, JOHNSON D L, LEGARRA A, TSURUTA S, LAWLOR T J. Hot topic: a unified approach to utilize phenotypic, full pedigree, and genomic information for genetic evaluation of Holstein final scoreJournal of Dairy Science, 2010, 931(2): 743-752.
[18] GUO X, CHRISTENSEN O F, OSTERSEN T, WANG Y, LUND M S, SU G. Improving genetic evaluation of litter size and piglet mortality for both genotyped and nongenotyped individuals using a single-step method. Journal of Animal Science, 2015, 93(2): 503-512. doi:10. 2527/jas.2014-8331.
[19] 周磊, 杨华威, 赵祖凯, 杨红杰, 刘剑锋. 基因组选择在我国种猪育种中应用的探讨. 中国畜牧杂志, 2018, 54(3): 4-8. doi:10. 19556/j.0258-7033.2018-03-004.
ZHOU L, YANG H W, ZHAO Z K, YANG H J, LIU J F. The application of genomic selection in Chinese pig breeding industry. Chinese Journal of Animal Science, 2018, 54(3): 4-8. doi:10.19556/ j.0258-7033.2018-03-004. (in Chinese)
[20] Antonio R, Richard C, Nabeel A, Andreia A, Alan A, Jonathan B, Christian B, Carol C, Richard C, Patrick D, et al. Design of a high density SNP genotyping assay in the pig using SNPs identified and characterized by next generation sequencing technologyPLoS One, 2009, 4l(8): e6524.
[21] YIN L L, ZHANG H H, TANG Z S, XU J Y, YIN D, ZHANG Z W, YUAN X H, ZHU M J, ZHAO S H, LI X Y, LIU X L. rMVP: A Memory-efficient, Visualization-enhanced, and Parallel-accelerated tool for Genome-Wide Association StudybioRxiv, 2020, 2020.08. 20.258491.
[22] MISZTAL I, LEGARRA A. Invited review: Efficient computation strategies in genomic selection. Animal, 2017, 11(5): 731-736. doi:10.1017/s1751731116002366.
[23] MISZTAL I, AGGREY S E, MUIR W M. Experiences with a single- step genome evaluation. Poultry Science, 2013, 92(9): 2530-2534. doi:10.3382/ps.2012-02739.
[24] Nielsen Bjarne, Ostersen Tage, Guosheng Su, Christensen O, Henryon M. Use Of Genomic SNP Information In Pig Breeding, 2010.
[25] 彭潇, 尹立林, 梅全顺, 王海燕, 刘小磊, 朱猛进, 李新云, 付亮亮, 赵书红. 猪主要经济性状的基因组选择研究. 畜牧兽医学报, 2019, 50(2): 439-445. doi:10.11843/j.issn.0366-6964.2019.02.023.
PENG X, YIN L L, MEI Q S, WANG H Y, LIU X L, ZHU M J, LI X Y, FU L L, ZHAO S H. A study of genome selection based on the porcine major economic traits. Acta Veterinaria et Zootechnica Sinica, 2019, 50(2): 439-445. doi:10.11843/j.issn.0366-6964.2019.02.023. (in Chinese)
[26] ZHANG H, YIN L, WANG M, YUAN X, LIU X. Factors affecting the accuracy of genomic selection for agricultural economic traits in maize, cattle, and pig populations. Frontiers in Genetics, 2019, 10: 189. doi:10.3389/fgene.2019.00189.
[27] 张金鑫, 唐韶青, 宋海亮, 高虹, 蒋尧, 江一凡, 弥世荣, 孟庆利, 于凡, 肖炜, 云鹏, 张勤, 丁向东. 北京地区大白猪基因组联合育种研究. 中国农业科学, 2019, 52(12): 2161-2170. doi:10.3864/j.issn. 0578-1752.2019.12.013.
ZHANG J X, TANG S Q, SONG H L, GAO H, JIANG Y, JIANG Y F, MI S R, MENG Q L, YU F, XIAO W, YUN P, ZHANG Q, DING X D. Joint genomic selection of Yorkshire in Beijing. Scientia Agricultura Sinica, 2019, 52(12): 2161-2170. doi:10.3864/j.issn.0578-1752.2019. 12.013. (in Chinese)
附件:猪基因组选择“两步走”策略效果评估
附表1 育种值估计的准确性
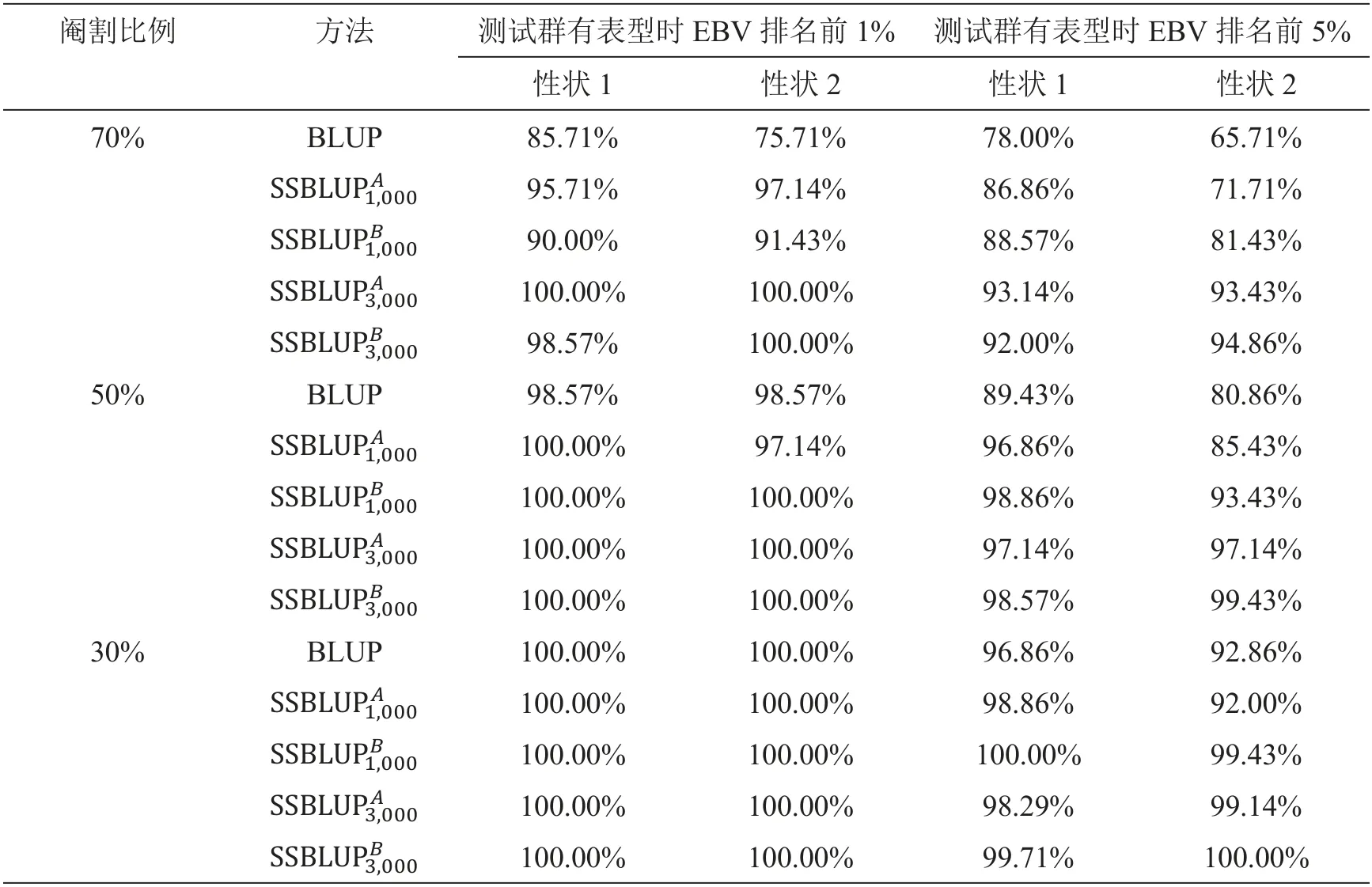
附表2 杜洛克早期选择的效果
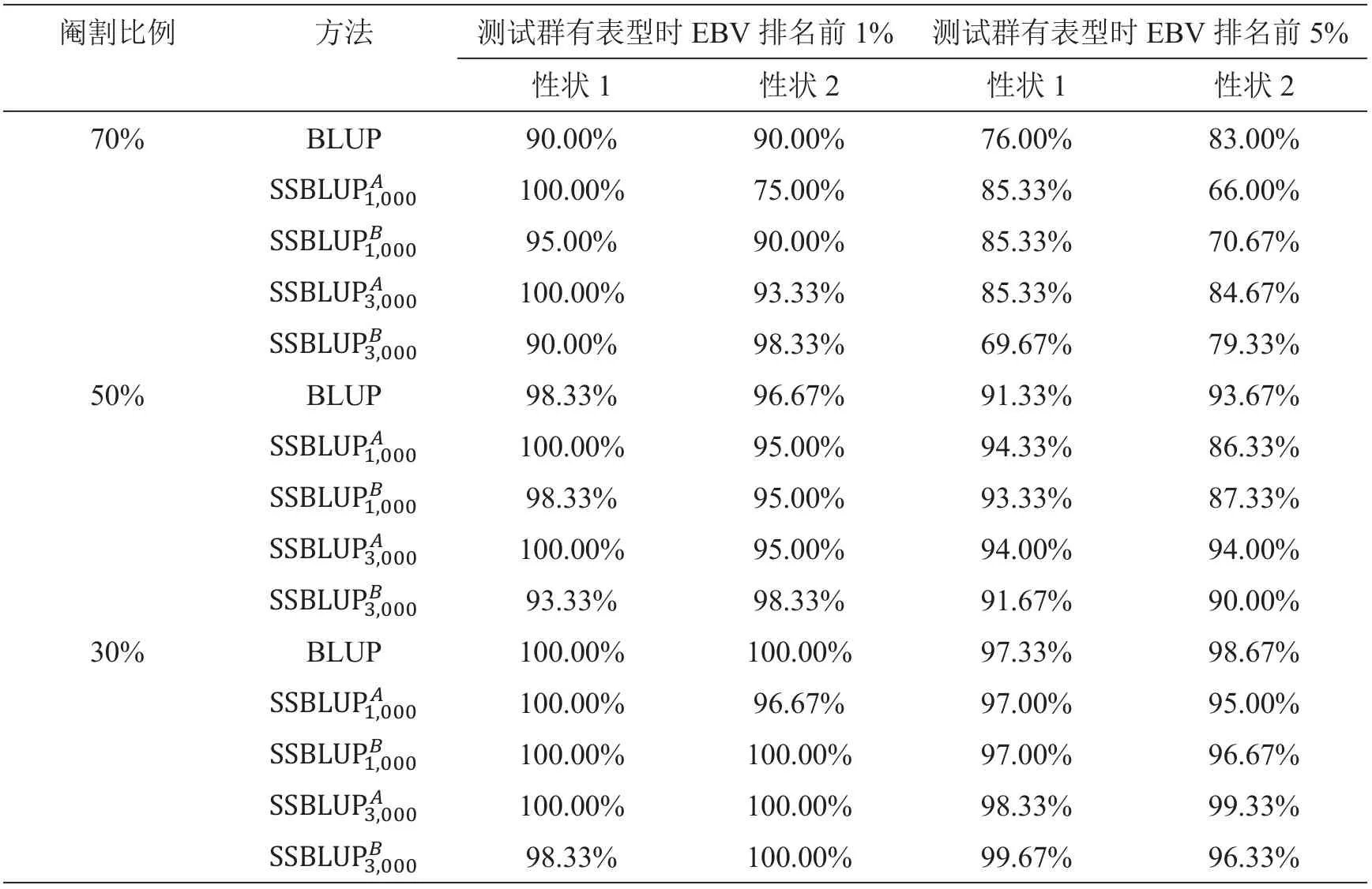
附表3 长白早期选择的效果
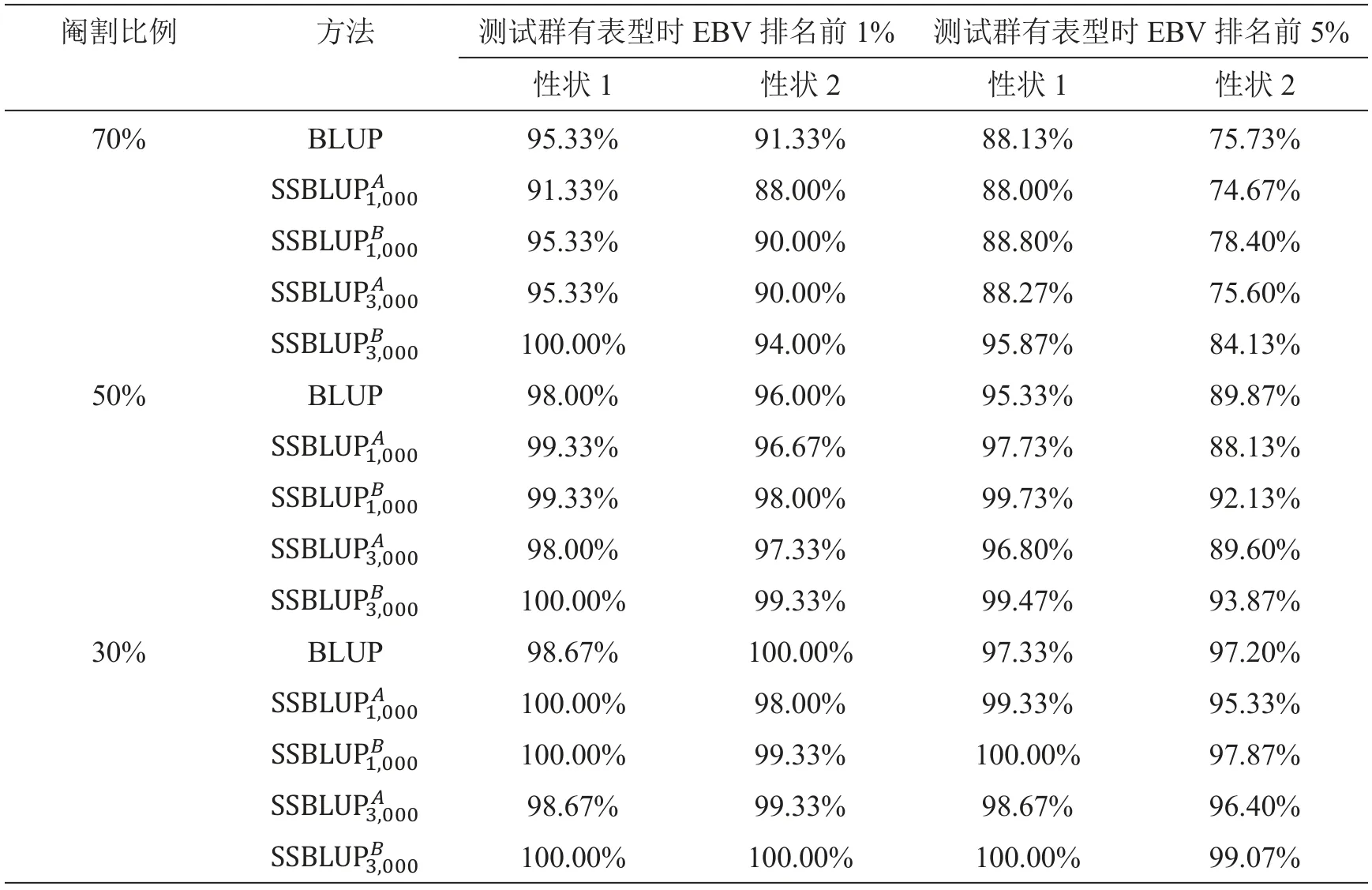
附表4 大白早期选择的效果
To Evaluate the “Two-Step” Genomic Selection Strategy in Pig by Simulation

1College of Animal Science and Technology, Huazhong Agricultural University /Key Laboratory of Agricultural Animal Genetics, Breeding and Reproduction of Ministry of Education/Key Laboratory of Swine Genetics and Breeding of Ministry of Agriculture and Rural Affairs/National Engineering and Technology Research Center for Livestock, Wuhan 430070;2National Animal Husbandry Service, Beijing 100107
【】Since genomic selection (GS) was proposed by Meuwissen et al. in 2001, it has been widely used in the breeding of dairy cows, pigs, and other livestock, and has significantly improved the speed of genetic gain of various economic traits. In 2017, with the organization and coordination of the National Grazing Headquarter Station and within the framework of the National Swine Improvement Program, the genomic selection platform for pig breeding was officially launched. Although genomic selection has made positive achievements in pig breeding, and the developing of advanced genotyping technology reduced the costs dramatically, some issues were still existed, including the insufficient number of genotyped individuals in majority of core breeding farms and the inappropriate implementation processes has restricted its wide application in practice.【】In combination with the actual situation of domestic pig breeding, the “two-step” strategy for genomic selection was proposed in this study, that is, the off-test evaluation and the early-stage prediction. Off-test evaluation referred to the genetic evaluation of replacement pigs by SSGBLUP after off-test, and early-stage prediction was carried out when the number of chips reached a certain scale. 【】In this study, the 50 k chip datasets of three breeds consisting of Duroc, Landrace, and Yorkshire were used as the base group to simulate the large-scale population of different breeds, respectively. The four generations were simulated: the first three generations were treated as the base population, and the fourth generation as the test population, two traits with medium and low heritability was simulated for each individual. The estimated breeding values of SSGBLUP and traditional BLUP model for different traits were calculated by the pig genomic selection platform based on the HIBLUP software. The predictive performance of early-stage was evaluated according to whether the individual’s testing records have influence their genomic estimated breeding values (GEBV) in test population. 【】The results showed that the predictive performance of off-test evaluation and early-stage for traits with medium heritability were better than those with low heritability. The selection accuracy of SSGBLUP was better than traditional BLUP. Moreover, with the increase of the number of chips and the expansion of the population size, the prediction accuracy was higher. The early-stage predictive performance of SSGBLUP was better than that of traditional BLUP, the early-stage prediction could be carried out when the number of genotyped pigs reached about 2 000, and castrating the last 30% individuals according to GEBV could ensure that the top 1% excellent individuals would not be mistakenly eliminated. And the prediction accuracy performance was increasing with the increased number of genotyped pigs. 【】The “two-step” strategy pretty was conformed to the state of domestic breeding program, and was easy to implement and promote the pig breeding in China. When the number of genotyped pigs was small, off-test evaluation could be carried out to improve the accuracy of selection, as well as efficiency, to a certain extent; when the number of genotyped pigs was large, early-stage prediction could be performed by castrating the pigs on the lower rank of GEBV, which could increase the amount of testing for more excellent pigs, and could also strength the selection intensity and accelerate the genetic gain. The “two-step” strategy was in line with the actual requirements of genomic selection in pig industry. The implementation of this strategy could further promote the application of genomic selection and speed up the genetic gain in pig breeding.
genomic selection; pig; two-step; off-test evaluation; early-stage prediction

10.3864/j.issn.0578-1752.2021.21.016
2020-10-19;
2021-02-04
国家自然科学基金面上项目(32072725)、国家生猪产业体系(CARS-35)、中央高校基本科研业务费专项资金资助项目(662020DKPYCFA006007)、猪基因组选择育种创新群体(2020CFA006)
唐振双,E-mail:zst@webmail.hzau.edu.cn。通信作者赵书红,E-mail:shzhao@mail.hzau.edu.cn。通信作者邱小田,E-mail:23753846@QQ.com
(责任编辑 林鉴非)
