谷子生长调节因子互作因子基因家族生物信息学分析
2021-11-15张立全张浩林李丛丛魏建华张杰伟
张立全 张浩林 李丛丛 魏建华 张杰伟


摘要:【目的】生长调节因子互作因子( GRF-interacting factor,GIF)是植物体内一类转录共激活因子,在植物生长发育和逆境胁迫中起重要作用。通过系统分析谷子GIF基因家族的组成、各成员的结构以及进化关系,为GIF基因调节机制研究提供参考。【方法】利用谷子基因组数据库,采用生物信息学的方法,鉴定谷子GIF基因家族的基因结构、染色体定位,编码蛋白相似性、二级结构、跨膜区和磷酸化位点预测,通过序列比对进行进化和分类分析。【结果】谷子含有3个GIF基因,均含有4个外显子,分布于第3、8和9號染色体上。编码SiGIF1蛋白和SiGIF2蛋白相似性最高,为72.04%,SiGIF1蛋白和SiGIF3蛋白相似性最低,为37.08%。二级结构分析显示,谷子GIF蛋白无规则卷曲占比最高( 41.56%-56.60%),其次为a-螺旋(34.43%-35.50%),再次为D-转角(5 19%~11.69%),p-折叠最低(3.23%- 11.26%)。TMHMM跨膜区进行分析显示,谷子GIF蛋白均不含有跨膜区。MEME保守基序分析显示,谷子GIF蛋白均含有保守的SSXT( PF05030)结构域。磷酸化位点预测分析表明谷子GIF蛋白均含有潜在磷酸化位点。【结论】谷子GIF基因家族的基因结构、磷酸化位点预测等生物信息学分析结果将为揭示谷子GIF基因家族在谷子生长发育过程中的功能提供重要的线索。
关键词:谷子;生长调节因子互作因子;基因家族;进化分析
中图分类号:S 515
文献标志码:A
文章编号:1008-03 84( 2021) 08-0878-06
Bioinformatics of Growth-interacting Factor Genes in Foxtail Millet
ZHANG Liquan 1, ZHANG Haolin 1.2. LI Congcong 1, WEI Jianhua 1*. ZHANG Jiewei 1*
(1. Agro-Biotechnology Research Institute, Beijing Acndemy ofAgriculture and Forestry ScienceslBeijing Key Laboratory of
Agricultural Genetic Resources and Biotechnology. Beijing 100097. China; 2. College ofLife Science.
Shanghai Normal University, Shanghai 200234, China )
Abstract: 【 Objective】 The composition, structure. and evolution of each member of the growth-interacting factors (GIF) ofthe growth-regulating factors (GRF) and the transcription cofactors that closely associate with the growth. development, andstress response of plants in Setaria italica were analyzed. 【Method】 Based on the S. italica genome database andbioinformatics. the structure. characteristics, position on the chromosome. proteins similarity, secondary structure,transmembrane domain, and phosphorylation sites of the GIF genes were obtained.【Result】The 3 SiGIFs in S. italicagenome contained 4 exons locating on the 3, 8. and 9 chromosomes. The greatest similarity between SiGIFl and SlGIF2 was72.04%. while the lowest was 37.08% between SiGIFl and SiGIF3. The secondary structure consisted of 41.56%- 56.60%random coils. 34.43 % -35.50% alpha helix, 5.l9% _ 11.69% beta turns and 3 .23 % - 11.26% extended strands. The TMHMMtransmembrane domain analysis showed no transmembrane domain in SiGIFs. MEME indicated that all SiGIFs containedconserved SSXT (PF05030) domain. And potential phosphorylation sites in the GIFs were predicted by analysis.【Conclusion】The bioinformatics revealed information on the structure, phosphorylation sites of SiGIF gene familyprovided crucial insights for the studies on the growth and development of plants.
Key words: Setaria italica; GIF; gene family; phylogeny analysis
0 引言
【研究意义】生长调节因子互作因子( GRF-interacting factor,GIF)是植物体内一类转录共激活因子,属于SSXT超家族基因,与人类SYT转录共激活因子同源,在植物生长发育和逆境胁迫中起重要作用。GIF与植物生长调节因子(Growth regulatingfactor,GRF)共同形成功能复合体,在植物侧生器官的细胞增殖等过程中发挥重要作用[1-2]。共转录激活因子GIFs通过高度保守的SNH结构域(SSXT)与生长调节因子GRFs的N端保守结构域QLQ特异性识别,形成特定时空特征的互作复合物,通过调节细胞增殖等生理过程调控植株生长发育进程[1-2]。解析谷子(Setaria italica)GIF基因的生物学功能,对更好理解C4作物的生长发育具有重要意义。【前人研究进展】目前,拟南芥(Arabidopsis thaliana)GIF的生物功能解析较为完整。拟南芥GIF基因家族共包含3个成员[3]。拟南芥AtGIF1(也被称为ANGUSTIFOLIA3,AN3)基因功能缺失突变体gifl因叶片细胞数减少导致其叶片变小变窄,而超量表达AtGIF1基因拟南芥因叶片细胞数增多导致叶片变大[4]。拟南芥AtGIF1蛋白不仅可以与拟南芥AtGRF1蛋白的N端保守结构域QLQ直接作用,也可以与拟南芥AtGRF5蛋白相互作用[5]。双分子荧光互补、免疫共沉淀等试验表明,所有检测的AtGIF和AtGRF成员相互作用,并且在植物细胞增殖和细胞分化中发挥重要作用[6-7]。拟南芥AtGIF通过不同通路调控根分生组织稳态中心( Quiescent center,QC)和根分生组织大小[8]。近年研究发现,拟南芥AtGIF2和AtGIF3基因的生物学功能与拟南芥AtGIF1基因的生物学功能类似,均在植物细胞分裂调控中具有重要作用[2]。GIF蛋白和GRF蛋白互作在单子叶植物如水稻和玉米中也得到证实,GIF蛋白和GRF蛋白相互结合形成功能复合体共同发挥作用[9-10]。【本研究切人点】谷子起源于中国,距今已有8700年的栽培历史。伴随我国社会经济的不断发展,作为最大的杂粮作物,谷子日益受到广大消费者的关注[11]。由中国科学家主导的谷子“Zhang gu”全基因组序列图谱的成功构建和美国科学家主导的谷子“豫谷1号”全基因组测序结果均于2012年同期在国际著名杂志《自然生物技术》发表[12-13]。最近,山西农业大学主导谷子中的模式植物“xiaomi”全基因组测序的完成[14],为后续生物信息学挖掘、鉴定和分析谷子功能基因提供重要基础。目前,谷子GIF基因家族的研究尚未见报道,迫切需要对其进行生物信息学分析。【拟解决的关键问题】本研究利用美国能源部谷子(豫谷1号)基因组数据库,对谷子GIF基因家族成员的基因结构、基本特征、推导编码蛋白、系统进化和潜在的磷酸化位点等进行分析,为下一步研究特定谷子SiGIF蛋白特定的生物学功能及其磷酸化修饰提供重要参考。
1材料与方法
1.1谷子GIF基因家族基因组、编码区和推导蛋白的获得
拟南芥GIF基因组、编码区和推导蛋白序列下载白TAIR数据库(http://www.arabidopsis.org),水稻(OryzasativaL. Spp.Japonica cv.Nipponbare)GIF基因组、编码区和推导蛋白序列下载自RAP数据库(http://rapdb.dna.affrc.go.jp/),谷子(S.italica)GIF基因组、编码区和推导蛋白序列下载白Phytozome数据库(https://phytozomej gi.doe.gov/p z/portal.html#!info? alias-OrgSitalica)。
分别以水稻GIF基因家族蛋白序列OsGIF1( LOC Os03952320.1) QsGIF2( LOC Osllg40100.3)和OSGIF3( LOCOs12931350.1)为参比序列,利用Phytozome数据库(Setaria italica v2.2)中Blast程序进行BlastP检索,再利用NCBI提供的CDS软件(http://www.ncbi.nlm.nih.gov/cdd)预测这些蛋白是否存在GIFs蛋白保守SSXT结构域,存在该结构域的推导蛋白序列属于谷子GIF基因家族成员。
1.2谷子GIF基因家族基本结构分析
将获得的谷子SiGIFs的基因组序列和编码区序列利用GSDS(http://gsds.cbi.pku.edu.cn/)进行基因结构分析[15]。将获得的谷子SiGIFs蛋白利用TMHMMServer v.2.0(http://www.cbs.dtu.dk/services/TMHMM-2.0/)进行跨膜结构域分析[16],将获得的谷子SiGIFs蛋白利用SOPMA(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page-npsasopma.html)进行a一螺旋、p一折叠、β-转角和无规则卷曲等二级结构分析[17]。谷子SiGIFs蛋白之间的氨基酸相似性利用欧洲生物信息研究所ClustalOmega T具(https://www.ebi.ac.uk/Tools/msa/clustalo/)进行分析[18]。
1.3谷子GIFs蛋白結构及系统进化关系分析
将3个拟南芥GIF蛋白、3个水稻GIF蛋白和鉴定出的谷子GIF蛋白保守基序应用MEME软件(http://meme.nbcr.net/meme/cgi-bin/meme.cgi)进行分析[15]。参数设置如下:基序的最大发现数目是2个,其他参数设置为程序默认值。
鉴定出的谷子、拟南芥和水稻的GIF蛋白利用ClustalX( 2.0)[19]软件进行多重序列比对,GIFs蛋白系统进化树使用MEGA 6.0[20]软件构建,程序采用邻接(Neighbor-joining,NJ)算法,进行1000次Bootstrap抽样。
1.4谷子GIFs蛋白潜在磷酸化位点分析
分别对鉴定出的谷子GIFs蛋白序列利用NetPhos3.1 Server软件[21](http://www.cbs.dtu.dk/services/Net-Phos/)进行潜在的磷酸化位点分析,程序中涉及的参数均选用默认值。
2 结果与分析
2.1谷子GIF基因家族成员的鉴定和命名
根据水稻中已经鉴定出来的3个GIF蛋白序列,分别利用JGI数据庫的Blast程序中进行BlastP,得到了3个谷子(S italica)候选GIF基因。利用保守域在线预测软件CDS进行验证GIF保守域SNH结构域( SSXT)的存在,据此确定谷子包含3个GIF基因。谷子GIF基因分布在第3、8和9号染色体上,根据他们在染色体上的基因座分别命名为SiGIF1~3(图1),对应的基因座分别为Seita.3G360000、Seita.8G193300和Seita.9G104500,转录本分别为Seita.3G360000.1、Seita.8G193300.1和Seita.9G104500.1,别名分别为Si023367m.g、Si026850m.g和Si037326m.g基因长度为561~696个碱基不等,外显子个数均为4个,编码蛋白长度为186~231个氨基酸不等(表1)。
2.2谷子GIF基因家族结构分析
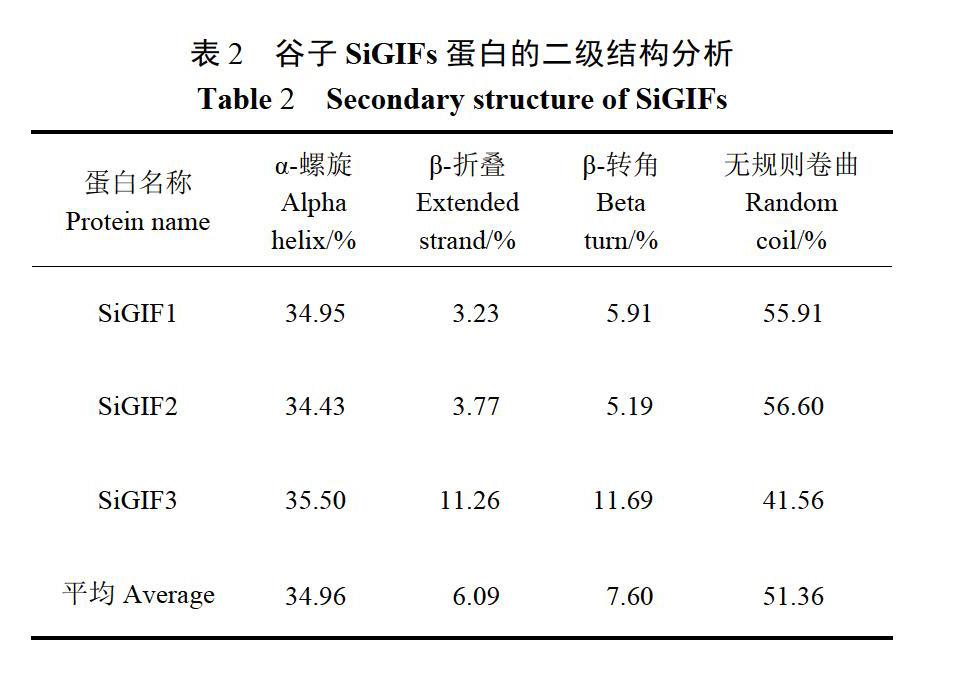
利用Clustal Omega对谷子SiGIFs推导编码蛋白的氨基酸序列的相似性进行分析,结果发现,谷子SiGIF1蛋白和谷子SiGIF2蛋白相似性最高,为72.04%;谷子SiGIFI蛋白和谷子SiGIF3蛋白相似性最低,为37.08%,谷子SiGIFI蛋白和谷子SiGIF2蛋白相似性为39.11%。利用SOPMA对谷子SiGIFs推导编码蛋白的二级结构进行分析,结果(表2)发现谷子SiGIFs蛋白中无规则卷曲的比例最高(41 56%~56.60%),其次为a-螺旋(34.43%~35.50%),再次为p一转角(5.19%~11.69%),p-折叠(3.23%~11.26%)最低。利用TMHMM对谷子SiGIFs推导蛋白的跨膜区进行分析,结果显示谷子SiGIFs蛋白均不含有跨膜区。
使用MEME保守结构域预测软件对拟南芥、水稻和谷子GIFs蛋白进行分析,结果表明,拟南芥、水稻和谷子GIFs蛋白均包含高度保守的SNH结构域。其中,第一个基序是位于蛋白N端的SNH结构域,P值为1.8×10_253。无论在单子叶植物水稻和谷子中,还是在双子叶植物拟南芥中,GIFs蛋白均含有SNH结构域结构域,表明GIFs蛋白在进化过程中具有高度保守的结构域(图2)。
2.3谷子GIFs系统进化分析
利用进化树分析软件MEGA 6.0对拟南芥、水稻、谷子共9个GIFs蛋白序列进行系统进化分析。结果(图3)显示:拟南芥、水稻和谷子GIFs蛋白并没有完全按照物种聚类在一起,而是混合分布。他们按照系统进化树可以分为3个亚家族,其中,水稻OsGIFl与谷子SiGIF3、拟南芥AtGIFI~3聚类在一起,推测谷子SiGIF3和水稻OsGIFI可能具有与双子叶植物拟南芥AtGIF1~3相似的进化过程和生物学功能。谷子SiGIF2和水稻的OsGIF2、谷子SiGIFI和水稻的OsGIF3聚类在一起,推测他们可能具有相似的生物学功能。
2.4谷子SiGIFs蛋白磷酸化位点分析
利用NetPhos 3.1 Serverl对谷子SiGIFs蛋白潜在的磷酸化位点进行分析,发现谷子SiGIFs蛋白中均存在潜在磷酸化位点,主要以丝氨酸和苏氨酸的形式存在,络氨酸的形式最少(图4)。其中,谷子SiGIF3的潜在磷酸化位点中是唯一仅含有2个酪氨酸位点。谷子SiGIFI含有15个潜在磷酸化位点,包括8个丝氨酸位点和7个苏氨酸位点。谷子SiGIF2含有12个潜在磷酸化位点,包括5个丝氨酸位点和7个苏氨酸位点(表3)。
3讨论
伴随着测序技术的不断发展与成熟,很多植物的高质量基因组被成功解析,为从全基因组水平分析和鉴定植物基因家族提供了便利[21]。GIF基因家族已经在拟南芥[3]、水稻[22]、小麦(Triticum aestivumL)[23]、野生二粒小麦(T.dicoccoides)[23]、乌拉尔图小麦(T. urartu)[23]、粗山羊草(Ae.tauschii)[23]中鉴定,截至目前,谷子GIF基因家族尚未鉴定。本研究鉴定出谷子GIF基因家族中包含3个成员,而已鉴定的拟南芥、水稻、小麦、野生二粒小麦、乌拉尔图小麦和粗山羊草GIF基因家族分别含有3、3、8、6、3和3个成员,推测在生物进化过程中,GIF基因可能在小麦和野生二粒小麦基因组中经历了不断发生谱系的特异扩张和拷贝。谷子SiGIFs均含有4个外显子,谷子SiGIFs蛋白均含有SNH结构域,表明GIF基因家族在植物中具有高度保守性,据此推测GIF转录因子作为GRF的共转录因子共同参与对下游靶基因的调节[24]。而不同植物中含有的成员数目各不相同,推测不同的GIF蛋白可能在特定的组织响应特定外界刺激从而精细调节植物各生理过程[24]。
进化分析表明,植物GIFs蛋白系统进化树基本上按照单子叶植物和双子叶植物分别聚类,据此推测GIF蛋白可能在单子叶植物和双子叶植物中各自具有类似的生物学功能,暗示植物GIF蛋白在进化过程中可能具有不同的进化过程。谷子SiGIFI和水稻OsGIF3、谷子SiGIF2和水稻OsGIF2同源性较高,结构域分析也表明,谷子SiGIF1和水稻OsGIF3、谷子SiGIF2和水稻OsGIF2具有类似的保守基序,表明他们可能具有有别于水稻OsGIF1的单子叶植物特有的生物学功能。谷子SiGIF3和OsPLC1、AtGIFI~3同源性较高。水稻OsGIF1与OsGRF4直接互作形成转录复合体,通过对下游靶基因作用,从而凋节水稻粒型[25]。最近研究发现,水稻Os GRF1可能通过精准调控OsGIFI特异性介导叶片生长[22],表明聚类在一起的同源基因可能具有不同的生物学功能,因此不能按照系统进化树中的聚类关系简单推测谷子SiGIFs各成员的生物学功能。谷子中特定SiGIF的功能还需要从谷子中克隆后逐一解析其生物学功能。
植物主要通過蛋白质来行使其生物学功能,磷酸化是最常见的一类蛋白质翻译后修饰,其主要通过蛋白激酶或磷酸酶在特定丝氨酸、苏氨酸和酪氨酸的羟基上增加或去除一个或多个磷酸基团,从而有效地改变蛋白质的结构和功能[26]。拟南芥生长素生物合成中的关键酶色氨酸转氨酶( Tryptophanaminotransferase of Arabidopsis,TAAl)的101位苏氨酸被转膜激酶4( Tans-membrane kinase 4.TMK4)磷酸化修饰后,调节了生长素生物合成,同时调节根分生组织和根毛的发育[27]。目前,谷子SiGIFs蛋白确切的生物学功能尚未见报道。磷酸化分析表明,谷子SiGIFs蛋白存在潜在磷酸化位点,暗示谷子SiGIFs蛋白可能通过磷酸化的方式来精细调节其生物学功能。
参考文献:
[1] DEBERNARDI J M MECCHIA M A,VERCRUYSSEN L et alPost-transcriptional control of GRF transcription factors by microRNAmiR396 and GIF co-activator affects leaf size and longevity [J]. PlantJournal. 2014. 79 (3): 413426
[2] KIM J H Biological roles and an evolutionary sketch of the GRF-GIFtranscriptional complex in plants[J]BMB Reports. 2019. 52(4):227 238
[3] LEE B H,KO J H,LEE s, et alThe Arabidopsis GRF-interactingfactor gene family performs an overlapping function in detenniningorgan size as well as multiple developmental properties [J].PlantPhysiology. 2009. 151(2):655-668
[4] KIM J H. KENDE H A transcriptional coactivator. AtGIFl. isinvolved in regulating leaf growth and morphology in Arobidopsis [J].PNAS. 2004. 101 (36) : 13374-13379.
[5]HORIGUCHI G. KIM G T. TSUKAYA H The transcription factorAtGRF, and the transcription coactivator AN3 regulate cellproliferation in leaf primordia of Arabidopsis thaliana[J]. The PlantJournal. 2005. 43 (l): 68-78
[6] VERCRUYSSEN L. VERKEST A. GONZALEZ N. et alANGUSTIFOLIA3 binds to SWUSNF chromatin remodelingcomplexes to regulate transcription during Arobidopsis leafdevelopment [J]. Ihe Plont Cell. 2014. 26 (l) : 210-229.
[7]LIANG G. HE H. LI Y. et al Molecular mechanism of microRNA396mediating pistil development in Arabidopsis [J]. Plant Physiology.2014. 164 (l): 249-258.
[8] ERCOLI M F. FERELA A. DEBERNARDI J M. et al GIFtranscriptional coregulators control root meristem homeostasis [J].Ihe Plant Cell. 2018. 30 (2): 347-359.
[9]GAO F. WANG K. LIU Y. et al. Blocking iniR396 increases rice vieldby shaping inflorescence architecture [J]. Nature Plants. 2016. 2:15196.
[10] NELISSEN H. EECKHOUT D. DEMUYNCK K. et al. Dynamicchanges in ANGUSTIFOLIA3 coI.lplex coI.lposltlon reveal a growthregulatory mecllanlsm in the maize leaf[J]. The Plont Cell. 2015.27(6): 1605-1619
[II]JIA G Q. HUANG X H. ZHI H. et al A haplotype map of genomicvariations and genoine-wide association studies of agronoinic traits infoxtail millet (Setaria italica) [J]. Nature Genetics. 2013. 45(8) :957961.
[12] BENNETZEN J L. SCHMUTZ J. WANG H. et al. Reference genomesequence of the model plant Setaria [J]. Nature Biotechnology, 2012.30 (6) : 555-561.
[13] ZHANG G Y. LIU X. QUAN Z W. et al. Genome sequence of foxtailmillet (Setaria italica) provides insights into grass evolution andbiofuel potential[J]. Nature Biotechnology, 2012, 30 (6): 549-554.
[14] YANG Z R. ZHANG H S. LI X K. et al. A mini foxtail millet with anArobidopsis-like life cycle as a C4 model 8ystem [J]. Arature Plants.2020. 6(9): 1167-1178.
[15]張杰伟 .丁莉萍.陈业娟.等.杨树磷酸肌特异性磷酶c基因家族鉴定与分析 [J] .福建农业学报. 2016. 31 (ll) : 1181-1186.
ZHANG J W. DING L P. CHEN Y J. et al. Genome-wide analysis andidentification of phosphoinositide-specific phospholipase C genefamily iii poplar(Populus trichocarpa) [J].Fujion Journal ofAZricultural Sciences. 2016. 3l (ll): ll8l-1186. ( in Chinese)
[16] BAILEY T L. JOHNSON J. GRANT C E. et al The MEME suite [J].Nucleic Acids Research. 2015. 43 (Wl): W39-W49.
[17] GEOURJON C. DELEAGE G SOPMA: significant improvements inprotein secondary structure prediction by consensus prediction frommultiple alignments [J]. Bioinformatics. 1995. 11 (6) : 681-684.
[18] SIEVERS F. WILM A. DINEEN D. et al Fast. scalable generation ofhigh-quality protein multiple sequence alignments using ClustalOmega[J]. Molecular Systems Biology, 201l . 7: 539.
[19] HUANG X Y. LIU G. ZHANG W W. Genome-wide analysis of LBD(LATERAL ORGAN BOUNDARIES domain) gene family iiiBrassico ropa [J]. Bracilion Archives of Biology and Technology.2018. 61: el8180049
[20] ZHANG J B. WANG X P. WANG Y C. et al. Genome-wideidentification and functional characterization of cotton (Gossypiumhirsuitum) MAPKKK gene family in response to drought stress [J].BMC Plont Biology. 2020. 20 (I):1-14
[21] MAO J X. ZHANG X S. ZHANG W J. et al. Genome-wideidentification. characterization and expression analysis of the MITFgene in Yesso scallops (Patinopecten yessoensis) with different shellcolors [J]. Gene. 2019. 688: 155-162.
[22] LU Y Z. MENG Y L. ZENG J. et al Coordination betweenGROWTH-REGULATING FAC.TORl and GRF-INTERACTINGFACTORl plays a key role in regulating leaf growth in rice [J]. BMCPlant Biology, 2020. 20 (l): 200
[23] ZAN T. ZHANG L. XIE T T. et al Genome-wide identification andanalysis of the growth-regulating factor (GRF) gene family and GRF-interacting factor family in Triticum aestivum L [J] BiochemicalGenetics. 2020. 58 (5): 705-724
[24] ZHANG J W. ZHANG Z B. ZHU D, et al. Expression and initialcharacterization of a Phosphoinositide-specific phospholipase C fromPopulus tomentosa [J] Journal of Plant Biochemistry andBiotechnology. 2015, 24(3): 338-346.
[25] LI S C . GAO F Y. XIE K L. et al. The OsmiR396c-OsGRF4-OsGIF,regulatory module detennines grain size and vield in rice [J]. PlantBiotechnology Journal. 20 16.14 (ll): 2134-2146
[26] LIU W Y. ZHANG B. HE W Y. et al. Characterization of in vivophosphorylation modification of differentially accumulated proteins incotton fiber-initiation process [J]. Acta Biochimica et BiophysicaSinica. 2016. 48 (8): 756-761
[27] WANG Q, QIN G C. CAO M. et al. A phosphorylation-based switchcontrols TAAl-mediated auxin biosynthesis in plants [J]. NatureCommunications. 2020. Il(l): 679.
(责任编辑:张梅)
收稿日期:2021-0307初稿;2021-0606修改稿
作者簡介:张立全( 1962-),男,副研究员,研究方向:谷子优质育种(E-mail: zhanglql962@163.com):共同第一作者:张浩林(1997-),男,
硕士研究生,研究方向:谷子优质育种(E-mail: 370842440@qq.com)
*通信作者:魏建华(1971-),男,研究员,研究方向:分子生物学(E-mail: weijianhua@baafsnetcn);张杰伟(1982-),男,副研究员,研究方向:谷子优质分子育种(E-mail: jwzhang919@163com)
基金项目:围家重点研发计划项日(2019YFDl000700、2019YFDl000703):北京市农林科学院科技创新能力建设专项(KJCX20200205、KJCX20210101)
