3个伞形科植物CPP基因家族的比较基因组学分析
2021-11-08巩克王希胤
巩克 王希胤
摘 要:CPP基因家族是一个转录因子家族,具有多种生物学功能。通过利用比较基因组学的方法,对芹菜、香菜和胡萝卜3个伞形科的物种进行分析,并将结果与莴苣、葡萄和拟南芥的CPP基因家族进行比较。结果显示,在3个伞形科物种中共鉴定了26个基因,其中芹菜、香菜和胡萝卜中分别有8个、12个和6个;香菜中的同源基因数量远大于芹菜和胡萝卜;全基因复制在CPP基因家族的扩张中发挥了重要的作用。同时,通过RNA-seq分析了不同组织中CPP基因的表达,将为伞形科中CPP和其他基因家族的比较和功能分析提供参考。
关键词:伞形科;基因家族;CPP-like
文章编号: 1005-2690(2021)16-0011-03 中国图书分类号: Q943.2 文献标志码: B
芹菜、香菜、胡萝卜是伞形科的3个代表性物种,生长在世界各地,以其香味和药用价值而闻名[1]。同时,这3种作物也具有很高的经济价值。
CPP转录因子在基因家族中是一个小型的转录因子家族。其成员典型特征是存在一个或两个类似的富含Cys的结构域,被称为CXC结构。从变形虫到植物,再到哺乳动物,CXC结构域表现出高度的保守性,CXC结构在不同的物种中表现出高度的一致性[2]。CPP-like基因在生殖组织的发育和控制植物细胞的分裂中发挥着重要作用。
目前,基于多个物种基因组数据的公布,CPP转录因子家族已经在拟南芥、水稻、茶树、陆地棉和大麦等多种植物中得到鉴定和分析[3-6],但对于伞形科物种的CPP基因家族的全面分析尚未有报道。
对伞形科的3个代表性物种的CPP基因家族进行鉴定和分析,以期阐明CPP家族进化规律,并探索芹菜和香菜不同组织中的基因表达模式,研究结果为进一步挖掘伞形科CPP转录因子家族的功能奠定基础。
1 结果与分析
1.1 基因组序列的获取与CPP家族基因的鉴定
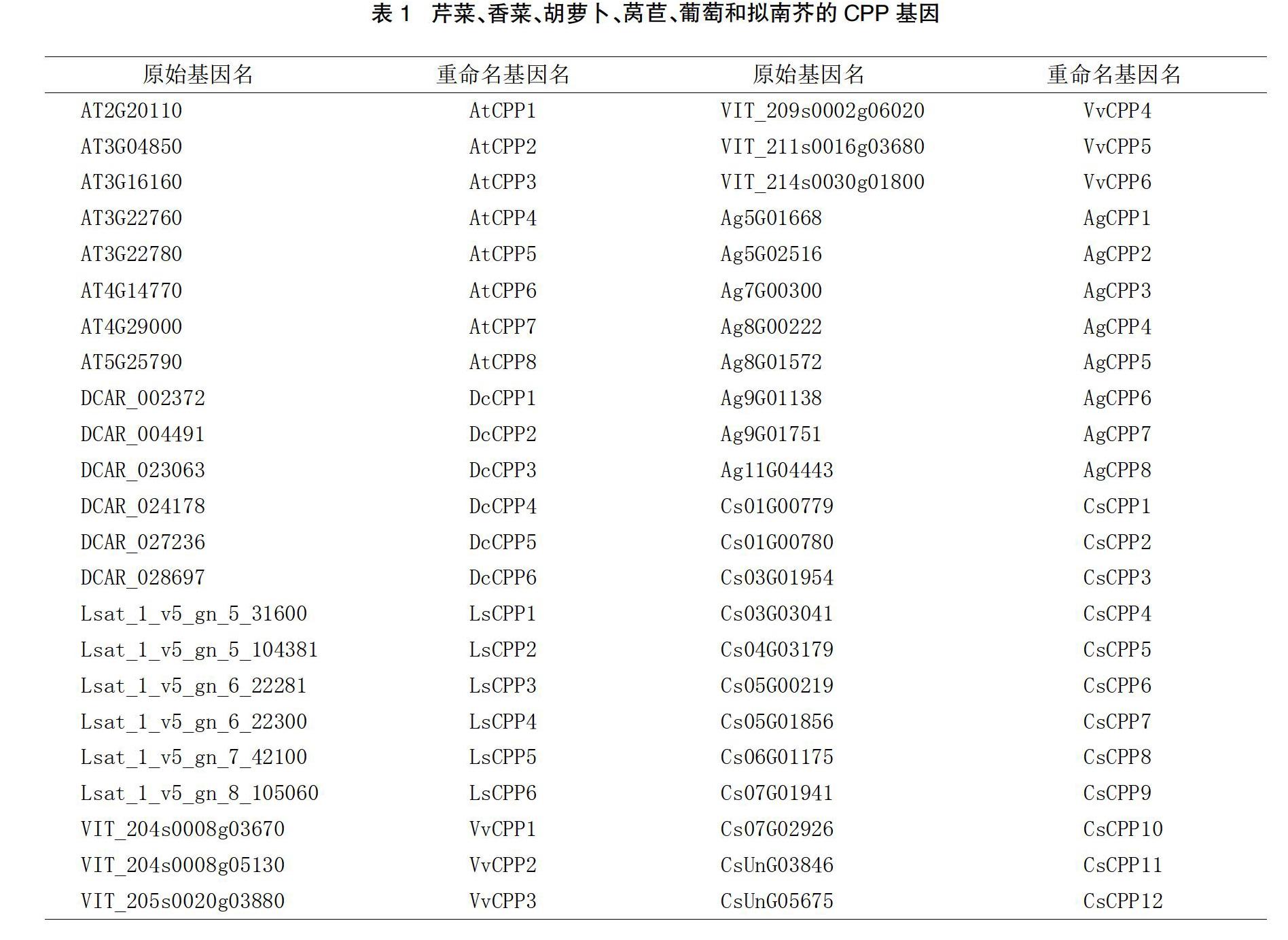
在葡萄、拟南芥、莴苣、芹菜、香菜和胡萝卜中共鉴定了46个CPP基因,并根据这些在染色体上的顺序重新进行了命名(见表1)。为探索CPP基因家族的进化历史和关系,利用上述6个物种的46个CPP氨基酸序列构建了系统发育树。根据系统进化树的拓扑结构,并参考拟南芥的分类标准,将CPP家族分为Ⅰ、Ⅱ两个类群。Ⅰ类包含19个基因,Ⅱ类包含27个基因。
1.2 CPP家族基因的结构分析和保守Motif分析
Motif分析显示,Ⅰ类和Ⅱ类家族拥有的motif存在较大的差异,Ⅰ类家族中普遍缺少motif3、motif5和motif10,Ⅱ类家族除AgCPP4外,普遍缺乏motif6、motif7和motif8。结构域分析显示,大部分的CPP基因家族都拥有两个TCR超家族结构域,而AgCPP1、AgCPP8、CsCPP4、CsCPP11和LsCPP3只拥有1个TCR超家族结构域,其中CsCPP4还拥有1个NT_Pol-beta-like超家族结构域以及1个TRF4超家族结构域。
1.3 CPP家族基因的同源性分析
通过对芹菜、香菜和胡萝卜的CPP家族基因同源性分析,芹菜与香菜之间发现了18对直系同源基因,其中有3个基因对不能被定位到任何染色体上。芹菜和胡萝卜之间发现12对直系同源基因。香菜和胡萝卜之间发现16对直系同源基因,其中有3个基因对不能被定位到任何染色体上。同时,在芹菜内部发现3对旁系同源基因。香菜内部发现10对旁系同源基因,其中有5对基因不能被定位到染色体上。胡萝卜内部发现3对旁系同源基因。
通过计算芹菜、香菜和胡萝卜直系同源基因对的Ks值和Ks/Ka的值,推断它们的分歧时间。结果显示,直系同源基因对的分化时间在芹菜和香菜之间为12.03~91.35百万年,在芹菜和胡萝卜之间为22.33~88.92百万年,在香菜和胡萝卜之间则为20.29~93.76百万年。
1.4 CPP家族基因的扩张与全基因组复制
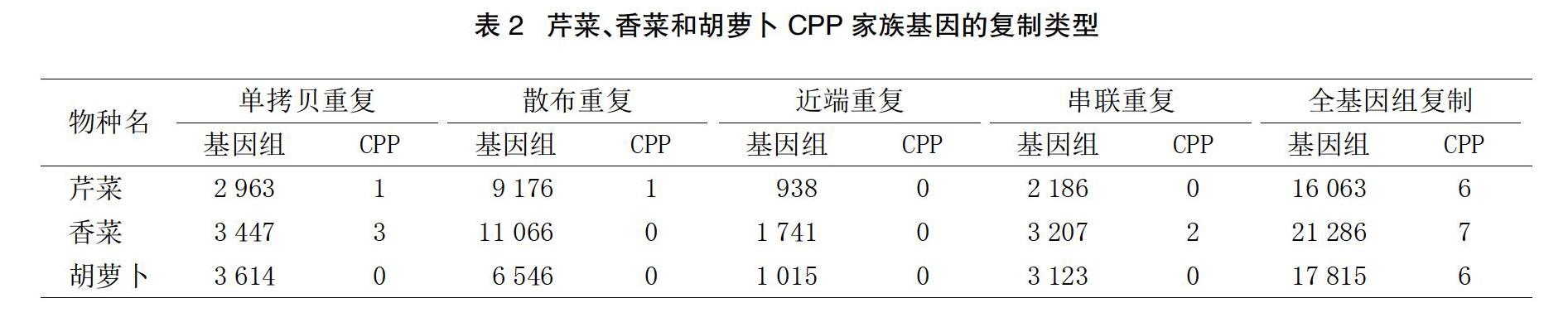
檢测5种基因复制的类型在芹菜、香菜和胡萝卜中分布。结果显示,全基因复制可能在伞形科物种CPP基因家族的扩张中起到了重要作用。在芹菜、香菜和胡萝卜中,分别有75%、58%、100%的CPP基因是通过WGD产生的。除此之外,在芹菜中,有1个CPP基因属于单拷贝复制类型,1个CPP基因属于散布复制类型。在香菜中,有3个CPP基因属于单拷贝复制类型,2个CPP基因属于串联复制类型,见表2。
1.5 CPP家族基因的表达分析
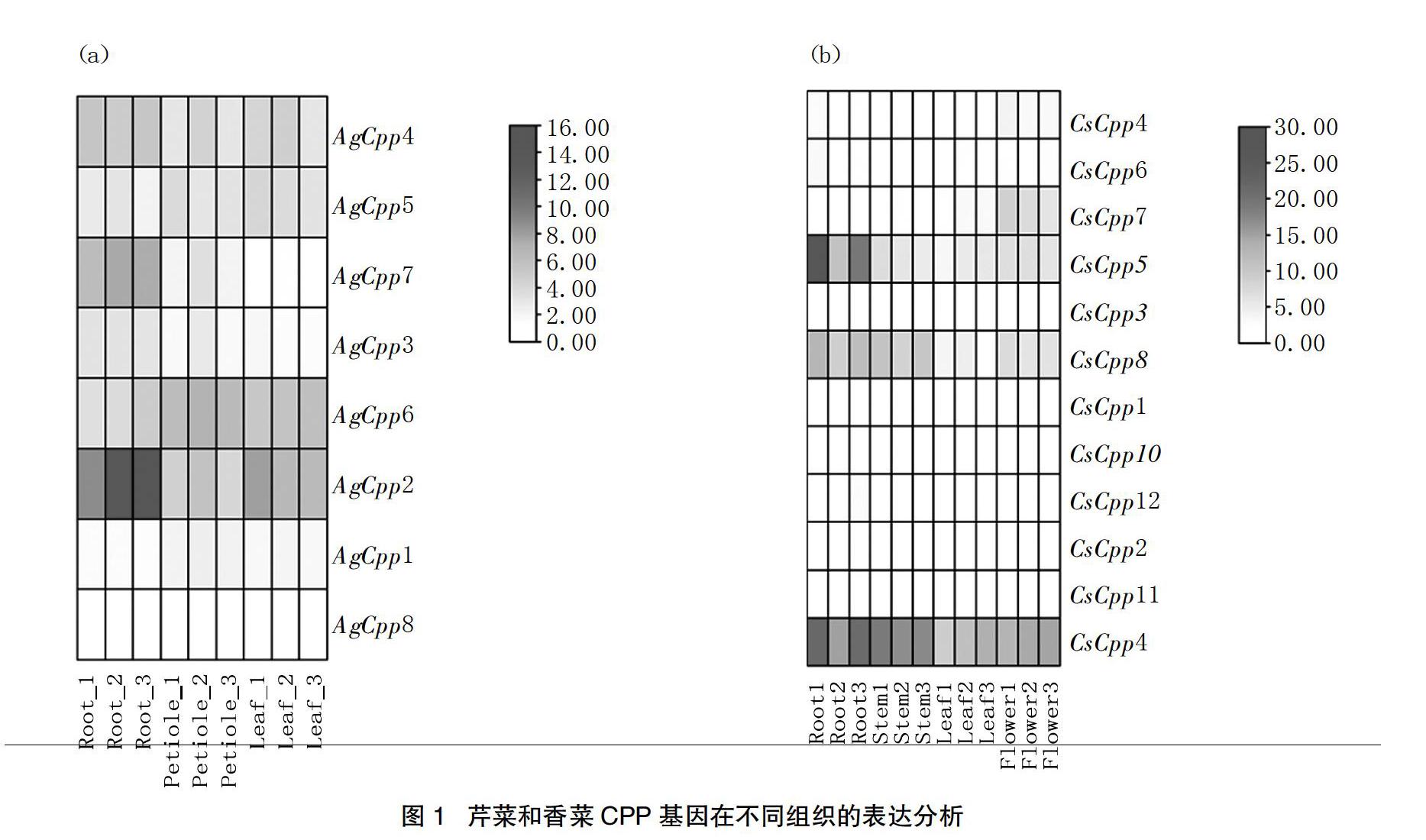
通过分析CPP基因在芹菜和香菜不同组织中的表达模式,结果显示,在芹菜的根、叶柄和叶3个组织中,AgCPP2在根部和叶中的表达水平最高,AgCPP6在叶柄中的表达水平最高,AgCPP8在3个组织的表达水平最低,不同组织之间的差异并不明显,这表明CPP家族基因在不同组织间的表达可能没有太大差异。在香菜根、茎、叶和花4个组织中,CsCPP4在所有组织中都有着最高的表达。而CsCPP1、CsCPP2、CsCPP3、CsCPP11、CsCPP12和CsCPP13在4个组织中的表达都非常低。CsCPP5在根部的表达水平较高,这可能暗示该基因与根部的生长发育有密切的联系,香菜在4个组织中也没有表现出明显的差异(见图1)。
2 讨论
CPP转录因子参与植物生长和发育过程,例如拟南芥AtCPP家族成员对芽、根及花序的发育发挥着重要的作用[7]。同时,CPP也受到多种非生物胁迫的调控,在不同的胁迫处理下,CCP基因的转录表达水平较对照处理均表现出显著提高。
尽管在CPP基因家族在许多植物中被研究过,但在伞形科中还没有相关文献及研究。最近发布了芹菜、香菜和胡萝卜的全基因组序列,促进了研究进程。为了解CPP基因的进化,利用芹菜、芫荽、胡萝卜、拟南芥、莴苣和葡萄的CPP氨基酸序列构建了一个系统发育树。基因结构和保守图案分析显示,同组或亚组的基因表现出类似的特征。香菜中的旁系同源基因对的数量明显多于芹菜和胡萝卜。此外,基于共线性分析,发现了WGD对芹菜科的ARF基因家族扩展有明显影响。通过对3个芹菜科物种的ARF基因家族进行全面分析,为植物中ARF基因家族的比较和功能分析奠定了坚实的基础。
3 材料与方法
3.1 基因组序列的获取与CPP基因的鉴定
芹菜和香菜的全基因组序列从香菜数据库中的获取[8],拟南芥、胡萝卜、莴苣和葡萄的基因组序列从JGI下载。使用CPP蛋白的保守结构域(PF03638)的隐马尔可夫模型来搜索,E值设定为1e-5。
3.2 系统发育树的构建
芹菜、香菜、胡萝卜、拟南芥、莴苣和葡萄CPP家族的氨基酸序列被用于系统发育分析。使用MAFFT[9]对所选物种的氨基酸序列进行比对,使用IQ-TREE[10]的最大似然法构建系统发育树,使用JTT+R3模型。
3.3 CPP基因的染色体位置、基因结构和保守基序分析
从基因组物理位置文件中检索每个CPP基因的染色体位置,并使用Perl脚本提取每个基因的染色体编号、起始位置和终止位置。使用TBtools绘制每个基因在染色体上的分布图。使用CDD网站分析CPP家族的保守结构域。利用MEME网站分析CPP家族的motif。根据基因组物理位置文件、CDD结构域预测结果和MEME预测结果,并对其进行可视化。
3.4 同源基因对的鉴定
使用OrthoMCL软件[11]鉴定直系同源基因和旁系同源基因,e值为1e-5。使用TBtools展示CPP基因在芹菜、香菜和胡萝卜中的关系。
3.5 CPP基因共线性分析和复制类型
使用MCScanX[12]进行串联分析。氨基酸序列使用Blastp分析,e值设置为1e-5。然后通过向MCScanX提交全基因组gff文件和blast的结果文件。使用duplicate_gene_classifier子程序鉴定复制类型。
3.6 CPP基因的进化分析
使用MAFFT对CPP基因对的编码序列(CDS)进行比对,并将比对文件转换为axt格式。使用Ka/Ks_calculator 2.0计算同义(Ks)和非同义(Ka)替换率。分歧时间使用公式T=Ks/2r估计。R表示中性替换率(5.2×10-9每个位点每年的替代次数)。
3.7 CPP基因表达分析
芹菜和香菜的不同组织中CPP基因表达的RNA-seq数据来自之前的研究[13-14]。以每百万碱基对转录物序列的每千字节片段(FPKM)表示的表达數据经过对数转换后用于聚类分析。使用TBtools进行层次聚类分析。
参考文献:
[ 1 ] Zefeng Yang,Shiliang Gu,Xuefeng Wang.et al. Molecular evolution of the CPP-like gene family in plants: insights from
comparative genomics of Arabidopsis and rice[J].J Mol Evol,2008,67(3):266-277.
[ 2 ] 王凯. 拟南芥和水稻CPP转录因子家族的生物信息学分析[J].生物技术通报,2010(2):76-84.
[ 3 ] Maljaei Mohammad Bagher,Moosavian Seyedeh Parisa,Mirmosayyeb Omid,et al. Effect of celery extract on thyroid function;Is herbal therapy safe in obesity[J].Int J Prev Med,2019(10):55.
[ 4 ] 杨如兴,王鹏杰,陈芝芝,等.茶树CPP转录因子家族的全基因组鉴定及分析[J].西北植物学报,2019,39(6):1024-1032.
[ 5 ] 黄静,郑晶,胡乐佳,等. 陆地棉CPP转录因子家族全基因组鉴定及分析[J/OL].分子植物育种:1-11[2021-08-24].http://kns.cnki.net/kcms/detail/46.1068.S.20210127.1655.012.html.
[ 6 ] 薛正刚,王树杰,郜战宁,等.大麦HvCPP转录因子家族的全基因组鉴定与分析[J/OL].分子植物育种,2021,19(9):2811-2818.
[ 7 ] Wang Wanpeng,Sijacic Paja,Xu Pengbo,et al. Arabidopsis TSO1 and MYB3R1 form a regulatory module to coordinate
cell proliferation with differentiation in shoot and root[J].Proc Natl Acad Sci USA,2018,115(13):3045-3054.
[ 8 ] Song X,Nie F,Chen W,et al.Coriander genomics database: a genomic, transcriptomic,and metabolic database for coriander[J].Hortic Res,2020,7(1):55.
[ 9 ] Finn Robert D,Tate John, Mistry Jaina,et al. The Pfam protein families database[J]. Nucleic Acids Res,2008,32(1):138.
[ 10 ] Rozewicki John, Li Songling,Amada Karlou Mar,et al. MAFFT-DASH: integrated protein sequence and structural alig-
nment[J]. Nucleic Acids Res,2019(W1):5-10.
[ 11 ] Nguyen Lam-Tung,Schmidt Heiko A,von Haeseler Arndt, et al. IQ-TREE: A fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies[J].Mol Biol Evol,2015(1):268-274.
[ 12 ] Chengjie Chen, Hao Chen,Yi Zhang ,et al. TBtools: An integrative toolkit developed for interactive analyses of big biological data[J]. Molecular Plant,2020,13(8):1194-1202.
[ 13 ] Wang Yupeng,Tang Haibao,Debarry Jeremy D,et al. MCScanX: a toolkit for detection and evolutionary analysis of gene
synteny and collinearity[J]. Nucleic Acids Res,2012,40(7):49.
[ 14 ] Dapeng Wang,Yubin Zhang,Zhang Zhang,et al. KaKs_Calculator 2.0:A toolkit incorporating gamma-series methods and sliding window strategies[J].Genomics,Proteomics & Bioinformatics,2010,8(1):77-80.
