基于COI和16S rRNA基因片段鉴定厦门海域的仔稚鱼
2021-11-04徐春燕庄之栋沈长春蔡建堤谢少卿
徐春燕,庄之栋,马 超,刘 勇,沈长春,蔡建堤,谢少卿
(福建省水产研究所,福建省海洋生物增养殖与高值化利用重点实验室,福建 厦门 361013)
鱼类早期个体(包括鱼卵、仔稚鱼)发育过程是鱼类生命周期中非常短暂、形态学和生理学等特征变化明显的过渡时期[1],鱼卵和仔稚鱼调查研究是鱼类生态学以及鱼类生活史研究的一部分,同时也是鱼类资源调查、早期补充机制和种群数量变动、渔业资源可持续利用等研究的重要基础[1-2]。目前,鱼卵和仔稚鱼种类的鉴定多是根据形态特征进行区别,但由于个体较小、用于鉴别的形态特征较少,不同种类很难进行准确区分。与已知的成鱼种类相比,已鉴别的鱼卵和仔稚鱼种类少之又少,鱼类早期生活史的相关研究相对滞后,相关形态学资料和图集十分有限,严重限制了鉴定工作的开展;另外国内缺少相关的分类学家,鉴定者的水平不一,很难保证鉴定结果的准确性[3-5]。
2003年,加拿大分类学家Hebert P D N[6]首次提出DNA条形码的概念,此后,DNA条形码技术被广泛应用于各种动物、植物及海洋生物的种类鉴定[7-9]。DNA条形码技术利用DNA序列对物种进行鉴定,突破了传统分类学只能依靠外部形态特征鉴定物种的限制,对产卵场相互交叠和产卵期相互交错、形态特征近似、形态学分类困难的鱼类早期个体种类的分类以及形态学分类上存有疑问的种类的验证提供了新的方法和手段[1]。线粒体细胞色素C氧化酶亚基I(Cytochrome C oxidase subunit I,COI)基因是鱼类常用的条形码基因,被广泛应用于物种鉴定和多样性分析[7,10-12]。然而,目前有研究指出单一的COI基因并不适用于所有鱼类物种的鉴别[13],结合其他条形码基因相互参照和验证是确保条形码技术鉴定准确性的必要手段[7,14-15]。
本研究选取应用比较广泛的COI和16S rRNA基因,对厦门海域采集的仔稚鱼样品进行种类鉴定,通过分析遗传距离及系统进化关系,探究其在仔稚鱼种类鉴定中的适用性,同时可以为仔稚鱼的准确鉴定和分类学研究提供依据和基础资料。
1 材料与方法
1.1 样品采集与形态学鉴定
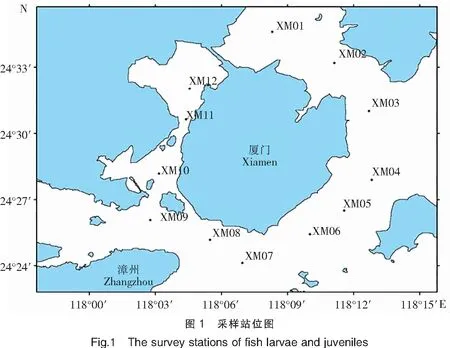
在厦门海域设置12个采样站位(图1),分别于2019年4月22日和2020年9月14日采集仔稚鱼样品。参照GB/T 12763.6—2007《海洋调查规范 第6部分:海洋生物调查》[16],利用大型浮游生物网(网口内径80 cm,网长280 cm,孔径0.505 mm)进行水平拖网采样,样品用95%酒精现场固定,带回实验室进行形态学和分子生物学鉴定。
采集到的仔稚鱼样品首先在奥林巴斯SZX7体视显微镜下根据形态特征进行鉴定和形态学归类,参照Okiyama M的《An Atlas of the Early Stage Fishes in Japan》[17]及张仁斋等的《中国近海鱼卵与仔鱼》[2]进行鉴定,之后根据初步的鉴定结果,每一种选取代表性个体进行基于COI和16S rRNA基因的DNA条形码分析。
1.2 DNA提取、PCR扩增及测序
选取的仔稚鱼样品用QIAGEN DNeasy Blood & Tissue Kit试剂盒进行DNA提取,DNA样品置于4℃保存备用。COI基因序列扩增采用通用引物[18],Fish F1:5′-TCAACCAACCACAAAGACATTGGCAC-3′,Fish R1:5′-TAGACTTCTGGGTGGCCAAAGAATCA-3′;16S rRNA基因序列扩增采用通用引物[19],16S AR:5′-CGCCTGTTTATCAAAAACAT-3′,16S BR:5′-CCGGTCTGAACTCAGATCACGT-3′。
PCR反应体系:2.5 μL 10×buffer,2 μL dNTPs(2.5 mmol/L),正反引物(10 μmol/L)各1 μL ,0.2 μL Taq酶,DNA模板1 μL,超纯水补足至25 μL。反应条件:94℃预变性5 min;94℃变性60 s,50℃退火50 s,72℃延伸60 s,30个循环;最后72℃延伸5 min。用1%的琼脂糖凝胶电泳对扩增产物进行检测,选取扩增效果好的PCR产物送至上海生工进行双向测序。
1.3 序列分析
测序获得的序列首先利用Chromas 2.6.5软件查看原始序列质量,质量好的序列利用Seqman软件进行双向拼接。将拼接所得COI和16S rRNA基因序列在NCBI(National Center for Biotechnology Information)网站进行比对,COI基因同时可以在BOLD(Barcode of Life Database)进行比对。比对后分别根据相似度进行种类鉴定,与库中物种序列相似度≥99%的即鉴定为同一个种,92%~99%之间的鉴定为同一属,85%~92%之间的鉴定为同一科[4],另外,两种基因鉴定结果一致时,最终将此种鉴定为一个物种,结果不一致时,则将其鉴定到科或属。鉴定后相关序列及信息递交至GenBank获取序列号。两种基因分别用MEGAX软件中Clustal W程序进行多重比对,比对后去除两端冗余序列,保留共有序列区域进行后续分析,统计序列的长度、碱基组成、保守位点、变异位点、简约信息位点、单突变位点,种内和种间遗传距离基于Kimura-2-parameter双参数模型(K2P)进行计算,分子系统进化树利用邻接法(Neighbor-joining)构建,以Bootstrap 1 000次重复抽样检验其分支置信度[20]。
2 结果与分析
2.1 测序结果及序列分析
本研究共选取80尾仔稚鱼样品分别进行COI和16S rRNA基因的条形码分析,由于在DNA提取、PCR扩增及测序过程中,均有可能出现结果不佳的情况,最终获得64条COI基因序列和74条16S rRNA基因序列。COI基因序列平均长度655 bp,16S rRNA基因序列平均长度575 bp,序列均递交至GenBank获取序列号,序列号见表1。
COI基因序列的平均碱基组成为T:30.4%、C:28.0%、A:23.4%、G:18.2%,A+T含量(53.8%)高于G+C含量(46.2%),符合多数鱼类COI序列的碱基组成特征。各密码子碱基组成表现出明显的差异性,第2个密码子平均GC含量最高,为56.5%;第3密码子次之,平均为42.7%;第1密码子平均GC含量最低,为39.5%。全部位点中包括保守位点379个、变异位点276个、简约信息位点265个、单突变位点11个。
16S rRNA基因序列的平均碱基组成为T:22.9%、C:25.4%、A:28.6%、G:23.1%,A+T含量(51.5%)高于G+C含量(48.5%),与多数鱼类线粒体DNA的碱基组成特征一致。各密码子碱基组成表现出明显的差异性,第1个密码子平均GC含量最高,为54.8%;第3密码子次之,平均为45.7%;第2密码子平均GC含量最低,为44.9%。全部位点中包括保守位点301个、变异位点298个、简约信息位点278个、单突变位点20个。
2.2 序列比对及仔稚鱼种类鉴定
所得COI基因同时在NCBI和BOLD数据库进行比对,16S rRNA基因在NCBI比对,比对后依据相似度进行鉴定,鉴定结果见表1。COI基因将仔稚鱼样品鉴定为26个种类,其中19个种类鉴定到种、6个鉴定到属、1个种类仅鉴定到科;16S rRNA基因将仔稚鱼样品鉴定为29个种类,其中23个种类鉴定到种、6个鉴定到属。
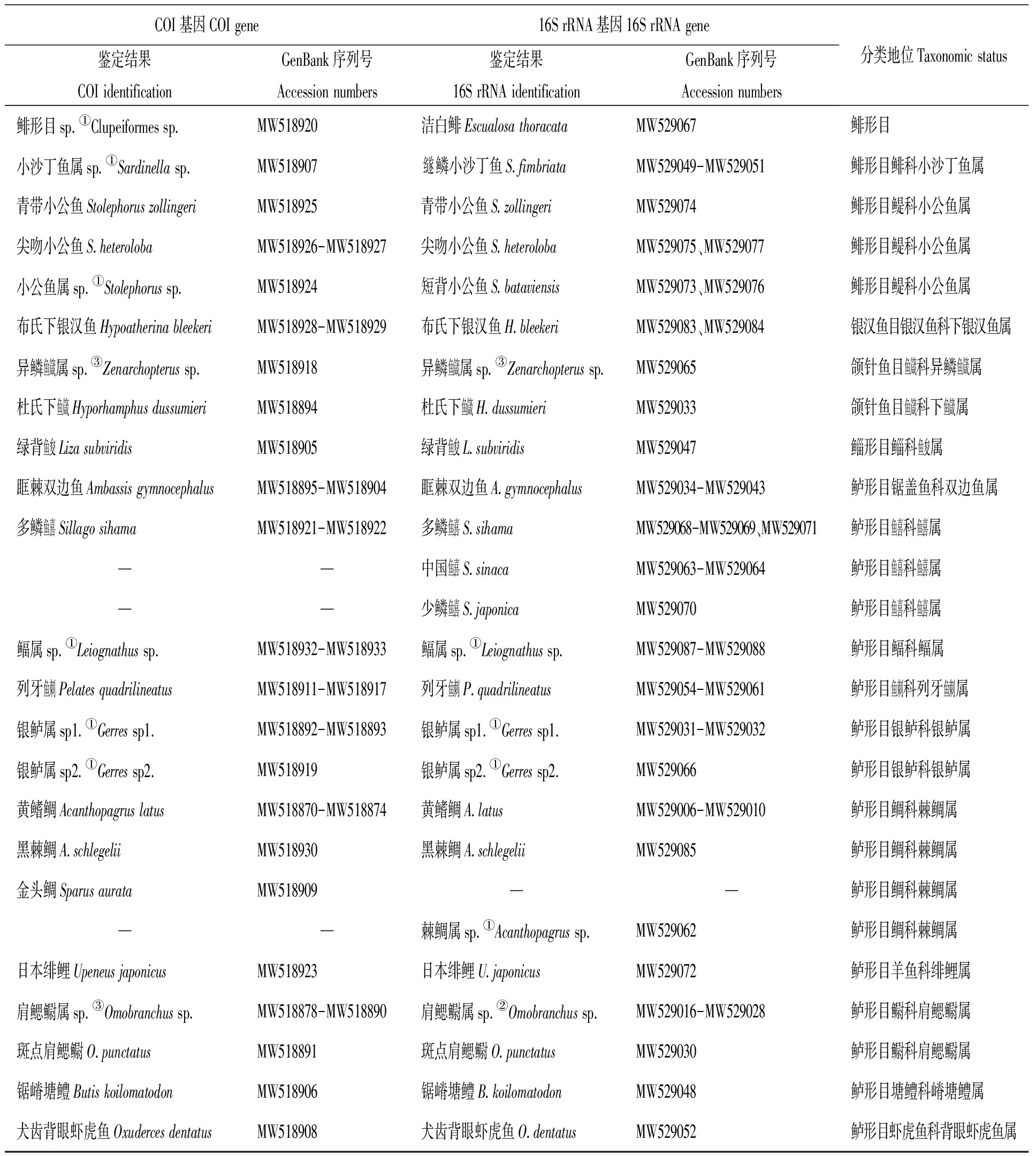
表1 基于COI和16S rRNA基因鉴定的厦门海域仔稚鱼
两种基因鉴定结果基本一致,仅有三处存在分歧。COI基因鉴定为鲱形目sp.(Clupeiformes sp.)的种类,在NCBI和BOLD中与洁白鲱(Escualosathoracata)和印度小公鱼(Stolephorusindicus)相似性均≥99%,不能确定具体为哪一个种类,仅将其鉴定到鲱形目sp.;而NCBI数据库中没有印度小公鱼16S rRNA基因,所以此种16S rRNA基因的比对结果仅与洁白鲱相似性≥99%,将其鉴定为洁白鲱;两种基因比对鉴定结果不同,本研究仅将其鉴定为鲱形目sp.。COI基因鉴定为小沙丁鱼属sp.(Sardinellasp.)的种类, 在NCBI和BOLD中与黑尾小沙丁鱼(S.melanura)、 白腹小沙丁鱼(S.albella)、 纟遂鳞小沙丁鱼(S.fimbriata)和多耙小沙丁鱼(S.jussieui)相似性均大于99%, 无法确定为哪一个种类, 仅鉴定到小沙丁鱼属sp.; 而其16S rRNA基因仅与纟遂鳞小沙丁鱼相似性大于99%, 其余小沙丁鱼属种类相似性均低于99%, 将其鉴定为纟遂鳞小沙丁鱼;本研究将其鉴定为小沙丁鱼属sp.。 COI基因鉴定为小公鱼属sp.(Stolephorussp.)的种类, 在NCBI和BOLD中与短背小公鱼(S.bataviensis)和江口小公鱼(S.commersonnii)相似性均大于99%, 无法确定为哪一个种类, 仅鉴定到小公鱼属sp.; NCBI数据库中没有江口小公鱼16S rRNA基因, 所以此种16S rRNA基因的比对结果仅与短背小公鱼相似性≥99%, 将其鉴定为短背小公鱼; 本研究将其鉴定为小公鱼属sp.。

续表1
2.3 遗传距离
利用MEGAX软件,基于K2P模型计算各分类阶元的遗传距离,结果见表2。
各种类COI基因的种内遗传距离在0~0.005 4之间,平均种内遗传距离为0.001 5(SD=0.001 75)。多个种类的种内遗传距离为0,褐菖鲉的种内遗传距离最大。同属不同物种间遗传距离在0.085 1~0.238 5之间,棘鲷属的黄鳍鲷和黑棘鲷的种间遗传距离最小,小公鱼属的青带小公鱼和小公鱼属sp.的种间遗传距离最大,平均种间遗传距离为0.197 6(SD=0.048 71),约为种内平均遗传距离的132倍,符合Hebert P D N等[21]提出的“10×规则”,即序列的种间遗传距离大于平均种内遗传距离的10倍以上时才能有效鉴别物种。同科不同属的仅鱼箴科的异鳞鱼箴属和下鱼箴属,属间遗传距离为0.216 4。目内科间遗传距离最小值出现在鲈形目弹涂鱼科和虾虎鱼科,为0.153 1;最大值出现在鲈形目羊鱼科和鱼喜科,为0.269 7;平均科间遗传距离为0.238 7(SD=0.018 24)。
16S rRNA基因种内的遗传距离在0~0.002 0之间,平均为0.000 3(SD=0.000 57),各种类的种内遗传距离明显小于其COI序列的遗传距离。多个种类的种内遗传距离为0,篮子鱼属sp.的种内遗传距离最大。同属不同物种间遗传距离在0.026 6~0.164 7之间,棘鲷属的棘鲷属sp.和黑棘鲷的种间遗传距离最小,肩鳃鳚属的斑点肩鳃鳚和肩鳃鳚属sp.的种间遗传距离最大,平均种间遗传距离为0.089 2(SD=0.045 42),同样超过种内平均遗传距离的10倍以上。同科不同属的鱼箴科的异鳞鱼箴属和下鱼箴属,属间遗传距离为0.182 3。目内科间遗传距离最小值同样出现在鲈形目弹涂鱼科和虾虎鱼科,为0.059 1;最大值出现在鲈形目鳚科和银鲈科,为0.227 5;平均科间遗传距离为0.162 3(SD=0.033 63)。
由表2可以看出,COI和16S rRNA基因遗传距离均随着分类阶元的提高而增大,但增幅随着分类阶元的提高越来越小。
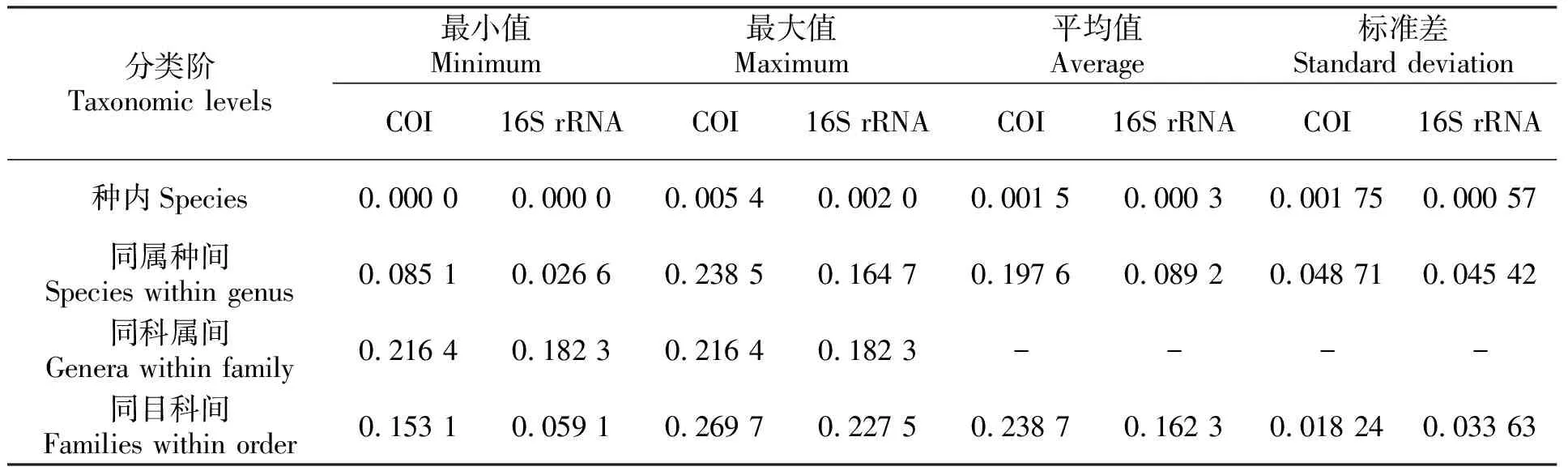
表2 不同分类阶元遗传距离
2.4 系统进化树
基于64条COI基因序列和74条16S rRNA基因序列分别构建系统进化树(图2、图3)。由图可以看出,基于COI和16S rRNA基因序列的系统进化树结果基本一致,在种的水平上,聚类效果清晰,所有物种都分别聚为独立的一支从而被有效区分,同一个种类的不同个体也都能聚在同一个分支。同属的个体在基于16S rRNA基因构建的系统进化树上聚类效果也很明显,如棘鲷属、银鲈属、属、肩鳃鳚属、小公鱼属的不同种类分别聚为一支,而基于COI基因构建的进化树上,肩鳃鳚属的斑点肩鳃鳚和肩鳃鳚属sp.并未聚在一支。在科和目的水平上,两条进化树的聚类效果都不清晰,聚类关系相互交叉、比较混乱。


3 讨论
3.1 COI和16S rRNA基因在仔稚鱼种类鉴定中的适用性及在系统进化分析中的局限性
Hebert P D N等[22]的研究表明,同一物种内的遗传距离多小于0.020,0.020常作为DNA条形码物种划分的阈值,另外指出只有种间遗传距离大于种内遗传距离的10倍以上时才能有效鉴别物种[21]。
本研究各个种类COI基因的种内遗传距离(0~0.005 4)均小于0.020,同属种间的遗传距离(0.085 1~0.238 5)均大于0.020,符合Hebert P D N等提出的种内遗传距离的标准,且种间和种内遗传距离分布范围没有交叉,存在明显的条形码间隙;平均种间遗传距离(0.197 6)为种内遗传距离(0.001 5)的约132倍;以上结果均表明,COI基因可以作为条形码对仔稚鱼进行种类鉴定。16S rRNA基因种内遗传距离(0~0.002 0)远小于0.020,同属种间的遗传距离(0.026 6~0.164 7)远远大于种内遗传距离分布范围,平均种间遗传距离(0.089 2)大于平均种内遗传距离(0.000 3)的10倍以上,16S rRNA基因同样也适合用来进行仔稚鱼的种类鉴定。另外,基于COI基因和16S rRNA基因构建的系统进化树也直观地反映出两种基因都能将所有物种区别开来,印证了两种基因在仔稚鱼种类鉴定中的适用性。
COI和16S rRNA基因在同科属间及同目科间的遗传距离的分布范围存在重叠,对基于COI和16S rRNA基因构建的系统进化树的分析也表明,在科和目的水平上,聚类关系不是很清晰。这说明,COI和16S rRNA基因不适合用来分析种以上高分类阶元的系统进化关系,这与其他诸多研究者的结论[12,23-24]相一致。
3.2 COI和16S rRNA基因对厦门海域仔稚鱼的鉴定结果分析
本研究共选取80尾仔稚鱼样品进行DNA提取、COI和16S rRNA基因的扩增、测序、序列比对与分析。通过序列比对,COI基因将仔稚鱼样品鉴定为26个种类(4个种类的样品PCR扩增失败),16S rRNA基因将仔稚鱼样品鉴定为29个种类(1个种类的样品PCR扩增失败),总的来说,16S rRNA基因通用引物扩增效率高于COI基因。除去扩增失败的种类,两种基因的鉴定结果基本一致,存在分歧的3处,多是由于COI基因比对结果与多个近缘种相似而无法确定具体哪个种类,而NCBI数据库中16S rRNA基因序列数据不足,16S rRNA基因比对结果仅与其中一个种类相似性大于99%。目前,DNA条形码数据库并不全面,尤其是一些非经济种和少见种的COI和16S rRNA基因序列还十分欠缺。另外,数据库中存在一些错误鉴定后提交的序列,如果未能加以甄别则会干扰后续研究者序列的比对和物种鉴定[25]。因此,DNA条形码基因序列库的完整和准确是物种鉴定的前提条件[26],除不断补充DNA条形码数据库外,还必须严格开展序列的审查工作,保证数据库中序列的质量,从而更好地发挥其在物种鉴定中的作用。
本研究中鉴定为鲱形目sp.、小沙丁鱼属sp.、小公鱼属sp.、鲾属sp.、银鲈属sp1.、银鲈属sp2.、棘鲷属sp.、篮子鱼属sp.的种类,经COI或16S rRNA基因比对后,均与数据库中多个种类高度匹配,相似性均大于99%,无法鉴定到种的水平。说明有些鱼类不仅形态上非常相似、无法区分,序列差异也较小,利用COI或16S rRNA基因均无法准确鉴定,这也是利用DNA条形码技术进行种类鉴定的难点[27],针对这种情况,仍需要结合更多分子标记(如线粒体Cytb基因、核基因分子标记RAG2等)作进一步研究。
综上所述,COI和16S rRNA基因均可以作为条形码对形态学上难以区分的仔稚鱼进行种类鉴定,两种基因鉴定结果基本一致,且多数能鉴定到种的水平,两种基因结合使用可以相互印证或补充,从而提高鉴定结果的准确性。由于本研究采集到的样品数量有限,有的种类仅1条序列,相关数据不够全面,下一步应采集更多季节或月份的仔稚鱼样品,对DNA条形码技术在仔稚鱼种类鉴定中的应用进行更充分的研究和探讨。
