甜瓜SLAF图谱构建及果实相关性状QTL分析
2021-10-20王岭才羿王桂超王迪盛云燕
王岭,才羿,王桂超,王迪,盛云燕
甜瓜SLAF图谱构建及果实相关性状QTL分析

1黑龙江八一农垦大学园艺园林学院,黑龙江大庆 163319;2黑龙江省农业科学院大庆分院,黑龙江大庆 163319
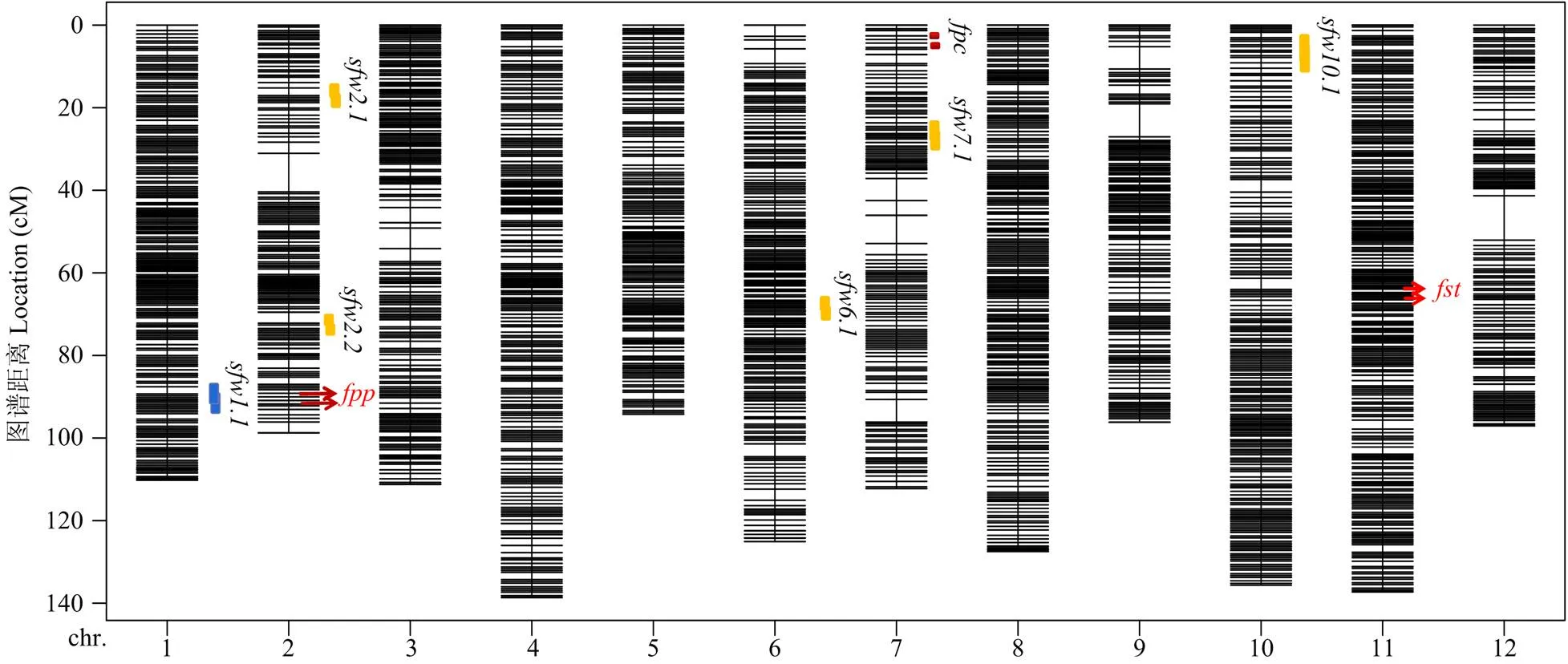
【】通过对甜瓜果实相关性状进行QTL定位及候选基因分析,为甜瓜品质育种及基因挖掘与功能验证提供理论基础。利用薄皮甜瓜1244为母本、厚皮甜瓜M5为父本配置杂交组合,结合SFLA测序技术开发分子标签,构建高密度遗传图谱,以F2:3表型数据为基础,采用Mapqtl进行QTL定位分析。获得了380 446 Mreads(83.12 Gb)数据,测序平均Q30为93.59%,平均GC含量为36.87%;开发了112 844个SLAF标签,3 274 879个SNP;构建了12个连锁群,共计10 596个上图标记,总图距为1 383.88 cM,标记间平均距离为0.13 cM,上图标记完整度平均为99.92%。将控制甜瓜果面沟性状基因()定位在第11条染色体末端Marker 1993423(62.18)—Marker 1998820(63.05),覆盖基因组0.72 Mb,包含33个候选基因;控制甜瓜果皮花纹基因()定位在第2条染色体Marker 459584(90.91)—Marker 459446(90.91),覆盖基因组0.08 Mb,包含5个候选基因,其中MELO3C026292(1-脱氧-D-木酮糖-5-磷酸还原异构酶)可能为控制果皮花纹的候选基因;同时检测到甜瓜果皮底色1个QTL位点,位于第7条染色体Marker 1229174(7.14)—Marker 1229973(7.14),贡献率为9.9%;检测到果型指数1个QTL位点,位于第9条染色体Marker 1705671(76.19)—Marker 1705915(79.23),贡献率为7.6%;在第1、2、6、7、10染色体检测到单果重相关6个QTL位点(、、、、、),贡献率在3.1%—17.0%,LOD值介于3.0—5.6。将甜瓜果面沟、果皮花纹基因分别定位在第11和第2条染色体,分别获得33个和5个候选基因;检测到1个果皮底色QTL位点、1个果型指数QTL位点和6个单果重QTL位点。
甜瓜;SLAF测序;遗传图谱构建;果实性状;QTL分析
0 引言
【研究意义】甜瓜具有悠久的栽培历史,作为葫芦科重要的经济作物,由于其具有丰富的多态性,逐渐成为研究果实性状的模式植物之一[1-2]。但是随着甜瓜产业的迅猛发展,消费需求与甜瓜特异品种缺乏的矛盾也日益突出[2]。甜瓜果实性状为消费者选择购买的首要依据,不同地区消费者对果皮花纹、颜色的认可标准存在巨大差异。本研究配置杂交组合,利用F2:3群体对甜瓜果实相关性状开展基因定位,对甜瓜种质资源创新与分子标记辅助选择育种具有一定的实践价值,对甜瓜果实相关性状的基因挖掘也具有一定的理论价值。【前人研究进展】甜瓜皮色变异丰富,黄、白、绿为主流颜色,而基于成熟度不同在底色上显现的花纹,又可分为斑点、斑块、斑条、条带等[3-5]。近年来,一些研究者通过QTL作图、BSA法、GWAS等方法对控制甜瓜果肉颜色的基因位点进行了定位分析[6-9],但是果皮颜色基因的研究报道相对较少。果皮果面沟(缝),在不同品种中呈现有或无的情况,该性状对果实品质存在一定影响[3]。2019年,Zhao等[1]利用GWS测序技术预测了果面缝的4个候选基因,这与邱果[10]将甜瓜果面沟基因定位在第11条染色体末端的结果相符合。相较于甜瓜其他农艺性状,甜瓜果实形状与重量的研究逐渐深入,目前已经报道QTL位点达到200余个,并且平均地分配在甜瓜12条染色上[2,8,11],国外研究者发现甜瓜果实形状基因与番茄同源[12],为甜瓜果实形状基因挖掘提供了依据[13]。【本研究切入点】前人在果实相关性状研究方面取得了较大进展,但仍有许多农艺性状定位遗传距离较远,QTL定位较分散,不利于甜瓜果实相关性状遗传机理研究。高密度分子遗传图谱有助于提高甜瓜的育种水平,加快育种进程。从1996年第一张甜瓜分子遗传图谱报道后,分子标记在甜瓜基因定位等方面显示出巨大的作用[14]。而甜瓜基因组(454 Mb)数据的公布,为甜瓜重要基因挖掘提供了依据[15]。葫芦科基因组数据库(Cucurbit Genomics Database,CuGenDB,http://cucurbitgenomics.org)数据的不断更新,为葫芦科物种基因组数据分析、遗传连锁图谱构建、基因的注释提供了重要的生物信息分析工具[1]。【拟解决的关键问题】本研究利用高通量测序技术对构建的F2群体单株开展SLAF测序技术开发标签并构建图谱,对甜瓜果实相关性状进行QTL分析及基因定位,为相关基因精细定位、功能基因挖掘提供理论基础。
1 材料与方法
1.1 试验材料
利用薄皮甜瓜1244为母本(薄皮甜瓜,无果面沟,成熟后果实底色为灰白色,有花纹,果实长圆形),厚皮甜瓜M5为父本(厚皮,有果面沟,成熟后果皮底色为金黄色,果皮无花纹,果实圆形)(图1),配置杂交组合,获得F1,自交、回交后获得195个F2:3家系和150个单株的BC1群体。
1.2 试验方法
2018—2019年在大棚种植亲本、F1、F2及BC1群体。2018年春季亲本及F1播种各30株,每个小区种植10株,3次重复;195株F2代群体随机种植,自交获得F3代种子;种植BC1P1和BC1P2两个回交群体各150株,调查相关性状。2019年3月,播种195个F2:3家系,每个家系10株,单蔓整枝,第7—10节子蔓留单果,采收果实和种子,用以田间表型数据调查及遗传分析。
1.3 果实相关性状调查
田间性状调查依据《甜瓜种质资源描述规范和数据标准》进行,在果实成熟期,对亲本及群体单株果实相关性状进行调查统计并拍照记录。根据表型鉴定结果,确定各性状在BC1、F2、F3各家系性状的分离情况,并进行卡方检验,用于遗传分析。
调查性状为:果面沟(fruit sutures,):有记为1,无记为0;果皮花纹(fuit pericarp pattern,):有记为1,无记为0;果皮底色(fruit pericarp color,):果实成熟后记录果皮底色,白绿色为1、绿色为2、黄绿为3、黄白色为4、黄色为5;果实单果重(single fruit weight,):果实成熟后利用天平称量单果重;果型指数(fruit shape,):果实纵径与果实横径的比值。
1.4 遗传图谱构建
利用CTAB改良法[16]提取基因组DNA后,运用SLAF-seq技术(specific-locus amplified fragment sequencing)构建甜瓜高密度遗传图谱,然后对果实相关性状基因进行QTL分析与基因定位。
1.4.1 酶切方案设计及测序 对参考物种基因组序列进行电子酶切预测,根据酶切方案选择原则,选取Hpy166II酶切组合,酶切片段长度在264—414 bp的序列定义为SLAF标签,预测可得到112 844个SLAF标签,SLAF-seq文库的测序原始测序读长为PE150 bp。
1.4.2 SNP标记开发 使用GATK软件工具包开发SNP标记。根据Clean Reads在参考基因组的定位结果,通过去重复(mark duplicates)、GATK进行局部重比对(local realignment)、碱基质量值校正(base recalibration)等预处理,再使用GATK进行单核苷酸多态性(single nucleotide polymorphism,SNP)的检测过滤,获得最终的SNP位点集。
1.4.3 遗传图谱构建 将筛选出的SNP标记通过与参考基因组的定位均匀分布到12个连锁群,通过标记间计算MLOD值,过滤掉与其他SNP的MLOD值均低于3的标记,定位为上图标记(Marker)。采用HighMap软件分析获得连锁群内Marker的线性排列,并估算相邻Marker间的遗传距离,构建甜瓜遗传图谱。
1.4.4 果实相关性状QTL分析 QTL分析基于表型数据值,采用Mapqtl方法对果实相关性状进行初步分析,LOD阈值为3.0,QTL命名采用英文缩写+染色体编号+位点序列号方法。
1.4.5 候选基因预测 利用Softberry(www.softberry. com)、国际葫芦科数据(www.icugi.org/pub/cucurbit/ genome)进行候选基因预测。
2 结果
2.1 甜瓜果实相关性状的遗传规律分析
对供试甜瓜亲本、F1及F2群体进行相关性状调查,母本1244为野生材料,果面光滑无果面沟,果实成熟后,果皮白绿色,单果重4.6 g,果实纵径与横径比值为1.7,椭圆形;父本M5为厚皮甜瓜,具有典型的果面沟,果实成熟后,果皮黄色,果肉黄色,果皮无花纹,单果重575 g,果实总径与横径比值为0.8,果实呈扁圆形(图1)。

图1 甜瓜供试亲本及F2单株表型
2018种植甜瓜F1及F2群体单株性状调查显示,F1具有果面沟,果皮有花纹,果皮底色白色偏黄,单果重165 g,果型指数为1.5,果型椭圆形;195个F2单株中果面沟有﹕无为145﹕50,符合3﹕1分离比率,果皮花纹有﹕无为152﹕43,分离比率符合3﹕1;回交群体BC1P1均具有果面沟,果皮有花纹,BC1P2果面沟有﹕无为76﹕74,果皮花纹有﹕无为81﹕69,符合1﹕1分离比率;卡方检测符合孟德尔遗传规律显著性分析标准。综上所述,甜瓜果面沟、果皮花纹均为1对显性基因控制的质量性状(表1)。
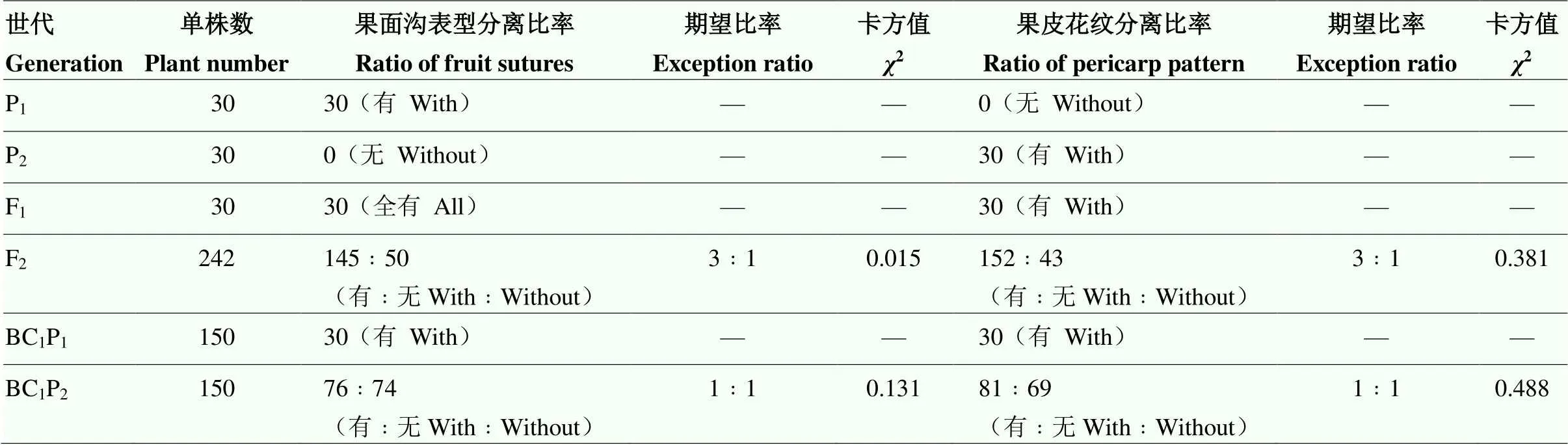
表1 甜瓜果皮相关性状分离比率
20.05=3.84
对F2群体果型指数与单果重进行测量分析,各性状F2单株呈正态分布,变异幅度较大,符合存在主效基因的遗传规律(图2)。
2.2 测序数据评估分析
利用Hiseq2500平台进行SLAF-seq文库的测序分析。结果显示,共获得80 446 Mreads(83.12 Gb)数据,测序平均Q30为93.59%,平均GC含量为36.87%,样本GC分布正常,亲本平均测序深度为23.21X,子代平均测序深度为25.50X。
1244和M5分别获得38 911 978 reads(11 653 958 274 bp)、31 878 110 reads(9 548 013 962 bp)数据,测序平均Q30为92.31%和92.67%,平均GC含量为37.01%和36.72%;子代群体获得了2 692 579 reads,538 400 027 bp,测序平均Q30为95.63%,平均GC含量为36.88%(表2)。对照数据的双端比对效率为91.26%,酶切效率为96.69%,建库数据符合要求。
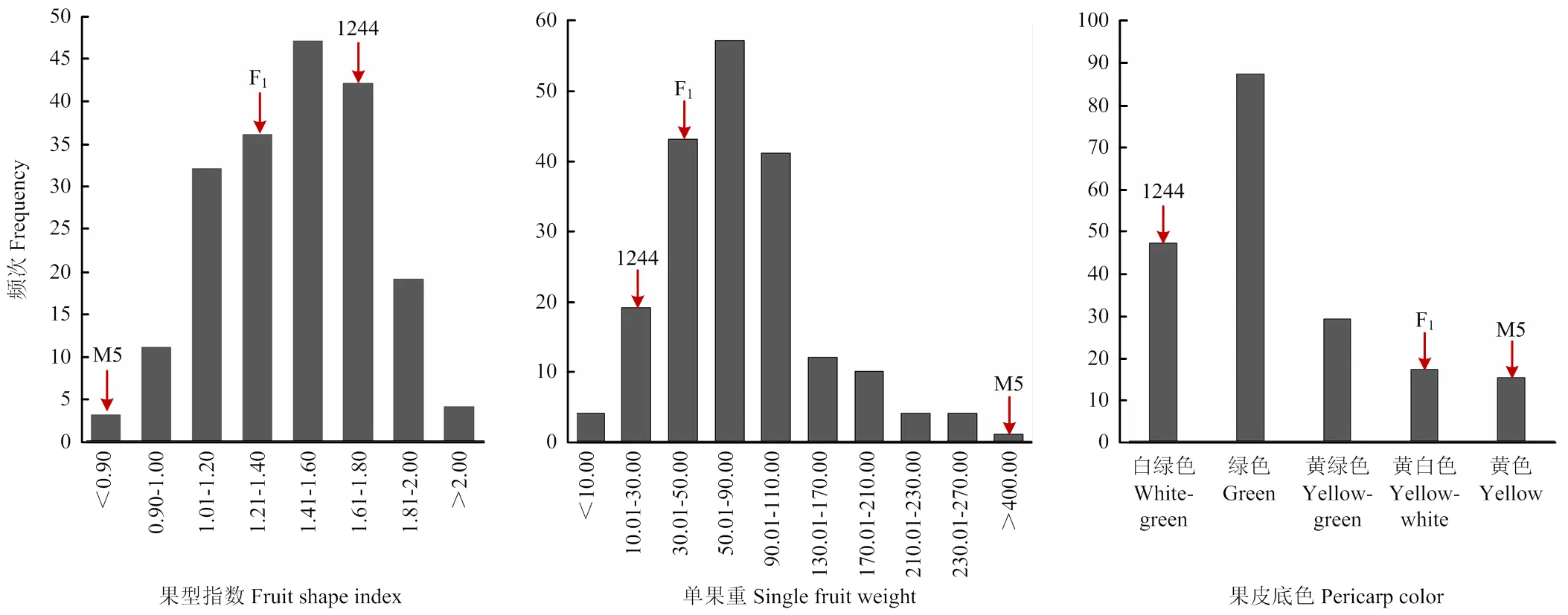
图2 甜瓜F2群体及亲本甜瓜单果重、果型指数与果皮底色表型的频率分布
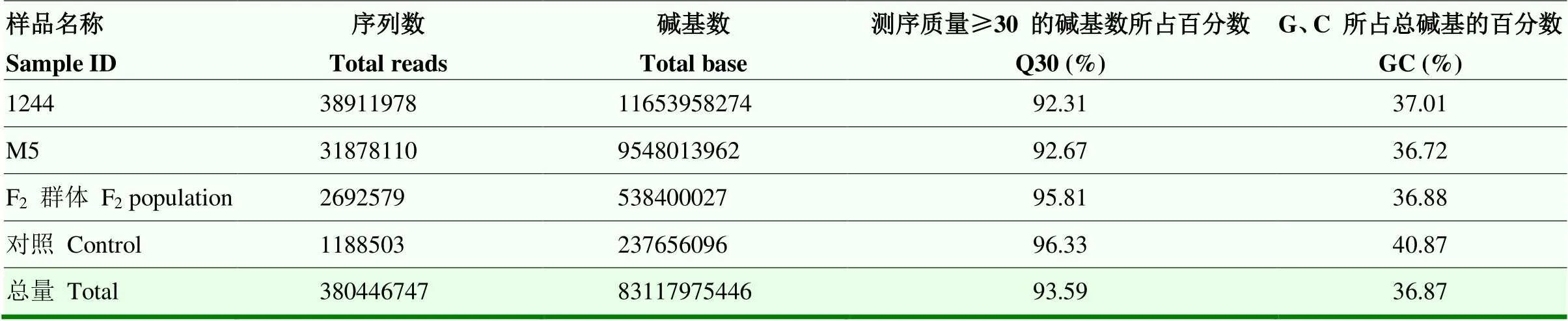
表2 测序质量分析
2.3 标记开发及SLAF 遗传图谱构建
利用GATK软件工具包分别对测序数据进行比对和SNP开发。共获得3 274 879个SNP标记,能成功分型的有2 218 102个,可以用于遗传图谱构建的标记有 1 898 620个。M5和1244分别获得SNP标记数量为2 250 862和1 489 769个,杂合率分别为12.55%和9.82%;子代测序获得SNP标记806 865个,杂合率为22.19%(表3)。
将筛选出的12 567个标记,通过与甜瓜参考基因组(www.icugi.org/pub/cucurbit/genome/melon/v3.5.1/)比对,共有10 596个标记上图,标记上图完整率平均为99.20%。构建了一张总图距为1 383.88 cM,包含12个连锁群,标记间平均遗传距离为0.13 cM的甜瓜遗传图谱(表4),标记数最多的为第11条染色体(1 579),标记数最少的为第7条染色体(300)(表4)。将建图所用的SLAF标记在基因组和遗传图谱进行共线性分析,各个连锁群上的标记顺序和基因组大部分保持一致,斯皮尔曼值位于0.963—0.996。
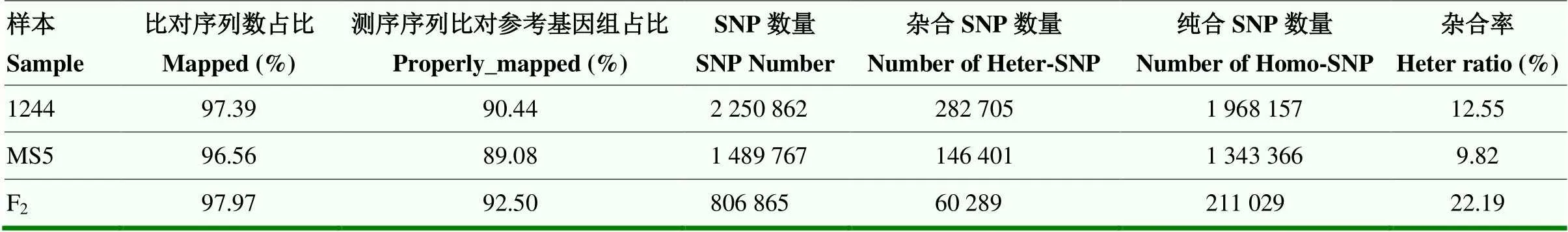
表3 测序数据比对和SNP统计

表4 甜瓜SLAF测序构建遗传图谱
2.4 果实性状QTL分析及基因定位
甜瓜果面沟为单个显性基因控制的性状,将该性状基因定位在第11条染色体尾端Marker 1993423(62.18)—Marker 1998820(63.05),基因组位置为20 408 304—21 129 670,覆盖0.72 Mb,包含33个候选基因(图3)。
甜瓜果皮花纹基因定位在第2条染色体,位于Marker459584(90.91)—Marker 459446(90.91),位于甜瓜基因组26 112 655—26 189 137,覆盖0.08 Mb,包含5个基因(图3、4)。
fsw:单果重 Single fruit weight;fpc:果皮底色 Pericarp color;fpp:果皮花纹
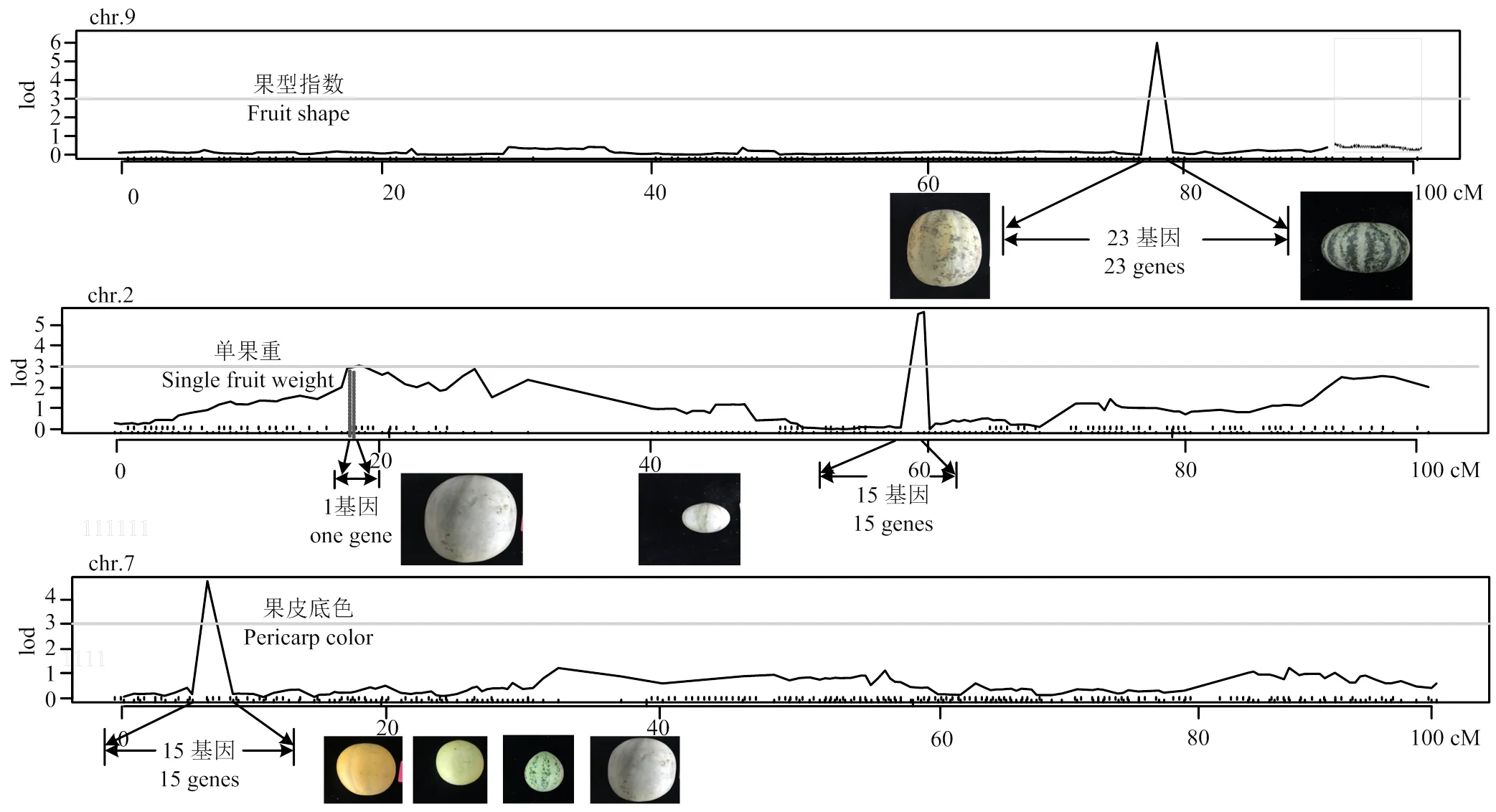
图4 QTL分析与候选基因
果型指数检测到1个QTL位点,位于第9条染色体,Marker 1705671(76.19)—Marker 1705915(79.23)之间,基因组起始位点为23 034 060— 23 259 152,该主效QTL贡献率为7.6%,LOD值为5.9(图4),加性效应为0.19,说明该性状QTL位点来源于母本效应,该候选区域覆盖基因组0.23 Mb,通过softberry、NCBI等在线软件分析,发现该候选区域包含23个候选基因,其中7个未知功能基因,剩余基因包含与乙烯结合转录因子、钙调蛋白、类糖载体蛋白基因及脱水效应元件结合蛋白等基因。
甜瓜单果重共检测到6个QTL位点,分别位于第1、2、6、7、10条染色体,其中第2条染色体贡献率最高,为17.0%,LOD值为5.6,加性效应为-45.10,主要受父本遗传效应影响。该区域位于Marker 414271(60.38)—Marker 413648(60.82),基因组1 626 517—1 635 504,共计0.54 Mb,包含15个候选基因,其中3个未知功能基因(图4)。第2条染色体前端的QTL位点,位于Marker 316077(18.35)—Marker 316123(18.35),贡献率为3.1%,LOD值为3.0,加性效应为17.81,为母本遗传效应,该候选区域位于甜瓜基因组18 344 184—18 881 371,覆盖基因组0.01 Mb,包含1个候选基因。其余检测到的QTL位点、、和贡献率分别为8.3%、10.2%、5.9%和8.3%,LOD值分别为3.3、4.2、3.1和3.2。加性效应均为负值,说明这些位点主要受父本遗传效应的影响(表5)。
果皮底色共检测到1个主效QTL位点,该位点位于第7染色体,位于Marker 1229174(7.145)—Marker 1229973(7.14),贡献率为9.9%,LOD值为4.7,加性效应为-0.44,受父本遗传效应影响。该QTL位点所在区域位于甜瓜基因组709 451—892 244,覆盖基因组0.18 Mb,通过基因比对,包含15个候选基因(表5)。

表5 甜瓜果实相关性状QTL位点与基因定位
3 讨论
构建高密度作物遗传图谱,可为基因定位、QTL分析提供重要的保障[2,6,17]。随着高通量测序技术的快速发展,甜瓜高密度遗传图谱的构建步入快速发展期,成为挖掘功能基因的重要途径之一[1-2]。GALPAZ[6]、张学军[17]、刘相玉[18]、张肖静[19]等通过构建甜瓜遗传图谱,实现了对甜瓜多种性状QTL位点候选基因的分析,为甜瓜分子遗传育种提供了重要的理论基础。本研究利用SLAF测序技术,以甜瓜F2:3群体构建了一张包含10 596个分子标记,标记间遗传距离为0.13 cM的遗传图谱,为甜瓜果实性状QTL分析及基因定位奠定了基础。
甜瓜众多农艺性状已被国内外研究报道,果面沟作为潜在影响甜瓜品质的重要性状,逐渐成为研究的热点之一。邱果[10]利用分离群体分组混合分析法(bulked segregant analysis,BSA)在第11号染色体上得到一个距离为1.1 cM的位点,并找到与其连锁的分子标记。Zhao等[1]利用GWAS测序技术,挖掘了一个与甜瓜果缝相关的位点,该位点位于甜瓜第11条染色体末端,覆盖基因组2 Mb,鉴定了4个候选基因,实现了果面缝的定位。综合前人的研究结果,从表型数据分析来看,果面沟(缝)属于典型的质量性状,候选目标区域位于甜瓜第11染色体末端,本研究与前人研究结果一致,但是在候选基因位置方面,仍需要构建永久群体,进一步深入研究,开展精细定位。
甜瓜果皮底色在白色、绿色及黄色的基础上,产生了巨大的变异,而果皮底色对甜瓜生产销售具有重要的影响。前人对果皮颜色遗传规律研究较深入,明确了黄色对白色、灰白色、绿色为不完全显性,绿色对白色、灰白色为显性的遗传规律[20]。近年来,相继报道了控制甜瓜绿色果皮基因[5]、橙色果肉的基因[19]和控制白色果肉的基因[6]。本研究将果皮底色性状按照数量性状分析,扫描到1个QTL位点,这与杨光华等[7]将果皮底色作为质量性状进行遗传研究的结果一致,但是本研究将控制果皮底色的基因定位在甜瓜第7染色体,这与前人的研究结果存在差异,进一步说明果皮颜色性状复杂,不能将该性状视为简单的质量性状,存在多个有效位点的可能性。甜瓜果皮覆纹在果皮成熟的基础上,形成条带纹、斑点纹、不规则花纹等不同特征。刘柳[3]对甜瓜果实条纹开展研究显示,条纹对纯色果皮为显性,并将控制条纹的基因定位在甜瓜第4条染色体上。本研究结果显示花纹对纯色底色为显性,单一基因控制,该基因位于甜瓜第2条染色体,与吕建春[4]研究候选染色体相同,并鉴定了、、和可能为候选基因。本研究在第2条染色体获得5个候选基因,其中与前人研究结果一致,推测该基因可能为控制果皮花纹的候选基因。下一步将加大群体,增加候选区间的标记数量,对果皮花纹基因开展精细定位,克隆候选基因。
目前报道的果实大小及果型指数相关QTL位点约有200多个[3-5,8,21-26]。甜瓜果实单果重与果型指数均为典型的数量性状,且易受环境条件影响。王贤磊[26]、张宁[27]、张雪娇[28]、栾非时[16]等定位了多个与果型指数、果实单果重相关的QTL位点,基本分布在甜瓜12条染色体。本研究挖掘到位于甜瓜第2条染色体中间靠后位置的,初步分析为本研究控制果实单果重的主效位点;该区域包含15个候选基因,其中5个未知功能基因,和为丝氨酸相关基因,该区域为DELLA蛋白家族基因,为DELLA蛋白家族基因,有研究指出蛋白介导各种激素调控,参与植物生长发育[29];此外,丝束蛋白()基因、类糖载体蛋白基因()、转录因子()等相关基因也位于候选基因中。另外,位于第2条染色体的,候选基因包含1个基因(),该基因为减数分裂相关基因,而减数分裂在植物形态建成方面具有重要的影响,因此该基因作为微效QTL位点的候选基因,下一步将开展基因功能验证,明确该基因的作用机制。此外,该位点作为唯一加性效应为正值的位点,说明母本在育种值中具有正向作用,因此该位点的深入研究,对甜瓜育种具有一定的实践指导意义。贡献率为10.2%,贡献率为5.9%,这两个候选区域较大,包含候选基因数量较多。贡献率为8.3%,候选区域包含2个候选基因,分别为和,功能注释显示为未知功能和氨基脱氧分支酶,相关基因在植物生长调节中的作用,还有待进一步的研究。
果实形状是重要农艺性状之一。美国威斯康星大学翁益群教授团队将78个控制葫芦科果实形状的QTL进行整合,发现多个区间内存在影响番茄果实大小的同源基因[30]。在调控球形甜瓜果实发育的基因(,OVATE-domain family protein)已被克隆,并证实与果实大小相关[21,31-32]。本研究母本为椭圆形,父本为扁圆形,通过QTL分析在甜瓜第9条染色体检测到1个QTL位点,如果按照母本为长圆形,父本为扁圆形的表型数据统计分析,长圆形对扁圆形为显性,符合前人认为甜瓜果实长形对圆形为显性的研究结果[31]。果型指数QTL位点候选区域包含的23个基因,除7个未知功能外,还包含各种类糖载体蛋白基因、钙离子通道相关基因及乙烯转录调控因子。乙烯参与植物的形态建成、生长发育等多个环节,因此加大群体,开展果型指数位点的精细定位,明确候选基因,可为葫芦科果实形状的深入研究提供理论基础。
4 结论
利用SLAF测序技术构建了包含12条染色体的甜瓜高密度遗传图谱,该图谱包含10 596个标记,总图距1 383.88 cM,相邻2个标记间的平均遗传图距为0.13 cM。利用F2:3家系在第9条染色体检测到果型指数1个QTL位点,包含23个候选基因;在第1、2、6、7、10染色体检测到单果重相关6个QTL位点(、、、、、),其中为主效QTL位点,包含15个候选基因;将控制甜瓜果面沟性状基因定位在第11条染色体末端Marker 1993423(62.18)—Marker 1998820(63.05),覆盖基因组0.72 Mb,包含33个候选基因,控制甜瓜果皮花纹基因定位在第2条染色体Marker 459584(90.91)—Marker 459446(90.91),覆盖基因组0.08 Mb,包含5个候选基因;检测到甜瓜果皮底色1个QTL位点,位于第7条染色体,包含15个候选基因。
[1] ZHAO G W, LIAN Q, ZHANG Z H, FU Q S, HE Y H , MA S W , RUGGIERI V, MONFORTE A J, WANG P Y, JULCA I, WANG H S, LIU J P, XU Y, WANG R Z, JI J B, XU Z H, KONG W H, ZHONG Y, SHANG J L, PEREIRA L,. A comprehensive genome variation map of melon identifies multiple domestication events and loci influencing agronomic traits. Nature Genetics, 2019, 51: 1607-1615.
[2] 高鹏, 刘识, 崔浩楠, 张泰峰, 王学征, 刘宏宇, 朱子成, 栾非时. 甜瓜基因组学、功能基因定位及基因工程育种研究进展. 园艺学报, 2020, 47(9): 1827-1844.
GAO P, LIU S, CUI H N, ZHANG T F, WANG X Z, LIU H Y, ZHU Z C, LUAN F S. Research progress of melon genomics, functional gene mapping and genetic engineering. Acta Horticulturae Sinica, 2020, 47(9): 1827-1844. (in Chinese)
[3] 刘柳. 甜瓜果皮条纹的遗传分析及其决定基因的精细定位[D]. 哈尔滨: 东北农业大学, 2019.
LIU L. Genetic analysis of rind stripe and fine mapping of the determinate gene in melon (L.) [D]. Harbin: Northeast Agricultural University, 2019. (in Chinese)
[4] 吕建春. 薄皮甜瓜(L. var.Pangalo)果皮花斑的遗传分析及基因定位[D]. 北京: 中国农业科学院, 2018.
LÜ J C. Inheritance and gene mapping of spotted trait in melon (L. var.Pangalo) [D]. Beijing: Chinese Academy of Agricultural Sciences, 2018. (in Chinese)
[5] 欧点点. 甜瓜果皮颜色遗传分析及绿色果皮基因定位[D]. 北京:中国农业科学院, 2019.
OU D D. Genetic Analysis and Gene Mapping for Green Peel Color in Melon (L.) [D]. Beijing: Chinese Academy of Agricultural Sciences, 2019. (in Chinese)
[6] Galpaz N, Gonda I, Shem-Tov D, Barad O, Tzuri G, Lev S, Fei Z J, Xu Y M, Mao L Y, Jiao C, Harel-Beja R, Doron-Faigenboim A, Tzfadia O, Meir E B A, Sa'ar U, Fait A, Halperin E, Kenigswald M, Fallik E, Kol N L G,. Deciphering genetic factors that determine melon fruit-quality traits using RNA seq-based high-resolution QTL and eQTL mapping. Journal of Molecular Cell Biology, 2018, 94(1): 169-191.
[7] 杨光华, 范荣, 杨小锋, 侯军亮, 袁士臣, 曹明, 王学林, 李劲松. 甜瓜果实颜色3个质量性状基因的定位. 园艺学报, 2014, 41(5): 898-906.
YANG G H, FAN R, YANG X F, HOU J L, YUAN S C, CAO M, WANG X L, LI J S. Construction of a highly dense genetic map using SNP and mapping of three qualitative traits in. Acta Horticulturae Sinica, 2014, 41(5): 898-906. (in Chinese)
[8] GUR A, TZURI G, MEIR A, SA'AR U, PORTNOY V, KATZIR N, SCHAFFER A A, LI L, BURGER J, TADMOR Y. Genome-wide linkage-disequilibrium mapping to the candidate gene level in melon (). Scientific Reports, 2017, 7: 9770.
[9] FEDER A, BURGER J, GAO S, LEWINSOHN E, KATZIR N, SCHAFFER A A, MEIR A, DAVIDOVICH-RIKANATI R, PORTNOYV, GAL-ON A, FEI Z J, KASHI Y, TADMOR Y. Focus on metabolism: A kelch domain-containing F-Box coding gene negatively regulates flavonoid accumulation in muskmelon. Plant Physiology, 2015, 169(3): 1714-1726.
[10] 邱果. 甜瓜果面沟相关基因定位与相关性分析[D]. 哈尔滨: 东北农业大学, 2018.
QIU G. Location of surface groove related gene and correlation analysis in melon [D]. Harbin: Northeast Agricultural University, 2018. (in Chinese)
[11] CHANG C W, WANG Y H, TUNG C W. Genome-wide single nucleotide polymorphism discovery and the construction of a high- density genetic map for melon (L.) using genotyping- by-sequencing. Frontiers in Plant Science, 2017, 8: 125.
[12] WU S, ZHANG B Y, KEYHANINEJAD N, RODRIGUEZ G R, KIM H J, CHAKRABARTI M, ILLA-BERENGUER E, TAITANO N K, GONZALO M J, DIAZ A, PAN Y P, LEISNERC P, HALTERMAN D, BUELL C R, WENG Y Q, JANSKY S H, VAN ECK H, WILLEMSEN J, MONFORTE A J, MEULIA T, VAN DER KNAAP E. A common genetic mechanism underlies morphological diversity in fruits and other plant organs. Nature Communications, 2018, 9: 4734.
[13] 王晨晖, 栾非时, 高鹏, 刘识, 解志强. 菜瓜×马泡瓜遗传连锁图谱构建及果实相关性状QTL分析. 园艺学报, 2019, 46(12): 2347-2358.
WANG C H, LUAN F S, GAO P, LIU S, XIE Z Q. Construction of melon genetic linkage map and QTL analysis of fruit related traits in snake melon × wild type melon genetic background. Acta Horticulturae Sinica, 2019, 46(12): 2347-2358. (in Chinese)
[14] 胡志程, 周梦迪, 吕建春, 付秋实, 王怀松. 甜瓜遗传图谱与基因定位研究进展. 分子植物育种, 2020, 18(7): 2290-2295.
HU Z C, ZHOU M D, LÜ J C, FU Q S, WANG H S. The research progress of genetic maps and genes mapping on melon. Molecular Plant Breeding, 2020, 18(7): 2290-2295. (in Chinese)
[15] GARCIA-MAS J, BENJAK A, SANSEVERINO W, BOURGEOIS M, MIR G, GONZÁLEZ V M, HÉNAFF E, CÂMARA F, COZZUTO L, LOWY E, ALIOTO T, CAPELLA-GUTIÉRREZ S, BLANCA J, CAÑIZARES J, ZIARSOLO P, GONZALEZ-IBEAS D, RODRÍGUEZ- MORENO L, DROEGE M, DU L, ALVAREZ-TEJADO M,. The genome of melon (L.). Proceedings of the National Academy of Sciences of the United States of America, 2012,109(29): 11872-11877.
[16] 栾非时, 矫士琦, 盛云燕, 朱子成. 甜瓜果实相关性状QTL分析. 东北农业大学学报, 2017, 48(3): 1-9.
LUAN F S, JIAO S Q, SHENG Y Y, ZHU Z C. Mapping of QTL for fruit traits in melon. Journal of Northeast Agricultural University, 2017, 48(3): 1-9. (in Chinese)
[17] 张学军, 杨文莉, 张永兵, 张健, 郭丽霞, 杨永, 李寐华, 伊鸿平. 采用GBS-seq技术构建甜瓜高密度遗传图谱. 新疆农业科学, 2019(10): 1828-1838.
ZHANG X J, YANG W L, ZHANG Y B, ZHANG J, GUO L X, YANG Y, LI M H, YI H P. Construction of high density genetic map and QTL mapping for downy mildew traits inssp.. Xinjiang Agricultural Sciences, 2019(10): 1828-1838. (in Chinese)
[18] 刘相玉, 张裕舒, 刘柳, 刘识, 高鹏, 王迪, 王学征. 基于CAPS标记的甜瓜单果重相关性状QTL分析. 中国农业科学, 2019, 52(9): 1601-1613.
LIU X Y, ZHANG Y S, LIU L, LIU S, GAO P, WANG D, WANG X Z. The QTL analysis of single fruit weight associated traits in melon based on CAPS markers. Scientia Agricultura Sinica, 2019, 52(9): 1601-1613. (in Chinese)
[19] 张肖静, 张凯歌, 朱华玉, 张宇, 胡倩梅, 程思源, 张敏娟, 胡建斌, 杨路明. 甜瓜侧枝相关性状的QTL定位. 园艺学报, 2019, 46(3): 519-528.
ZHANG X J, ZHANG K G, ZHU H Y, ZHANG Y, HU Q M, CHENG S Y, ZHANG M J, HU J B, YANG L M. QTL mapping of lateral branch associated traits in. Acta Horticulturae Sinica, 2019, 46(3): 519-528. (in Chinese)
[20] TZURI G, ZHOU X J, CHAYUT N, YUAN H, PORTNOY V, MEIR A, SA'AR U, BAUMKOLER F, MAZOUREK M, LEWINSOHN E, FEI Z J, SCHAFFER A A, LI L, BURGER J, KATZIR N, TADMOR Y. A ‘golden’ SNP in CmOr governs the fruit flesh color of melon (). The Plant Journal, 2015, 82(2): 267-279.
[21] ZHANG H, LI X M, YU H Y, ZHANG Y B, LI M H, WANG H J, WANG D M, WANG H S, FU Q S, LIU M, JI C M, MA L M, TANG J, LI S, MIAO J S, ZHENG H K, YI H P. A high-quality melon genome assembly provides insights into genetic basis of fruit trait improvement. IScience, 2019, 22:16-27.
[22] DIAZ A, FERGANY M, FORMISANO G, ZIARSOLO P, BLANCA J, FEI Z J, STAUB J E, ZALAPA J E, CUEVAS H E, DACE G, OLIVER M, BOISSOT N, DOGIMONT C, PITRAT M, HOFSTEDE R, VAN KOERT P, HAREL-BEJA R, TZURI G, PORTNOY V, COHEN S, SCHAFFER A, KATZIR N, XU Y, ZHANG H Y, FUKINO N, MATSUMOTO S, GARCIA-MAS J, MONFORTE A J. A consensus linkage map for molecular markers and quantitative trait loci associated with economically important traits in melon (melo L.). BMC Plant Biology, 2011, 11(1): 111.
[23] DÍAZ A, ZAROURI B, FERGANY M, EDUARDO I, ALVAREZ J M, PICÓ B, MONFORTE A J. Mapping and introgression of QTL involved in fruit shape transgressive segregation into ‘piel de sapo’ melon (L.). PLoS One, 2014, 9(8): e104188.
[24] PERPIÑÁ G, ESTERAS C, GIBON Y, MONFORTE A J, PICÓ B. A new genomic library of melon introgression lines in a cantaloupe genetic background for dissecting desirable agronomical traits. BMC Plant Biology, 2016, 16(1): 154.
[25] RAMAMURTHY R K, WATERS B M. Identification of fruit quality and morphology QTLs in melon (L.) using a population derived from flexuosus and cantalupensis botanical groups. Euphytica, 2015, 204: 163-177.
[26] 王贤磊, 高兴旺, 李冠, 王惠林, 耿守东, 康锋, 聂祥祥. 甜瓜遗传图谱的构建及果实与种子QTL分析. 遗传, 2011, 33(12): 1398-1408.
WANG X L, GAO X W, LI G, WANG H L, GENG S D, KANG F, NIE X X. Construction of a melon genetic map with fruit and seed QTLs. Hereditas, 2011, 33(12): 1398-1408. (in Chinese)
[27] 张宁, 张显, 张勇, 马建祥, 杨小振, 王永琦. 甜瓜果实糖含量相关性状QTL分析. 西北植物学报, 2015, 35(2): 252-257.
ZHANG N, ZHANG X A, ZHANG Y, MA J X, YANG X Z, WANG Y Q. QTL analysis of fruit sugar content correlated traits in melon. Acta Botanica Boreali-Occidentalia Sinica, 2015, 35(2): 252-257. (in Chinese)
[28] 张雪娇, 高鹏, 栾非时. 甜瓜果实相关性状QTL分析. 中国蔬菜, 2013(18): 35-41.
ZHANG X J, GAO P, LUAN F S. QTL analysis of fruits related traits in melon (L.). China Vegetables, 2013(18): 35-41. (in Chinese)
[29] 周鹏, 李钱峰, 熊敏, 范晓磊, 赵冬生, 张昌泉, 刘巧泉. DELLA蛋白介导的激素互作调控植物生长发育研究进展. 植物生理学报, 2020, 56(4): 661-671.
ZHOU P, LI Q F, XIONG M, FAN X L, ZHAO D S, ZHANG C Q, LIU Q Q. Advances in DELLA protein-mediated phytohormonal crosstalk in regulation of plant growth and development. Plant Physiology Communications, 2020, 56(4): 661-671. (in Chinese)
[30] PAN Y P, WANG Y H, MCGREGOR C, LIU S, LUAN F S, GAO M L, WENG Y Q. Genetic architecture of fruit size and shape variation in cucurbits: A comparative perspective. Theoretical and Applied Genetics, 2020, 133(1): 1-21.
[31] MONFORTE A J, DÍAZ A, CAÑO-DELGADO A, KNAAP E. The genetic basis of fruit morphology in horticultural crops: Lessons from tomato and melon. Journal of Experimental Botany, 2014, 65(16): 4625-4637.
[32] 范文林, 王贤磊, 李群, 俞志杰, 李冠. 甜瓜果长基因与性别表达基因的遗传分析及定位. 新疆农业科学, 2018(10): 1765-1774.
FAN W L, WANG X L, LI Q, YU Z J, LI G. Genetic analysis and primary localization of fruit length geneand sex expression genein melon (L.). Xinjiang Agricultural Sciences, 2018(10): 1765-1774. (in Chinese)
Specific Length Amplified Fragment (SFLA) Sequencing Mapping Construction and QTL Analysis of Fruit Related Traits in Muskmelon
WANG Ling1, CAI Yi1, WANG GuiChao1, WANG Di2, SHENG YunYan1
1College of Horticulture and Landscape Architecture, Heilongjiang Bayi Agricultural University, Daqing 163319, Heilongjiang;2Daqing Branch of Heilongjiang Academy of Agricultural Sciences, Daqing 163319, Heilongjiang
【】Through the QTL mapping and candidate gene analysis of muskmelon (L.) fruit-related traits, so as to provide a theoretical basis for muskmelon quality breeding, gene mapping and functional verification.】 The F1plant was made cross by 1244 as female parent with thin-skinned melon and M5 with thick-skinned melon as male parent, combined with SFLA sequencing technology to develop molecular tags and construct high-density genetic maps. Based on F2:3phenotype data, Mapqtl analysis method was used to detect QTL location. 【】Totally 380 446 Mreads (83.12 Gb) data were obtained, the average Q30 and GC content was 93.59% and 36.87%, respectively; totally 112 844 SLAF tags and 3 274 879 SNPs were obtained; a genetic map contained 12 linkage groups with a total of 10 596 markers were constructed. The genetic map spanned 1 383.88 cM with the average distance was 0.13 cM between markers, and totally 99.92% developed markers were mapped. The gene controling fruit sutures () of melon was located at the end of chromosome 11, between Marker 1993423 (62.18) and Marker 1998820 (63.05), covering 0.72 Mb of the genome, and containing 33 candidate genes. The gene for pericarp pattern () was located at chromosome 2 between Marker 459584 (90.91) and Marker 459446 (90.91) which covered the genome of 0.08 Mb and contained 5 candidate genes, among them MELO3C026292 (1-deoxy-D-xylulose-5-phosphate reductoisomerase) might be the candidate gene for pericarp pattern. At the same time, QTL locus of pericarp color () was detected which located between Marker 1229174 (7.14) and Marker 1229973 (7.14) on the chromosome 7, with the contribution rate was 9.9%. One QTL locus of fruit shape ()was detected between Marker 1705671 (76.19) and Marker 1705915 (79.23) on chromosome 9 with a contribution rate of 7.6%. Six QTL loci related to single fruit weight (,,,,,) were detected on chromosomes 1, 2, 6, 7, and 10 with the contribution rate were between 3.1% to 17%, and the LOD value were between 3.0 to 5.6.【】The fruit sutures and pericarp pattern genes were located on the chromosomes11 and 2, and then 33 and 5 candidate genes were obtained, respectively; One QTL for pericarp color, one QTL for fruit shape and 6 QTLs for single fruit weight were also detected.
melon; SLAF sequencing; genetic map construction; fruit related traits; QTL analysis

10.3864/j.issn.0578-1752.2021.19.014
2020-11-25;
2021-03-16
国家自然科学基金(31772330)、中央引导地方科技发展专项(ZY19C02)
王岭,E-mail:wlbynd@163.com。通信作者盛云燕,Tel:0459-6819610;E-mail:shengyunyan@byau.edu.cn
(责任编辑 赵伶俐)
