细胞周期同步化方法在细菌细胞周期研究中的应用
2021-10-13傅雄飞黄雄亮
傅雄飞 黄雄亮 夏 霖
1(中国科学院深圳先进技术研究院 合成生物学研究所定量合成生物学研究中心 深圳 518055)
2(中国科学院大学 北京 100049)
1 引 言
细胞周期是细胞生长、繁殖和发育过程中的一个重要的调控过程,也是生命活动中普遍存在的一个复杂过程。细胞周期是由一系列与细胞周期相关的调控蛋白组成的调控网络,这一调控网络能确保细胞周期相关事件正确、有序地进行。细胞的生长、细胞形态的发生、染色体的复制以及细胞的分裂等事件的开始与结束都受到细胞周期的调控,每个事件都在细胞周期调控网络的监测下环环相扣。
对细胞周期的研究并不仅仅为了满足人类对生命本质的好奇。除了提高人们对生命的认知以外,细胞周期研究对于疾病治疗也有着极大的促进作用。细胞周期调控网络对细胞的生长过程起监控作用,使细胞能够正常、稳定地把遗传物质传递给子代细胞。当遗传物质染色体 DNA 出现损伤时,细胞将停止生长直到损伤得以修复。而当细胞周期调控网络发生紊乱时,细胞的生长分裂也会出现问题从而导致疾病的发生[1]。中国儒家创始人孔子曾说:“未知生,焉知死?”,其认为只有真正了解了“生”,才能去解析“死”,而科学家们对细胞周期的研究正是对生命本质的探索。只有当人们知道了细胞生长繁殖的过程,才能对细胞的死亡和疾病的发生有更加直观的认识。
合成生物学是近十年来迅速发展的新兴学科,通过使用分子生物学工具和技术,对细胞进行正向工程化改造,用于研究细胞系统并促进其对生产性目的的应用[2-3]。在对细胞进行人为改造的过程中,复杂的细胞系统使得许多理性设计变得不可预期,阻碍了该领域的快速发展。因此,对细胞周期的研究是该领域发展不可避免的一环。
2 细胞周期的定义
细胞周期是指细胞在连续分裂的过程中,从一次分裂结束到下一次分裂开始的这段时间所经历的全过程,包括一系列特定的、有序的且周期性发生的事件。其中,最为重要的事件是染色体复制和细胞分裂。根据不同的细胞类型,细胞周期的研究可以分为真核细胞和原核细胞两部分。
在真核细胞中,细胞周期可以简单地分为分裂间期和分裂期两个阶段。在分裂间期,细胞持续生长,体积变大,并执行染色体复制,确保细胞在进入分裂期所需的营养充足以及遗传物质的倍增。该期还可进一步地分为染色体复制前的准备阶段 G1期、染色体复制阶段 S 期以及染色体复制结束后的 G2期。在有丝分裂过程中,当细胞进入分裂期后,有丝分裂开始,细胞核、细胞质依次一分为二,从而形成两个子细胞。真核细胞的细胞周期以 cyclin/CDK(cyclin/Cyclin-Dependent Kinase)复合体为调控核心。细胞外部和内部的各种信号通过不同的信号通路传递给cyclin/CDK 复合体,从而激活或抑制该复合体的活性,进而激活或抑制受其调控的蛋白,调控下游相应事件的发生[4-7]。
在原核细胞中,细胞周期主要由 C 期和 D期组成。其中,C 期是指从染色体复制起始到复制完成这段时间,而 D 期则是从染色体复制完成到细胞分裂这一阶段。当原核细胞的生长速率较慢时,上一轮分裂结束至下一轮分裂开始的间隔要大于 C 期与 D 期的时间之和,此时在上一轮分裂结束至染色体复制开始会有一段间隙期,将这一阶段定义为 B 期[8-9]。与真核细胞的细胞周期最大的区别是,在营养较为丰富的培养条件下,部分原核细胞如大肠杆菌、沙门氏菌等能够以较快的速率进行生长分裂,此时 C 期与 D 期的时间之和要远大于细胞分裂的间隔时间,多次染色体复制事件将发生重叠,正在进行的细胞分裂所对应的染色体复制起始发生在上一轮甚至更早的分裂周期。
3 细胞周期研究方法
细胞在培养过程中是相对独立的个体,每一个细胞的生长分裂都由其独自的细胞周期调控网络进行调节。因此,细胞群体由一个个处于细胞周期不同阶段的细胞组成。目前,对细胞周期的研究主要可以分为单细胞水平和群体水平上的研究两大类。
在单细胞水平上对细胞周期进行研究主要依赖于显微技术,可对群体中的每个细胞进行独立的观测研究。然而,许多传统的生化分析方法无法在显微成像的同时对单细胞进行分析,大部分显微成像所用到的分子标记也具有一定的局限性,这使得显微成像技术在细胞周期研究中也受到了一定的限制[10-11]。
在群体水平上研究细胞周期,一方面是通过条件突变株来研究细胞周期相关的关键基因及其在细胞周期中的调控作用;另一方面则是通过数学模型的辅助,对细菌在培养过程中的细胞周期相关参数的群体平均值进行计算,进而在宏观上对细胞周期进行研究[12-15]。
除此之外,还可以通过同步化的方法,使细胞群体中的每一个细胞都处于细胞周期的同一阶段,然后同步地进行生长分裂,如图 1 所示。通过这一独特的策略来研究细胞周期大大地提高了研究目标的样本量,使得许多只能在群体水平上使用的研究方法也可以用于细胞周期研究。
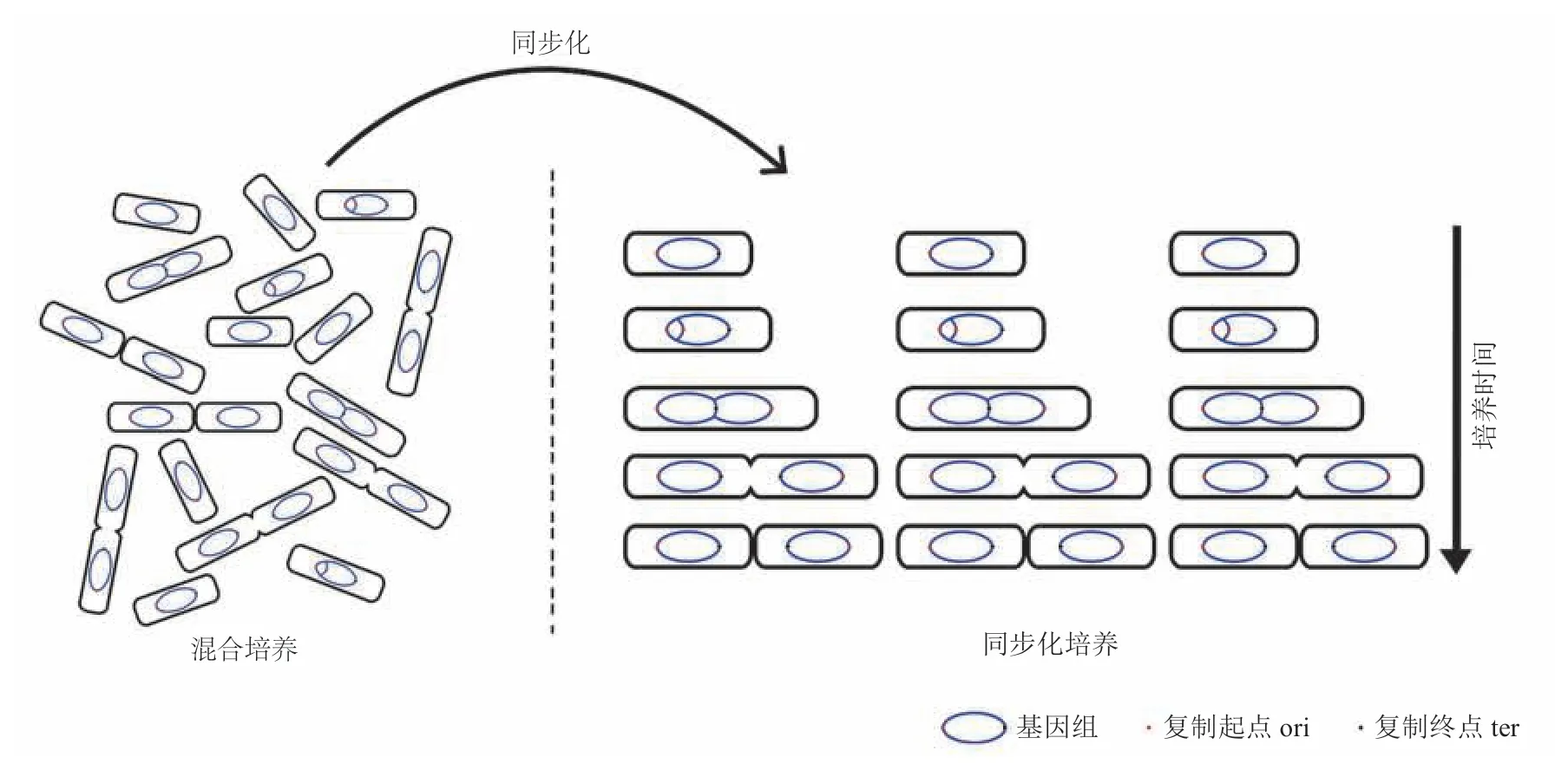
图1 细胞周期同步化Fig.1 Cell cycle synchronization
4 同步化方法的标准
自 19 世纪 50 年代以来,科学家们一直致力于细胞周期同步化方法的研究和探索,尤其是在细胞周期研究领域尝试了各种各样的方法来获得同步化的细胞。然而,并不是所有的方法都适用于细胞周期研究:有的方法获得的同步化效果较差,有的甚至会引入与细胞周期不相关的影响因素,从而导致人们对细胞周期的机理产生错误的理解。因此,使用同步化方法对细胞周期进行研究需要满足以下几个基本标准:(1)同步化方法可以获得大量的同步化细胞;(2)经同步化方法处理后获得的同步化细胞能够继续正常地进行生长分裂;(3)获取的同步化细胞应集中在细胞周期的某个特定阶段,同步化程度需要足够高,能够对细胞周期事件有较高的分辨率,如大部分的同步化细胞能够在差异较小的一个时间段内进行分裂;(4)同步化方法应尽可能少地引入与细胞生理条件不一致的影响因素,对细胞周期产生较少的干扰,使同步化细胞能较好地反映正常细胞周期的特性;(5)利用同步化方法获得的同步化细胞在同步生长分裂的过程中,细胞周期相关的周期性事件至少在 2~3 个分裂周期中能有较好的重复性[16-20]。这些标准对同步化方法具有较为严格的限制,目的是为了降低研究方法对细胞周期研究带来额外的不可预估的影响。针对不同的研究目标和对象,不同的同步化方法具有不同的优缺点,熟悉了解不同方法的优缺点可以合理地避免方法对研究产生的不良影响。
5 同步化方法在细菌细胞周期研究中的发展与应用
由于不同细菌在形态、分裂模式等生理特性上的差异,同步化方法的应用在一定程度上存在菌种限制性。
5.1 新月柄杆菌
新月柄杆菌(Caulobacter crescentus)的细胞分裂是不对称分裂,形成一个一端带鞭毛的可游动的细胞和一个一端带柄状茎杆的茎细胞。其中,只有茎细胞可以进行 DNA 复制。与其他细菌不同的是,新月柄杆菌的一轮细胞周期中只会起始一次 DNA 复制,这可能更有助于其适应在营养相对匮乏的环境下生存。对新月柄杆菌细胞周期的研究可以帮助人们更好地理解细胞分化和发育,同时,因为新月柄杆菌在细胞周期伴随着独特的形态变化,这使得人们可以更容易地分离处于不同细胞周期阶段的新月柄杆菌,从而获得同步化的新月柄杆菌。因此,新月柄杆菌已成为细胞周期研究的一个重要的模式工具菌株。
1962 年,根据新月柄杆菌分裂所产生的两种不同形态子细胞在浮力密度上的差异,Stove 和Stanier[21]使用差速离心的方法分离出新月柄杆菌分裂后产生的两种不同形态的细胞:游动细胞和叶状柄细胞,将二者分别接种至新鲜培养基中可以观测到两类细胞的同步化生长和分裂以及在生长分裂过程中的形态变化。1977 年,参考真核细胞中的血细胞和裂殖酵母的同步化方法,Evinger和 Agabian[22]利用密度梯度离心的方法对新月柄杆菌进行同步化。与差速离心的方法相比,密度梯度离心的方法分离获得的同步化细胞的同步化程度更高。1993 年,Stephens 和 Shapiro[23]使用这一同步化方法对新月柄杆菌鞭毛形成的相关基因在细胞周期不同时期的转录调控进行检测。1997 年,Mohl 和 Gober[24]在同步化培养的新月柄杆菌中研究了 ParA 和 ParB 在细胞周期不同时期的转录表达模式以及细胞定位,发现 ParA 和ParB 在新复制 DNA 迁移至准备分裂的细胞两端这一事件中起重要作用。Domian 等[25]还利用同步化方法研究了新月柄杆菌的细胞周期核心调控蛋白 CtrA 在细胞周期不同阶段的磷酸化水平和空间分布,进一步阐释了 CtrA 在新月柄杆菌细胞周期中的调控模式。随着技术的发展,Shapiro实验室于 2000 年使用微阵列芯片对新月柄杆菌在细胞周期不同时期的转录组进行了全局分析,发现新月柄杆菌中有 553 个基因(占所有基因的19%)的转录水平在细胞周期中并不是恒定不变的,而是随着细胞周期的进行发生周期性变化,分别对应细胞周期中的不同事件,在特定的时期发挥其功能。通过与 CtrA 温度敏感型菌株的比较,他们还确定了其中 26% 的细胞周期依赖的基因直接或间接地受到 CtrA 蛋白的调控[26]。随后,更多的文章报道了通过新月柄杆菌同步化方法结合组学分析从全局的角度对其细胞周期进行分析[27-30]。
另外,结合微流控芯片技术,Madren 等[31]开发出了一个可以在长达 4 天的时间内持续产生新分裂游动细胞的微流控芯片。该方法利用叶状柄细胞易于黏附在材料表面的特性,使之黏附在微流控芯片中,而新分裂的游动细胞则会被流动的新鲜培养基带出芯片,基本原理与后面将提到的大肠杆菌膜洗脱同步化装置原理类似。相比于传统的离心分离法,该方法的好处在于需要的培养基较少,避免了复杂繁琐的离心过程,减少了对同步化细胞的干扰,并且该同步化芯片可以在4 天内连续不断地产生新分裂的同步化细胞,更有利于时间较长的实验持续进行。传统的离心分离法根据细胞的形态和浮力密度来进行同步化,无法将新分裂的游动细胞与快要分化成叶状柄细胞的流动细胞分离,而该方法洗脱获得的都是新分裂的子细胞,因此只要将收集时间控制在较短的区间,就可获得同步化程度更高的子细胞。然而,由于微流控芯片技术更适用于实时观察分析,所以微流控芯片同步法在同步化细胞的产量上要远小于传统的离心分离法。
5.2 大肠杆菌
大肠杆菌(Escherichia coli)是目前科学研究和工业生产中都普遍使用的模式细菌,人们对于大肠杆菌的研究可以追溯至一百多年前。科学家们对大肠杆菌的同步化方法也做了许多的尝试,然而,与新月柄杆菌不同的是,大肠杆菌的分裂是对称分裂,母细胞在分裂后形成两个相同大小、相同形态的子细胞,而且也不会形成像新月柄杆菌的叶状柄类似的结构,因此,大肠杆菌的同步化要相对更难一些。
1956 年,Barner 和 Cohen[32]通过去除培养基中的胸腺嘧啶(T)使胸腺嘧啶营养缺陷型大肠杆菌停止在 DNA 合成阶段,并在合适的时间后补充胸腺嘧啶使这些被捕获在 DNA 合成阶段的大肠杆菌同步地开始 DNA 合成以及生长分裂。这种通过改变细胞的生长条件(如营养、温度等)来使处于细胞周期不同阶段的细胞同步在同一阶段,获得的同步化生长的细胞群体在生长分裂过程中的生理特性往往会受到生长条件变化所带来的影响,因而并不能很好地反映正常细胞周期中的细胞生理性质[16-18]。为了解决这一问题,Maruyama 和 Yanagita[17]通过物理的方法,在不改变细胞生理条件的情况下获得同步化的细胞。他们利用堆叠的滤纸过滤处于对数生长期的 B 系大肠杆菌,分离获得了较大与较小的两类细胞,分别接种到新鲜培养基中时,均可获得阶梯状增长的生长曲线。从生长曲线可以看出,较大的一类细胞是更接近细胞分裂的成熟细胞,较小的一类则是处于细胞周期中相对不成熟的状态。相比于之前的同步化方法,通过堆叠滤纸过滤的方法可以获得同步化效果较好的 B 系大肠杆菌。1963年,Helmstetter 和 Uretz[33]使用滤纸堆叠过滤的方法获得同步化生长的大肠杆菌,并发现该菌对于 X 射线和紫外射线的敏感性并不是恒定不变的,而是随着细胞周期的变化呈周期性变化。
虽然不少研究表明通过滤纸堆叠方法获得的同步化大肠杆菌的同步化效果较好,但 Helmstetter等[19]认为仅仅是根据大小筛选的原理并不能获得如此高的同步化效果。随后,Helmstetter 通过减少滤纸堆叠厚度发现,减少堆叠的滤纸可以获得更好的同步化效果,从而搭建了基于膜洗脱原理的子细胞洗脱同步化装置(Membrane-Elution Baby Machine)。该方法的实现要求细菌具有黏附在膜表面的能力,并可以在膜表面进行正常的生长分裂,而新分裂的新生子细胞则会被流动的培养基洗脱,通过收集较短时间内分裂的子细胞可以获得同步化程度较高的细胞群体。由于该方法没有改变细菌生长过程中的生理条件,也不需要过于繁琐的样品处理步骤,黏附在膜上的细菌细胞经过一定时间的培养可以达到一个稳定的生长状态,因此通过这一方法获得的同步化细胞受到的干扰较小,可以较好地反映细胞在稳态生长条件下的生理特性[34-36]。使用多聚赖氨酸对硝酸纤维素滤纸进行预处理可以将这一方法应用到黏附性较差的 K 系大肠杆菌上,但富含氨基酸的培养基中带电氨基酸的存在会极大地影响多聚赖氨酸介导的黏附作用,大大降低了同步化细胞的产量[37]。
2005 年,Bates 等[38]通过改造大肠杆菌鞭毛,使得大肠杆菌可在诱导剂的作用下黏附在玻璃微球表面,随后将黏附了大肠杆菌的玻璃微球装载至色谱柱中,可使用新鲜培养基进行洗脱。新分裂的细胞在没有诱导剂的情况下将形成正常鞭毛,因此无法黏附在玻璃微球表面,将被流动的培养基带出,通过收集洗脱下来的新生子细胞即可获得同步化大肠杆菌。这一方法适用于任意培养基以及大多数含有鞭毛的细菌,其虽然进一步扩大了膜洗脱同步化方法的适用范围,但同步化的效果相对较低,同步化程度约为 79%。2019年,结合微流控芯片技术,Chang 等[39]在大肠杆菌 MG1655 一端表面展示绿色荧光蛋白 GFP,通过生物素化的 anti-GFP 抗体与链霉亲和素包被的磁珠结合,得到人造磁细菌。使用磁场将人造磁细菌吸附在微流控芯片的通道顶部,新分裂的子细胞则可以被流动的新鲜培养基洗脱,从而获得同步化细胞。这一方法充分利用了合成生物学的方法,结合微流控芯片技术,将基于膜洗脱原理的同步化方法应用于黏附力较弱的 K 系大肠杆菌 MG1655,进一步扩大了膜洗脱同步化方法的应用。
基于膜洗脱原理的同步化方法为大肠杆菌细胞周期研究提供了一个更为直接的方法。1968年,Cooper 和 Helmstetter[40]使用膜洗脱获取新分裂子细胞的同步化方法研究大肠杆菌在细胞周期不同阶段的 DNA 复制情况,直接测定了大肠杆菌在不同生长速率下的 C 期和 D 期,验证了大肠杆菌快速生长状态下的多复制叉同时进行DNA 合成以及缓慢生长状态下存在 DNA 复制停止的间隙期两种 DNA 复制模式,并提出了稳态生长的细菌群体中每个细胞复制起点的群体平均数与生长速率的定量关系。2005 年,Bates等[41]使用显微成像技术对同步化大肠杆菌进行实时监测,观察染色体和 DNA 复制复合体在细胞周期过程中的动态变化。
5.3 其他细菌
除了新月柄杆菌和大肠杆菌以外,科学家们同样尝试过许多其他细菌的同步化。例如,枯草芽孢杆菌可以通过芽孢的同步萌芽来获得同步化细胞[42]。De Nisco 等[43]通过转换培养基诱导细菌严谨反应调控的方法获得苜蓿中华根瘤菌的同步化细胞,并对同步化培养的根瘤菌进行了转录组分析。海洋中的蓝细菌细胞周期与昼夜循环相关联,因此通过周期性光照即可使蓝细菌细胞周期同步化。Waldbauer 等[44]对同步化的蓝细菌进行转录组和蛋白质组分析,探索蓝细菌在白天光合作用固碳到晚上呼吸作用这一过程中所涉及的基因转录表达调控。使用dnaA温度敏感型突变株可以获得结核分支杆菌的同步化细胞,Nair 等[45]和Bandekar 等[46]通过该方法分别分析了该菌细胞周期相关的转录表达模式,预测其细胞周期相关基因的功能。
6 总结与展望
总体来说,细菌细胞周期同步化法可以分为条件转变法、物理分离法和基于膜洗脱原理的子细胞装置(Baby Machine)三大类[16,36]。条件转变法包括周期性转变温度、转变培养基营养、周期性光照等,其中根据条件转变次数又可以分为周期性条件转变和预转变。周期性转变是指周期性变换细菌生长的生理条件,使细菌细胞周期同步化。预转变则是指通过化学试剂、营养下调等条件改变将细菌细胞可逆地阻断在细胞周期的某个时期,去除化学阻断剂或重新上调营养后,将不再改变细菌生长的生理条件,使细菌细胞在适宜的条件下同步地生长分裂。物理分离法则是利用细胞周期不同阶段的细胞具有不同大小、密度或表面抗原等特性,通过离心、流式细胞技术、微流控芯片等物理方法分选出处于特定时期的细胞。例如,新月柄杆菌的密度梯度离心法就是根据新分裂的两种子细菌的浮力密度差异对其进行分离。相比于条件转变法,物理分离法是直接对混合培养的细菌进行分类分离,因为不改变细菌生长过程中的生理条件,不会对细菌细胞周期造成干扰,由此获得的同步化细胞在同步化培养时的细胞周期特性更接近于正常稳态培养时的状态。基于膜洗脱原理的子细胞装置因为“母细胞”固定在装置上,在流动的新鲜培养基中持续地生长分裂,经一定的时间后可以达到稳态生长,而且省去了物理分离法中繁琐的样品处理步骤,收集短时间内新分裂的子细胞可以认为是受到干扰最小的。
尽管条件转变法对细菌的生理状态干扰最大,但当研究目标是与改变的条件相关联的事件时,使用该法进行同步化研究也可获得有用的参考信息[47-52]。通过子细胞装置获得的同步化细胞受到的干扰最小,但其要求细菌能够很好地黏附在材料表面,这极大地限制了该方法的适用性,同时其同步化细胞产量也不如物理分离法[35,38,53]。三种方法都有各自的优缺点,不同的细菌适用不同的方法,在选择使用方法时应根据实验目标、细菌种类等实际情况仔细斟酌。
随着显微成像技术的发展,在单细胞水平上对细胞周期进行研究成为该类研究的一个重要方法[54-56]。但细胞周期同步化方法受到细菌类型、同步化产量、同步化质量等因素的限制,使得同步化方法的研究及应用难以普及。然而,细胞周期同步化方法不仅可以为研究提供大量的组学信息,也使许多生化与分子实验技术,如 Western Blot、荧光实时定量 PCR 等技术得以应用到研究当中,同时细胞群体的同步化也能提高单细胞显微成像分析过程中处于同一细胞周期阶段的细胞数,使数据分析更具有统计意义[41,57-58]。大肠杆菌的细胞周期调控网络仍然有许多不清晰的地方,DNA 复制与细胞分裂通过什么机制来调控偶联在一起是目前人们比较关心的一个问题。基于膜洗脱技术的大肠杆菌细胞周期同步化装置——子细胞装置,可以为研究人员提供干扰极小的同步化细胞,对于稳态生长条件下的大肠杆菌细胞周期研究具有重要的意义。尽管这一方法受到菌株黏附能力的限制而不能很好地应用到不同的菌株当中,而随着技术的发展,人们通过对细菌进行定向地改造,将逐渐克服这些限制。目前,合成生物学的快速发展也为克服细菌细胞周期同步化方法的限制带来了新的技术和思路[38-39],只要攻克了其中的短板问题,同步化方法必然会给细菌细胞周期研究带来极大的推动作用。
