基于TCGA数据库分析肝细胞癌组织UCK2、PRIM1和DNTM1基因水平对患者预后的影响*
2021-09-22卢秉久
赵 艺,卢秉久
肝细胞癌(hepatocellular carcinoma,HCC)的发生是一个多因素作用的过程,早期临床缺乏特殊症状,肿瘤具有侵袭性,可能发生转移,患者就诊时多已发展到晚期[1,2]。现阶段,治疗HCC的方法有多种,其中应用较多的包括肝移植、肿瘤切除、栓塞化疗、消融等,但这些治疗方法的疗效不一,患者预后差。因此,从遗传学角度研究HCC发病的分子机制,寻找新的肿瘤标志物,有助于早期诊断、高精准度的靶向治疗和预防性治疗[3-5]。研究肝癌的发病原因及分子机制一直是研究者们努力的方向。研究揭示了肝癌发病的分子机制及其与病理学特征之间的紧密联系,他们发现有PRAF2和GRB2等多种类型的基因突变导致了HCC的发生[6,7],同时也有学者基于基因组学研究,利用癌症基因图谱(The Cancer Genome Atlas,TCGA)和基因水平综合数据库(Gene Expression Omnibus, GEO)等数据库对HCC的关键基因组变化进行了研究[8-10],通过对TCGA数据库的挖掘,可以发现多种HCC致癌基因和抑癌基因,这些基因与HCC的发生发展及预后关系密切,利用这些数据库可开展基因组学相关研究。本文通过对TCGA数据库中HCC癌组织和癌旁组织进行基因富集分析(gene set enrichment analysis, GSEA)及对京都基因和基因组百科全书(Kyoto Encyclopedia of Genes and Genomes, KEGG)的通路注释和富集分析,筛选出HCC组织差异水平密切相关的DNTM1、PRIM1和UCK2基因,以探讨这三种基因对HCC的早期诊断和临床治疗提供支持的价值。
1 资料与方法
1.1 数据采集 打开TCGA (https://cancergenome.nih.gov/)数据库,自《癌症基因组图集-肝细胞癌数据集》(TCGA-LIHC)收集基因数据和临床数据。 从TCGA数据库中提取HCC癌组织和癌旁组织DNTM1、PRIM1和UCK2 水平的有关资料,得到癌和癌旁组织的测序数据,由MD Anderson Cancer Center对所有数据进行标准化预处理。同时,下载患者的临床资料,其中临床数据为Level 3 等级。在TCGA 数据库中设定的检索条件如下:①Cases选项,Primary Site: Liver;其余选项以系统默认的设置;②Files 选项,Data Category: Transcriptome Profiling,Biospecimen,Clinical;Data Type: Gene Expression Quantification,Biospecimen Supplement,Clinical Supplement; Experimental Strategy: RNA - Seq;Work flow type: HTSeq - Counts,其余选项以系统默认的设置。筛选得到424例有效数据文件,其中HCC组织数据374 例,癌旁组织数据50例。随后,通过R软件( https: / /www.r-project.org /) 对所下载的数据进行整理,筛选数据库,进行临床病理学参数分类,寻找相关的差异基因,并绘制火山图和热图。
1.2 基因的筛选和生存分析绘制 将差异基因通过除异均值化M值法( trimmed mean of M values,TMM) 对基因水平进行标准化处理,应用edgeR包筛选差异基因,采用错误发现率 ( false discovery rate,FDR) 对基因的显著性进行校正。差异水平基因的截断点(cut-off value)设定为: fdr=0.05 ,lgFC=1。应用单因素COX分析发现差异基因对肝癌患者预后的影响,并绘制生存曲线。以基因水平的中位数为分界,定义HCC组织DNTM1、PRIM1和UCK2基因水平的高低,据此将样本分为低水平组和高水平组,同时绘制Kaplan-Meier曲线。
1.3 统计学方法 应用R3.5.2软件进行统计学分析和相应图形的绘制,应用Survival包进行单因素和多因素Cox比例回归模型的筛选,并建立多基因预后模型。应用 Survival ROC包绘制ROC曲线,并计算曲线下面积(AUC),判断Cox回归模型预测HCC患者5 a生存率的准确性。
1.4 差异基因的富集分析 根据基因水平将患者分成低水平组和高水平组,对两组样品行GSEA富集分析。我们选择基因集“c2.cp.kegg.v6.2.symbols.gmt”行KEGG富集分析,得到KEGG富集分析结果。
2 结果
2.1 差异基因的筛选 在TCGA-LIHC数据库,收集374例HCC组织和50例癌旁组织所对应的临床和病理学参数。同时对检索到的153个HCC相关的差异基因 (其中51个水平下调,102个水平上调)进行了分析,应用R软件的edgeR软件包研究上述差异基因。对满足以下条件:即筛选fdr=0.05和lgFC=1者,根据筛选结果绘制火山图(图1),图中红色数据点为满足筛选条件得到的HCC相关的差异基因,并对这些基因进行生存分析。
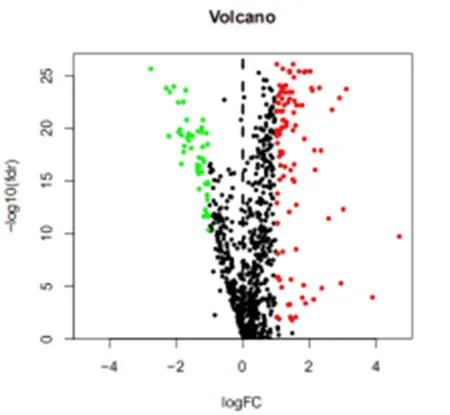
图1 差异水平基因火山图横坐标是-lg(P value),纵坐标是lgFC;绿色代表下调基因,红色代表上调基因
2.2 生存分析 应用R软件的 Survival软件包对生存行单因素COX分析,对筛选得到的差异基因进行生存分析。根据生存率指标风险比值(hazard ratio, Hr),绘制得出森林图(forest plot,图2)。除了CYP2C9基因人群生存率降低外,其他基因人群生存率不受影响(Hr>1)。对单因素分析得到的影响预后的相关基因进行多因素逐步回归,以风险值=∑(基因系数×基因水平)构建风险模型,将基因水平的中位数值设为阈值,根据此阈值将患者划分为高风险组和低风险组,根据高低风险值绘制Kaplan-Meier曲线,结果高风险组HCC患者总体生存率显著低于低风险组患者(图3)。

图2 生存率相关的基因森林图P<0.001为显著性差异

图3 HCC患者K-M生存曲线横坐标是生存时间,纵坐标是生存率。根据基因水平的中位值,将患者分为高低两组。红色代表基因高水平组,蓝色代表低水平组P<0.001为显著性差异
2.3 诊断性ROC曲线情况 根据生存分析的结果,绘制ROC曲线,基于多因素COX回归模型预测的HCC患者预后风险因素,其AUC = 0.649,大于其他临床因素的AUC值,进一步确认了多因素 Cox回归模型预测HCC患者预后风险的可信性,可以为肝癌患者进行个性化治疗提供参考,从而降低患者病死率,改善预后(图4)。
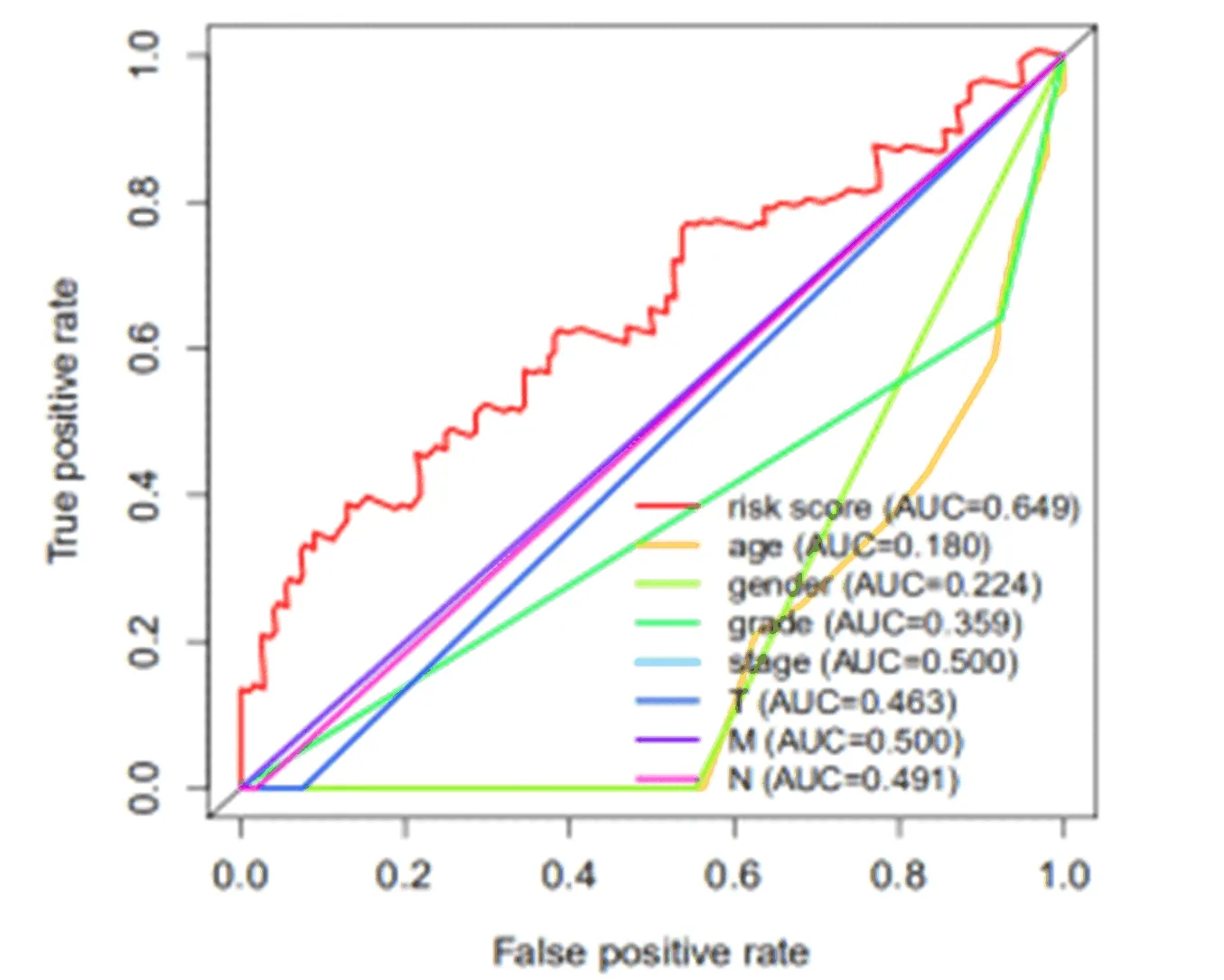
图4 多因素Cox模型分析的针对性ROC曲线
2.4 对差异基因进行GSEA富集分析的结果 通过前述研究筛选出的显著水平性基因DNTM1、PRIM1和UCK2,进行GSEA富集分析。GSEA显示了许多显著丰富的信号通路,进一步证明了上述基因与HCC发生及与患者预后的显著性关系,从而揭示了HCC组织DNTM1、PRIM1和UCK2基因水平对生存的影响(图5)。

图5 GSEA富集分析结果图形的上半部分是ES值富集的过程,图形的下半部分是基因在每个功能的分布情况。在图形中,不同的颜色代表不同的功能
3 讨论
癌症基因图谱(TCGA)数据库容纳了比较全面的基因测序数据。在美国政府的支持下,国家癌症研究所和人类基因组研究所联合创建了该数据库。在数据库中除了包含大量肿瘤基因数据外,还有众多多维度的基因组变化的图谱。库中数据涉及到一万多名患者的病变组织和正常组织信息,其中病变组织的类型有三十多种,包括 10种罕见肿瘤类型。该数据库收集的数据很丰富,包括miRNA 序列、mRNA 序列、基因水平和DNA 甲基化相关数据等[11,12]。
中国的肝癌发病率居于全球的前列。近年来,高通量测序技术的发展在揭示癌症发生发展过程中基因的异常水平和识别与癌症诊断和预后相关的生物标记物方面起到了至关重要的作用[13,14]。本研究通过筛选 GSE10186 基因芯片得到差异基因153个,其中上调基因102个,下调基因51个。其中促癌基因 UCK2、DNTM1和PRIM1在GSE10186 芯片癌组织中呈现高水平,表明UCK2、PRIM1和DNTM1基因具有促进癌变的作用。抑癌基因CYP2C9在GSE10186 芯片癌组织中呈现低水平,表明CYP2C9基因对肝癌有抑制作用。在对 89个差异水平基因进行单因素Cox回归分析筛选出与肝癌患者预后显著相关的基因,在对374例肝癌患者进行风险比值的评分后,将患者划分为高风险组和低风险组并进行 Kaplan-Meier 生存分析,结果表明高风险组肝癌患者总体生存率显著低于低风险组患者。ROC曲线确认了基于多因素COX回归模型预测肝癌患者预后风险的可信性,可以为肝癌患者进行个性化治疗提供参考,从而降低患者的病死率,改善预后。
迄今为止,已鉴定出三种人尿苷胞苷激酶基因,包括UCK1、UCKL1和UCK2,其中UCK1和UCK2共有约70%的序列同一性[15-16]。UCK1在多种正常人体组织中均有存在,如骨骼肌、心脏、肝脏和肾脏,而UCK2仅在正常人胎盘和睾丸中检测到,但其在胰腺肿瘤组织、结直肠癌组织、神经母细胞瘤和乳腺癌组织上调[17,18]。因此,UCK2被认为是癌症预后的生物标志物。肝癌组织UCKL1和UCK2水平比邻近肝组织高,推断UCK2上调可能是肝癌的一个有价值的预后指标[19]。
DNMT1是哺乳动物基因组表观遗传修饰中DNA甲基化的关键基因,其编码的蛋白是一种分子量大且功能复杂的酶,具有多种调控功能,参与机体发育过程中干细胞生长、细胞增殖、器官发育、衰老和肿瘤发生等多个生物学过程[20]。学者在实验中发现DNMT1蛋白的高水平能有效预测早期胃肠道癌和严重癌前病变,具有潜在的临床应用价值。DNA在真核细胞中的复制是由一个复杂的染色体复制装置完成的,其中DNA聚合酶α和DNA引物酶是两个关键的酶活性成分。DNA引物酶含有PRIM1和PRIM2,前者携带有酶和引物,因此具有催化和延伸功能;后者则缺乏酶的活性。DNA在合成过程中,PRIM1 mRNA水平与DNA的复制进程存在着密切的相关性。在没有PRIM1酶的情况下,DNA的复制也难以进行,故而此种物质对细胞增殖具有促进作用。目前,该领域的研究在不断增多,一些报道中已经出现PRIM1基因在不同肿瘤细胞中作用的相关内容。
根据TCGA数据分析结果显示,在肝癌形成过程中,我们发现了与其密切相关的信号通路,确定了UCK2、PRIM1和DNTM1这3个基因水平与肝癌的相关性,可作为预测肝癌患者预后的指标,为肝癌的理论研究和治疗提供一定的参考。然而,本文也存在一定的局限性,研究仅基于生物信息学分析,以后应该同时开展一些临床和实验研究,从而证实这些指标的应用价值,为肝癌的诊治提供支持。
