基于高通量测序技术的北冰洋典型环境原生生物多样性研究进展
2021-09-16陈一璇孔菏君徐大鹏
陈一璇, 孔菏君, 徐大鹏
基于高通量测序技术的北冰洋典型环境原生生物多样性研究进展
陈一璇1, 2, 孔菏君1, 2, 徐大鹏1, 2
(1. 厦门大学近海海洋环境科学国家重点实验室, 厦门大学海洋与地球学院, 福建 厦门 361102; 2. 厦门大学福建省海洋碳汇重点实验室, 福建 厦门 361102)
原生生物指可独立生存的单细胞或群体真核生物, 其多样性高、结构功能复杂、分布广泛, 是海洋微型浮游生物群落的重要组成部分, 在维持海洋生态系统结构与功能中发挥着重要作用。早期使用显微镜、流式细胞术等传统方法对原生生物多样性的研究具有若干局限性, 随着分子生物学技术的发展, 测序技术(尤其高通量测序)为深入研究原生生物多样性、群落组成、时空变化及甄别其环境驱动因素等做出重要贡献。北冰洋是受全球气候变暖影响最为严重的大洋, 作为水体生态系统的重要组成者, 原生生物所受影响仍待深入探讨。因此, 探究北冰洋典型环境(如海水、海冰、融池等)中原生生物的群落组成、动态变化等, 有助于了解全球变暖过程中北冰洋生态系统的变化。然而, 由于极地自然环境恶劣、样品采集困难等原因, 北冰洋原生生物多样性的相关研究十分欠缺, 许多研究空白亟待补充。本文对基于高通量测序技术的北冰洋原生生物多样性研究进行了文献搜集与整理, 以期为全球变化背景下原生生物多样性研究提供参考。
北极; 原生生物; 群落组成; 海冰; 微型真核生物; 全球变暖; 融池
原生生物经典定义为植物、动物、真菌以外的所有真核生物[1]。由于单细胞与多细胞类群在生理生态等方面的巨大差异, 目前被大多数学者所接受的定义为:“能够独立生存的单细胞或群体真核生物”[2]。海洋浮游原生生物个体微小、丰度高、在浮游生物总生物量中有巨大贡献。其营养模式主要包括光合自养型、异养型、混合营养型、寄生型4种, 粒径跨越至少3个数量级, 在生态系统中可占据不同生态位, 构成微食物环, 与海洋生态系统中的物质循环和能量流动密切相关[3-5]。
海冰常年漂浮于北冰洋, 秋季到晚春期间甚至能基本覆盖整片海域[6]。近年来, 由于全球气温上升, 北冰洋海冰覆盖度大幅缩减[7], 最小覆盖面积从7.0× 106~7.5×106km2下降至3.5×106~4.5×106km2 [8]。2012年末, 海冰总面积仅3.6×106km2 [6]。同时, 北冰洋海冰正经历从多年冰向当年冰转变: 卫星数据显示, 从1980年到2011年, 多年冰比例已从75%降至45%, 存在时间较久的“老冰”从50%降至10%[9]; 据推测, 到2050年, 北冰洋中心海域的多年冰将完全消失[10]。温度的升高还导致冰上融池面积增大、数量增多[11]。Lüthje等[12]发现融池在浮冰上的覆盖率已超过80%, 很多融池正逐渐转变为冰洞。此外, 夏季融冰期延长、冰盖变薄、卤道结构改变等环境变化可加速海冰融化[13],将进一步加剧北冰洋所受全球变暖的冲击。
变化的北冰洋可能导致其中的原生生物在多样性、群落结构、分布模式等方面发生改变, 从而影响该海域食物网结构及元素循环。因此, 开展全球变暖背景下北冰洋原生生物多样性研究、了解群落结构及其变动规律具有重要现实意义。
1 原生生物分类系统与生态功能
原生生物主要类群包括: 不等鞭毛类, 主要由硅藻、金藻等单细胞藻类和双并鞭虫组成; 囊泡虫, 主要包括纤毛虫、甲藻和顶复虫; 根足虫, 包括丝足虫、放射虫; 泛植物超群, 包括灰胞藻、红藻及绿藻等; 后鞭毛超群, 主要包括领鞭毛虫; 待定类, 包括隐藻和定鞭藻等[14](表1)。上述类群均为北冰洋常见类群。
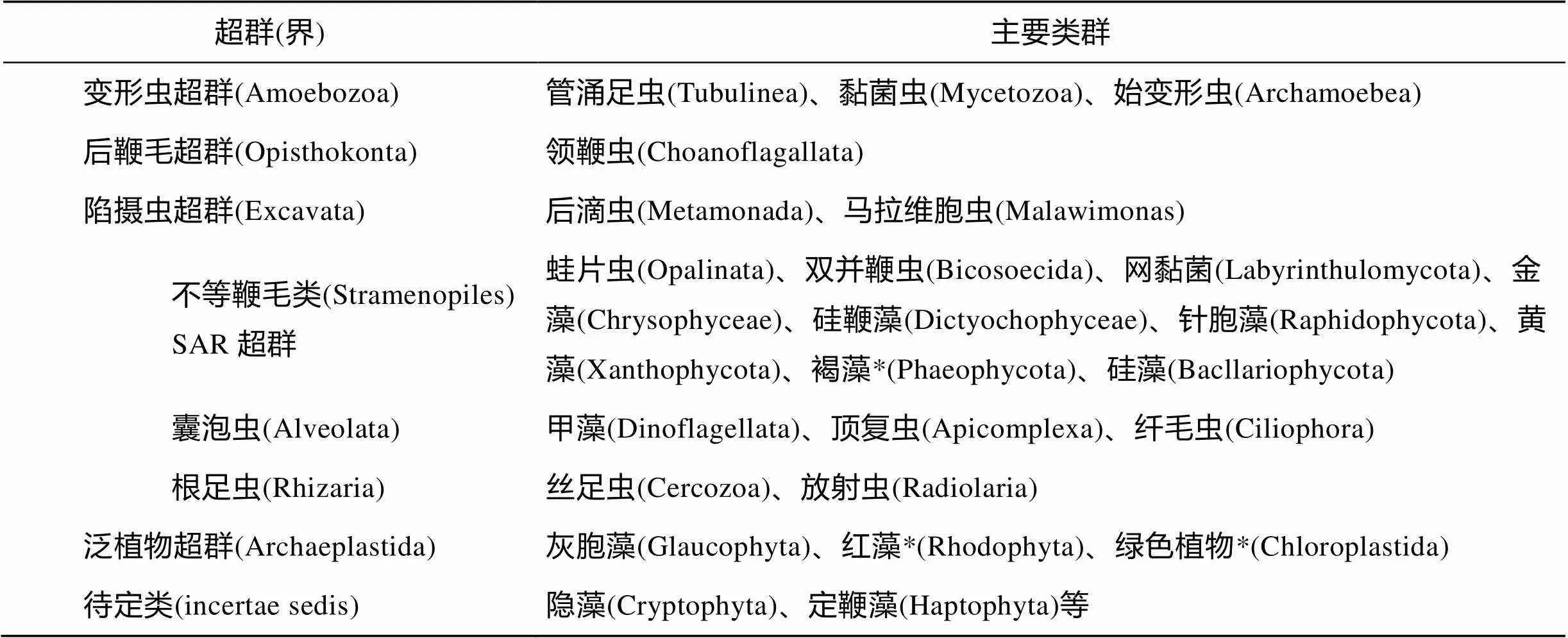
表1 原生生物主要类群[14]
注: *表示同时包含多细胞类群
原生生物类群间及与其他微型生物间构成了复杂的营养关系: 自养类群通过光合作用所产生的有机物经异养/混合营养类群摄食传递至更高营养级(图1中红色箭头示); 寄生类群寄生于其他微型/中型浮游生物体内, 其大量繁殖可“裂解”宿主, 将宿主体内物质释放到水体中(图1中蓝色箭头示); 细菌等原核生物可通过矿化作用为自养原生生物初级生产力的形成和积累提供二氧化碳和无机营养盐, 同时作为异养/混合营养型原生生物的食物(图1中黑色箭头示), 参与微食物环元素循[2, 5, 15]。
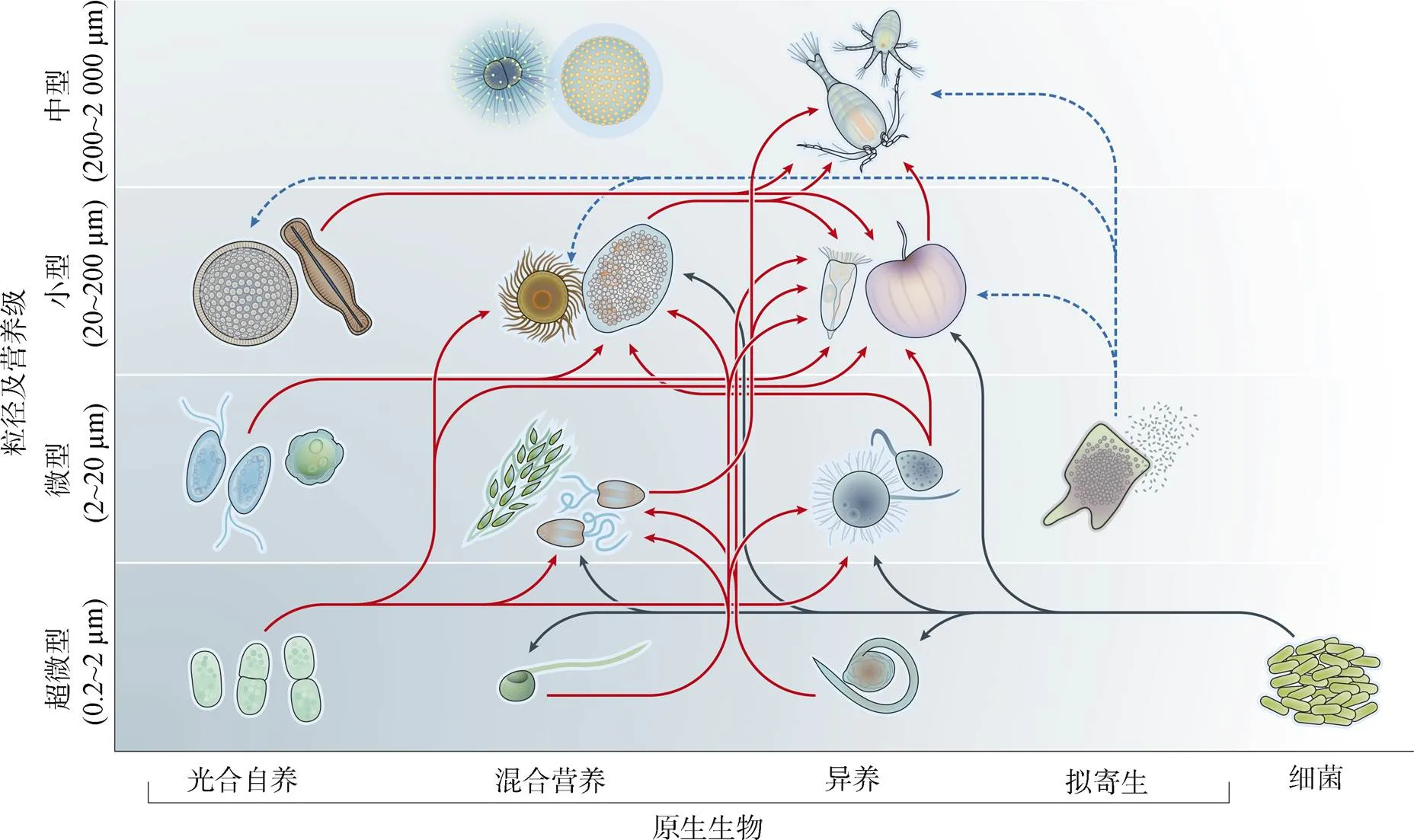
图 1 原生生物在海洋浮游生态系统中的生态及生物地球化学作用[5]
2 原生生物多样性研究方法
显微镜观察是原生生物多样性研究中广泛使用的手段, 能定性、定量地了解样品组成。由于一些原生生物类群个体微小(如最小的类群仅0.9 μm)、形态学特征不明显(或不易观察)等原因, 显微镜难以对其进行准确的种类鉴别[16]。电子显微镜分辨率更高, 可对细胞形态结构进行精准观察, 然而样品需进行特殊处理, 操作繁复、耗时较长且不能在定性的同时很好地完成定量工作。流式细胞术能针对特定原生生物类群进行快速定量分析, 但难以完成对低阶元、低丰度、粒径较大类群的研究。因此, 上述经典研究手段均存在一定局限性。
聚合酶链式反应技术的广泛应用, 使得基于测序技术的分子生物学方法逐渐成为(包括原生生物在内的)微型生物多样性研究的重要手段[17]。第二代测序(又称高通量测序, high-throughput sequencing, HTS)相较于经典研究技术及第一代测序(如Sanger测序法)具有明显优势: 高通量, 单次反应可产生高达几百G数据; 高覆盖度, 可获得样品中低丰度类群; 所得单位数据成本更低; 无需专业的分类学技术与知识, 可对原生生物整体进行研究[18-20]等。同时, 该技术也存在若干不足, 如: 由于真核生物小亚基核糖体基因多拷贝数现象的存在[21-22], 该方法无法获得原生生物类群细胞丰度; 由于引物的偏好性, 该方法无法客观反映类群间的相对丰度[23]; 由于DNA在体外仍可保存较长时间, 环境中休眠、死亡个体甚至生物碎片(碎屑)会对结果造成一定影响[24-25]。即便如此, 该技术的应用仍对原生生物多样性研究产生了革命性影响[26-28]。
3 基于高通量测序技术的北冰洋原生生物多样性
3.1 海水
北冰洋气候低、海冰常年覆盖、太阳辐射不足。冰雪融水、陆地径流的汇入使得北冰洋表层水具有低温低盐的特性。高温高盐的大西洋水经海峡汇入北冰洋后冷却、下沉形成中层水。两层水间形成盐跃层[29]。真光层中营养盐有限, 次表层叶绿素最大水层(subsurface chlorophyll maximum layer, SCM)中的营养盐及光照条件达到最优, 生产力较高[30], 原生生物大都分布于上层混合水体中[31]。
北冰洋中心海域(图2a), 囊泡虫、不等鞭毛类、根足虫、Telonemia、绿藻、隐藻、定鞭藻、丝足虫等组成原生生物群落, 其结构和多样性随季节改变: 夏季,、等较活跃, 而异养型、混合营养型类群主要以孢囊等休眠形式存在[13]; 秋季, 海水中既存在独特类群, 如冰下海水中的鞭毛藻(主要为Pedinellales)、纤毛虫和SCM中的囊泡虫(主要为Syndiniales)、MAST, 又存在各栖息地共有类群, 如甲藻; 冬季, 寄生型Marine Alveolate Groups I/II (MAG I/II)及变形虫超群中的混合营养类群是北极点附近海水原生生物群落的主要组分[32]。混合营养方式是原生生物度过极夜的重要策略。
白令海夏季高温高盐。由于太平洋海水和沿岸淡水的注入, 楚科奇海(图2a)营养盐丰富, 硅藻、金藻、甲藻等浮游植物丰度较高, 主要分布在营养盐跃层和温盐跃层之间, 并影响纤毛虫旋毛纲等异养类群的分布[17]。因此, 太平洋水注入会影响楚科奇海中上层水体原生生物群落组成, 异养及混合营养型原生生物的增殖将促进复杂微食物环的形成, 进而影响西北冰洋生态系统的元素循环和能量传递。
波弗特海(图2a)于秋季形成冰间水道(flaw lead), 水道及周围海冰原生生物生物量及丰富度较低。克隆文库测序结果显示, 阿蒙森湾冬末冰间水道中纤毛虫等异养型类群居多; 随着冬春交替、温度上升、光照增强, 硅藻、青绿藻等自养类群逐渐增多; 春末, 硅藻为优势类群, 但异养型类群比例逐步升高,、等寄生/异养类群表现活跃, 形成了复杂的微食物网[33]。气候变暖令北冰洋冬季变暖、海冰面积和厚度减少, 有利于自养微型生物类群生长, 而纤毛虫等异养类群可捕食自养类群, 可能在未来北冰洋成为优势类群进而影响微食物网结构和功能[34]。
东北冰洋(图2a)接受来自北冰洋中心海域的贫营养低温水及与经弗拉姆海峡汇入的高温高盐的大西洋水, 加之近年来气温上升、海冰消减、盐跃层衰减, 该海域呈现“大西洋化”趋势[35]。夏末秋初, 欧亚海盆(图2a)的原生生物群落组成与不同水体的输送密切相关: 冰下海水以甲藻为主, 混合营养型居多; 无冰覆盖的表层水中, 异养型非鞭毛类为主、兼有来自大西洋和北冰洋中心区域类群[36]。因此, 洋流、水团的输送可直接影响北冰洋原生生物的组成及地理分布, 可推测气温上升会将更多太平洋或大西洋种类带至北冰洋[37]。
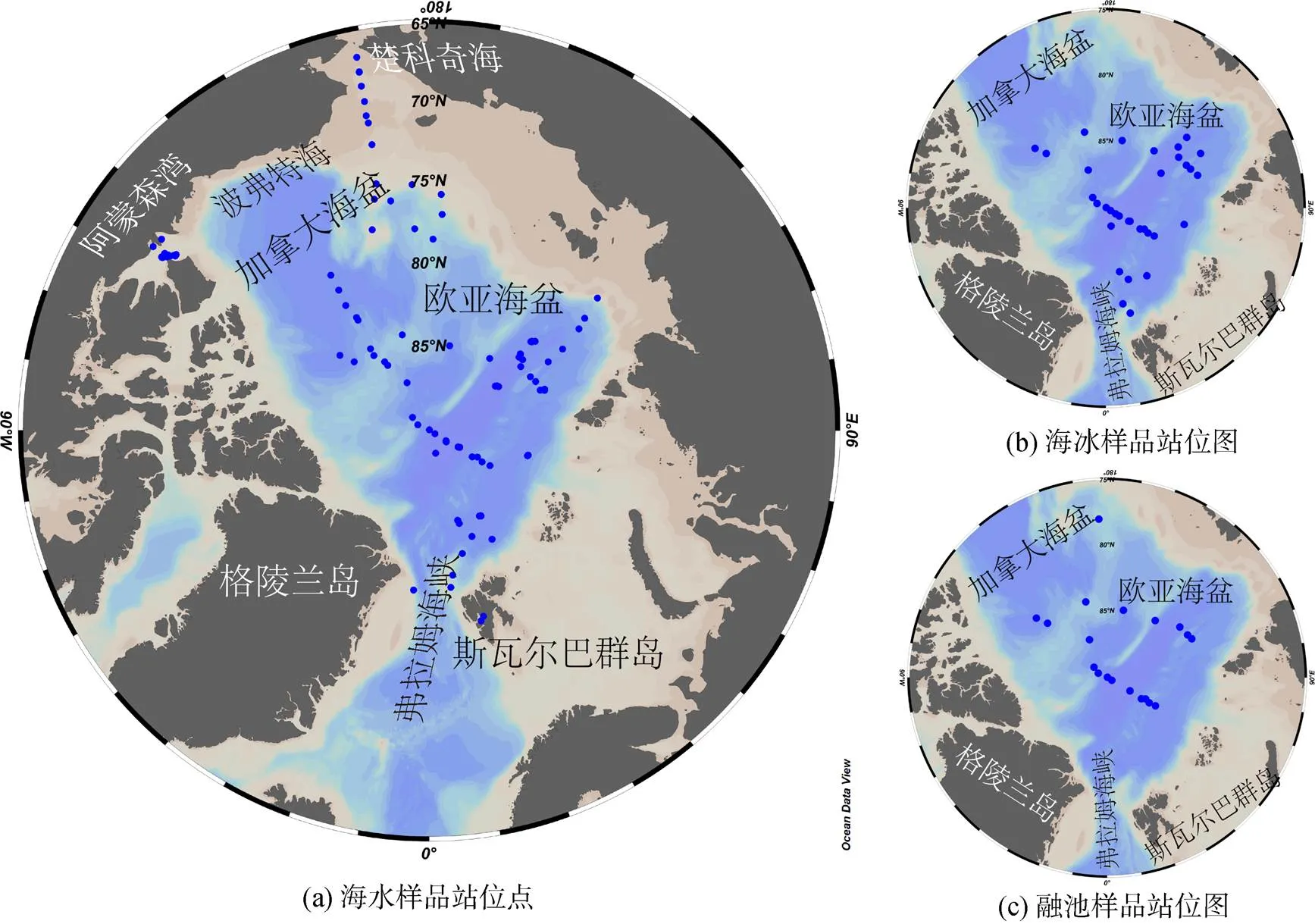
图2 基于高通量测序技术的北冰洋原生生物多样性研究站位分布。(a) 海水样品站位点[16-17, 32-34, 36, 38-41, 49]; (b) 海冰样品站位图[13, 16, 32, 36, 44-45]; (c) 融池样品站位图[37, 45, 49-50]
斯瓦尔巴群岛和格陵兰岛附近(图2a)具有较多峡湾。较高纬峡湾中, 原生生物群落与海冰覆盖度、叶绿素、光照相关; 较低纬峡湾中, 与水温、营养盐种类及浓度的关系更为密切[38]。冬末, 克隆文库测序结果显示, 较北部的Billefjorden峡湾海冰覆盖率较高, 中层水体原生生物多样性高于表层, 自养型以绿藻中的为主, 异养型以不等鞭毛类和Syndiniales为优势类群[39]。
北冰洋深层水体中基于高通量测序技术的原生生物多样性工作相对较少。冬末春初, 斯瓦尔巴北部海域(图2a)的原生生物丰富度随海水深度的增加(5 m至250 m)先增加后减少, 甲藻和Syndiniales为优势类群[40]; 春季, 该海区上层水体中, 纤毛虫在较北部、包含北冰洋中心成分的海水中最多, 而硅藻等典型的春季藻华常见种则是南部、包含大西洋来源的海水中原生生物群落的重要组分; 该海域深层水来自大西洋, 群落组成则为定鞭藻, 硅藻、隐藻、Picozoa等大西洋水同期常见原生生物。因此, 大西洋可能是北冰洋深层水中原生生物某些种类的来源之一[41]。
3.2 海冰
海冰主要由冰晶混合物、封闭或半封闭盐泡、卤道(brine channel)组成42]。海冰中原生生物群落主要取决于其中原有类群及从附近水体迁入类群的生长及死亡[43]。在北冰洋中心海域(图2b), 夏季海冰中融化程度较高的卤道更多、温度及营养盐含量更高, 不等鞭毛类-囊泡虫-根足虫(即SAR超群)为优势类群, 其中异养型、混合营养型类群更为活跃[13]。秋季海冰中原生生物多样较高, 定鞭藻、绿藻、纤毛虫、不等鞭毛类、Picobiliphyta、甲藻、Protalveolata等为优势类群[16]。硅藻(如、、)、金藻(如和)、丝足虫(如NAMAKO- 15和)等种类更偏好海冰而非海水和融池环境[44]。夏季海冰融水中类群经卤道与海水群落发生交换, 海水和海冰的原生生物组成具有一定相似性[45]。因此, 气候变暖导致北冰洋海冰覆盖面积降低的同时, 将减少原生生物的冬季栖息地[46]。
理化性质的差异导致不同年龄海冰中原生生物群落具有差异: 一年冰中, 最初的群落组成和海水群落相似, 随着海冰形成时间延长, 受海冰中环境条件、食物来源等因素影响, 原生生物群落组成发生改变[43]。北冰洋中心海域(图2b)夏季多年冰和当年冰的主要类群虽均为不等鞭毛类、囊泡虫和根足虫, 但多年冰中的不等鞭毛类占比最高、双并鞭虫更活跃, 而囊泡虫和根足虫在当年冰中更多[13]; 秋季多年冰中的原生生物多样性更高[45]。
3.3 融池
夏季冰雪融水在海冰表面汇集为融池[12]。相较于海水, 融池更接近于淡水环境, 其中的原生生物类群也多在其他淡水环境中出现。Bursa[47]利用显微镜观察发现金藻中为融池中最具代表性类群。Hegseth等[37]利用高通量测序方法报道了融池中存在更多的类群, 如金藻中,等, Chlamydomonadaceae中sp.和sp.等(图2c); Sørensen等[48]基于显微镜观察的研究发现, 格陵兰岛东北部融池中, 捕食者主要为纤毛虫, 如sp.,sp.,和sp.。Xu等[49]利用高通量测序方法在封闭性融池中报道了更多包括寡毛类在内的旋毛类纤毛虫及前人未发现的侧口类纤毛虫(图2c)。融池中原生生物与周围海冰、海水中群落也可产生交换: 秋冬季冰晶形成, 海冰卤道中的高盐水向下排入周围水体; 冰晶在逐渐生长的同时, 可能困住融池中的原生生物[50]。例如, Xu等[49]发现开放性融池和周围海水的原生生物群落组成极为相似, 融池中原生生物群落更多受到生态漂变(ecological drift)的控制。
4 研究展望
全球环境变化(如气温升高、海洋酸化等)可能会导致全球生物多样性降低, 北冰洋作为全球最为脆弱的生态系统之一首当其冲。然而, 我们目前对海洋, 尤其是极端环境(如极地、深海等)中原生生物多样性依然知之甚少[51]。现已有研究团队在北冰洋多个海区, 如加拿大海盆、楚科奇海海域、波弗特海、阿蒙森海盆等, 利用传统或(和)现代分子生物学方法开展了原生生物多样性研究[52]。由于样品采集、经费、研究手段等限制, 目前北冰洋原生生物多样性研究仍局限在易到达海区、研究多集中于样品易获得的夏季、研究重点大都聚焦于海水真光层等。
基于目前研究进展, 北冰洋原生生物多样性亟需更多研究: 1) 在典型海区, 尤其是以往研究较少涉及的区域, 如大陆架、深海水体等, 开展广覆盖、全水深的样品采集; 2) 开展覆盖全季节(尤其是样品采集难以进行的冬春季)的样品采集; 3) 与DNA不同, RNA在胞外可快速降解[53], 基于RNA测序可反映环境中的“活跃”类群[54-59], 因此, 未来研究应据此加强对活跃类群的阐释; 4) 加强对代表性海区/环境的时间序列(如以小时、天、周、月、季节等为频率的样品采集)研究, 如对融池不同发育阶段的研究将更好的揭示其中原生生物群落的演化过程; 5) 在前期工作基础上对北冰洋原生生物多样性数据的整合(如模型的构建), 将更全面揭示原生生物多样性的变动规律。总之, 北冰洋原生生物多样性数据的获得, 将为了解全球变化下该环境中原生生物多样性组成、地理分布、对环境因素变化的响应以及在生物地球化学循环中的功能等方面提供重要参考数据。
[1] WHITTAKER R H. New concepts of kingdoms of organisms[J]. Science, 1969, 163(3863): 150-160.
[2] CARON D A, COUNTWAY P D, JONES A C, et al. Marine protistan diversity[J]. Annual Review of Marine Science, 2012, 4(1): 467-493.
[3] AZAM F, FENCHEL T, FIELD J G, et al. The ecological role of water-column microbes in the sea[J]. Marine Ecology Progress Series, 1983, 10: 257-263.
[4] AZAM F, MALFATTI F. Microbial structuring of marine ecosystems[J]. Nature Reviews Microbiology, 2007, 5(10): 782-791.
[5] CARON D A, ALEXANDER H, ALLEN A E, et al. Probing the evolution, ecology and physiology of marine protists using transcriptomics[J]. Nature Reviews Microbiology, 2016, 15: 6-20.
[6] SMOLA Z T, KUBISZYN A.M, ROZANSKA M, et al. Protists of Arctic Sea Ice[C]//ZIELINSKI T, SAGAN I, SUROSZ W. Interdisciplinary approaches for sustainable development goals. GeoPlanet: Earth and Planetary Sciences. Berlin: Springer, 2018: 133-146.
[7] KWOK R, ROTHROCK D A. Decline in Arctic sea ice thickness from submarine and ICESat records: 1958– 2008[J]. Geophysical Research Letters, 2009, 36(15): L15501.
[8] ARRIGO K R. Sea ice ecosystems[J]. Annual Review of Marine Science, 2014, 6(1): 439-467.
[9] MASLANIK J, STROEVE J, FOWLER C, et al. Distribution and trends in Arctic sea ice age through spring 2011[J]. Geophysical Research Letters, 2011, 38(13): L13502.
[10] Arctic Council. Arctic marine shipping assessment 2009 report[R]. 2009.
[11] SANKELO P, HAAPALA J, HEILER I, et al. Melt pond formation and temporal evolution at the drifting station Tara during summer 2007[J]. Polar Research, 2015, 29(3): 311- 321.
[12] LUTHJE M, FELTHAM D L, TAYLOR P D, et al. Modeling the summertime evolution of sea-ice melt ponds[J]. Journal of Geophysical Research: Oceans, 2006, 111: C02001.
[13] STECHER A, NEUHAUS S, LANGE B, et al. rRNA and rDNA based assessment of sea ice protist biodiversity from the central Arctic Ocean[J]. European Journal of Phycology, 2015, 51(1): 1-16.
[14] ADL S M, SIMPSON A G B, LANE C E, et al. The revised classification of eukaryotes[J]. Journal of Eukaryotic Microbiology, 2012, 59(5): 429-514.
[15] LIMA-MENDEZ G, FAUST K, HENRY N, et al. Determinants of community structure in the global plankton interactome[J]. Science, 2015, 348(6237): 874-875.
[16] KILIAS E, KATTNER G, WOLF C, et al. A molecular survey of protist diversity through the central Arctic Ocean[J]. Polar Biology, 2014, 37(9): 1271-1287.
[17] 吴月. 南北极微微型真核浮游生物群落结构及其环境相关性分析[D]. 上海: 上海海洋大学, 2018.
WU Yue. Community structure of picoeukaryotes in polar region sea and relationgship with environment factors[D]. Shanghai: Shanghai Ocean University, 2018.
[18] 秦楠, 栗东芳, 杨瑞馥. 高通量测序技术及其在微生物学研究中的应用[J]. 微生物学报, 2011, 54(4): 445-457.
QIN Nan, LI Dongfang, YANG Ruifu. Next-generation sequencing technologies and the application in microbiology[J]. Acta Microbiologica Sinica, 2011, 54(4): 445-457.
[19] 王兴春, 杨致荣, 王敏, 等. 高通量测序技术及其应用[J]. 中国生物工程杂志, 2012, 32(1): 109-114.
WANG Xingchun, YANG Zhirong, WANG Min, et al. High-throughput sequencing technology and its application[J]. China Biotechnology, 2012, 32(1): 109-114.
[20] 王绍祥, 杨洲祥, 孙真, 等. 高通量测序技术在水环境微生物群落多样性中的应用[J] 化学通报, 2014, 77(3): 196-203.
WANG Shaoxiang, YANG Zhouxiang, SUN Zhen, et al. Application of high throughput sequencing in the diversity of water microbial communities[J]. Chemistry, 2014, 77(3): 196-203.
[21] FEI Z, RAMON M, FABRICE N, et al. Mapping of picoeucaryotes in marine ecosystems with quantitative PCR of the 18S rRNA gene[J]. Fems Microbiology Ecology, 52(1): 79-92.
[22] GONG J, DONG J, LIU X, et al. Extremely high copy numbers and polymorphisms of the rDNA operon estimated from single cell analysis of Oligotrich and Peritrich Ciliates[J]. Protist, 2013, 164(3): 369-379.
[23] VAULOT D, ROMARI K, NOT F. Are autotrophs less diverse than heterotrophs in marine picoplankton?[J]. Trends in Microbiology, 2002, 10(6): 266-267.
[24] JOSEPHSON K L, GERBA C P, PEPPER I L. Polymerase chain reaction detection of nonviable bacterial pathogens[J]. Applied & Environmental Microbiology, 1993, 59(10): 3513-3515.
[25] DELL’ANNO A, DANOVARO R. Extracellular DNA plays a key role in deep-sea ecosystem functioning[J]. Science, 2005, 309, 2179-2179.
[26] LOPEZ-GARCIA P, RODRIGUEZ-VALERA F, PEDROS- ALIO C, et al. Unexpected diversity of small eukaryotes in deep-sea Antarctic plankton[J]. Nature, 2001, 409: 603- 607.
[27] DUNTHORN M, OTTO J, BERGER S A, et al. Placing environmental next-generation sequencing amplicons from microbial eukaryotes into a phylogenetic context[J]. Molecular Biology & Evolution, 2014, 31(4): 993-1009.
[28] BESCOT N L, PROBERT I, CARMICHAEL M, et al. Eukaryotic plankton diversity in the sunlit ocean[J]. Science, 2015, 348: 1261605.
[29] 史久新, 赵进平. 北冰洋盐跃层研究进展[J]. 地球科学进展, 2003, 18(3): 351-357.
SHI Jiuxin, ZHAO Jinping. Advances in studies on the Arctic halocline[J]. Advances in Earth Science, 2003, 18(3): 351-357.
[30] MARTIN J, TREMBLAY J É, GAGNON J, et al. Prevalence, structure and properties of subsurface chlorophyll maxima in Canadian Arctic waters[J]. Marine Ecology Progress Series, 2010, 412: 69-84.
[31] LOVEJOY C. Changing views of arctic protists (marine microbial eukaryotes) in a changing Arctic[J]. Acta Protozoologica, 2014, 53(1): 91-100.
[32] BACHY C, LOPEZ-GARCIA P, VERESHCHAKA A, et al. Diversity and vertical distribution of microbial eukaryotes in the snow, sea ice and seawater near the North Pole at the end of the polar night[J]. Frontiers in Microbiology, 2011, 2(106): 106.
[33] TERRADO R, MEDRINAL E, DASILVA C, et al. Protist community composition during spring in an Arctic flaw lead polynya[J]. Polar Biology, 2011, 34(12): 1901-1914.
[34] ONDA D F L, EMMANUELLE M, COMEAU A M, et al. Seasonal and interannual changes in ciliate and dinoflagellate species assemblages in the Arctic Ocean (Amundsen Gulf, Beaufort Sea, Canada)[J]. Frontiers in Marine Science, 2017, 4: 16.
[35] POLYAKOV I V, PNYUSHKOV A V, ALKIRE M B, et al. Greater role for Atlantic inflows on sea-ice loss in the Eurasian Basin of the Arctic Ocean[J]. Science, 2017, 356(6335): 285-291.
[36] FLORES H, DAVID C, EHRLICH J, et al. Sea-ice properties and nutrient concentration as drivers of the taxonomic and trophic structure of high-Arctic protist and metazoan communities[J]. Polar Biology, 2019, 42(7): 1377-1395.
[37] HEGSETH E N, SUNDFJORD A. Intrusion and blooming of Atlantic phytoplankton species in the high Arctic[J]. Journal of Marine Systems, 2008, 74(1/2): 108-119.
[38] LUO W, LI H, GAO X, et al. Microbial eukaryotic diversity with emphasis on picoprasinophytes under the sea ice of the central Arctic Ocean in summer[J]. Current Science, 2018, 115(9): 1709-1713.
[39] SORENSEN N, DAUGBJERG N, GABRIELSEN T M. Molecular diversity and temporal variation of picoeukaryotes in two Arctic fjords, Svalbard[J]. Polar Biology, 2012, 35(4): 519-533.
[40] DE S, ANTONIO G G, TOMASION M P, et al. Diversity and composition of pelagic prokaryotic and protist communities in a thin Arctic sea-ice regime[J]. Microbial Ecology, 2019, 78(2): 388-408.
[41] MESHRAM A R, VAFER A, KRISTIANSEN S, et al. Microbial eukaryotes in an arctic under-ice spring bloom north of Svalbard[J]. Frontiers in Microbiology, 2017, 8: 1099.
[42] KREMBS C, GRADINGER R, SPINDLER M. Implications of brine channel geometry and surface area for the interaction of sympagic organisms in Arctic sea ice[J]. Journal of Experimental Marine Biology and Ecology, 2000, 243(1): 55-80.
[43] CARON D A, GAST R J, GARNEAU M V. Sea ice as a habitat for micrograzers[M]. New York, US: John Wiley & Sons, Ltd, 2016.
[44] HARDGE K, PEEKEN I, NEUHAUS S, et al. The importance of sea ice for exchange of habitat-specific protist communities in the Central Arctic Ocean[J]. Journal of Marine Systems, 2017, 165: 124-138.
[45] KILIAS E S, PEEKEN I, METFIES K. Insight into protist diversity in Arctic sea ice and melt-pond aggregate obtained by pyrosequencing[J]. Polar Research, 2014, 33: 23466.
[46] NIEMI A, MICHEL C, HILLE K, et al. Protist assemblages in winter sea ice: setting the stage for the spring ice algal bloom[J]. Polar Biology, 2011, 34(12): 1803-1817.
[47] BURSA A. Phytoplankton in coastal waters of the Arctic Ocean at Point Barrow, Alaska[J]. Arctic, 1963, 16(4): 239-262.
[48] SORENSEN H L, THAMDRUP B, JEPPESEN E, et al. Nutrient availability limits biological production in Arctic sea ice melt ponds[J]. Polar Biology, 2017, 40(8): 1593- 1606.
[49] XU D, KONG H, YANG E J, et al. Contrasting community composition of active microbial eukaryotes in melt ponds and sea water of the Arctic Ocean revealed by high throughput sequencing[J]. Frontiers in Microbiology, 2020, 11: 1170.
[50] LEE S H, MCROY C P, JOO H M, et al. Holes in progressively thinning Arctic sea ice lead to new ice algae habitat[J]. Oceanography, 2011, 24(3): 302-308.
[51] HOLLAND M M, BITZ C M, 2003. Polar amplification of climate change in coupled models. Climate dynamics, 21(3/4): 221-232.
[52] LIU Q, ZHAO Q, MCMINN A, et al. Planktonic microbial eukaryotes in polar surface waters: recent advances in high- throughput sequencing[J]. Marine Life Science & Technology, 2021, 3: 94-102.
[53] JONES S E, LENNON J T. Dormancy contributes to the maintenance of microbial diversity[J]. Proceedings of the National Academy of Sciences USA, 2010, 107(13): 5881-5886.
[54] CAMPBELL B J, YU L, HEIFRLBERG J F, et al. Activity of abundant and rare bacteria in a coastal ocean[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(31): 12776.
[55] KARL D M, BAILIFF M D. The measurement and distribution of dissolved nucleic acids in aquatic environments[J]. Limnology & Oceanography, 1989, 34(3): 543- 558.
[56] LOGARES R, AUDIC S, BASS D, et al. Patterns of rare and abundant marine microbial eukaryotes[J]. Current Biology, 2014, 24: 813-821.
[57] MASSANA R, GOBET A, AUDIC S, et al. Marine protist diversity in European coastal waters and sediments as revealed by high‐throughput sequencing[J]. Environmental Microbiology, 2015, 17(10): 4035-4049.
[58] XU D, LI R, HU C, et al. Microbial eukaryote diversity and activity in the water column of the South China Sea based on DNA and RNA high throughput sequencing[J]. Frontiers in Microbiology, 2017, 8: 1121.
[59] XU D, SUN P, ZHANG Y, et al. Pigmented microbial eukaryotes fuel the deep sea carbon pool in the tropical Western Pacific Ocean[J]. Environmental microbiology, 2018, 20: 3811-3824.
Advances in protist diversity studies in representative environments of the Arctic Ocean based on high-throughput sequencing
CHEN Yi-xuan1, 2, KONG He-jun1, 2, XU Da-peng1, 2
(1. State Key Laboratory of Marine Environmental Science, College of Ocean and Earth Sciences, Xiamen University, Xiamen 361102, China; 2. Fujian Key Laboratory of Marine Carbon Sequestration, Xiamen University, Xiamen 361102, China)
Arctic; protist; community composition; sea ice; microbial eukaryotes; global warming; melt ponds
The term protist refers to a diverse array of eukaryotic organisms capable of living as single cells, although many species form colonies ranging from a few to many cells. Early protist diversity studies usually applied conventional methods such as microscopic observation and flow cytometry. However, due to the limitations associated with these approaches, the diversity of protists as a whole cannot be studied thoroughly. Recent advances in sequencing-based techniques, e.g., high-throughput sequencing, have enabled in-depth assessment of protist diversity from diverse environments. The Arctic Ocean has
much attention because it is one of the most vulnerable environments affected by global warming. However, compared with their prokaryotic counterparts, our understanding of the diversity, community composition, and environmental driving factors of protists has largely lagged behind. In this review, published articles on protist diversity from diverse Arctic environments (e.g., seawater, sea ice, melt ponds, etc.) have been synthesized, and we intend to provide useful data for future studies on the diversity of Arctic protists, especially in the context of global warming.
National Natural Science Foundation of China, Nos. 41876142; 91751207; 41861144018]
Nov. 24, 2020
P735
A
1000-3096(2021)08-0081-08
10.11759/hykx20201124001
2020-11-24;
2021-02-02
国家自然科学基金项目(41876142, 91751207, 41861144018)
陈一璇(1998—), 女, 广东潮阳人, 硕士研究生, 主要从事海洋原生生物生态学研究。E-mail: yixuan.chen20@imperial.ac.uk; 孔菏君(1997—), 共同第一作者, 女, 山东菏泽人, 硕士研究生, 主要从事海洋原生生物生态学研究。E-mail: 22320181152124@stu.xmu.edu.cn; 徐大鹏(1980—), 通信作者, 男, 副教授, 从事海洋原生生物生态学研究, E-mail: dapengxu@xmu.edu.cn
(本文编辑: 赵卫红)
