基于聚合酶链置换反应的2D-LASM混沌文本加密算法
2021-09-10王夕远殷志祥崔建中徐如解
王夕远, 殷志祥,2*, 唐 震, 杨 静, 崔建中, 徐如解
(1. 安徽理工大学 数学与大数据学院, 安徽 淮南 232001;2. 上海工程技术大学 数学与统计学院, 上海 201620; 3. 淮南联合大学 计算机系, 安徽 淮南 232001)
近年来由于科技的迅猛发展,传统电子计算机已经难以满足人们的需求.DNA计算机是近些年最有独创性和出乎意料的发现之一,其具有高度的并行性、运算速度快、存储容量大、耗能低和DNA分子资源丰富等优点.为了制造出DNA计算机,人们开始对DNA计算进行研究.DNA计算的第一个例子解决了一个7城市哈密顿路径问题[1].2000年,Head等[2]提出了使用DNA质粒的一种新的计算方法,列出了潜在的优势.通过报告计算图顶点集最大独立子集基数的NP完备算法题的一个实例计算,说明了新方法的有效性.2003年,殷志祥等[3]提出了在基于表面的DNA计算采用荧光标记策略,解决了简单的0-1规划问题.2011年,Qian等[4]提出了DNA链置换级联的神经网络计算.此外,DNA计算还可以用来构建半加器、半减器、全加器、全减器[5-9].2019年,Chao等[10]在DNA折纸上利用DNA单分子导航仪求解迷宫问题.同年,唐震等[11- 12]利用了DNA折纸术解决了一类特殊的整数规划问题,还提出了在DNA折纸基底上的一种动态的与非门计算系统.
随着计算机技术的发展和运用,人们通过网络进行信息传输的频率越来越频繁.为了防止传输的信息被截获破解,人们越来越重视对信息的传输加密.由于混沌系统具有有界性、遍历性、伪随机性、对初值条件和控制参数敏感性等特点,人们把混沌系统用于信息加密领域.2010年,王林林[13]提出了基于混沌的密码算法设计与研究.2015年,贾嫣等[14]提出了基于改进混沌映射的图像加密算法,该算法采用的是雅克比椭圆映射对初始密钥进行迭代产生新的密钥.2020年,严利民等[15]提出了混沌映射和流密码结合的图像加密算法.2021年,蔡敏等[16]设计并实现了基于混沌时间序列的图像加密算法.同年,曾祥秋等[17]提出了基于改进的Logistic映射的混沌图像加密算法.考虑到DNA可以存储大量信息的特点,人们开始将DNA和混沌系统进行结合,并用于信息加密领域.2016年,Wang等[18]提出使用CML和DNA序列操作的彩色图像加密方案.该方案将图像的每一个像素进行DNA编码,并给出了8种相对应的DNA编码规则.2017年,孙倩等[19]提出了基于DNA编码与统计信息优化的图像加密算法.2020年,朱凯歌等[20]设计了基于DNA动态编码和混沌系统的彩色图像无损加密算法.
之前,人们所提出的将DNA用于信息加密的算法中,并没有涉及到DNA计算中的化学反应,只是将数据转换成了DNA编码的形式.本文提出了一种基于聚合酶链置换反应的2D-LASM混沌映射文本加密算法,成功将DNA计算中的化学反应结合到加密过程中,并对该算法进行了密钥空间分析,表明该算法具有较好的加密效果.
1 准备工作
1.1 二维Logistic-Adjusticed-sine (2D-LASM)映射
本文的加密算法采用结构简单、性能优良的二维Logistic-Adjusticed-sine(2D-LASM)映射,其数学表达式如下:
其中,μ∈[0,1],Xn,Yn∈(0,1).这里给定初值,设X1=0.5,Y1=0.5,为了消除瞬态效应迭代n次,产生两个长度为n的伪随机数组X和Y.
1.2 DNA序列编码
DNA(脱氧核糖核酸)是染色体的主要组成成分,同时也是主要遗传物质.DNA是双螺旋结构,有两条脱氧核苷酸链,一个脱氧核苷酸分子由三个分子组成:一分子含氮碱基、一分子脱氧核糖及一分子磷酸.脱氧核苷酸共有4种含氮碱基分别为:A(腺嘌呤)、T(胸腺嘧啶)、G(鸟嘌呤)和C(胞嘧啶).按照Watson-Crick碱基互补配对原则,A和T互补,C和G互补.在计算机中,信息的存储是用二进制0和1进行表示.二进制中0和1是互补的,因此00和11,10和01也是互补的.若将A、T、C、G分别用00,01,10,11进行表示共有24种,而符合互补原则的只有8种,见表1.
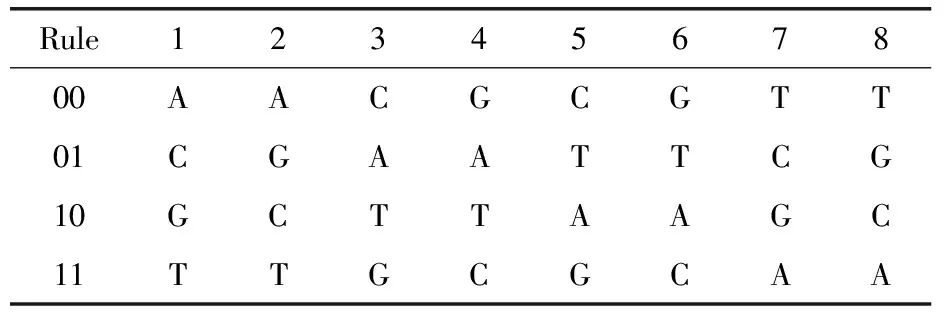
表1 DNA编码规则
1.3 聚合酶链置换反应原理
聚合酶链置换反应(Polymerase Strand Displacement, PSD)是一种基于聚合酶的链置换反应,其和一般的链置换反应不同的是该反应需要有酶的参与.反应原理见图1,这里的A和A*互补,B和B*互补,C和C*互补,D和D*互补.这种反应类似于聚合酶链式反应(PCR).首先,引物和部分互补的双链DNA粘性末端按碱基互补配对原则结合,即A和A*相结合;其次,将温度调至DNA聚合酶最适反应温度,在DNA聚合酶的作用下,从引物的3′端开始以5′→3′端的方向延伸,合成与模板5′-D-C-B-A-3′互补的DNA链,将部分互补的双链DNA中的单链5′-C*-D*-3′置换出来.
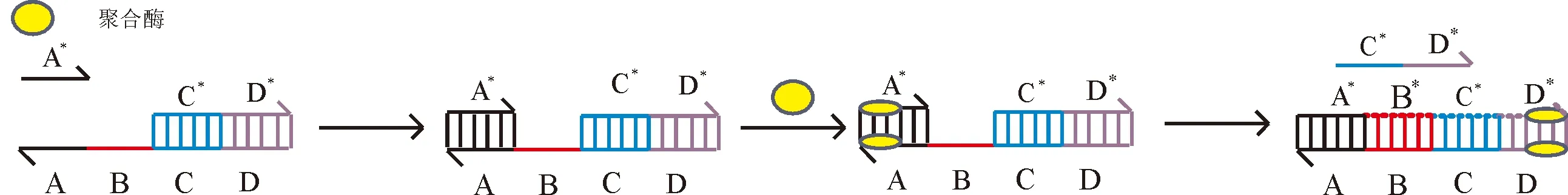
图1 聚合酶链置换反应原理Fig.1 Principle of polymerase strand displacement reaction
2 本文的加密算法
2.1 算法框架
(1)将待加密的文本信息进行置乱处理,随后和2D-LASM混沌映射产生的伪随机序列进行异或操作,之后再根据产生的随机种子选择相应的DNA编码规则得到相对应的DNA序列;
(2)将所得到的DNA序列进行PSD反应,得到一组新的DNA序列.根据随机种子选择相应的DNA解码规则,得到加密的文本.
本文的加密算法具体框架如图2所示.
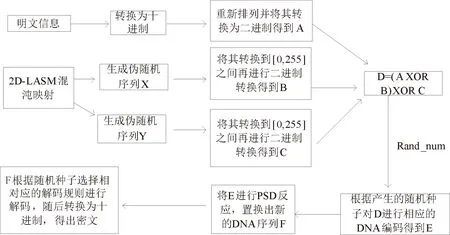
图2 算法框架Fig.2 Algorithm framework
2.2 加密算法的工作步骤
Step1:输入一段明文,将其转换为十进制的数,长度为L;
Step2:采用randperm函数将十进制数进行重新排列,并将其转换为二进制数,记为A;
Step3:通过二维Logistic-Adjusted-Sine混沌映射迭代1 000+L次,得到两个伪随机序列X,Y;
Step4:分别取X,Y的后L位,经过mod(floor(k*108),256)将其转换到[0,255]之间并进行二进制处理得到B,C,这里mod表示取余,floor表示向下取整;
Step5:对Step2中的A和Step4中所得到的B,C进行异或操作得到D;
相关的地方性立法目前还不健全,特别是自然地理环境特殊的省份,例如西藏自治区发展高原农业和河谷农业,东北三省有广袤的东北平原发展专业化农业,都没有出台相关文件。“各地可结合当地的优势农产品布局,形成各具特色的农业机械化区域,进一步拓展农业机械化的服务和作业领域,突出综合性、多样性、优质高效性,实现农业机械化的跨越式发展,促进农业机械化与地区经济协调统一发展。”[5]即形成地理单元与行政区划紧密连接的农业机械化发展格局。伴随着我国经济的发展和全面深化改革进程的加快,农业机械化立法已经明显滞后于现实的发展,制约了农业机械化在发展农业现代化中的作用。
Step6:根据产生的随机种子选择相应的DNA编码规则对D进行编码,得到DNA序列E;
Step7:将所得到的DNA序列E按照长度为m进行分割,得到u条DNA序列.再将这u条序列分别进行PSD反应,可以置换出u条新的DNA序列;
Step8:将Step7所得到的u条DNA序列在DNA连接酶的作用下组成一条新的DNA序列F;
Step9:根据随机种子选择相应的DNA解码规则对F进行解码,并将其转换为十进制数G;
Step10:对G使用char函数得到相应的密文.
解密过程是加密过程的逆过程,这里由于篇幅问题就不再阐述.
2.3 实例分析
该实例的仿真是在Matlab 2016a仿真软件上进行的,这里所采用的明文为you are a better man;将其转换为十进制数得到[121,111,117,32,97,114,101,32,97,32,98,101,116,116,101,114,32,109,97,110],可知L为20.并将其进行重新排列得到[97,101,98,114,110,121,97,109,116,32,32,32,117,32,101,116,111,97,101].随后转换为二进制数得到A;
通过二维Logistic-Adjusted-Sine混沌映射迭代1 000+20次,得到两个伪随机序列X,Y;
分别取X,Y的后20位,经过mod(floor(k* 108),256)将其转换到[0,255]之间并进行二进制处理得到B,C,这里mod表示取余,floor表示向下取整;
对A,B,C进行异或操作得到D;这里产生的随机种子为2,即选择第二种DNA编码规则进行编码得到DNA序列E: CGACCGTCGTTTCTACAAAGGGAGTAATTCGGCTATACTTTGTTGACGG-GCGAAGCCTAGCCCTGTGACTCCTTCCCCAT长度为80 nt;对DNA序列E按照长度为16 nt进行分割,可得到5条DNA序列,见图3.

图3 DNA序列E的分割Fig.3 Segmentation of DNA sequence E
将所得到的5条DNA序列进行PSD反应,具体过程见图5.为了保证和DNA序列E相对应,生成一组随机数,根据上述的方法进行DNA编码得到DNA序列F:CCGCAGTTCATGGACCGCTCGGTTAGACTCGAGATCTTTCATTAAAAGATTCCGACG-CCACCTCGGTGTTTGGCGTACCT长度为80 nt;对F按照16 nt进行分割得到5条DNA序列,见图4.

图4 DNA序列F的分割Fig.4 Segmentation of DNA sequence F
经过PSD反应之后得到5条长度为16 nt的DNA单链,将这5条单链按照m1-m2-m3-m4-m5的顺序进行合并得到F.这样通过PSD反应将文本信息转换的DNA序列E成功变成了DNA序列F;接下来根据随机种子选取相对应的DNA解码规则将其转换为二进制数G,将G转换成十进制数H,之后得到密文“|Jn_N<>h®]g+”.
通过以上步骤成功将明文you are a better man转换为|□Jn_N<>□h®]g+.需要注意的是这里的DNA序列F是根据产生的随机数进行DNA编码的,产生的随机数不同这里的F就不同,最终得到的密文也就有所不同.
3 算法安全性分析
若想对密文进行解密需要知道明文所对应的DNA序列E和所产生的随机种子,但由于DNA序列F是由产生的随机数所得到的,再加上PSD反应是不可逆的,因此,得到DNA序列E就显得很困难,此外还需要知道明文信息转换为十进制之后是如何进行重新排列的.本文将经过PSD反应(图5)所置换出来的DNA单链设置为16 nt,由于DNA具有4种含氮碱基,因此,每条DNA单链共有416种可能性.由于满足互补条件的DNA编码规则共有8种,因此,相对应的DNA解码规则也有8种.对明文信息转换成十进制的数进行重新排列共有20!种,因此,该算法的密钥空间为(20!)*8*8*416*416*416*416≈2184远大于2100,这说明该算法可以抵抗穷举攻击,安全性能高.
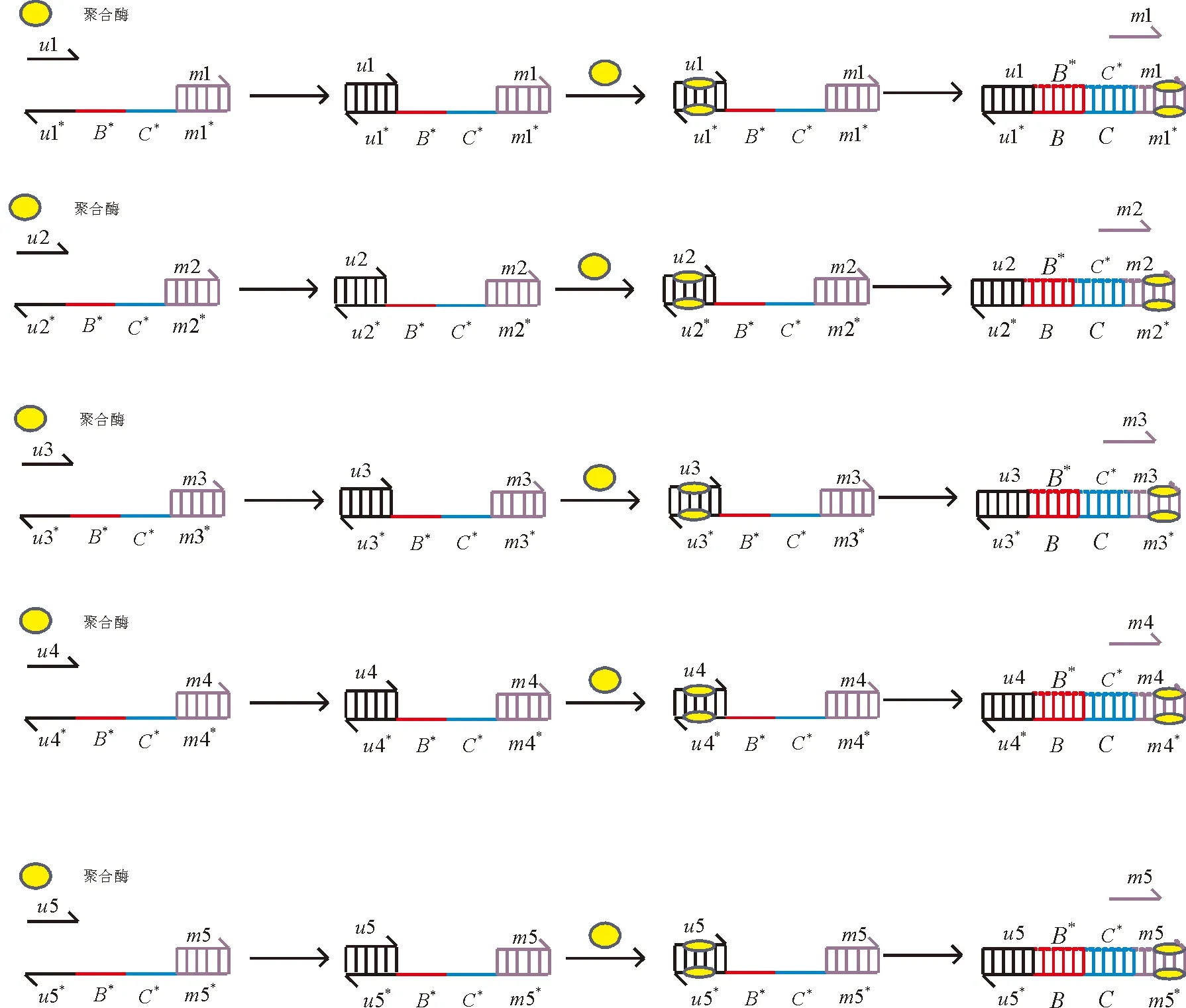
图5 PSD反应Fig.5 Polymerase stand displacement reation
4 结 论
本文提出了一种基于聚合酶链置换反应的2D-LASM混沌映射文本加密算法,克服了之前DNA加密算法中没有涉及到DNA计算加入化学反应这一缺点.加入化学反应之后,算法的密钥空间有了很大的改善.同时,这一算法还为图像加密提供了思路.如何将DNA计算中的化学反应用于图像加密,这是以后需要解决的问题.
