棉花转录因子GhSPL1生物信息学分析*
2021-09-01杨笑敏谢业涛李永旗孙亮庆
杨笑敏,谢业涛,李永旗,孙亮庆
(江西省棉花研究所,江西 九江 332105)
棉花是我国重要的经济作物和战略储备物资,棉纤维是纺织工业的基本原料。衣食住行穿衣居于首位,随着人民生活水平的不断提高,人民对纺织品的需求达到了发达国家水平[1]。我国作为14亿的人口大国,拥有960万平方千米国土,可耕地面积为124.8万平方千米,其中棉花的耕种面积为4.9万平方千米,主要分布在长江流域、黄河流域及新疆。近几年,由于棉花高投入,低收益,农民植棉积极性降低,内地植棉面积大幅度少,国产棉花产量难以满足人们对棉花产量的需求,棉花的供求出现失衡,矛盾日益突出[2-3]。棉花纤维的品质,决定棉花的价格,决定农民的收益。随着棉花全基因组测序的完成[4-7],挖掘功能基因,从分子层面提高棉花品质,培育优良品种已成为研究的热点。
棉花品种布局、土壤和栽培技术等因素通过影响棉花生长发育而影响纤维品质[8]。研究发现SPL在植物的整个生命周期中都发挥着重要的调控作用,参与植株的形态建成[9],调控植株发育阶段的转变[10],调控叶片发育[11],促进植物开花[12-13],参与果实的发育成熟过程[14-15],应答生物与非生物胁迫[16-17],参与激素信号转导[18]多个生理生化过程。SPL1基因是SPL(SQUAMOSA promoter-binding protein-like)家族中的一员,编码植物中转录调控因子[19]。SPL蛋白具有高度保守的DNA结合结构域[20]。SPL转录因子最早是在金鱼草中鉴定出来的,随后在拟南芥[21]、玉米[22-23]、水稻[24-25]、小麦[26]、苹果[27]、番茄[28]等植物中陆续被发现。植物间隔期的长短能够反映其叶原基分化速率及其叶片形成的快慢。过表达AtSPL9和AtSPL10的拟南芥,叶片间隔期增加,叶片的生成速率降低,从而使植株整个生命周期中叶片总量减少,单个叶面积增加[29],过表达AtSPL13的拟南芥幼苗,与对照相比第1片真叶的形成所用时间更长[30]。玉米沉默LG1会使叶片不能正常发育,形成异常的叶舌与叶耳[31]。过表达OsSPL14的水稻,分蘖数减少,增加花序分枝、穗粒数、千粒重,通过调控水稻的株型提高产量[32]。过表达AtSPL3拟南芥开花期提前,如果受到光照增强、光照时间增加,拟南芥的花和花序无法正常发育[33-34]。过表达LeSPL3载体转化烟草后,会导致花柄离区离层细胞层数增加,花容易从植株上脱落[35]。过表达OsSPL16的水稻,与对照相比籽粒更宽,单粒种增加,籽粒更加饱满充实;降低OsSPL16的水稻,籽粒宽降低,变得细长,胚乳透明度增加,水稻垩白度降低,稻米外观明显改变[36-37]。沉默AtSPL8的拟南芥植株,主要是对植株花和果实产生影响,会导致花药变小,花粉量减少,果荚变短小,出现半不育表型[38-39]。
通过比较GhSPL1在棉花生长发育过程中的FKPM值,发现GhSPL1在棉花胚珠形成过程中的数值较高,具有较高的表达,本研究对GhSPL1进行了基因结构分析,对GhSPL1进行了蛋白的亲水性/疏水性分析、跨膜结构分析、信号肽预测、亚细胞位置预测、二级结构预测、同源建模,对物种之间的亲缘关系进行分析,对棉花胚珠发育过程中的表达情况进行了分析,为进一步验证GhSPL1的功能奠定了基础。SPL参与调控棉花整个生长发育周期,但本课题没有针对GhSPL1进行的深入研究和挖掘GhSPL1的功能,有特于以后进一步扩展。
1材料与方法
1.1生物信息学分析
从cottonFGD中下载GhSPL1的基因序列、蛋白序列、基因组序列。在Cell-PLoc2.0中进行亚细胞定预测;GhSPL1的蛋白序列在ExPASY、ProtScale、TMHMM、SignalP4.0、GOR IV、SMART、Phyre2中进行生物信息学分析。
1.2进化分析
分别以GhSPL1的CDS序列和氨基酸序列为目标序列,在cottonFGD网站用Blastn比对检索,获得海岛棉、亚洲棉、雷蒙德氏棉三个棉种的同源序列,在Phytozome用Blastp比对检索,得到柏树、杨树、可可、茄子、拟南芥、玉米、水稻、炭藓中的同源序列,用SMART对比对获得的基因进行基因结构域进行分析,留下含有一个SBP结构域,2个ANK结构域,1个跨膜区域的基因。把最终得到的氨基酸序列输入Clustal W 软件进行多重序列比对,在MEGA7. 0 软件中选择 Neighbor-Join 法,设定Bootstrap的值为1000,构建物种系统进化树。
1.3表达分析
从Gossypium Resource And Network Database网站中的Expression Visualization中下载GhSPL1基因胚珠发育过程中的FPKM值(Fragments Per Kilobase Million)。
FRKM值越大,基因表达量越高,FRKM值越小,基因的转录水平越低。利用Excel工具得到基因表达柱形图。
2结果与分析
2.1 GhSPL1蛋白生物信息学分析
本研究在cottonFGD(https://cottonfgd.org/profiles/gene)网站中获得GhSPL1基因序列和其编码的蛋白序列,GhSPL1基因位于D12染色体上的43027984~43035100处,基因长度为7117 bp,CDS长度为2964 bp。利用GSDS软件(http://gsds.gao-lab.org/)对绘SPL1基因结构进行分析,结果如图1,GhSPL1基因含有10个外显子。在Cell-PLoc2.0(http://www.csbio.sjtu.edu. cn/bioinf/Cell-PLoc-2/)中对GhSPL1蛋白进行亚细胞定位预测,结果显示GhSPL1蛋白在细胞核上。

图1 GhSPL1基因的结构分析
在ExPASY网站ProtParam(https://web.expasy.org/protparam/)程序中对GhSPL1基因编码的蛋白质进行蛋白组分分析,结果显示GhSPL1基因编码987个氨基酸分子,其中精氨酸(arg)和赖氨酸(lys)是带正电的氨基酸残基有113个,天冬氨酸(asp)和谷氨酸(glu)是带负电的氨基酸残基有129个。
GhSPL1蛋白的相对分子质量是109667.79 kDa,预测的等电点(PI)是6.02,GhSPL1蛋白的分子式是C4783H7564N1380O1487S46,GhSPL1蛋白的不稳定系数是49.67,脂肪系数为81.80,总平均亲水性是-0.425,预测该蛋白为不稳定蛋白。
利用在线网站ProtScale(http://web.expasy.org/protparam/)预测了GhSPL1蛋白的亲疏水性,将GhSPL1的蛋白序列输入ProtScale,选择默认的Hphob. / Kyte & Doolittle,滑窗大小为9,线性加权模型,预测结果如图2,结果表明:第227位精氨酸(R)的所测分值最低为-3.578,则S亲水性最强,第960位缬氨酸(V)的所测分值最高为3.311,则F疏水性最强,GhSPL1蛋白序列中亲水性氨基酸的总分大于疏水性氨基酸的总分,该蛋白为亲水性蛋白。综上所述,GhSPL1蛋白是一种亲水性的不稳定蛋白。

图2 GhSPL1蛋白的亲水性/疏水性分析
通过在线网站TMHMM(http://www.cbs.dtu.dk/services/TMHMM/)对GhSPL1进行跨膜结构域预测分析(图3),结果表明:1~964的氨基酸位于细胞膜外部,942~964的氨基酸为跨膜区,965~987的氨基酸位于细胞膜内部,该蛋白存在跨膜区域,属于跨膜蛋白。
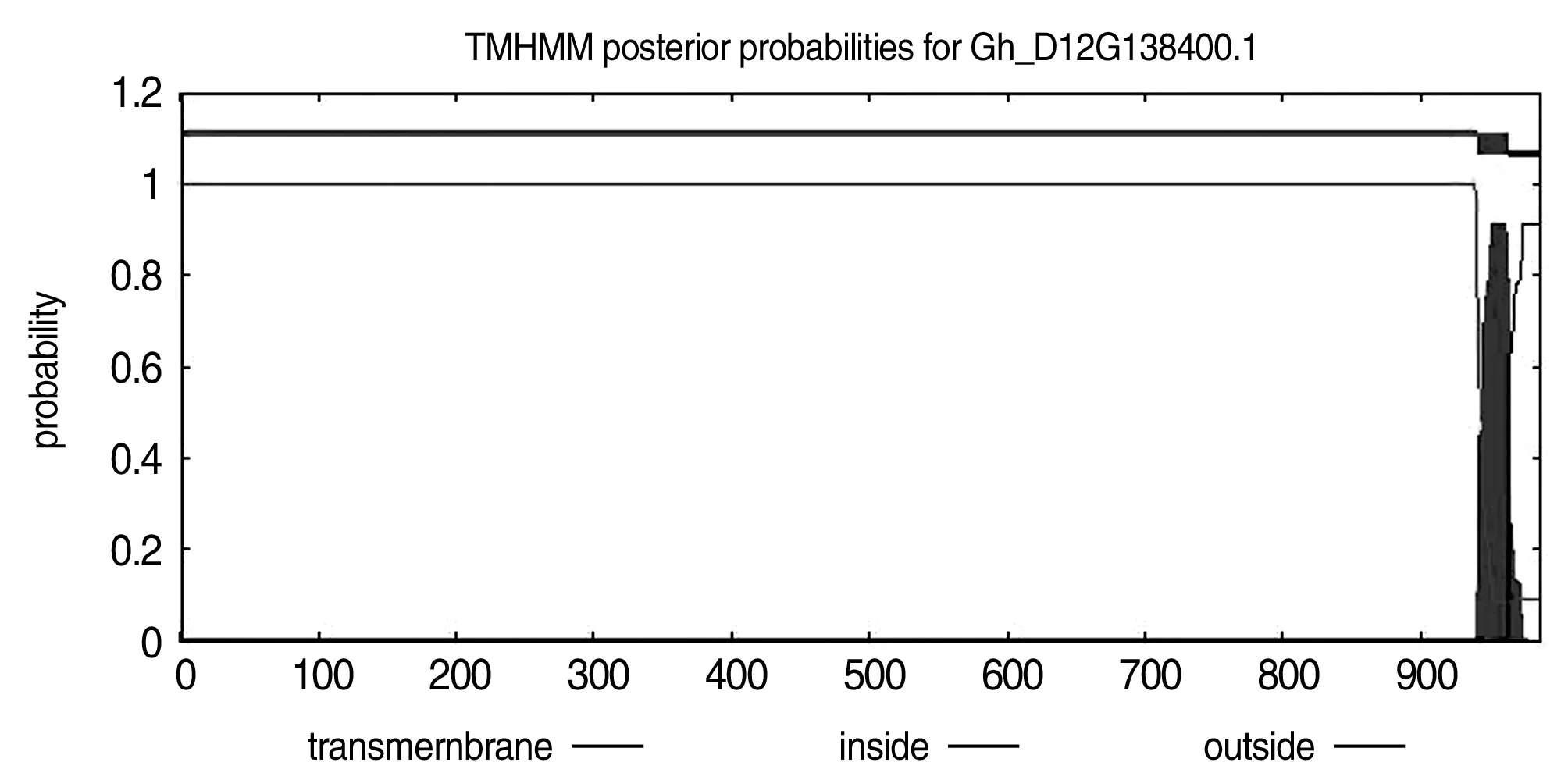
图3 GhSPL1蛋白的跨膜结构分析
利用SignalP4.0网站(http://www.cbs.dtu. dk/services/SignalP-4.0/)预测GhSPL1蛋白的信号肽,结果如图4所示:信号肽区域处,S值较高,GhSPL1蛋白的S值变化相对比较平稳,波动不大,在第1个蛋氨酸(Met)处是最大值,分值为0.168;GhSPL1蛋白的C值(剪切位点值)在第54位天冬氨酸(Asp)处是最大值,为0.453,GhSPL1蛋白的Y值(综合剪切位点值)在第54位天冬氨酸(Asp)处是最大值,为0.212,D值为0.155。综上所述,GhSPL1不是分泌蛋白。
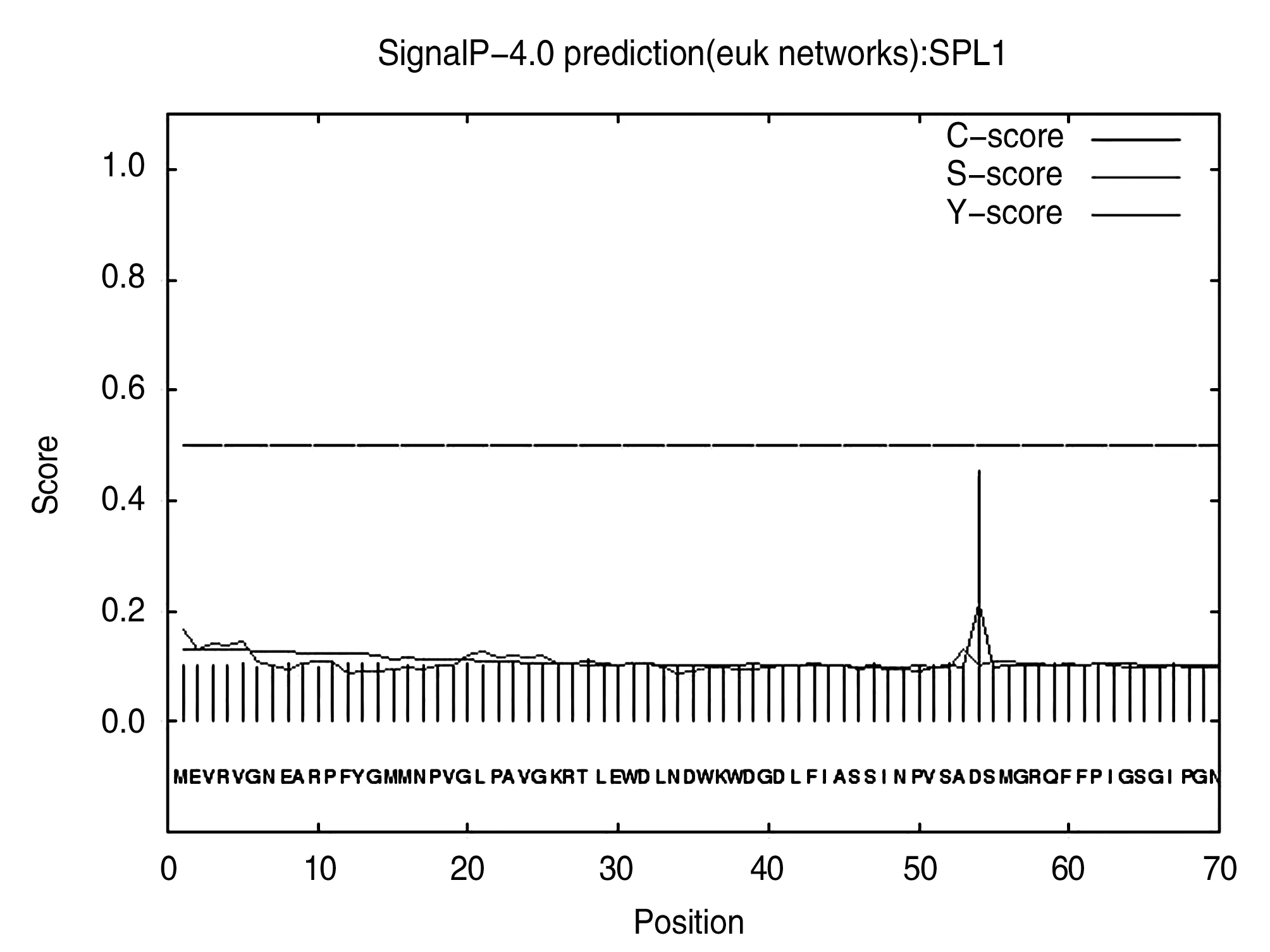
图4 GhSPL1蛋白的信号肽预测

利用GOR IV软件(https://npsa-prabi.ibcp.fr/cgi-bin/)对GhSPL1的二级结构进行预测分析,结果如图5所示,该蛋白主要由无规则卷曲、α螺旋、延伸连构成,其中α螺旋有297个氨基酸,占30.09%;延伸连有148个氨基酸,占14.99%;无规则卷曲有542个氨基酸,占54.91%。

图5 GhSPL1蛋白的二级结构预测
利用在线软件SMART(http://smart.embl-heidelberg.de/)对GhSPL1蛋白的结构域进行分析,结构如图6:该蛋白从156位氨基酸开始到230为氨基酸结束,编码植物蛋白中发现的序列特异性DNA结合结构域SBP,该结构域含有2个锌指结构;从754位氨基酸开始到783位氨基酸,811位氨基酸到841位氨基酸,编码两个锚蛋白重复序列区域,即ANK结构域;从942位氨基酸开始到964为氨基酸是TMHMM程序检测到的跨膜螺旋区域。
利用软件Phyre2(http://www.sbg.bio.ic. ac.uk/phyre2)对GhSPL1蛋白的三维结构进行建模,结果如图7所示,GhSPL1蛋白主要由无规则卷曲、α螺旋构成,预测分析结果与GhSPL1蛋白二级分析结果一致。

图7 GhSPL1蛋白的三维建模
2.2系统进化分析
在cottonFGD网站中用Blastn程序检索,获得海岛棉、亚洲棉、雷蒙德氏棉的同源序列,在Phytozome在Blastp程序中检索,获得柏树、杨树、可可、茄子、拟南芥、玉米、水稻、炭藓的同源序列,用软件SMART工具分析蛋白保守结构域,筛选出于GhSPL1基因所含有的结构域相同的基因序列,共获得25个基因,其中海岛棉有5个基因,亚洲棉有2个基因,雷蒙德氏棉有2个基因,柏树有2个基因、杨树有3个基因、可可有2个基因、茄子有1个基因、拟南芥有2个基因、玉米有2个基因、水稻有1个基因、炭藓有3个基因。通过MEGA7.0构建进化树如图8所示:可可的基因与棉花中的基因在同一个分支上,所以可可与棉花的亲缘关系较近,该基因在水稻、玉米、茄子等中都含有该类型的基因,所以基因出现在单子叶植物和双子叶植物分化之前。

图8 GhSPL1蛋白的三维建模
2.3基因表达分析
从网站Gossypium Resource And Network Database(http://grand.cricaas.com.cn/home)下载GhSPL1基因在胚珠发育过程中的FPKM值,该值可以代表基因的表达情况。如图9所示:GhSPL1基因在整个胚珠形成过程都有较高的表达,在胚珠形成的第20天有较高的表达,在第25天表达量较低。
3讨论与结论
SPL是植物中特有的一类转录因子,参与调控植物的生长发育、次生代谢、响应生物与非生物胁迫。GhSPL1作为SPL家族中的一个成员,含有一个在进化上高度保守的SBP结构域,该区域是DNA结合区域。本研究在海岛棉、亚洲棉、雷蒙德氏棉、柏树、杨树、可可、茄子、拟南芥、玉米、水稻、炭藓中鉴定出GhSPL1的同源基因,但是由于很多的物种基因测序工作还没有开展,所以GhSPL1基因的结构、数量、进化机制、生物学功能还不明确,有待进一步研究。
花是植物的繁殖器官,开花是植物营养生长与生殖生长的分水岭,开花受到光照、温度、植物激素、生物胁迫、非生物胁迫及自身内部情况等多种因素的影响。棉花开花的已知途径有光周期诱导途径,春花途径,赤霉素途径,自主途径,GhSPL1在棉花胚珠发育过程中有较高的表达,由于GhSPL1在植株的开花过程中发挥重要的调控作用,猜测GhSPL1可能参与棉花开花诱导过程,但是其具体作用机制还不清楚,仍需进一步验证挖掘GhSPL1的基因功能。
