基于mtDNA和cpDNA序列的甘薯栽培种及近缘野生种分析
2021-07-24王连军田小海柴沙沙焦春海杨新笋
王 崇,王连军,田小海,雷 剑,柴沙沙,焦春海,杨新笋
(1 湖北省农业科学院 粮食作物研究所/湖北省甘薯工程技术研究中心/粮食作物种质创新与遗传改良湖北省重点实验室,湖北 武汉 430064; 2 长江大学 农学院,湖北 荆州 434000; 3 湖北省农业科学院,湖北 武汉 430064)
甘薯Ipomoea batatas是旋花科Convolvulaceae番薯薯Ipomoea双子叶植物,主要起源于南美洲等热带、亚热带地区,是世界第七大粮食作物、中国第五大粮食作物[1]。甘薯富含蛋白质、脂肪、多糖、维生素、胡萝卜素、花青素、氨基酸、钙、磷、钾等多种物质,营养价值丰富[2-3]。甘薯抗旱性强,耐贫瘠能力强,具有极强的适应性,在中国具有悠久的栽培历史[4]。甘薯是无性繁殖作物,染色体数目多,遗传背景复杂,在遗传上高度杂合,中国的甘薯栽培种94%以上具有‘南瑞苕’和‘胜利百号’的遗传信息,造成中国甘薯遗传背景狭窄,严重制约甘薯品种的遗传改良[5]。甘薯近缘野生种种质资源丰富,在世界范围内分布有600~700种,具有丰富的遗传多样性和潜在的基因资源,如高淀粉含量、抗病虫、抗病毒、抗旱等优良特质,可为改良甘薯品种和拓宽甘薯基因库提供重要的基因资源[6-7]。
伴随现代生物技术不断发展、更新,DNA分子标记技术在植物研究领域中广泛应用于植物种质鉴定、遗传多样性分析、指纹图谱构建、亲缘关系鉴定和遗传图谱构建等[8]。Yang等[9]使用SSR标记分析了380个甘薯品种的遗传多样性和遗传结构;苏一钧等[10]筛选出30对多态性丰富的SSR引物,对303份甘薯地方材料进行了遗传多样性和群体结构分析,发现303份甘薯材料间亲缘关系较近,遗传多样性丰富;季志仙等[11]选用6个ISSR标记对17份甘薯品种进行分析,显示同类型品种间的遗传相似性较高,亲缘关系密切。SSR和ISSR等分子标记技术主要是基于植物基因组DNA开发,在植物亲缘鉴定和遗传背景研究中具有极好的应用价值。随着现代生物技术的发展,基于线粒体DNA(Mitochondrial DNA,mtDNA)和叶绿体DNA(Chloroplast DNA,cpDNA)开发的分子标记技术也越来越广泛地应用于植物遗传分析[12-13]。
mtDNA具有细胞质遗传特性,一些mtDNA参与编码具有重要生物学功能的蛋白或亚基,如核糖体和生物氧化链上某些重要酶的部分亚基,还与细胞生命周期、真核细胞抗药性有关[14]。在植物细胞中,mtDNA参与调控子叶大小、叶片形状、植物细胞质雄性不育,还影响拟南芥、玉米和黄瓜等作物的叶绿体结构以及光合作用[15-16]。mtDNA具有进化速度快、缺少重组以及严格母系遗传等特点,且线粒体基因组较小、易于测序,已经成为研究植物遗传进化和系统发育的理想工具[17]。cpDNA具有单倍型、母系遗传、缺乏杂合细胞质、不发生等位基因重组等特性,非编码区不受选择影响,核酸位点表现出丰富的多态性和变异,与mtDNA序列广泛用于研究植物的系统发育、种间或种内的亲缘关系鉴定等研究[18]。Godbout等[19]采用nad3、nad5 等mtDNA序列对北美短叶松进行了系统发育研究,结果表明mtDNA可反映北美短叶松的地理关系;Hu等[20]使用mtDNA非编码区序列分析薯蓣属的遗传背景,结果表明线粒体基因重组时存在丰富的变异位点,可用于鉴别区分不同薯蓣品种;马丽等[21]使用matK基因序列对59个石榴品种进行了鉴定和分类,结果表明matK可用于石榴的系统发育分析。
本研究通过对3个甘薯栽培种及8个近缘野生种进行mtDNA和cpDNAmatK序列分析,综合分析3个甘薯栽培种及8个近缘野生种的亲缘关系和遗传多样性,为甘薯近缘野生种的保护及利用提供参考,为甘薯遗传育种提供一定的理论依据。
1 材料与方法
1.1 试验材料
试验所用材料为3个甘薯栽培种(‘浙紫薯3号’‘鄂薯6号’‘皖薯7号’)及8个近缘野生种(何鲁牵牛I. holubii、‘黄茑萝’I. hederifoliavar. lutea、空心菜I. aquaticaForsk、‘月光花’I.alba、‘黄色朝颜’I. obscuraKeniak、变色牵牛I.ndica、旋转牵牛I. digitata huge caudex、树牵牛I.carnea),种植于湖北省农业科学院粮食作物研究所。长势旺盛时进行样品采集。
1.2 DNA 提取
采取试验样品新鲜、幼嫩叶片后,参考Yang等[9]的方法,使用改良CTAB法提取甘薯栽培种及近缘野生种的基因组DNA。使用分光光度计测量DNA的浓度,用15 g/L琼脂糖凝胶电泳检测DNA的质量。将 DNA 质量浓度稀释到 70~80 ng/μL,在−20 ℃冰箱中保存,备用。
1.3 PCR 扩增
本研究选用6对mtDNA和1对cpDNA通用引物(表1),引物由武汉天一辉远生物科技有限公司合成。将7对引物分别在试验材料中进行扩增。PCR反应扩增体系:10× PAGE Buffer(Mg2+)2.5 μL,NTPs(10 mmol/L)2.0 μL,正向引物和反向引物各.0 μL,基因组 DNA(70~80 ng/uL)1.0 μL,EasyTaq®DNA Polymerase 0.5 μL,ddH2O 补至总体积为5 μL。PCR 扩增程序:94 ℃ 预变性 5 min;94 ℃ 变性 30 s,56~57 ℃ 退火 30 s,72 ℃ 延伸 1 min,35 个循环;72 ℃ 延伸 5 min;在 4 ℃ 条件下保存 min。使用15 g/L的琼脂糖凝胶电泳检测PCR扩增结果,以单一、清晰、明亮条带作为检测标准,将检测合格的扩增产物送到武汉天一辉远生物科技有限公司进行双向测序。

表 1 试验采用引物信息Table 1 The information of primers used in this experiment
1.4 数据分析
用软件DNAstar对测序结果进行手动校对,删除两端冗余碱基,将正向序列和反向序列进行拼接,使用软件MEGA X[25]对拼接好的序列进行比对,使用软件 DnaSP 6.0[26]对序列进行多态性 (π)和单倍型(Hd)等序列特征分析。使用MEGA X软件采用邻接距离矩阵法(Neighbor-joining distance matrix method)构建系统发育进化树,使用自展法(Bootstrap)进行可信度检测,重复1000次。使用软件NetWork的中介邻接网络(Median-joining network,MJ)算法构建单倍型关联图。
2 结果与分析
2.1 序列特征分析
对6个mtDNA序列和cpDNAmatK序列的测序结果进行多态性分析,mtDNA序列ccb256在3个甘薯栽培种及8个近缘野生种中无碱基差异,其余6个序列表现出多态性。对表现出多态性的序列进行分析,将ccb203、nad2/1-2、nad2/4-5、nad5/4-5、nad7/1-2和matK在11个材料中的扩增序列进行对位排列后,上述序列的长度分别为494、1 247、1 503、1 665、964 和 840 bp。6 个序列扩增片段GC碱基占比分别为46.52%~47.17%、53.90%~54.69%、48.02%~48.54%、45.93%~46.50%、56.39%~57.23%和32.90%~34.09%。在5个线粒体序列中,序列nad5/4-5的核酸多态性最高(0.003 95),含有20个变异位点、10个单一突变位点、10个简约信息位点、6个插入/缺失位点。ccb203、nad2/1-2、nad2/4-5和nad7/1-2序列的核苷酸多态性、变异位点数、单一突变位点数、简约信息位点数和插入/缺失位点数分别为 0.003 20、0.000 17、0.003 43、0.001 86,5、1、12、4,2、1、3、0,3、0、9、4,6、11、63、24。matK序列的核苷酸多态性、变异位点数、单一突变位点数比mtDNA序列的高,简约信息位点数和插入/缺失位点数比mtDNA序列的低。6个序列合并后,序列长度为 6 713 bp,共有变异位点 69 个,其中单一突变位点39个、简约信息位点30个,插入/缺失位点111个(表2)。

表 2 试验材料的序列多态性信息Table 2 Sequence polymorphism information of experiment materials
ccb203、nad2/1-2、nad2/4-5、nad5/4-5、nad7/1-2和matK序列中,单倍型数量最多的是cpDNA序列matK,9 个;ccb203、nad2/1-2、nad2/4-5、nad5/4-5和nad7/1-2的单倍型数量分别是5、2、8、8和5。6个序列的单倍型多样性分别为0.818、0.182、0.927、0.927、0.818 和 0.945,matK 区域的单倍型多样性最高。单倍型多样性方差和单倍型多样性标准差最大的区域是nad2/1-2,分别为0.020 61和0.144。6个序列合并后,单倍型数量、单倍型多样性、单倍型多样性方差和单倍型多样性标准差分别是 9、0.945、0.004 34 和 0.066(表 3)。

表 3 试验材料的单倍型多样性Table 3 Haplotype diversity of experiment materials
对 6 个序列进行 Tajima’sD测验和 Fu and Li’sD*/F*测验,在5个mtDNA序列中,序列nad7/1-2 的 Tajima’sD值、Fu and Li’sD*值和 Fu and Li’sF*值最大,序列nad2/1-2 的 Tajima’sD值、Fu and Li’sD*值和 Fu and Li’sF*值最小,且 5 个mtDNA 序列的 Tajima’sD值、Fu and Li’sD*值和Fu and Li’sF*值均差异不显著 (P>0.10),符合中性进化模型。cpDNAmatK 的 Tajima’sD值、Fu and Li’sD*值和 Fu and Li’sF*值分别为−1.627 84、−2.009 96 和−2.168 40,在 0.05

表 4 Tajima’s D 测验和 Fu and Li’s D*/F*测验Table 4 Tajima’s D test and Fu and Li’s D*/F* test
2.2 系统发育树分析
对3个甘薯栽培种及8个近缘野生种进行遗传多样性分析,11份试验材料间的遗传距离均在0.000 00~0.005 84 之间,‘月光花’与‘黄色朝颜’2个品种间的遗传距离最大,为0.005 84,3个栽培种间的遗传距离为0,3个栽培种及8个近缘野生种间的平均遗传距离为 0.003 26 (表 5)。
基于邻接法构建甘薯栽培种及近缘野生种的系统发育进化树,可将3个甘薯栽培种及8个近缘野生种分为2大类(Ⅰ和Ⅱ) (图1),第一大类包括7个近缘野生种,第二大类包括3个栽培种和1个近缘野生种。第一大类又可分为2类,何鲁牵牛、‘黄色朝颜’、树牵牛和旋转牵牛被归为一类,‘黄茑萝’‘月光花’和变色牵牛被归为一类;第二大类可分为2类,第一类为空心菜,第二类为‘浙紫薯3号’‘鄂薯6号’和‘皖薯7号’(图 1)。

图 1 邻接法构建系统发育进化树Fig. 1 The phylogenetic tree constructed by neighbor-joining method
2.3 单倍型中介网络
使用软件NetWork中介邻接网络算法对3个甘薯栽培种及8个近缘野生种的单倍型绘制网络图,11份材料表现出9种单倍型,图中无明显主体单倍型(图2)。单倍型H_1~H_8分别对应何鲁牵牛、‘黄茑萝’、空心菜、‘月光花’‘黄色朝颜’、变色牵牛、旋转牵牛和树牵牛,单倍型H_9对应‘紫薯3号’‘鄂薯6号’和‘皖薯7号’。单倍型之间呈线状排列,无主体单倍型,单倍型H_1与H_5有共同节点,单倍型H_3与H_8有共同节点,单倍型H_4与H_6有共同节点。中介邻接网络算法分析结果与邻接聚类分析结果一致,甘薯栽培种与野生种之间存在界限。
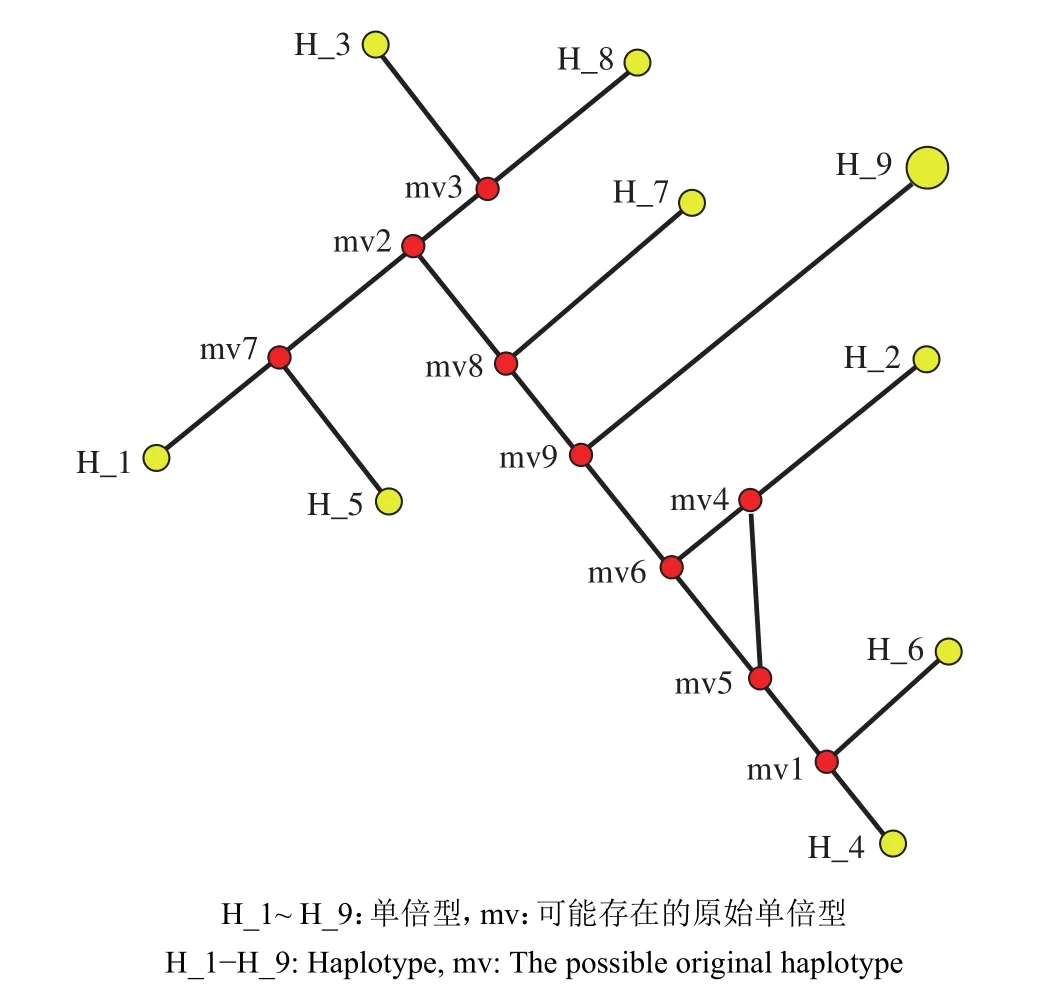
图 2 单倍型中介邻接网络图Fig. 2 Median-joining network for haplotypes
3 讨论与结论
甘薯近缘野生种是重要的育种资源,基于mtDNA和cpDNA序列特征对甘薯近缘野生种进行分析,可为甘薯育种提供新的资源。序列特征分析在甘薯及近缘野生种分析中被广泛应用,刘峥等[27]以及俞立璇等[28]使用ITS序列对甘薯栽培种和近缘野生种进行序列特征分析,显示ITS序列可用于旋花科的系统演化关系分析,为甘薯野生种的利用提供了理论依据。matK序列是叶绿体中进化速率较快的基因之一,存在一定程度的分化,由于叶绿体基因组较为保守,其进化速率远低于核基因的进化速率[29]。低进化速率限制了matK及其他叶绿体基因在低级分类层面(如属、亚属)中的应用,matK序列不能应用于甘薯属内间的亲缘关系研究。线粒体基因在系统学研究中应用较为广泛,如nad1基因应用于探讨稻族Oryzeae的系统发育[30];nad5序列应用于秦艽组植物的分子系统发育分析及物种鉴定[31];nad7/1-2序列用于分析突尼斯柑橘砧木种质资源的遗传多样性[32]。与核基因相比,线粒体基因如nad1序列在较高等级类群的系统发育重建中有更高的信息量,变异水平更适于在高等级层面的系统发育重建。相较于核基因和叶绿体基因,线粒体基因变异速率低,结构不稳定,但筛选出合适的线粒体序列片段仍能为植物的系统学研究和亲缘关系鉴定提供新的证据。在植物系统学研究中,采用mtDNA对甘薯近缘野生种进行分析的报道较少,通过mtDNA和cpDNA对甘薯近缘野生种进行分析,可为研究旋花科的系统演化提供新的方法和指导。
本研究选用6个mtDNA序列和1个cpDNA序列的引物在3个甘薯栽培种及8个近缘野生种中进行扩增,结果显示,在11个材料中,mtDNA序列ccb256无多态性,表现出多态性的序列占比为85.7%,表明基于mtDNA和cpDNA开发的通用引物在甘薯栽培种及近缘野生种的多态性分析中具有良好的多态性。具有多态性的5个mtDNA序列中,nad2/1-2和nad7/1-2序列中的GC占比大于AT占比,ccb203、nad2/4-5和nad5/4-5序列中的GC占比小于AT占比,表明nad2/1-2和nad7/1-2序列更为稳定。matK序列的GC占比小于AT占比,matK序列的核苷酸多样性高于mtDNA序列的,与Yang等[33]基于核基因组和叶绿体基因组分析中国橡树的试验结果基本保持一致。对mtDNA序列的变异位点进行分析,产生的变异位点以单一突变位点和插入/缺失突变为主,序列nad5/4-5的单一突变位点最多(10个),序列nad2/4-5插入/缺失位点在6个序列中最多(63个)。与mtDNA序列相比,cpDNA序列matK的单一突变位点(23个)比mtDNA序列的多,而插入/缺失位点(1个)远少于mtDNA序列的。对6个序列的单倍型多样性进行分析,mtDNA序列中nad2/1-2序列的单倍型数量和单倍型多样性最小(2、0.182)。matK序列的单倍型数量(9)和单倍型多样性(0.945)比5个mtDNA序列的高,表明相较于mtDNA序列,cpDNA序列反映的序列特征信息更为丰富。对甘薯近缘野生种进行分析时,mtDNA和cpDNA序列结合获得的结果更为准确。
中性检验(Neutrality test)是通过统计学的检验方法分析DNA数据,进而检验自然选择[34]。本研究采用 Tajima’sD和 Fu and Li’sD*/F*检验,结果显示基于 mtDNA 序列分析的 Tajima’sD值、Fu and Li’sD*值和 Fu and Li’sF*值均差异不显著(P>0.10),符合中性进化模型;matK序列的Tajima’sD值、Fu and Li’sD*值和 Fu and Li’sF*在0.05
3个甘薯栽培种及8个近缘野生种的mtDNA和cpDNA序列分析显示,mtDNA序列和cpDNA序列可用于甘薯栽培种及近缘野生种的遗传多样性和亲缘关系研究,为旋花科植物的分类和系统发育研究提供指导,还可为甘薯种质资源的开发利用和筛选提供理论依据。
