脑神经胶质瘤替莫唑胺耐药circRNA-miRNA-mRNA调控网络的构建
2021-07-07朱琳
朱琳
脑神经胶质瘤是中枢神经系统最常见的原发 性恶性肿瘤。目前,最大限度的手术切除,术后辅以放化疗是神经胶质瘤治疗的主要方法。其中替莫唑胺(Temozolomide,TMZ)在脑胶质瘤,尤其是神经胶质母细胞瘤(WHOⅣ)治疗上取得了成功,其明显降低了术后复发率,大大提高了脑胶质瘤的生存率。目前替莫唑胺是临床治疗脑神经胶质瘤的一线化疗药物。但随着用药周期的延长,几乎所有初始治疗有效的病人都会出现病情进展,产生替莫唑胺耐药(Temozolomide Resistance,TR)。其耐药机制尚未完全明确。
环状RNA(Circular RNA,circRNA)是一类新型的内源性非编码RNA(noncoding RNA,ncRNA),其特征是3'和5'端以闭环结构共价连接,因此circRNA不易受核酸内切酶的影响,在体内表达稳定,是目前较为理想的疾病诊断生物标志物,以及分子生物治疗靶点。circRNA可通过多种途径发挥肿瘤调控作用,但目前研究较为清楚的是其微小RNA(microRNA,miRNA)海绵作用,circRNA通过miRNA海绵竞争性的与靶miRNA结合,使miRNA降解或者失去调控功能,进而调控miRNA下游靶基因的表达水平。目前研究表明,circRNA在非小细胞肺癌、结直肠癌、乳腺癌、肝癌、胃癌等多种肿瘤中异常表达,而且环状RNA(circRNA)-微小RNA(miRNA)-信使RNA(mRNA)调控网络也参与了多种肿瘤的发生、发展、放疗抵抗,以及化疗耐药等过程。但目前circRNA以及circRNA-miRNA-mRNA调控网络在脑神经胶质瘤替莫唑胺中研究较少。
因此,本研究以人脑神经胶质瘤替莫唑胺耐药细胞株U87/TR以及其亲本细胞株U87为细胞模型,通过高通量circRNA芯片比较其circRNAs表达谱差异,并初步筛选关键circRNAs,并预测circRNAmiRNA-mRNA调控网络,为进一步深入研究circRNA在脑神经胶质瘤替莫唑胺耐药中的作用及其分子机制提供依据。本研究符合《世界医学协会赫尔辛基宣言》相关要求。
1 材料与方法
1.1 主要材料试剂
人脑神经胶质瘤替莫唑胺耐药U87/TR细胞株以及其亲本U87细胞株购买于上海弘顺生物科技有限公司(规格BW124577)。RPMI-1640培养液、胎牛血清、胰蛋白酶购自美国GIBCO公司。RNeasy Mini Kit购自德国Qiagen公司,RT-PCR试剂盒等购自美国Sigma公司。circRNA芯片的制备与杂交由上海吉凯生物科技公司完成。1.2 方法
1.2.1 circRNA芯片检测及数据分析
用RNeasy Mini Kit(Qiagen,Hilden,德国)提取人脑神经胶质瘤替莫唑胺耐药U87/TR细胞株以及其亲本U87细胞株总RNA(n=3)。使用NanoDrop ND-1000量化每个样品的总RNA。样品制备和微阵列杂交是根据美国Arraystar公司的标准方案进行的。总RNA用Rnase R消化以除去线性RNA并富集环状RNA。然后,将富集的环状RNA扩增并转录为荧光cRNA(Arraystar超级RNA标记试剂盒;Arraystar)。标记的cDNA(complementary DNA)探针与美国Arraystar人源性环形RNA芯片2.0版Arraystar Human circRNA Array V2(8x15K,Arraystar)杂交。使用安捷伦功能提取软件(版本11.0.1.1)来分析获取的阵列图像。使用R软件Limma软件包执行分位数归一化和后续数据处理。通过火山图(Volcano Plot)过滤鉴定了两组之间表达circRNA差异有统计学意义。通过倍数变化过滤鉴定了两个样品之间差异表达的circRNA。进行了层次聚类,以显示样品之间可区别的circRNA表达模式。1.2.2 RT-PCR芯片结果验证
将circRNA芯片分析结果中差异表达倍数及信号值较高的4个circRNAs行RT-PCR分析,分布为上调的circRNAs:人环 状 RNAhsa_circRNA_009054,hsa_circRNA_104338;和下调的circRNAs:hsa_circRNA_016459,hsa_circRNA_101975,进一步验证芯片检测结果的可靠性。利用软件Primer Premier 5.0设计引物(表1)。以U87和U87/TR细胞总RNA 2μg为模板,进行逆转录,以GAPDH作为内参照。按照试剂盒操作进行PCR反应,条件为:95℃30 s;95℃5 s,60℃31 s,扩增40个循环。以2表示circRNA的表达水平,实验重复3次。
表1 引物设计
1.2.3 生物信息学分析
运用Miranda v5数据库(http://www.ebi.ac.uk/enright-srv/microcosiTi/htdocs/targets/v5)预测circRNA-miRNA结合关系以及结合位点。miRNA靶基因预测软件:miRanda v5(http://www.ebi.ac.uk/enright-srv/microcosiTi/htdo/targets/v5),TargetScan(http://www.targetscan.org)和mi-Base(http://pictar.bio.nyu.edu)。circRNA-miRNAmRNA调控网络通过Cytoscape软件进行可视化(Version 3.6.1:http://cytoscape.org/)。1.3 统计学方法
应用SPSS 18.0进行统计学分析,两组细胞间各circRNAs水平差异分析采用配对样本t
检验,P
<0.05为差异有统计学意义。2 结果
2.1 circRNA差异表达谱
circRNA芯片数据经过标准化处理和统计学分析,结果显示,与亲本U87细胞株相比,在U87/TR细胞株中,共鉴定出313个差异有统计学意义表达的circRNAs,其中包括145个明显上调的circRNA和168个明显下调的circRNA,成功建立神经胶质瘤替莫唑胺耐药相关circRNA差异表达谱。层次聚类分析筛选显示了显著上调的下调的前10位circRNAs。表2,3列出了显著上调和下调的前10位circRNAs详细信息。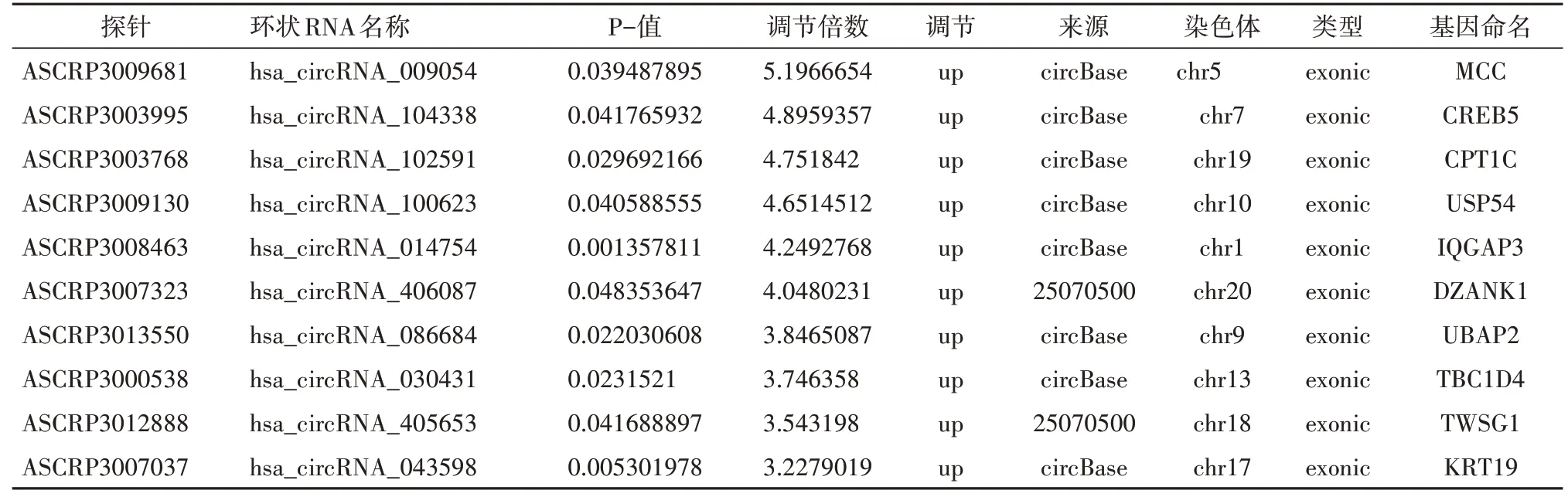
表2 在U87/TR细胞株中鉴定出较亲本U87细胞株上调的前10位circRNAs
2.2 RT-PCR验证芯片结果
为了进一步证实circRNA芯片的准确性,我们将circRNA芯片分析结果中差异表达倍数及信号值较高的4个关键circRNAs行RT-PCR分析验证(上下调circRNAs各2个),RTPCR结果显示,相比较于U87细胞,hsa_circRNA_009054和hsa_circRNA_104338在U87/TR细胞中显著高表达(n
=3,t
=12.09、9.52,P
=0.007、0.011),hsa_circRNA_016459和hsa_circRNA_101975在U87/TR细胞中明显低表达(n
=3,t
=13.86、7.75,P
=0.005、0.016),而且RT-qPCR结果与circRNA芯片结果相符(表4),证实了circRNA芯片的准确性。从而鉴定了hsa_circRNA_009054,hsa_circRNA_104338(上调)以 及hsa_circRNA_016459,hsa_circRNA_101975(下调)4个神经胶质瘤替莫唑胺耐药相关的关键circRNAs。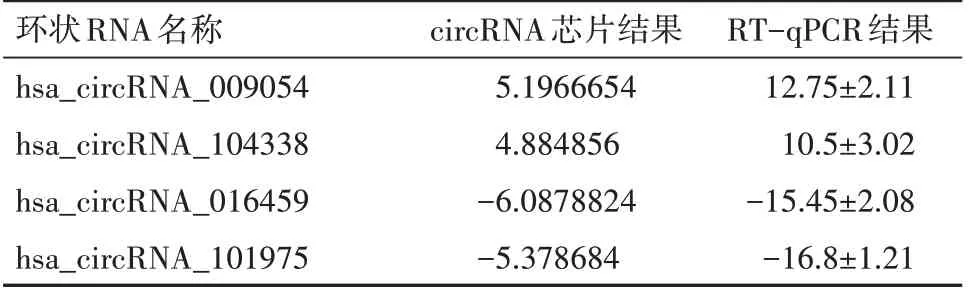
表4 RT-PCR验证结果与circRNA芯片检测结果对比
2.3 circRNA-miRNA调控网络的构建
同时,我们通过miRanda v5数据库对鉴定的关键circRNAs进行miRNA靶位点预测。miRNA靶位点预测结果显示,hsa_circRNA_009054,hsa_circRNA_104338以及hsa_circRNA_016459,hsa_circRNA_101975均可与多个miRNAs靶向结合,hsa_circRNA_009054排名前五位的miRNA分别为:hsa-miR-33a-5p,hsamiR-4685-5p,hsa-miR-6855-5p,hsa-miR-2467-5p,hsa-miR-4446-3p;hsa_circRNA_009054排名前五位的miRNA分别为:hsa-miR-15b-3p,hsa-miR-550a-5p,hsa-miR-509-5p,hsa-miR-1224-3p,hsa-miR-758-5p;hsa_circRNA_009054排名前五位的miRNA分 别 为:hsa-miR-193b-5p,hsa-miR-134-5p,hsamiR-4439,hsa-miR-6820-5p,hsa-miR-5190;hsa_circRNA_009054排名前五位的miRNA分别为:hsa-miR-23a-5p,hsa-miR-423-5p,hsa-miR-875-3p,hsamiR-877-3p,hsa-miR-30b-3p。通过生物信息学分析,我们进一步预测了hsa_circRNA_009054,hsa_circRNA_104338以及hsa_circRNA_016459,hsa_circRNA_101975与其排名第一位的靶向miRNA的具体结合位点(分别为:hsa-miR-33a-5p,hsamiR-15b-3p以 及hsa-miR-193b-5p,hsa-miR-23a-5p),见图1。从而鉴定了hsa-miR-33a-5p,hsa-miR-15b-3p以及hsa-miR-193b-5p,hsa-miR-23a-5p 4个神经胶质瘤替莫唑胺耐药相关的关键miRNAs,构建了神经胶质瘤替莫唑胺耐药相关的circRNA-miRNA调控网络。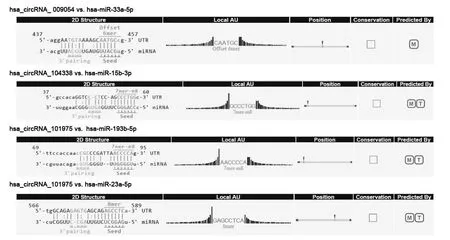
图1 关键circRNAs与关键miRNAs靶向结合位点的详细注释图

表3 在U87/TR细胞株中鉴定出较亲本U87细胞株下调的前10位circRNAs
2.4 circRNA-miRNA-mRNAs调控网络的构建
进一步我们通过miRanda v5、TargetScan、miBase数据库对关键miRNAs.进行靶基因预测,并通过Cytoscape软件对circRNA-miRNA-mRNA调控网络进行可视化分析。从而构建了神经胶质瘤替莫唑胺耐药相关的circRNA-miRNA-mRNAs调控网络,我们预测的circRNA-miRNA-mRNAs调控网络,包括4个关键circRNAs,20个关键miRNAs,以及278个关键mRNAs。这些关键circRNAs,miRNAs,以及mRNAs通过网络互相作用,影响神经胶质瘤替莫唑胺耐药过程。3 讨论
脑神经胶质瘤替莫唑胺耐药仍是临床亟待解决的关键问题。目前研究认为,DNA损伤修复机制,如O-甲基鸟嘌呤-DNA-甲基转移酶(MGMT),DNA错配修复(mismatch repair,MMR),碱基切除修复(base excision repair,BER);肿瘤微环境;肿瘤细胞自噬;p53、EGFR、PI3K/AKT、JAK2/STAT3等信号通路等异常激活与失活、非编码RNA调控均可能与脑神经胶质瘤替莫唑胺耐药相关,但其具体机制尚未完全阐述。其中非编码RNA调控与脑神经胶质瘤替莫唑胺耐药的相关性越来越受到重视。
circRNAs表达较线性RNA稳定,而且在体内的表达量是线性RNA的10倍以上。研究表明,circRNA的异常表达与调控作用与肿瘤的发生发展密切相关,而circRNA-miRNA-mRNA调控网络成为肿瘤研究的重要方向。在非小细胞肺癌中,有研究 表 明,hsa_circ_0087862可 通 过 靶 向miR-1253/RAB3D轴在非小细胞肺癌中发挥癌基因作用,circ_FGFR1可通过miR-381-3p促进在非小细胞肺癌细胞的进展以及PD-1耐药。在乳腺癌中,有研究 发 现,Hsa_circ_0091074可 通 过microRNA-1297促进三阴性乳腺癌细胞的进展,circ-ABCB10可通过Let-7a-5p/DUSP7轴促进乳腺癌的紫杉醇耐药。在胃癌中,有报道称,circRNA_100876可通过抑制miR-136上调MIEN1的表达增强胃癌的增殖和转移能力,circ_CCDC66可通过miR-618/BCL2轴促进胃癌的顺铂耐药。这些研究表明circRNA以及circRNA-miRNA-mRNA调控网络参与了多种肿瘤的发生发展,以及耐药等演进过程。但目前circRNA以及circRNA-miRNA-mRNA调控网络在脑神经胶质瘤替莫唑胺中未见相关研究。
在我们研究中,我们选取了人脑神经胶质瘤替莫唑胺耐药U87/TR细胞株以及其亲本U87细胞株为细胞模型,通过高通量circRNA芯片筛选其circRNAs表达谱,结果发现俩细胞株之间存在313个差异有统计学意义表达的circRNAs,其中包括145个明显上调的circRNA和168个明显下调的circRNA,说明脑神经胶质瘤替莫唑胺耐药伴随着circRNA的改变,为筛选关键circRNAs,我们选取上下调差异倍数最高的两个circRNA进行PR-PCR验证,其结果与芯片相符,我们通过miRNA预测,以及靶基因预测,构建了circRNA-miRNA-mRNA调控网络,这个包括hsa_circRNA_009054,hsa_circRNA_104338 和 hsa_circRNA_016459,hsa_circRNA_101975 4个 关 键circRNAs,hsa-miR-33a-5p,hsamiR-15b-3p以及hsa-miR-193b-5p,hsa-miR-23a-5p等20个关键miRNAs,以及THOC2,TP53,STAT5B,RORC等与肿瘤发生发展密切相关278个关键mRNAs。这些关键circRNAs,miRNAs,以及mRNAs可能通过网络互相作用,影响神经胶质瘤替莫唑胺耐药过程。但目前的研究仍无法直接证明circRNAs对脑神经胶质瘤替莫唑胺耐药的调控作用,接下来,我们课题组将向U87/TR细胞株转染关键circRNA过表达和干扰质粒,建立过表达或干扰circRNA细胞模型,鉴定circRNA对U87/TR细胞株生物学行为的影响,明确circRNA下游的具体的miRNA-mRNA调控网络及其机制。
综上所述,我们的研究构建了替莫唑胺耐药相关的关键circRNA-miRNA-mRNA调控网络,为深入研究circRNA在脑神经胶质瘤替莫唑胺耐药中的作用及其分子机制提供了实验依据和前期基础。
