MADS-box基因家族调控植物花器官发育研究进展
2021-06-30穆艳霞
王 莹,穆艳霞,王 锦
(西南林业大学 园林园艺学院,云南 昆明 650224)
花器官是被子植物生殖繁育的重要器官,也是植物最为多样化的器官之一。因生长环境和繁衍策略不同,各类植物的花器官在进化中产生多样化,从而最大限度确保繁殖成功。虽然部分植物花型较特殊,如兰科植物、金鱼草等,但大多数花器官具有典型的四轮花部结构,即从外向中心依次为花萼、花瓣、雄蕊、雌蕊(心皮)。花器官是研究植物进化、分类的重要依据,一直以来都是被关注的热点。随着第一个与花发育相关的MADS-box基因从模式植物拟南芥中被克隆,花发育调控和相关基因的研究有了深入推进。分子水平的研究显示,花器官的启动、分化与建成是各类基因转录调控、相互作用共同完成的联合机制,在该机制下MADS-box基因家族起着关键调控作用[1]。
MADS-box基因编码的转录因子广泛存在于所有真核生物中[2],影响着植物各个器官的形态发生与生长发育,如根系、果实的生长调控[3-4]等,对于MADS-box基因的深入研究是由于其在花器官中的重要功能而引起。MADS得名于酵母的MCMI(Minichromosomemaintenancegene)基因、拟南芥的AGAMOUS(AG)基因、金鱼草的DEFICIENCE(DEF)基因和人类血清应答因子SRF(Serumresponsefactor),这些基因均在N端含有一个约180 bp的MADS-box保守区域,同时编码相同的DNA结合域并识别相似的目标DNA序列[5]。目前对MADS-box基因在植物体内的功能研究较为全面,在模式植物拟南芥、金鱼草、矮牵牛、水稻和玉米中发现并验证了大量MADS-box基因及其功能[6];但不同植物中MADS-box基因的表达模式并非完全相同,结合同源基因功能探索不同种类植物花器官发育的模式系统仍需大量研究工作。本文通过总结近年来MADS-box基因对植物花器官发生发育调控的研究进展,对其花发育调控网络机制进行梳理,以便进一步开展对MADS-box同源基因的挖掘与蛋白功能分析,为完善花发育调控网络和探索不同植物花发育模式系统奠定基础。
1 MADS-box基因分类与结构
早期对MADS-box基因系统发育的分析根据基因功能将其分为不同亚家族,随后通过拟南芥基因组序列比对,确定该家族基因主要演化支之间的系统发育关系,并确认在植物、动物和真菌中是由1个祖先复制产生2个谱系(Type Ⅰ和Type Ⅱ)[7]。植物中的Type Ⅰ型基因与动物中的SRF-like基因聚为一支,其基因结构显示只含有1~2个外显子,编码蛋白具有1个核心保守的MADS-domain和1个扩展而又高度可变的Carboxy-terminal domain[8]。因Type Ⅰ型基因的结构特点和功能重要性,越来越多研究学者开展了对其的研究[9]。从拟南芥中克隆到的Ⅰ型基因包括AGL23、AGL28、AGL61、AGL62、AGL37、AGL80等,这些基因作用于拟南芥的胚乳,对雌配子体和胚胎的发生发育起重要作用[10-11]。在毛竹中发现6个Type Ⅰ型基因对花序发育起重要作用,其中PeMADS5在拟南芥中异位表达可导致早花现象[12]。
被子植物中发现的MADS-box基因多属于Type Ⅱ型基因。Type Ⅱ型分为MEF2型和MIKC型2个分支,MEF2型主要存在于动物和真菌中;MIKC型为植物所特有,包括6个内含子和7个外显子,编码蛋白由MADS-domain、I-domain(intervening domain)、K-domain(keratin-like domain)和C-terminal domain 4个域组成(图1)。K-domain是MIKC型的特征序列,C-terminal domain是最不保守区域,不同的C结构可能导致MADS-box蛋白功能不同。研究表明,I和K结构域参与MADS-box转录因子的蛋白互作,C结构域作为转录激活域可能对稳定蛋白互作起作用[13-14]。MIKC型进一步分为MIKCC型和MIKC*型,其中,MIKCC型占大多数,包括所有已知表达模式或突变表型的植物MADS-box基因[15](表1)。

图1 MADS-box 蛋白分类与结构[7,8,13-14]Fig.1 Classification and structure of MADS-box proteins

表1 MADS-box基因及其功能[2,14,16-18]
2 MADS-box基因家族调控花器官发育
从细胞学和解剖学分析,花发育起始于分生组织细胞的转变,这种转变由一系列内源因素和环境因素共同诱导产生,由此启动开花,细胞从茎尖分生组织(shoot apical meristem,SAM)转变为花序分生组织(inflorescence meristem,IM),进而花序分生组织周边产生花分生组织(floral meristem)分化成各轮器官;单子叶植物中花序分生组织先产生穗分生组织,再分化花分生组织[2,19]。这一系列过程由开花整合因子(FT、SOC1)、花原基响应特征基因(AP1、LFY、CAL、FUL、AGL24),以及花器官特征基因(ABCDE类基因)作为主要调控因子联合其他因子共同完成。花器官形态构成由多条反应通路和众多家族基因共同调控,少数影响较大的因子决定表型差异。MADS-box基因作为关键调控因子,不同水平的基因表达,会特化成不同形态结构。当然,该类基因编码转录因子,通过与DNA结合或蛋白间互作对花结构形成也起到不可忽视的作用。此外,MADS-box基因下游靶基因的响应同样导致花器官多样化的发生。
2.1 开花启动中MADS-box基因的作用
开花启动的本质是信号传导与植物细胞特性转变。目前确定存在6条开花信号途径,包括光周期途径、春化途径、自主途径、赤霉素途径、温敏途径和年龄途径[2],这些途径感知到开花信号并进行传导,通过调控两类作用相反的MADS-box基因启动开花,一类是开花强抑制因子FLOWERINGLOCUSC(FLC)和SHORTVEGETATIVEPHASE(SVP),另一类是调控花芽分化的开花整合因子和花原基特征基因。春化途径、自主途径和温敏途径主要通过转录后调控、组蛋白修饰的表观遗传调控抑制FLC和SVP基因的表达,以解除开花抑制状态。FLC是MADS-box基因家族成员,可以直接抑制开花整合因子FT和SOC1的转录表达抑制开花[20-21]。开花前FLC的本底转录水平较高,接收开花启动信号后,一些基因促进FLC反义mRNA的表达,导致其正义mRNA的表达减弱,同时染色质修饰复合物通过修饰组蛋白改变FLC染色质区域状态,达到抑制FLC表达的目的[22-23]。在温敏途径中,高温环境促进FT表达量上升,SVP则抑制表达,它能与FLC相互作用负调控开花整合因子的转录活性[24],进而影响开花,但目前具体分子机制尚不清楚。有趣的是,SVP与花原基特征基因AGL24同为MADS-box基因家族SVP/AGAMOUS-LIKE谱系的旁系同源基因,SVP抑制花器官形成,AGL24却是开花激活子[25]。单子叶植物小麦中发现SVP同源基因TaVRT2,它与TaVRN1是春化途径中的开花启动子,TaVRT2蛋白可以直接与TaVRN1的启动子结合,介导TaVRN1正反馈环[18,26],这与双子叶植物中SVP调控不相同,因此SVP及其同源基因对花器官形成的调控作用仍需进一步研究。光周期途径、赤霉素途径和年龄途径中根据信号直接或间接激活FT、SOC1、AP1、AGL24等基因的表达以启动开花。
2.2 MADS-box基因调控细胞分化
开花启动的6大信号途径汇聚于2个关键开花整合因子,即FLOWERINGLOCUST(FT)和SUPPRESSOROFOVEREXPRESSIONOFCONSTANS1(SOC1)。FT不是MADS-box基因,而是一种可移动蛋白,被长距离运输至茎顶端激活下游关键基因AP1的表达,引发花分生组织在花序分生组织的特定位置产生[27]。SOC1可以将多种途径的开花信号进行整合,并与AGL24转录因子相互结合到对方启动子上,形成基因转录调节的自我反馈环,SOC1与AGL24还可形成异源二聚体,协同调节LFY基因的表达[28]。茎尖分生组织内SOC1和其他因子上调引发营养生长向花序分生组织特性的转变[29]。
花分生组织的产生需要花原基特征基因(AP1、LFY、CAL、FUL、AGL24)的决定性作用,这些基因除LFY外均为MADS-box基因。APETALA1(AP1)与CAULIFLOWER(CAL)为旁系同源基因,它们的高表达决定着花分生组织的分化。在拟南芥中,ap1-cal双突变体在单花位置上产生大量芽增殖,也被称为“花椰菜表型”,花序分生组织不能分化成花分生组织[30]。虽然FRUITFULL(FUL)只在花序分生组织中表达,不在花分生组织表达,但其对花分生组织的起始分化有重要作用,拟南芥中FUL与AP1对花分生组织的决定性存在功能冗余[31]。
MADS-box转录因子通过协调各类分生组织中特征基因的表达变化来控制花发育的进程与开关,建立新发育程序的同时抑制先前的发育程序。从营养生长到花序分生组织的转变由开花整合因子控制,它整合不同途径的信号输入并诱导转变;随后开花整合因子SOC1等激活花分生组织特征基因的表达,在花序分生组织侧面形成花分生组织并抑制花序分生组织特征基因的表达[16]。有研究表明,SOC1、AGL24和AGL15与染色质复合物相互作用以防止花器官特征基因SEP3被过早激活,从而抑制花分生组织细胞的过早分化[32];AP1建立花分生组织后逐渐下调前期发育程序并下调自身激活子和SOC1[33],当然它也上调基因,如LFY等,共同作用于花分生组织的器官特化;之后AP1与MADS-box基因家族的E类蛋白SEP3形成二聚体,激活参与花器官特化的基因表达。
2.3 MADS-box基因调控花器官建成
花分生组织细胞增殖和扩大,经花器官特征基因(ABCDE类基因)的调控在原基侧翼按时间顺序依次从外向内形成不同花器官。花器官特征基因除A类基因的AP2是AP2/ERF家族外,均为MADS-box基因。对大多数基因而言,它们的表达区反映功能区,A类基因在花萼与花瓣中表达,单独调控花萼形成,与B类基因协同调控花瓣发育;B类与C类基因在雄蕊中共同表达调控发育分化,C类与D类基因调控心皮与胚珠的形成。A类基因与C类基因具有拮抗作用,相互限制在花原基的表达位置,从而对花器官的边界进行限制[34];也就是说当A类基因突变后,C类基因会在1、2轮的花萼和花瓣中表达,反之亦然,这是经典的“花发育ABC模型”理论(图2-a)。据此分析单子叶植物小花结构与双子叶植物花器官的对应关系,为内外稃片与萼片同源、浆片与花瓣同源提供证据[17]。基因的异位表达可能导致花器官结构的改变,如拟南芥三类突变体,而自然界花器官构造千差万别,单子叶百合科植物第1轮花萼瓣化源于B类基因在外三轮中异位表达(图2-b),降低B类基因表达量影响花被特征形成[35]。在一些低等真双子叶植物中,如毛茛纲中,B类基因只在部分花瓣中表达[36](图2-c)。E类基因在所有花器官形成中功能冗余,如拟南芥的E类基因SEP,sep1-sep2-sep3三突变体只有花萼,SEP4在形成花萼中与其他3个SEP基因功能冗余,同时参与其他花结构发育,sep四突变体花结构整体变为具有心皮状特征的叶状结构。不同物种中,E类同源基因产生不同的亚功能化和新功能化,梅花SEP1和SEP4在花分生组织和萼片中表达,与拟南芥相比,分生组织中缺少SEP2,萼片中多了SEP1,SEP2、SPE3对于梅花瓣、雄蕊和雌蕊的形成都非常重要[37]。由此可见,MADS-box基因家族的花器官特征基因表达模式在不同植物中有所差异,拟南芥、金鱼草等模式植物作为普遍性代表可以帮助理解花形成过程,进一步发掘更多MADS-box基因在不同类型花器官中的表达模式和功能作用将有助于完善花发育理论。
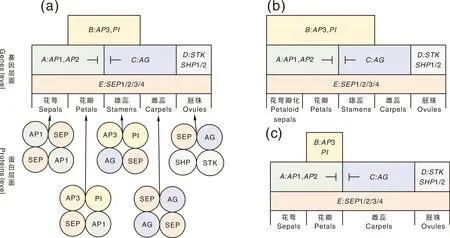
a,不同层面阐释花发育模型:基因层面为“ABC(D)E模型”——花萼:A类基因调控;花瓣:A+B类基因调控;雄蕊:B+C类基因调控;雌蕊:C类基因单独调控;胚珠:D类基因调控(包括部分C类基因调控);E类基因对所有花器官形成起辅助调控。蛋白层面为“四聚体模型”,由同源或异源二聚体蛋白再聚合形成四聚体——花萼:AP1-SEP-AP1-SEP;花瓣:AP3-PI-SEP-AP1;雄蕊:AP3-PI-SEP-AG;雌蕊:AG-SEP-AG-SEP;胚珠:AG-SEP-SHP-STK。b,单子叶百合科中B类基因覆盖前3轮,导致花萼瓣化。c,毛茛纲部分植物中B类基因只在花瓣中表达。(注: AP2基因不属于MADS-box基因,其他ABCDE类基因均为MADS-box基因。)a,Interpreted floral development models at different levels. “The ABC(D)E model” was at genes level. Sepals: A-class genes alone specified; Petals: A-and B-class genes combined to specified; Stamens: B-and C-class genes combined to specified; Carpels: C-class genes alone specified; Ovules: D-class genes specified (partially involved C-class genes); E-class genes were required to co-regulated the formation of floral organs. “The quartet model” was at proteins level. Tetramers were formed by homodimer or heterodimer. Sepals: AP1-SEP-AP1-SEP; Petals: AP3-PI-SEP-AP1; Stamens: AP3-PI-SEP-AG; Carpels: AG-SEP-AG-SEP; Ovules: AG-SEP-SHP-STK. b,Petaloid sepals in Liliaceae of monocotyledon resulted as B-class gene covered the first three whorls. c,B-class genes were expressed only in parts of the petals in some Ranunculus. AP2 gene was not a MADS-box gene,the other ABCDE genes were all MADS-box genes.图2 花器官发育模型[34-36]Fig.2 Floral organ development models
MADS-box基因的表达量调节花器官形态建成,在生化层面的调控即转录因子编码的蛋白与目标DNA序列相结合或与蛋白结合产生互作效应。MADS-box转录因子在植物中调控复杂基因,需要高度特异性靶向不同目标基因。所有MADS-box转录因子会以二聚体形式识别DNA上的CArG(CC[A/T]6GG)motif,其中MIKCC型MADS-box基因的蛋白二聚体可在两个位点与DNA结合,再以形成四聚体的方式使DNA成环并改变其结构,抑制或激活靶基因表达,这种调控形式具有植物特异性[8]。以B类基因蛋白为例,AP3和PI蛋白仅以异源二聚体形式结合DNA,它们在一个自调节反馈环中以自身启动子维持转录[38]。酵母双杂交实验显示,B类同源蛋白DEF和GLO形成的异源二聚体具有很强的DNA结合活性。而E类基因编码蛋白通过形成复合物作为辅助因子帮助激活ABC基因,并与这些基因的同源或异源二聚体蛋白形成四聚体,发挥调控花器官形成的功能,这是四聚体模型的调控机制[39](图2-a)。不同MADS-box蛋白激活靶基因转录的能力不同,AP3-PI异源蛋白二聚体在AP3启动子下与CArG-box结合并不能激活转录,需与AP1或SEP3蛋白形成三聚体才可激活AP3基因启动子[40]。
2.4 MADS-box基因调控下游靶基因
近年来,MADS-box转录因子调控下游靶基因的研究进展迅猛,其通过激活下游靶基因诱导花器官形成。首个鉴定的MADS-box蛋白靶基因是NAC-LIKEACTIVATEDBYAP3/PI(NAP)。NAP编码植物特有的NAC家族蛋白,在花瓣和雄蕊发育过程中,由AP3-PI诱导并控制细胞从分裂到伸长的过渡[41];同时随着花器官形成和果实的成熟、衰老,NAP类基因的表达量上调,推测其与MADS-box基因相互作用以调控花到果实的发育转变[42]。雄蕊与心皮边界的限制需要SUPERMAN(SUP)基因,AP3、PI和AG基因可调控SUP在这两轮花器官边界的表达,其调控机制目前尚未明确;但有研究提出,SUP也可以抑制B类基因AP3、PI在第4轮心皮中表达,同时可以平衡第3、4轮花器官的细胞增殖[43]。AG蛋白可与SPOROCYTELESS(SPL)基因3’UTR区的CArG-box类似序列结合而激活SPL转录;因此,SPL作为AG的直接靶基因编码转录因子调控胚珠模式和早期小孢子体的发生[44]。水稻E类蛋白OsMADS8的靶基因是编码bZIP转录因子的OsTGA10,该基因在雄蕊发育期间优先表达,突变体会产生雄性不育。OsTGA10对绒毡层发育极为重要,可通过与已知绒毡层基因互作发挥作用[45]。研究MADS-box转录因子下游靶基因可以更全面地了解花发育调控网络,通过基因芯片技术找到47个AP3和PI的靶基因,其中11个主要或仅在花中表达。在耧斗菜中找到7 049个AP3-3的直接靶基因[40]。MADS-box转录因子下游靶基因的类型、作用对综合理解花器官发育与多样化机制极为重要,随着生物技术的进步,从酵母双杂交到染色质免疫共沉淀,发现更多MADS-box转录因子的靶标基因将有助于完善花发育调控网络。
3 讨论与展望
花器官发生发育研究的几十年间,研究学者从细胞、分子和基因层面探索花器官构造形成的内在机制,基因表达研究并不优于形态学或解剖学,基因研究的大量数据将有助于解决隐藏在表型下的相关问题;通过基因水平研究人们对于花发育的调控网络已有基础框架(图3),并在不断完善和发掘更多调控节点,在这些节点当中MADS-box基因家族作为主要调控因子备受关注。

→表示调控具有促进作用;─┤表示调控具有抑制作用;─表示已经研究确定的调控路径;表示未确定的调控路径;*表示该基因不是MADS-box基因;未标记的基本均为MADS-box基因。→indicated acceleration; ─┤indicated inhibition; ─ represent this regulatory path has been studied; represent an unspecified regulatory path; * meant the gene was not a MADS-box gene; Genes unmarked were all MADS-box genes.图3 MADS-box基因调控花发育网络、花发育过程和相关基因[14,20,21,28,43-45]Fig.3 MADS-box genes regulate floral development network,floral development processes and related genes
随着测序技术和生物信息学的发展,越来越多植物通过基因组或转录组测序得到深入研究,围绕不同物种花器官,MADS-box基因家族众多成员及其功能被一一揭示。如欧洲白桦中BpAP1基因过量表达可引起早花现象[46];兰科植物蝴蝶兰B类基因PhalPI对侧瓣和唇瓣发育起重要调控作用[47];青钱柳花发育雌雄异熟研究显示,CpAG与GA协同作用打破花芽休眠,通过GA途径调控花发生[48];菊花中异位表达向日葵C类基因HAM59,转基因株系显示雄性不育与花结构转变,花盘内小花雄蕊转化成花瓣状结构形成重瓣花表型[49]。这些研究仅揭开MADS-box基因家族冰山一角,还有大量成员等待发掘,未来研究无疑将继续提高人们对花器官发生发育复杂调控的认知,并加强对MADS-box基因家族更高层次功能的理解,即MADS-box转录因子形成蛋白复合物如何通过表观遗传修饰调控靶基因活性,这也是后续研究MADS-box基因家族的重点之一。
通过拟南芥基因组序列比对,MADS-box基因家族主要演化支间的系统发育关系得到确认[7],在不同植物物种中测定该类基因有助于进一步评价植物进化关系。从松柏科植物[50-51]和买麻藤目(Gnetales)[52]中克隆出MADS-boxB类和C类基因,研究显示这些基因存在于被子植物和裸子植物最后一个祖先中,它们的同源基因在生殖结构中表达可能早于两个类群分化(约3亿a前)[38]。虽然裸子植物和被子植物生殖器官同源性尚不明确,裸子植物MADS-box基因将是进一步研究系统进化的有力工具。此外,MADS-box基因在较远距离的苔藓、蕨类植物中被发现[52],虽与花结构中MADS-box基因不是直系同源,但若要建立完整植物进化图,仍需对各类植物中MADS-box基因功能进行分析。
尽管大量MADS-box基因主要在生殖发育中发挥作用,部分该家族基因在营养生长和果实发育中也发挥重要作用。番茄中JOINTLESS基因与叶、花、果实等器官的脱落有关[53];拟南芥中Arabidopsisnitrateregulated1(ANR1)基因控制硝酸盐以调节根的生长[54]。MADS-box基因在被子植物根、茎、叶和胚胎中广泛表达,结合这些表达模式和基因功能,将有望阐明MADS-box基因在植物形态进化和构建中的综合作用。
参考文献(References):
[1] THEIEN G. Development of floral organ identity: stories from the MADS house[J].CurrentOpinioninPlantBiology,2001,4(1): 75-85.
[2] 许智宏,种康. 植物细胞分化与器官发生[M]. 北京: 科学出版社,2015: 217-270..
[3] KIM S H,MIZUNO K,FUJIMURA T. Isolation of MADS-box genes from sweet potato [Ipomoeabatatas(L.) Lam.]expressed specifically in vegetative tissues[J].Plant&CellPhysiology,2002,43(3): 314-322.
[4] ZHAO H B,JIA H M,WANG Y,et al. Genome-wide identification and analysis of the MADS-box gene family and its potential role in fruit development and ripening in red bayberry (Morellarubra)[J].Gene,2019,717: 144045.
[5] SCHWARZ-SOMMER Z,HUIJSER P,NACKEN W,et al. Genetic control of flower development by homeotic genes inAntirrhinummajus[J].Science,1990,250(4983): 931-936.
[6] 严海燕. 植物发育分子生物学[M]. 北京: 科学出版社,2012: 147-177.
[7] ALVAREZ-BUYLLA E R,PELAZ S,LILJEGREN S J,et al. An ancestralMADS-boxgene duplication occurred before the divergence of plants and animals[J].ProceedingsoftheNationalAcademyofSciences,2000,97(10): 5328-5333.
[8] LAI X L,DAHER H,GALIEN A,et al. Structural basis for plant MADS transcription factor oligomerization[J].ComputationalandStructuralBiotechnologyJournal,2019,17: 946-953.
[9] BEMER M,HEIJMANS K,AIROLDI C,et al. An atlas of type IMADSboxgene expression during female gametophyte and seed development inArabidopsis[J].PlantPhysiology,2010,154(1): 287-300.
[10] MASIERO S,COLOMBO L,GRINI P E,et al. The emerging importance of type I MADS box transcription factors for plant reproduction[J].ThePlantCell,2011,23(3): 865-872.
[11] KANG I H,STEFFEN J G,PORTEREIKO M F,et al. The AGL62 MADS domain protein regulates cellularization during endosperm development inArabidopsis[J].ThePlantCell,2008,20(3): 635-647.
[12] ZHANG Y T,TANG D Q,LIN X C,et al. Genome-wide identification of MADS-box family genes in moso bamboo (Phyllostachysedulis) and a functional analysis ofPeMADS5 in flowering[J].BMCPlantBiology,2018,18(1): 1-18.
[13] RIECHMANN J L,MEYEROWITZ E M. MADS domain proteins in plant development[J].BiologicalChemistry,1997,378(10): 1079-1101.
[14] THEIEN G,GRAMZOW L. Structure and evolution of plant MADS domain transcription factors[M]//Plant transcription factors. Amsterdam: Elsevier,2016: 127-138.
[15] BECKER A,THEIßEN G. The major clades ofMADS-boxgenes and their role in the development and evolution of flowering plants[J].MolecularPhylogeneticsandEvolution,2003,29(3): 464-489.
[16] SRI T,GUPTA B,TYAGI S,et al. Homeologs ofBrassicaSOC1,a central regulator of flowering time,are differentially regulated due to partitioning of evolutionarily conserved transcription factor binding sites in promoters[J].MolecularPhylogeneticsandEvolution,2020,147: 106777.
[17] KONG L,DUAN Y L,YE Y F,et al. Screening and analysis of proteins interacting with OsMADS16 in rice (OryzasativaL.)[J].PLoSOne,2019,14(8): e0221473.
[18] XIE L,ZHANG Y,WANG K,et al.TaVrt2,anSVP-like gene,cooperates withTaVrn1 to regulate vernalization-induced flowering in wheat[J].NewPhytologist,2019: 16339.
[19] CHANDLER J W. Founder cell specification[J].TrendsinPlantScience,2011,16(11): 607-613.
[20] SHARMA N,RUELENS P,D’HAUW M,et al. A flowering locus C homolog is a vernalization-regulated repressor inBrachypodiumand is cold regulated in wheat[J].PlantPhysiology,2017,173(2): 1301-1315.
[21] SHARMA N,GEUTEN K,GIRI B S,et al. The molecular mechanism of vernalization inArabidopsisand cereals: role ofFLOWERINGLOCUSCand its homologs[J].PhysiologiaPlantarum,2020,170(3): 373-383.
[22] LI Z C,JIANG D H,HE Y H. FRIGIDA establishes a local chromosomal environment forFLOWERINGLOCUSCmRNA production[J].NaturePlants,2018,4(10): 836-846.
[23] LI Z C,OU Y,ZHANG Z C,et al. Brassinosteroid signaling recruits histone 3 lysine-27 demethylation activity toFLOWERINGLOCUSCchromatin to inhibit the floral transition inArabidopsis[J].MolecularPlant,2018,11(9): 1135-1146.
[24] HONG J K,PARK S R,SUH E J,et al. Effects of overexpression ofBrassicarapaSHORTVEGETATIVEPHASEgene on flowering time[J].KoreanJournalofBreedingScience,2020,52(3): 244-251.
[25] LIU X,SUN Z C,DONG W,et al. Expansion and functional divergence of theSHORTVEGETATIVEPHASE(SVP) genes in eudicots[J].GenomeBiologyandEvolution,2018,10(11): 3026-3037.
[26] LIU H R,LI G,YANG X J,et al. Transcriptome profiling reveals phase-specific gene expression in the developing barley inflorescence[J].TheCropJournal,2020,8(1): 71-86.
[27] CHAE E,TAN Q K G,HILL T A,et al. AnArabidopsisF-box protein acts as a transcriptional co-factor to regulate floral development[J].Development(Cambridge,England),2008,135(7): 1235-1245.
[28] LEE J,OH M,PARK H,et al.SOC1 translocated to the nucleus by interaction withAGL24 directly regulatesLEAFY[J].ThePlantJournal,2008,55(5): 832-843.
[29] TORTI S,FORNARA F. AGL24 acts in concert withSOC1 andFULduringArabidopsisfloral transition[J].PlantSignaling&Behavior,2012,7(10): 1251-1254.
[30] GOSLIN K,ZHENG B B,SERRANO-MISLATA A,et al. Transcription factor interplay betweenLEAFYandAPETALA1/CAULIFLOWERduring floral initiation[J].PlantPhysiology,2017,174(2): 1097-1109.
[32] HILL K,WANG H,PERRY S E. A transcriptional repression motif in the MADS factorAGL15 is involved in recruitment of histone deacetylase complex components[J].ThePlantJournal,2008,53(1): 172-185.
[33] AGLIASSA C,NARAYANA R,BERTEA C M,et al. Reduction of the geomagnetic field delaysArabidopsisthalianaflowering time through downregulation of flowering-related genes[J].Bioelectromagnetics,2018,39(5): 361-374.
[34] YIFENG X,NATHANA⊇L P,ENG‐SENG G,et al.SUPERMANregulates floral whorl boundaries through control of auxin biosynthesis[J].TheEMBOjournal, 2018,37 (11): e97499.
[35] OTANI M,AOYAGI K,NAKANO M. Suppression of B function by chimeric repressor gene-silencing technology (CRES-T) reduces the petaloid tepal identity in transgenicLiliumsp[J].PLoSOne,2020,15(8): e0237176.
[36] KRAMER E M,DORIT R L,IRISH V F. Molecular evolution of genes controlling petal and stamen development: duplication and divergence within theAPETALA3 andPISTILLATAMADS-boxgene lineages[J].Genetics, 1998,149 (2): 765-783.
[37] ZHOU Y Z,XU Z D,YONG X,et al. SEP-class genes inPrunusmumeand their likely role in floral organ development[J].BMCPlantBiology,2017,17(1): 1-11.
[38] GIOPPATO H A,DORNELAS M C. When Bs are better than As: the relationship between B-classMADS-boxgene duplications and the diversification of perianth morphology[J].TropicalPlantBiology,2019,12(1): 1-11.
[39] XIANG L,CHEN Y,CHEN L P,et al. B and EMADS-boxgenes determine the perianth formation inCymbidiumgoeringiiRchb.f[J].PhysiologiaPlantarum,2018,162(3): 353-369.
[40] JIANG Y C,WANG M M,ZHANG R,et al. Identification of the target genes of AqAPETALA3-3 (AqAP3-3) inAquilegiacoerulea(Ranunculaceae) helps understand the molecular bases of the conserved and nonconserved features of petals[J].NewPhytologist,2020,227(4): 1235-1248.
[41] HUANG F Y,ZHANG Y H,HOU X L.BcAP3,a MADS box gene,controls stamen development and male sterility in Pak-choi (Brassicarapassp. chinensis)[J].Gene,2020,747: 144698.
[42] 李芳. NAP家族成员克隆及其在桃果实发育与成熟期间的表达特性研究[D]. 杨凌: 西北农林科技大学,2016.
LI F. Cloning and expression characterization of NAP family members during development and ripening of peach[D]. Yangling: Northwest A & F University,2016. (in Chinese with English abstract)
[43] PRUNET N. My favourite flowering image: anArabidopsisinflorescence expressing fluorescent reporters for theAPETALA3 and SUPERMAN genes[J].JournalofExperimentalBotany,2019,70(21): e6499-e6501.
[44] UEMURA A,YAMAGUCHI N,XU Y F,et al. Regulation of floral meristem activity through the interaction of AGAMOUS,SUPERMAN,and CLAVATA3 inArabidopsis[J].PlantReproduction,2018,31(1): 89-105.
[45] CHEN Z S,LIU X F,WANG D H,et al. Transcription factorOsTGA10 is a target of the MADS protein OsMADS8 and is required for tapetum development[J].PlantPhysiology,2018,176(1): 819-835.
[46] 黄海娇,李慧玉,姜静.BpAP1转基因白桦中开花相关基因的时序表达[J]. 东北林业大学学报,2017,45(1): 1-6.
HUANG H J,LI H Y,JIANG J. Quantitative expression analysis of several flowering-related genes inBpAP1 transgenic birch(Betulaplatyphylla×Betulapendula)[J].JournalofNortheastForestryUniversity,2017,45(1): 1-6.(in Chinese with English abstract)
[47] 袁秀云,许申平,王莹博,等. 蝴蝶兰PhalPI基因的克隆及在花器官突变体中的表达分析[J]. 植物研究,2017,37(3): 416-423.
YUAN X Y,XU S P,WANG Y B,et al. Cloning ofPhalPIgene fromPhalaenopsisand its expression in floral organ mutants[J].BulletinofBotanicalResearch,2017,37(3): 416-423.(in Chinese with English abstract)
[48] CHEN,MAO,HUANG,et al. Morphological characterization of flower buds development and related gene expression profiling at bud break stage in heterodichogamousCyclocaryapaliurus(Batal.) lljinskaja[J].Genes,2019,10(10): 818.
[49] SHULGA O A,MITIOUCHKINA T Y,SHCHENNIKOVA A V,et al. Chrysanthemum modification via ectopic expression of sunflowerMADS-boxgeneHAM59[J].ActaHorticulturae,2015: 105-111.
[50] MOURADOV A,GLASSICK T V,HAMDORF B A,et al. Family ofMADS-boxgenes expressed early in male and female reproductive structures of Monterey pine[J].PlantPhysiology,1998,117(1): 55-62.
[51] WINTER K U,BECKER A,MUNSTER T,et al.MADS-boxgenes reveal that gnetophytes are more closely related to conifers than to flowering plants[J].PNAS,1999,96(13): 7342-7347.
[52] THANGAVEL G,NAYAR S. A survey of MIKC typeMADS-boxgenes in non-seed plants: algae,bryophytes,lycophytes and ferns[J].FrontiersinPlantScience,2018,9: 510.
[53] MAO L,BEGUM D,CHUANG H W,et al. JOINTLESSis aMADS-boxgene controlling tomato flower abscissionzone development[J].Nature,2000,406(6798): 910-913.
[54] GAN Y B,BERNREITER A,FILLEUR S,et al. Overexpressing theANR1MADS-boxgene in transgenic plants provides new insights into its role in the nitrate regulation of root development[J].PlantandCellPhysiology,2012,53(6): 1003-1016.
