古基因组解密末次盛冰期前后的东亚北部人群动态
2021-06-28毛晓伟平婉菁付巧妹
毛晓伟,平婉菁,付巧妹
古基因组解密末次盛冰期前后的东亚北部人群动态
毛晓伟,平婉菁,付巧妹
中国科学院古脊椎动物与古人类研究所,北京 100044
现代人的起源与演化一直是地球与生命科学领域的研究焦点。过去10多年,围绕世界各地考古遗址人群开展的古基因组研究,在旧石器时代晚期以来欧洲、北亚等区域人群的演化问题上取得重大进展。然而,东亚人群古基因组研究则相对滞后,尤其是对中国人类古基因组的研究极度匮乏,使东亚人群遗传演化和迁徙融合的历史成为现代人演化进程里的重要“缺环”。但是,近几年中国科学家在这方面的研究取得了突破性进展。
2017年,中国科学院古脊椎动物与古人类研究所(简称“中科院古脊椎所”)付巧妹团队从我国北京4万年前的田园洞人化石中获取中国以至东亚最古老现代人的基因组序列,数据分析表明以其为代表的古东亚人群与同时期某一古欧洲人群存在特殊的遗传联系,首次为探索整个欧亚大陆人群的遗传历史开启东亚篇章[1]。2020年,其团队首次系统性研究距今5000年青藏高原人群的线粒体基因组揭示青藏高原人群由低海拔向高海拔地区少量的人群流动和之后一段时间高海拔地区内部的人群扩张[2]。同年,其团队又相继发表我国南方1.1万年前人类古线粒体基因组及东亚自9500年来南北方人群的大规模、系统性古基因组研究,表明万年来东亚南北方人群在内频繁迁徙互动、南岛语族人群和南方内陆古人群的联系、向外通过沿海地带不断扩散交流的复杂历史[3,4]。2021年,其团队再度针对我国北方沿海的山东人群及河南仰韶遗址进行规模性古线粒体基因组研究,揭示其人群自新石器时代以来的遗传连续性[5,6]。这些研究不仅从时间和空间上填补了东亚人类古基因组的巨大空缺,揭开东亚人群与欧亚其他人群此前未知的遗传联系以及迁徙互动在其演化过程中的重要作用,更为人们解析了东亚北部自4万年前田园洞人至近万年前古北方沿海人群之间长达数万年的遗传历史断层。
冰河时期(距今约7~1.15万年)的气候变化对欧洲人群的迁徙和种群大小变化构成影响[7],考古证据表明其对亚洲高纬度和高海拔地区的人群历史也有类似的影响[8],但缺少相关的遗传学证据与针对性研究。在这一时期,尤其是末次盛冰期前后的东亚北部人群是否因气候的剧烈变化而发生相应人群的更替或迁徙交流?东亚人群是否携有与环境适应性相关的特有基因?这些都是古基因组研究亟待探索而颇具挑战的关键问题。
2021年5月27日,杂志在线发表了中科院古脊椎所付巧妹团队与云南大学张虎才研究组(第一作者为中科院古脊椎所毛晓伟副研究员,通讯作者为中科院古脊椎所付巧妹研究员和云南大学张虎才教授)合作开展的东亚北部人群的古基因组研究[9]。在该研究中,团队通过最前沿的古基因组实验技术获取我国东北地区黑龙江省距今33,000~3400年的25个早期人类样本的古基因组数据(其中11例个体超过1万年,图1),填补东亚北部人群遗传历史的相关时间断层,并结合北京田园洞人基因组数据展开遗传演化与群体遗传学分析,系统绘制出4万年来东亚北部人群的群体动态演化图谱。此外,研究发现东亚人群特有的环境适应性基因至少在1.9万年前出现,对此前利用当今现代人基因组所估算出的突变时间予以精确修正,为探究东亚北部人群的适应性遗传变异情况及相关基因的选择机制提供了新的思路和证据。
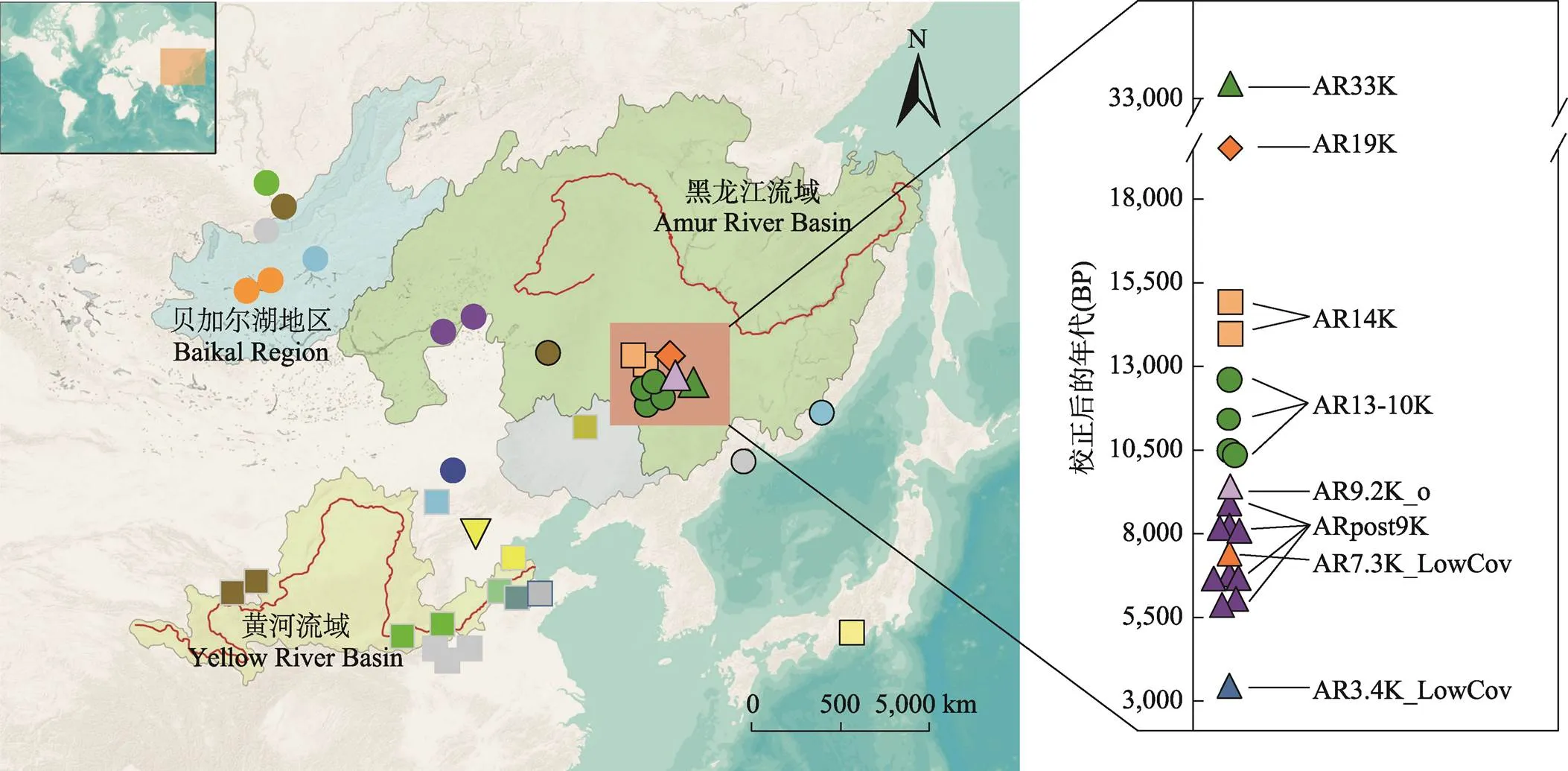
图1 新样本和已发表的东亚北方个体信息地理和时间分布图[9]
该研究指出生活在末次盛冰期前、距今约3.3万年的AR33K个体与距今4万年田园洞人的遗传成分相似。此前有研究表明同时期来自蒙古东北部、距今3.4万年的索尔吉特人(Salkhit)同时携有中国田园洞人和古西伯利亚北方人群“亚纳人”(Yana)相关的祖源成分[10],而本研究显示AR33K个体与同时期索尔吉特人(Salkhit)不一样,他不含有“亚纳人”(Yana)相关的遗传成分,与田园洞人是聚类在一起。这一结果说明在末次盛冰期之前,田园洞人相关人群在东亚北部广泛存在;同时,田园洞人相关人群在蒙古地区与亚那人(Yana)相关人群混合,而此相关人群在黑龙江地区存在并保持着连续性(图2)。
末次盛冰期约在2.65万年前开始,一直持续到约1.9万年前,这是距现在最近的极寒冷时期。为进一步探究末次盛冰期气候变化如何影响东亚北部人群,该研究针对生存在末次盛冰期末期、距今1.9万年的AR19K个体的古基因组展开分析。这是东亚首例末次盛冰期前后的人类基因组,分析表明在末次盛冰期前后(1.9万年)北部发生了人群特殊变化,即之前广泛存在的田园洞人相关人群可能消失,东亚古北方人群此时已在黑龙江出现。一方面,其数据显示AR19K个体相较于4万年前田园洞人,他并没有与同一地区的3.3万年前的个体更近,间接反映了人群的不连续。另一方面,研究显示AR19K个体相比于东亚古南方沿海人群(距今8400~4200年的奇和洞、亮岛、溪头村、昙石山遗址个体)来说,与东亚古北方沿海人群(距今9500~7700年的扁扁洞、小荆山、小高遗址、博山遗址个体)的遗传关系更近。由此表明,在末次盛冰期即将结束的时候,东亚南北方人群的差异就已经形成,并且最早的东亚古北方人群(以AR19K个体为代表的人群)在黑龙江地区出现。这一结果将此前研究所发现的东亚南北方人群遗传分化的时间(9500年前[3])向前推进了近万年。
末次盛冰期之后,东亚北部人群的流动情况又是如何?此前有关新石器时代早期黑龙江流域鬼门洞的狩猎采集人群(来自俄罗斯远东地区、距今7700年的DevilsCave_N个体)的古基因组研究表明,黑龙江流域人群在距今约8000年以来存在遗传连续性[11,12],然而距今8000年之前的人群情况尚不可知。本研究通过获取末次盛冰期之后、距今8000年之前黑龙江地区的16例个体基因组数据(距今约1.4~0.9万年),对该地区这段空白的人群遗传历史展开研究并获得有价值的发现。研究显示,末次盛冰期后的黑龙江地区人群相比于其他东亚人群而言,与新石器时代早期鬼门洞人群的遗传成分最为接近。这一结果表明,黑龙江流域人群的遗传连续性可能早在1.4万年前就已经开始,这比此前假说所提出的时间向前推进了6000年。该结论与黑龙江地区陶器和玉器最早出现在1.5万年前的考古记录相符[13]。另外,本研究发现生活在末次盛冰期后、距今1.4万年前的AR14K个体与古西伯利亚人群(Ancient Paleo- Siberians)[12]有特殊的遗传联系。古西伯利亚人群是在美洲之外与美洲原住民最相关的人群,而数据显示AR14K个体比鬼门洞人群更符合古西伯利亚人群所混有东亚祖源成分的来源。

图2 末次盛冰期前后的东亚北部人群动态示意图[9]
此外,研究对东亚北部人群的适应性遗传变异情况展开探索。此前有全基因组关联分析表明基因的V370A突变与东亚典型体征如更粗的毛发、更多的汗腺和铲形门齿有关[14,15],因此理解该基因突变的自然选择机制,对研究东亚人群的适应性变异至关重要。目前对该基因的选择机制有两种假说:一是在约2万年前的低紫外线环境中,该选择增加了母乳中的维生素D含量[16];二是在约3万年前的温暖潮湿气候中,该选择可调控体温调节性出汗[17]。此外,相关研究利用当今现代人基因组估算出基因V370A的突变时间在距今1.14万年(置信区间在距今4300~43,700年)。然而,本研究通过对大规模的古东亚人群基因组研究,更直接、深入地揭示该基因的出现时间和可能的选择机制。研究发现,
除田园洞人及末次盛冰期之前的AR33K个体以外,基因的V370A突变出现在包括AR19K在内的所有已知的古东亚人群之中。这一结果表明,基因的V370A突变率在末次盛冰期阶段或之后不久的时间里可能升高,此外也证明了原假说二的观点:3万年前基因的V370A突变在温暖潮湿环境中被选择的可能性较小。该结论使得所估算突变基因出现时间的上限提早并更为精确(图2)。
随着古基因组技术与遗传学分析手段的不断进步及国内专业团队与考古机构多方、跨学科合作的大力开展,针对东亚人群的古基因组研究已然迎来飞速发展的时刻。本研究首次在东亚地区开展跨度为4万年的大规模人类古基因组研究,且首次利用古基因组在适应性方面探究东亚人群重要表性特征的演化来源,揭示东亚北部距今40,000~3400年的的人群动态遗传历史,并为进一步探索东亚人群与环境的关系提供了重要遗传学证据。此外,该合作研究团队在通过古基因组技术与遗传学分析手段的深度融合揭秘东亚人群演化方面树立新的研究范式,为推进跨学科学术生态的建立提供借鉴和范本。
前沿技术的探索运用,是本研究得以取得重要进展与发现的前提,而在未来的东亚人群演化研究中亦将继续发挥重要作用。在古基因组获取方面,付巧妹团队通过延展应用或共同创新的古基因组关键技术,极大扩展了古基因组研究样本的时间轴线与区域范围:如共同开发古核DNA捕获技术,实现从大量环境微生物DNA里“钓取”出极其微量的内源DNA,使古基因组研究进入全基因组时代;如参与开发的新一代古DNA片段提取技术,将非冰冻层人类古DNA破译的时间推进到40万年前;如延展应用沉积物古DNA富集与种属鉴定方法,成功从青藏高原“土”中获取东亚首例已灭绝古人类DNA等等,成为世界上可实施操作这些古基因组关键技术的仅有的几个国际顶尖实验室之一,为国内科研团队独立、自主开展中国人群遗传演化研究提供了技术保障。在遗传学分析手段方面,该研究所运用的D统计量、人群混合结构、古老型人遗传成分比例等分析,都为挖掘、准确解读与深度分析不断增长的、更大规模、海量而复杂的东亚人类古基因组数据提供了方法基础。东亚有着极其丰富的考古遗存与复杂的人群历史,古基因组研究与考古学、病理学、微生物学、计算机科学等更多学科的结合,将为人们从更多维度、更精确、细节性地了解东亚人类神秘的过去——他们的饮食、环境、迁徙、健康、功能性基因的选择机制与适应性结果等打开更多新的窗口。
[1] Yang MA, Gao X, Theunert C, Tong H, Aximu-Petri A, Nickel B, Slatkin M, Meyer M, Pääbo S, Kelso J, Fu Q. 40,000-year-old individual from Asia provides insight into early population structure in Eurasia., 2017, 27(20): 3202–3208.
[2] Ding M, Wang T, Min-Shan Ko A, Chen H, Wang H, Dong G, Lu H, He W, Wangdue S, Yuan H, He Y,Cai L, Chen Z, Hou G, Zhang D, Zhang Z, Cao P, Dai Q, Feng X, Zhang M, Wang H, Yang MA, Fu Q. Ancient mitogenomes show plateau populations from last 5200 years partially contributed to present-day Tibetans., 2020, 287(1923):20192968.
[3] Yang MA, Fan XC, Sun B, Chen CY, Lang JF, Ko YC, Tsang CH, Chiu Hunglin, Wang TY, Bao QC, Wu XH, Hajdinjak M, Ko AMS, Ding MY, Cao P, Yang RW, Liu F, Nickel B, Dai QY, Feng XT, Zhang LZ, Sun CK, Ning C, Zeng W, Zhao YS, Zhang M, Gao X, Cui YQ, Reich D, Stoneking M, Fu QM. Ancient DNA indicates human population shifts and admixture in northern and southern China., 2020, 369(6501): 282–288.
[4] Bai F, Zhang X, Ji X, Cao P, Feng X, Yang R, Peng M, Pei S, Fu Q. Paleolithic genetic link between Southern China and Mainland Southeast Asia revealed by ancient mitochondrial genomes., 2020, 65(12): 1125–1128.
[5] Liu JC, Zeng W, Sun B, Mao XW, Zhao YS, Wang F, Li ZG, Luan FS, Guo JF, Zhu C, Wang ZM, Wei CM, Zhang M, Cao P, Liu F, Dai QY, Feng XT, Yang RW, Hou WH, Ping WJ, Wu XH, Bennett EA, Liu YC, Fu QM. Maternal genetic structure in ancient Shandong between 9500 and 1800 years ago., 2021, doi: 10.1016/j.scib.2021. 01.029.
[6] Miao B, Liu Y, Gu W, Wei Q, Wu Q, Wang W, Zhang M, Ding M, Wang T, Liu J, Liu F, Cao P, Dai Q, Yang R, Feng X, Ping W, Hou W, Yuan H, Fu Q. Maternal genetic structure of a Neolithic population of the Yangshao culture., doi:10.1016/j.jgg.2021.04.005.
[7] Fu Q, Posth C, Hajdinjak M, Petr M, Mallick S, Fernandes D, Furtwängler A, Haak W, Meyer M, Mittnik A, Nickel B, Peltzer A, Rohland N, Slon V, Talamo S, Lazaridis I, Lipson M, Mathieson I, Schiffels S, Skoglund P, Derevianko AP, Drozdov N, Slavinsky V, Tsybankov A, Cremonesi RG, Mallegni F, Gély B, Vacca E, Morales MR, Straus LG, Neugebauer-Maresch C, Teschler-Nicola M, Constantin S, Moldovan OT, Benazzi S, Peresani M, Coppola D, Lari M, Ricci S, Ronchitelli A, Valentin F, Thevenet C, Wehrberger K, Grigorescu D, Rougier H, Crevecoeur I, Flas D, Semal P, Mannino MA, Cupillard C, Bocherens H, Conard NJ, Harvati K, Moiseyev V, Drucker DG, Svoboda J, Richards MP, Caramelli D, Pinhasi R, Kelso J, Patterson N, Krause J, Pääbo S, Reich D. The genetic history of Ice Age Europe., 2016, 534(7606): 200–205.
[8] Rybin EP, Khatsenovich AM, Gunchinsuren B, Olsen JW, Zwyns N. The impact of the LGM on the development of the Upper Paleolithic in Mongolia., 2016, 425: 69–87.
[9] Mao X, Zhang H, Qiao S, Liu Y, Chang F, Xie P, Zhang M, Wang T, Li M, Cao P, Yang R, Liu F, Dai Q, Feng X, Ping W, Lei C, Olsen JW, Bennett EA, Fu Q. The deep population history of northern East Asia from the Late Pleistocene to the Holocene., 2021, doi: 10.1016/ j.cell.2021.04.040.
[10] Massilani D, Skov L, Hajdinjak M, Gunchinsuren B, Tseveendorj D, Yi S, Lee J, Nagel S, Nickel B, Devièse T, Tom H, Meyer M, Kelso J, Peter BM, Pääbo S. Denisovan ancestry and population history of early East Asians., 2020, 370(6516): 579–583.
[11] Siska V, Jones ER, Jeon S, Bhak Y, Kim HM, Cho YS, Kim H, Lee K, Veselovskaya E, Balueva T, Gallego-Llorente M, Hofreiter M, Bradley DG, Eriksson A, Pinhasi R, Bhak J, Manica A. Genome-wide data from two early Neolithic East Asian individuals dating to 7700 years ago., 2017, 3(2): e1601877.
[12] Sikora M, Pitulko VV, Sousa VC, Allentoft ME, Vinner L, Rasmussen S, Margaryan A, de Barros Damgaard P, de la Fuente C, Renaud G, Yang MA, Fu Q, Dupanloup I, Giampoudakis K, Nogués-Bravo D, Rahbek C, Kroonen G, Peyrot M, McColl H, Vasilyev SV, Veselovskaya E, Gerasimova M, Pavlova EY, Chasnyk VG, Nikolskiy PA, Gromov AV, Khartanovich VI, Moiseyev V, Grebenyuk PS, Fedorchenko AY, Lebedintsev AI, Slobodin SB, Malyarchuk BA, Martiniano R, Meldgaard M, Arppe L, Palo JU, Sundell T, Mannermaa K, Putkonen M, Alexandersen V, Primeau C, Baimukhanov N, Malhi RS, Sjögren KG, Kristiansen K, Wessman A, Sajantila A, Lahr MM, Durbin R, Nielsen R, Meltzer DJ, Excoffier L, Willerslev E. The population history of northeastern Siberia since the Pleistocene., 2019, 570(7760): 182–188.
[13] Yue JP, Li YQ, Yang SX. Neolithisation in the southern Lesser Khingan Mountains: lithic technologies and ecological adaptation., 2019, 93(371): 1144–1160.
[14] Tan J, Yang Y, Tang K, Sabeti PC, Jin L, Wang S. The adaptive variant EDARV370A is associated with straight hair in East Asians., 2013, 132(10): 1187–1191.
[15] Fujimoto A, Ohashi J, Nishida N, Miyagawa T, Morishita Y, Tsunoda T, Kimura R, Tokunaga K. A replication study confirmed the EDAR gene to be a major contributor to population differentiation regarding head hair thickness in Asia., 2008, 124(2): 179–185.
[16] Hlusko LJ, Carlson JP, Chaplin G, Elias SA, Hoffecker JF, Huffman M, Jablonski NG, Monson TA, O'Rourke DH, Pilloud MA, Scott GR. Environmental selection during the last ice age on the mother-to-infant transmission of vitamin D and fatty acids through breast milk., 2018, 115(19): E4426–E4432.
[17] Kamberov YG, Wang S, Tan J, Gerbault P, Wark A, Tan L, Yang Y, Li S, Tang K, Chen H, Powell A, Itan Y, Fuller D, Lohmueller J, Mao J, Schachar A, Paymer M, Hostetter E, Byrne E, Burnett M, McMahon AP, Thomas MG, Lieberman DE, Jin L, Tabin CJ, Morgan BA, Sabeti PC. Modeling recent human evolution in mice by expression of a selected EDAR variant., 2013, 152(4): 691–702.
URI: https://kns.cnki.net/kcms/detail/11.1913.R.20210528.1013.002.html
2021-05-27
毛晓伟,博士,副研究员,研究方向:群体遗传、数量遗传。E-mail: maoxiaowei@ivpp.ac.cn
平婉菁,硕士,中级工程师,研究方向:实验技术。E-mail: pingwanjing@ivpp.ac.cn
毛晓伟和平婉菁并列第一作者。
付巧妹,博士,研究员,研究方向: 演化遗传、群体遗传。E-mail: fuqiaomei@ivpp.ac.cn
10.16288/j.yczz.21-187
2021-05-28 16:32:40
