基于生物信息学方法分析Cornelia de Lange综合征中失调的基因
2021-06-07陆迪明赵奕宁李溱何咏菁高学仁
陆迪明 赵奕宁 李溱 何咏菁 高学仁
摘 要:Cornelia de Lange综合征(CdLS)是一组罕见的累及多器官系统的遗传异质性疾病,其发病机制尚未完全阐明。本研究采用生物信息学方法综合分析CdLS患者与健康个体的基因表达谱数据,发现63个在CdLS患者中失调的基因,其中37个基因在CdLS患者中上调,26个基因在CdLS患者中下调。GO和KEGG通路富集分析显示,失调的基因显著富集在“整合素介导的细胞粘附”“细胞对生长因子刺激的负调节”“JAK-STAT级联”“钙信号通路”等生物学过程和信号通路。对失调基因表达的蛋白质进行相互作用网络分析,结果显示IL-6是网络中的关键基因。总之,当前研究发现了一些与CdLS有关的基因、生物学过程和信号通路。研究结果将有助于阐明CdLS的发病机制,为以后的治疗提供理论基础。
关键词:CdLS; 生物信息学; 基因; 罕见病
中图分类号:R596.02 文献标识码:A 文章编号:1006-3315(2021)4-196-002
Cornelia de Lange综合征(CdLS)是一组罕见的累及多器官系统的临床及遗传异质性疾病,其临床表型主要包括特殊面容、肢体缺陷、生长发育落后等[1,2]。据报道CdLS的全球患病率为1/30000~3/30000,但由于患者临床表型的多样性和临床医生对本病的认识不足,实际患病率可能更高[3]。目前已知的CdLS相关基因有5个:NIPBL、SMC1A、SMC3、RAD21和HDAC8基因,其中SMC1A、SMC3和RAD21基因编码的蛋白共同构成黏连蛋白环的核心亚基,NIPBL和HDAC8基因编码的蛋白分别调控黏连蛋白环的乙酰化和去乙酰化[1,4]。黏连蛋白环的主要功能是调控细胞分裂期姐妹染色单体的凝聚,同时黏连蛋白环还能影响细胞中DNA修复、基因表达和骨的形成[5-8]。在临床诊断的CdLS患者中,以NIPBL基因突变最多见(>60%),SMC1A和HDAC8基因突变各占约5%,SMC3和RAD21基因突变较罕见(<1%),而约30%的患者仍未明确分子病因[9]。为了进一步探討与CdLS有关的基因,本研究采用生物信息学方法综合分析CdLS患者与健康个体的基因表达谱数据。
1.材料和方法
从GEO数据库(https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE64034)中获得2个CdLS患者(NIPBL 742_743delCT,L248TfsX6和NIPBL 2969delG;G990DfsX2)和4个正常个体的成纤维细胞基因表达谱数据。采用GEO2R在线工具(https://www.ncbi.nlm.nih.gov/geo/geo2r/)分析基因表达谱数据,获取在Cornelia de Lange综合征中失调的基因。利用clusterProfiler R包对失调基因进行GO和KEGG通路富集分析。通过STRING数据库(https://string-db.org/)对失调基因表达的蛋白质进行相互作用网络分析。利用Cytoscape软件绘制蛋白质相互作用网络图,并分析网络中每个节点的度。
2.结果
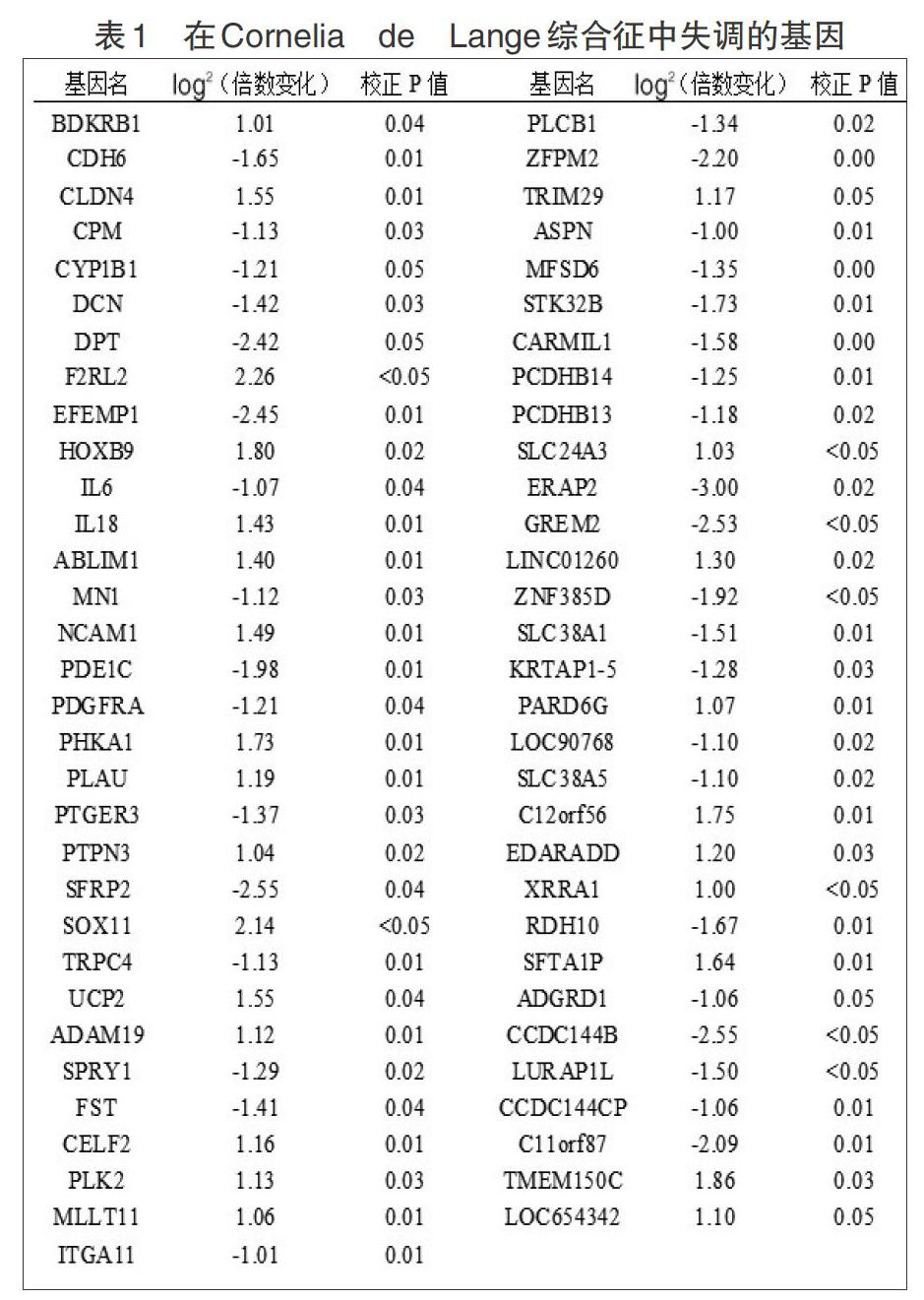
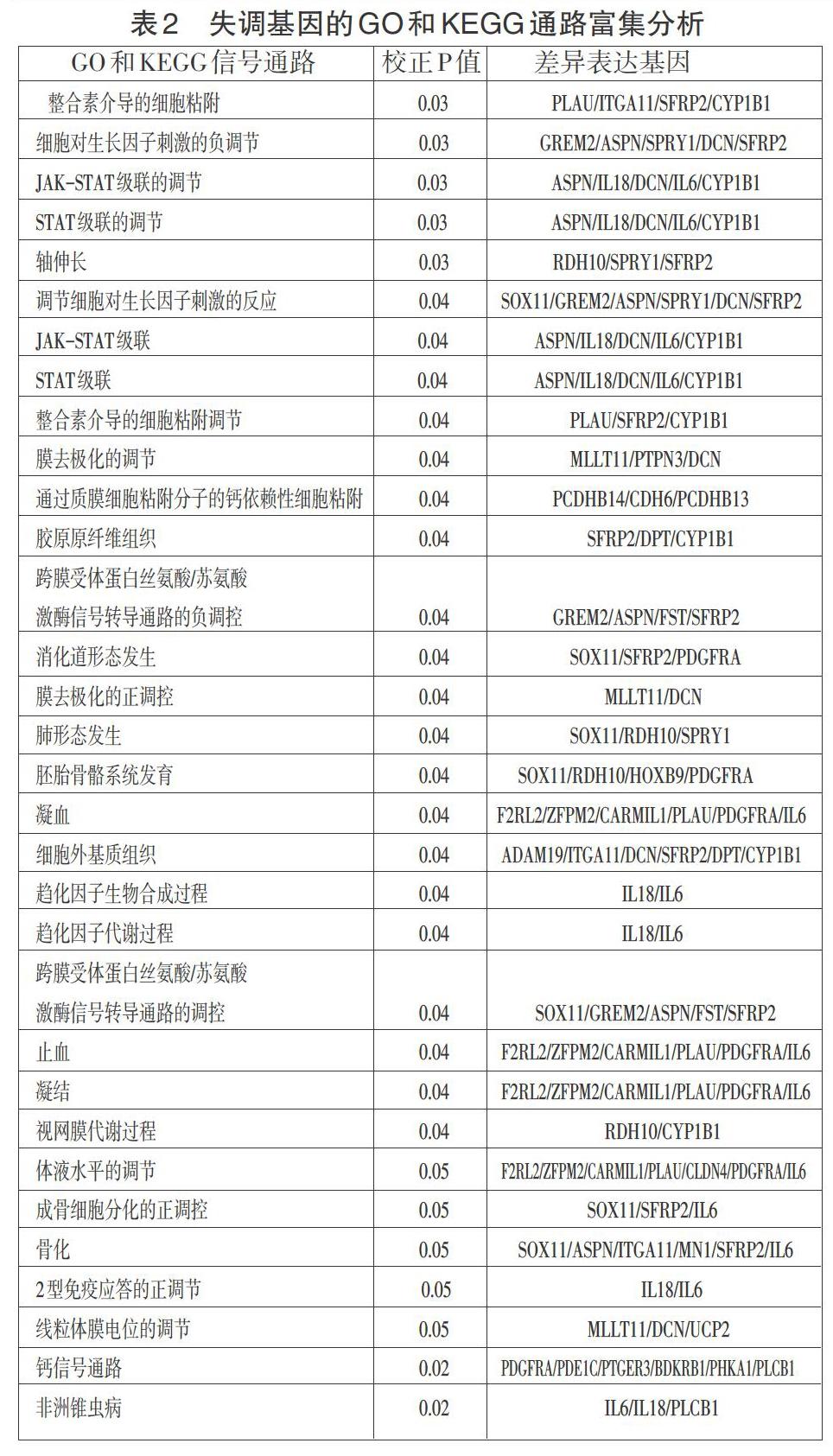
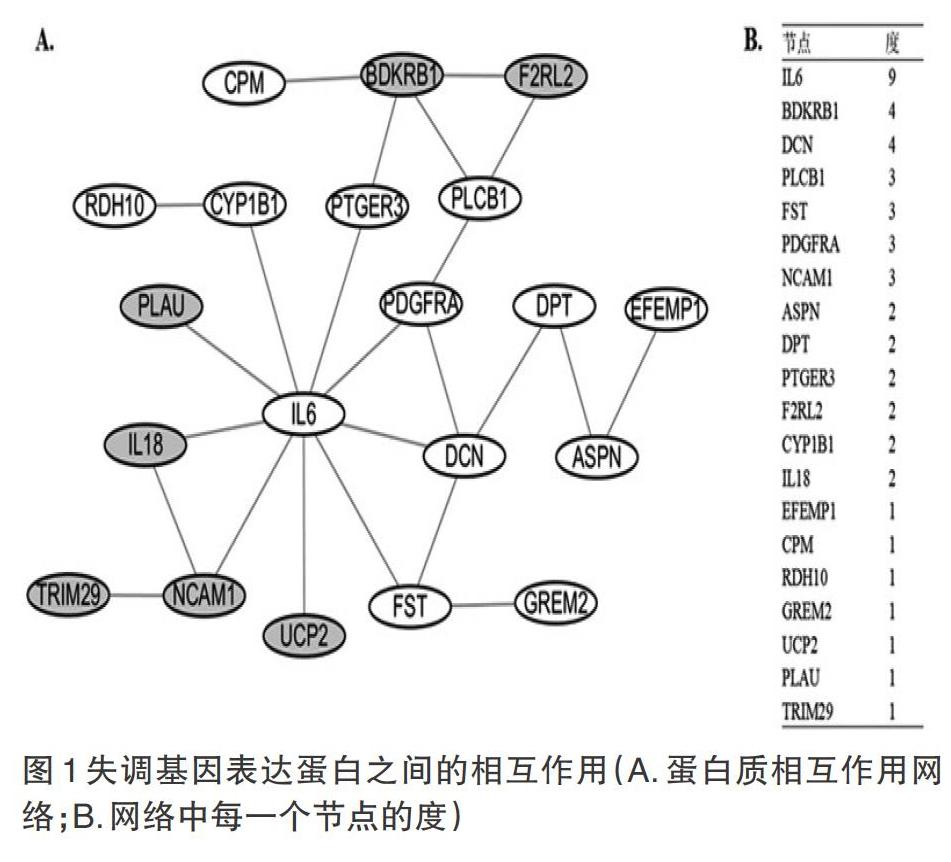
通过分析CdLS患者与健康个体的基因表达谱数据,共发现63个在CdLS患者中失调的基因,其中37个基因在CdLS患者中上调,26个基因在CdLS患者中下调(表1)。GO和KEGG通路富集分析显示,失调的基因显著富集在“整合素介导的细胞粘附”,“细胞对生长因子刺激的负调节”,“JAK-STAT级联”,“钙信号通路”等生物学过程和信号通路(表2)。对失调基因表达的蛋白质进行相互作用网络分析,结果显示IL-6具有最高的度,是网络中的关键基因(图1)。
3.讨论
随着医学科学,特别是分子生物学、医学遗传学的迅猛发展,研究人员对CdLS的病因和发病机制已有深入的了解。然而,CdLS的发病机制目前尚未阐明。当前研究通过生物信息学方法综合分析CdLS患者与健康个体的基因表达谱数据,发现了一些与CdLS有关的基因、生物学过程和信号通路。研究结果将有助于阐明CdLS的发病机制,为以后的治疗提供理论基础。
基金项目:国家自然科学基金青年科学基金项目,项目编号:81800780
参考文献:
[1]Ramos FJ, Puisac B, Baquero-Montoya C, et al. Clinical utility gene card for: Cornelia de Lange syndrome[J] Eur J Hum Genet, 2015;23
[2]Gillis LA, McCallum J, Kaur M, et al. NIPBL mutational analysis in 120 individuals with Cornelia de Lange syndrome and evaluation of genotype-phenotype correlations[J] Am J Hum Genet, 2004;75:610-23
[3]Mannini L, Cucco F, Quarantotti V, Krantz ID, Musio A. Mutation spectrum and genotype-phenotype correlation in Cornelia de Lange syndrome[J] Hum Mutat, 2013;34:1589-96
[4]Deardorff MA, Porter NJ, Christianson DW. Structural aspects of HDAC8 mechanism and dysfunction in Cornelia de Lange syndrome spectrum disorders[J] Protein Sci, 2016;25:1965-76.
[5]Nasmyth K, Haering CH. Cohesin: its roles and mechanisms[J] Annu Rev Genet, 2009;43:525-58
[6]Gause M, Schaaf CA, Dorsett D. Cohesin and CTCF: cooperating to control chromosome conformation[J] Bioessays, 2008;30:715-8
[7]Brooker AS, Berkowitz KM. The roles of cohesins in mitosis, meiosis, and human health and disease[J] Methods Mol Biol, 2014;1170:229-66
[8]Cohen-Zinder M, Karasik D, Onn I. Structural maintenance of chromosome complexes and bone development: the beginning of a wonderful relationship[J] Bonekey Rep, 2013;2:388
[9]Boyle MI, Jespersgaard C, Br?ndum-Nielsen K, Bisgaard AM, Tümer Z. Cornelia de Lange syndrome[J] Clin Genet, 2015;88:1-12
