柑橘叶斑驳病毒的逆转录重组酶聚合酶扩增检测
2021-05-12段玉许建建马志敏宾羽周常勇宋震
段玉,许建建,马志敏,宾羽,周常勇,宋震
柑橘叶斑驳病毒的逆转录重组酶聚合酶扩增检测
段玉,许建建,马志敏,宾羽,周常勇,宋震
西南大学/中国农业科学院柑桔研究所,重庆 400712
【】建立柑橘叶斑驳病毒(citrus leaf blotch virus,CLBV)逆转录重组酶聚合酶扩增(reversetranscription-recombinase polymerase amplification,RT-RPA)的快速检测体系,为该病毒提供快速、简便的检测方法。以CLBV ORF1保守序列为靶标,通过设计、筛选特异性检测引物、优化反应温度和反应时长等条件建立CLBV的RT-RPA检测体系。分别以感染CLBV、柑橘衰退病毒(citrus tristeza virus,CTV)、柑橘黄化脉明病毒(citrus yellow vein clearing virus,CYVCV)、柑橘裂皮类病毒(citrus exocortis viroid,CEVd)、柑橘碎叶病毒(citrus tatter leaf virus,CTLV)、柑橘鳞皮病毒(citrus psorosis virus,CPsV)、温州蜜柑萎缩病毒(satsuma dwarf virus,SDV)、柑橘溃疡病菌(subsp.,)和柑橘黄龙病菌亚洲种(Liberibacter asiaticus,Las)的柑橘核酸为模板,应用该体系进行RT-RPA扩增,评价检测特异性;利用CLBV阳性样品核酸的10倍浓度梯度稀释模板,比较RT-RPA、RT-PCR和RT-qPCR的检测灵敏度;通过比较上述3种方法对72份柑橘样品的CLBV检出率,检验所建立RT-RPA法的适用性。以感染了CLBV的柑橘核酸样品为模板,通过对设计的3对引物筛选试验、6个温度和5个反应时长的梯度试验,建立了CLBV的RT-RPA检测体系,明确引物RCLBV-F/RCLBV-R2能够特异扩增CLBV ORF1序列,且扩增效果良好,目的片段大小为144 bp,反应最适温度为40℃,反应最佳时长为30 min。在感染CLBV、CTV、CYVCV、CEVd、CTLV、CPsV、SDV、和Las的柑橘样品、健康对照和空白对照中,该体系仅在CLBV侵染样品中扩增出预期大小的特异性目的条带,说明建立的RT-RPA检测体系特异性强;灵敏度检测结果表明,RT-RPA和RT-PCR的检测灵敏度相当,但低于RT-qPCR;所建立的RT-RPA方法在72份柑橘样品中检测出11份样品为CLBV阳性,检出率为15.28%,该结果与普通RT-PCR和RT-qPCR检测方法的检测结果一致。建立了CLBV的RT-RPA检测体系,该体系具有耗时短、操作简便、特异性强等优点,适用于田间一定规模柑橘样品的检测。
柑橘叶斑驳病毒;逆转录重组酶聚合酶扩增;快速检测
0 引言
【研究意义】柑橘叶斑驳病毒(citrus leaf blotch virus,CLBV)是线性病毒科()柑橘病毒属()的正义单链RNA病毒[1-2],是柑橘病毒属的代表种[3]。CLBV于1984年首次在金橘中被检测并报道[4],后来在澳大利亚、日本、美国和西班牙的几个柑橘品种中亦检测到[5]。我国于1995年在对福建猕猴桃进行调查中首次发现CLBV[6],随后的检测表明此病毒在猕猴桃上分布广且危害有加重的趋势[7-8]。该病毒侵染几乎所有柑橘属植物及其近缘种,多为隐症,但可引起金橘和枳壳的芽接合部失调、橘橙叶片斑驳和香橼茎痘等症状[3]。在自然条件下,该病毒主要通过嫁接等无性繁殖途径传播,也可通过柑橘种子传播,但传播率很低[9]。目前,早期检测、应用脱毒苗木是有效防止此病毒危害的重要手段,因此建立简便、快捷的CLBV检测方法,对有效防控该病毒具有重要意义。【前人研究进展】随着对CLBV研究的深入,多种CLBV的鉴定方法被建立,从指示植物鉴定到斑点杂交和组织印迹杂交[10],再到分子检测,如RT-PCR检测[3,11]、多重RT-PCR检测[12]、实时荧光定量PCR[13-15]等。然而,现有的分子检测方法大多耗时费力、不够简便。RT-LAMP[16]检测法突破了热循环的局限,但其复杂的引物设计和较长的反应时长使其不够简便快捷。近年来,一种新型的等温核酸扩增技术——重组酶聚合酶扩增(recombinase polymerase amplification,RPA),因其快速、灵敏、操作简便而受到关注。RPA使用一种含有重组酶、单链DNA结合蛋白和具有置换活性的DNA聚合酶的酶促混合物进行恒温扩增,目标产物可在凝胶电泳中呈现。早在2006年,此技术就被应用于DNA的快速灵敏检测[17]和快速检测转基因作物[18]等,现已广泛应用于植物的病原检测,如马铃薯帚顶病毒(potato mop-top virus,PMTV)[19]、香蕉束顶病毒(banana bunchy top virus,BBTV)的RPA检测[20]。在柑橘病害检测方面,应用于柑橘黄龙病菌的RPA荧光检测技术[21]也已建立,整个检测反应能在15 min内完成,操作简单便捷,适用于该病害的田间快速检测。RT-RPA用于检测RNA病毒,如马铃薯Y病毒(potato virus Y,PVY)[22]、苹果茎痘病毒(apple stem pitting virus,ASPV)[23]、日本山药花叶病毒(Japanese yam mosaic virus,JYMV)[24]、玫瑰花叶病毒(rose rosette virus)[25]、辣椒脉斑驳病毒(chilli veinal mottle virus,ChiVMV)的RT-RPA检测[26]和RNA病毒高灵敏度RT-RPA核酸测流分析技术[27]等。【本研究切入点】关于CLBV的检测方法尽管已有报道,但在操作实施时均耗时费力,对操作技巧、试验仪器的要求较高。RT-LAMP能够实现CLBV的恒温检测,操作简便,但需要复杂的探针设计和较长的反应时间,仍有进一步优化的必要。【拟解决的关键问题】通过设计特异引物,建立CLBV的RT-RPA快速检测体系,为CLBV的检测提供新的技术手段。
1 材料与方法
试验于2019—2020年在西南大学柑桔研究所脱毒课题组完成。
1.1 试验材料与主要试剂
试验材料:本研究涉及的柑橘病害阳性材料包括CLBV、柑橘衰退病毒(citrus tristeza virus,CTV)、柑橘黄化脉明病毒(citrus yellow vein clearing virus,CYVCV)、柑橘裂皮类病毒(citrus exocortis viroid,CEVd)、柑橘碎叶病毒(citrus tatter leaf virus,CTLV)、柑橘鳞皮病毒(citrus psorosis virus,CPsV)、温州蜜柑萎缩病毒(satsuma dwarf virus,SDV)、柑橘溃疡病菌(subsp.,)和柑橘黄龙病菌亚洲种(Liberibacter asiaticus,Las),均由西南大学柑桔研究所国家柑桔苗木脱毒中心提供。
主要试剂:Biospin全能型植物基因组DNA提取试剂盒、多糖多酚植物总RNA提取试剂盒、Prime ScriptTMOne Step RT-PCR Kit Ver.2试剂盒购自北京宝日医生物技术有限公司;GoTaq® qPCR Master Mix试剂盒购于普洛麦格公司;RT-RPA核酸扩增试剂盒的品牌为众测生物。其余试剂均为分析纯。
1.2 核酸制备
取50 mg柑橘叶片组织样品,按照RNA提取试剂盒的说明书提取样品核酸,用CLBV1F/CLBV5R引物进行CLBV检验,将阳性样品核酸保存于-20℃备用。其余柑橘病毒阳性核酸也按照RNA提取试剂盒的说明指导提取核酸并检测为病毒阳性后存于-20℃备用。柑橘溃疡病菌和柑橘黄龙病菌则按照DNA试剂盒的说明指导提取核酸并检测为阳性后存于-20℃备用。
1.3 引物设计与引物特异性的筛选
根据CLBV基因ORF1的gRNA基因保守序列(登录号MG572236.1),通过Premier 5.0软件设计了3对引物(表1)。引物设计时注意避免错配、二聚体和发卡结构,最佳引物长度在30—35 bp,产物在100—150 bp。引物的RT-RPA筛选体系:上下游引物各2 μL(10 μmol·μL-1),加入RT-RPA试剂盒里的A缓冲液41.5 μL和B缓冲液2.5 μL,RNA 2 μL,盖上单元盖,充分振荡混匀,瞬时离心10 s,37℃ 30 min,加入上述反应体系等体积的三氯甲烷,充分混匀,12 000 r/min离心5 min,吸取10 µL上清产物,进行2.5%的琼脂糖凝胶电泳分析。将检测为阳性的RT-RPA反应产物进行测序,由华大基因公司完成。CLBV的RT-PCR检测引物CLBV1F/CLBV5R参考HARPER等[28]报道的序列合成,扩增片段大小为425 bp;RT-qPCR引物CLBVF/CLBVR参考Ruiz-Ruiz等[14]报道的序列合成。

表1 CLBV检测所用引物序列
1.4 反应温度和反应时长优化
设定从34℃至44℃的温度差,以2℃为温度梯度,检测体系条件按照1.3中所述,进行RT-RPA反应体系的温度优化,反应产物进行琼脂糖凝胶电泳分析,筛选扩增效率最佳的反应温度。
设定从26 min至34 min的反应时间差,以2 min为时间梯度,检测体系条件按照1.3中所述,进行RT-RPA反应体系的反应时长优化,反应产物进行琼脂糖凝胶电泳分析,筛选扩增效率最佳的反应时长。
1.5 RT-RPA检测体系的特异性评价
将1.2制备的CLBV、CTV、CYVCV、CEVd、CTLV、CPsV、SDV、和Las阳性样品核酸,设置CLBV阳性对照、健康对照和水对照,按照1.4建立的RT-RPA方法进行检测,评价所建RT-RPA检测体系的特异性。
1.6 RT-RPA检测体系的灵敏度比较
选取现提的CLBV阳性核酸为模板,按10倍梯度稀释,分别采用所建立的RT-RPA及文献报道的RT-PCR[28]、RT-qPCR[14]进行平行检测,比较其检测灵敏度。
RT-RPA检测体系:RCLBV-F/RCLBV-R2各2 μL(10 μmol·μL-1),A缓冲液41.5 μL,B 缓冲液2.5 μL,RNA 2 μL,40℃ 30 min,加入等反应体系体积的三氯甲烷,12 000 r/min离心5 min,取上清产物4℃保存备用。
RT-PCR检测体系:CLBV1F/CLBV5R各0.2 μL(10 μmol·μL-1),Prime Script 1 Step Enzyme Mix 0.4 μL,2×1 Step Buffer 5 μL,去核酸酶水2.2 μL,RNA 2 μL。检测程序:50℃反转录30 min,94℃预变性3 min,94℃变性30 s,58℃解链30 s,72℃延伸30 s,循环35次,72℃再延伸5 min,12℃结束反应,反应产物4℃保存。
RT-qPCR检测体系:CLBVF/CLBVR各0.4 μL(10 μmol·μL-1),GoTaq® qPCR Master Mix 10 μL,去核酸酶水7.2 μL,cDNA 2 μL。检测程序:95℃预变性5 min,95℃变性15 s,58℃解链30 s,72℃延伸30 s,荧光收集15 s,35个循环。
1.7 柑橘样品的CLBV检测
将72份疑似CLBV的柑橘样品,按照RNA提取试剂盒提取核酸,分别采取RT-PCR、RT-RPA和RT-qPCR 3种方法按照1.6的反应体系和程序进行检测,统计各检测方法的CLBV阳性检出率。
2 结果
2.1 RT-RPA检测体系的建立与优化
2.1.1 特异性引物的筛选 选用CLBV阳性样本的核酸为模板,设置健康对照和水对照,使用所设计的3对RT-RPA引物进行扩增。结果表明(图1),引物RPACF1/RPACR1和引物RCLBV-F/RCLBV-R2能在感染CLBV的柑橘样品中扩增出单一的目的条带,健康对照和水对照则无扩增条带的扩增,表明这两对引物具有特异性;而引物RCLBV-F/RCLBV-R1无明显目的条带扩增,表明该引物扩增效果不佳。引物RCLBV-F/RCLBV-R2扩增出目的条带较引物RPACF1/ RPACR1的明亮,表明引物RCLBV-F/RCLBV-R2的扩增效率较优,将其扩增出的产物送去华大基因公司进行测序,结果表明该扩增产物大小为144 bp,符合目的条带大小;测序结果在NCBI上进行序列比对,结果显示其与目的基因序列一致性100%,表明引物RCLBV-F/RCLBV-R2具有扩增CLBV的特异性,用于后续试验。
2.1.2 反应温度和反应时长条件的筛选 在6个不同温度条件下,RCLBV-F/RCLBV-R2均表现出良好的扩增效率。在34℃时,存在弱的非特异性弱条带,随着反应温度的升高,非特异带逐渐消失;扩增效率随温度升高而升高,在40℃时,扩增效率达到最大,40℃后,随着反应温度的升高,扩增效率变化不明显。因此,40℃为RT-RPA反应体系反应的最优温度(图2)。
在5个不同反应时长条件下,RT-RPA检测体系均能进行特异性扩增。在反应30 min之前,扩增产量随反应时间的延长而增加,在反应时间为30 min时,产物的积累量达到最大,在反应30 min后,随着反应时间的延长,扩增产量逐渐下降,可能与相关酶的失活或产物DNA发生降解有关。因此,30 min为RT-RPA反应体系的最佳反应时长(图3)。
2.2 RT-RPA检测体系的特异性
利用优化后的RT-RPA检测体系对分别感染了CTV、CYVCV、CEVd、CTLV、CPsV、SDV、、Las和CLBV的柑橘阳性样品进行RT-RPA检测,电泳结果如图4所示,所建立的RT-RPA体系仅在CLBV的柑橘阳性样品检测出CLBV特异条带,对其他柑橘病原物侵染样品及健康对照、水对照的检测则均呈阴性。可见,所建立的RT-RPA检测体系特异性良好。
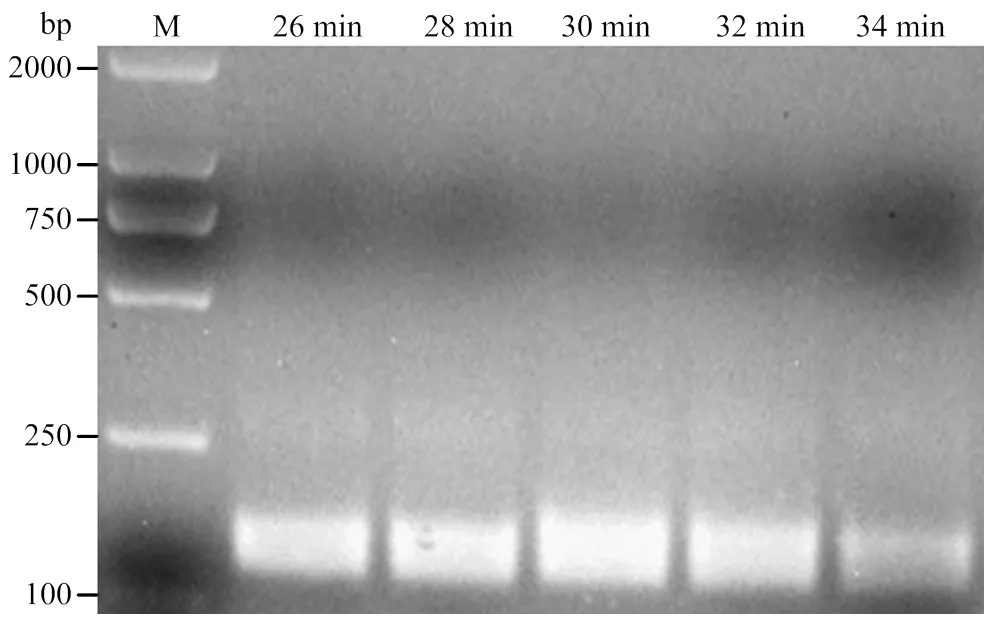
M:DNA分子质量标记DL2000 Marker;26—34 min:反应时长梯度Time gradient of reaction
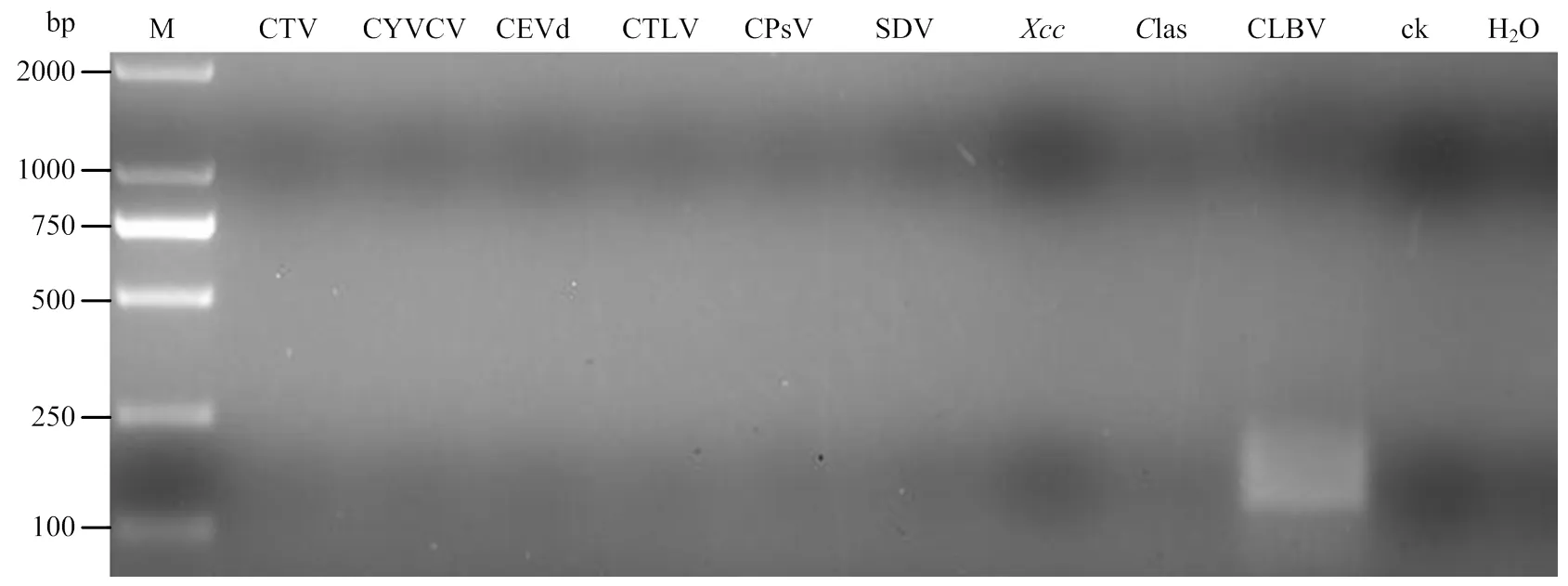
M:DNA分子质量标记DL2000 Marker;CTV—CLBV:不同柑橘病害阳性样本Different citrus disease positive samples;ck:健康对照Health control;H2O:水对照Water control
2.3 RT-RPA检测体系的灵敏度
提取CLBV阳性样品核酸,检测表明其浓度为1.17 µg·µL-1。通过10倍梯度稀释,获得系列浓度梯度模板,平行进行RT-PCR、RT-RPA和RT-qPCR检测,比较灵敏度。结果显示RT-RPA和RT-PCR能检测到的最小CLBV核酸浓度为1.17×10-3µg·µL-1,RT-qPCR能检测到浓度为1.17×10-6µg·µL-1的CLBV核酸(图5)。由此表明,RT-RPA检测的灵敏度和RT-PCR相当,比RT-qPCR的检测灵敏度低1 000倍。
2.4 柑橘样品检测
同时运用RT-PCR、RT-RPA和RT-qPCR 3种检测方法检测72份柑橘样品核酸,统计结果表明,3种检测方法均检测出11份带有CLBV的柑橘样品,检出率均为15.28%,3种检测方法的检测结果一致(图6)。由此可见本研究所建立的RT-RPA检测方法稳定可靠,可用于柑橘样品CLBV的检测。
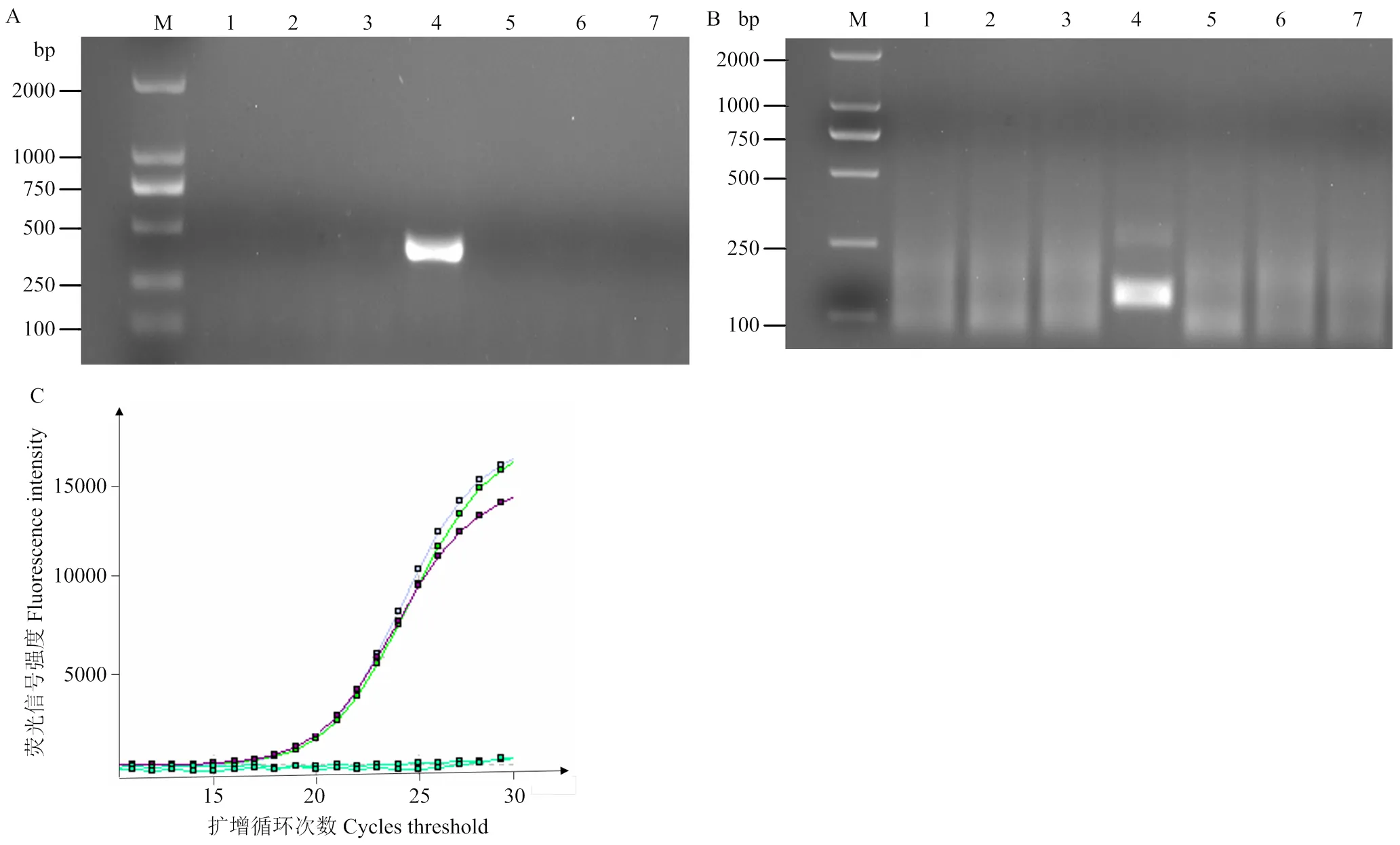
A:RT-PCR;B:RT-RPA;C:RT-qPCR。M:DNA分子质量标记DL2000 Marker;1—7:柑橘样品核酸Sample nucleic acid
3 讨论
本研究通过在CLBV ORF1保守序列上设计特异引物,优化反应时间和反应温度等,建立了CLBV的RT-RPA快速检测体系。该体系操作简便,反应迅速灵敏,特异性强,可快速呈现检测结果,不仅突破了普通RT-PCR的温度限制,也不需要复杂仪器,对操作者的技术要求较低,便于基层人员对CLBV的快速检测,应用前景广阔。目前检测CLBV普遍采用RT-PCR和RT-qPCR法,其中RT-qPCR需要避光操作和精密的仪器进行荧光采集,建立标准曲线和分析复杂的荧光数据,要消耗大量的时间和精力,不够简洁便利[13];普通的RT-PCR检测体系耗时较长,对仪器具有很强的依赖性。RT-RPA检测与上述两种检测方法相比,具有无需避光、不依赖于精密的仪器,不需要单独的反转录步骤且能在恒温条件进行检测反应的优点。与另一种恒温检测方法逆转录环介导等温扩增(RT-LAMP)相比,本研究所建立的RT-RPA方法不需要设计复杂的探针和4—6条引物,反应温度更低,反应时长缩短了一半,只需要35 min就能完成所有的检测反应,且产物单一,结果清晰明了,而RT-LAMP的反应产物成分复杂,不利于后续的产物测序分析。因而,RT-RPA比RT-LAMP法具有一定优势。
RNA容易降解,在逆转录过程中也存在逆转录效率问题。本研究为了更为准确地比较RT-RPA、普通RT-PCR和RT-qPCR检测方法的检测灵敏度,采用从柑橘样品中直接抽提的RNA作为模板,既反映了逆转录效率也反映了扩增效率,具有更高的真实性。在本研究中,RT-RPA与普通RT-PCR检测到的最小CLBV核酸浓度为1.17×10-3µg·µL-1,说明RT-RPA与普通RT-PCR的检测灵敏度在一个数量级;而RT-qPCR检测法则能检测到的最小CLBV核酸浓度为1.17×10-6µg·µL-1,比RT-RPA与普通RT-PCR灵敏1 000倍,是3种方法中灵敏度最高的检测法。本研究的目的是建立一种简便可靠的CLBV检测体系,而非追求最灵敏的检测方法,且本研究所涉及的RT-RPA检测技术已成功应用于多种植物病毒的检测,例如玉米绿斑驳病毒(maize chlorotic mottle virus)[29]、黄瓜绿斑驳花叶病毒(cucumber green mottle mosaic virus)[30]等。本研究建立的RT-RPA检测体系能特异检测出带有CLBV的柑橘样品,灵敏度和RT-PCR相当,虽灵敏度不及RT-qPCR,却比RT-qPCR更省时便捷,不需要复杂的操作和精密仪器的加持,可以在基层等条件不足的地方进行检测应用,实用度较高。
本研究在对72份疑似携带CLBV的柑橘样品的检测中,RT-RPA检测出11株CLBV阳性样品,结合普通RT-PCR和RT-qPCR验证,证实了该方法适用于柑橘植株CLBV的检测。本研究中关于CLBV的检出率为15.28%,该检出率不代表田间柑橘植株普遍携带CLBV的概率,仅代表本研究涉及的柑橘样品中携带CLBV的植株数量。
4 结论
通过特异性引物设计、反应温度和反应时长等条件优化,建立了CLBV的RT-RPA检测体系。该检测方法特异性强、灵敏度高,操作简单便捷,整个检测能在35 min内完成,适合于柑橘样品CLBV的快速检测。
[1] VIVES M C, Galipienso L, Navarro L, Moreno P, Guerri J. Characterization of two kinds of subgenomic RNAs produced by citrus leaf blotch virus. Virology, 2002, 295(2): 328-336.
[2] Rubino L, Russo M, De Stradis A, Martelli G P., a novel genus in the family. Archives of Virology, 2012, 157(8): 1629-1633.
[3] 朱晨熹, 王国平, 郑亚洲, 杨作坤, 王利平, 徐文兴, 洪霓. 来源于猕猴桃的柑橘叶斑驳病毒的RT-PCR检测及外壳蛋白基因序列分析. 植物病理学报, 2016, 46(1): 11-16.
ZHU C X, WANG G P, ZHENG Y Z, YANG Z K, WANG L P, XU W X, HONG N. RT-PCR detection and sequence analysis of coat protein gene of citrus leaf blotch virus infecting kiwifruit trees. Acta Phytopathologica Sinica, 2016, 46(1): 11-16. (in Chinese)
[4] NAVARRO L, PINA J A, BALLESTAROLMOS J F, MORENO P, CAMBRA M. A new graft transmissible disease found in nagami kumquat//Ninth IOCV Conference Proceedings. Riverside Springer, 1984: 234-240.
[5] CHE X B, Piestun D, Mawassi M, Yang G, Satyanarayana t, Gowda s, DAWSON W O, Bar-Joseph m. 5′-Coterminal subgenomic RNAs in citrus tristeza virus-infected cells. Virology, 2001, 283(2): 374-381.
[6] 林尤剑, 高日霞. 福建猕猴桃病害调查与鉴定. 福建农业大学学报, 1995, 24(1): 49-53.
LIN Y J, GAO R X. Survey and identification ofspp. diseases in Fujian, China. Journal of Fujian Agricultural University, 1995, 24(1): 49-53. (in Chinese)
[7] Galipienso L, VIVIES M C, MORENO P, MILNE R G, Navarro L,GUERRI J. Partial characterisation of citrus leaf blotch virus, a new virus from Nagami kumquat. Archives of virology, 2001, 146(2): 357-368.
[8] LIU H, SONG S, WU W, MI W, SHEN C, BAI B X, WU Y F. Distribution and molecular characterization of citrus leaf blotch virus fromin Shaanxi province, China. European Journal of Plant Pathology, 2019, 154(3): 855-862.
[9] GUERRI J, PINA J A, VIVES M C, NAVARRO L, MORENO P. Seed transmission of citrus leaf botch virus: Implications in quarantine and certification programs. Plant Disease, 2004, 88(8): 906.
[10] Galipienso L, VIVES M C, Navarro L, Moreno P, Guerri J. Detection of citrus leaf blotch virus using digoxigenin-labeled cDNA probes and RT-PCR. EuropeanJournal of Plant Pathology, 2004, 110(2): 175-181.
[11] PENG Q D, QIU L, YANG T, NING J C, XU Q Y, DONG J H, XI D H. A multiple reverse transcription PCR assay for simultaneous detection of four main viruses in kiwifruit. European Journal of Plant Pathology, 2020, 156(4): 1207-1212.
[12] 黄爱军, 王莹, 丁敏, 卢占军, 易龙. 柑橘4种病毒多重PCR检测技术的建立及应用. 园艺学报, 2019, 46(8): 1616-1622.
HUANG A J, WANG Y, DING M, LU Z J, YI L. Establishment and application of multiple PCR rapid detection of four citrus viruses. Acta horticulturae Sinica, 2019, 46(8): 1616-1622. (in Chinese)
[13] 刘欢, 米伟丽, 刘斐, 吴薇, 吴宽, 吴云锋. 猕猴桃植株中柑橘叶斑驳病毒实时荧光定量PCR检测技术的建立及应用. 植物病理学报, 2019, 49(2): 167-173.
LIU H, MI W L, LIU F, WU W, WU K, WU Y F. Development and evaluation of a real-time fluorescent quantitative PCr assay for detection of citrus leaf blotch virus in kiwifruit plants. Acta Phytopathologica Sinica, 2019, 49(2): 167-173. (in Chinese)
[14] RUIZ-RUIZ S, AMBROS A, VIVES M D G, NAVARRO L, MORENO P, GUERRI J. Detection and quantitation of citrus leaf blotch virus by TaqMan real-time RT-PCR. Journal of Virological Methods, 2009, 160(1/2): 57-62.
[15] OSMAN F, HODZIC E, KWON S J, WANG J B, VIDALAKIS G. Development and validation of a multiplex reverse transcription quantitative PCR (RT-qPCR) assay for the rapid detection of citrus tristeza virus, citrus psorosis virus, and citrus leaf blotch virus. Journal of Virological Methods, 2015, 220: 64-75.
[16] LIU H, WU W, TAN J Q, LI Y, MI W L, JIANG L J, WU Y F. Development and evaluation of a one-step reverse transcription loop-mediated isothermal amplification for detection ofcitrus leaf blotch virus. Journal of Virological Methods, 2019, 270: 150-152.
[17] PIEPENBURG O, WILLIAMS C H, STEMPLE D L, ARMES N A. DNA detection using recombination proteins. PLoS biology, 2006, 4(7): 1115-1121.
[18] LIU H, WANG J B, LI P, BAI L, JIA J W, PAN A H, LONG X Q, CUI W D, TANG X M. Rapid detection of P-35S and T-nos in genetically modified organisms by recombinase polymerase amplification combined with a lateral flow strip. Food Control, 2020, 107: 106775.
[19] DESHIELDS J B, MOROZ N, BRELEY L E, MORA-ROMERO G A, TANAKA K. Recombinase polymerase amplification (RPA) for the rapid isothermal detection off. sp.and potato mop-top virus. American Journal of Potato Research, 2019, 96(6): 617-624.
[20] KAPOOR R, SRIVASTAVA N, KUMAR S, SAEITHA R K, SHARMA S K, JAIN R K, BARANWEL V K. Development of a recombinase polymerase amplification assay for the diagnosis of banana bunchy top virus in different banana cultivars. Archives of Virology, 2017, 162(9): 2791-2796.
[21] QIAN W J, LU Y, MENG Y Q, YE Z Z, WANG L, WANG R, ZHENG Q Q, WU H, WU J. Field detection of citrus Huanglongbing associated with ‘Liberibacter asiaticus’ by recombinese polymerase amplification within 15 min. Journal of Agricultural and Food Chemistry, 2018, 66(22): 5473-5480.
[22] WANG Y, CHEN R H, NIE X Z, ZHONG Z Y, LI C Y, LI K, HUANG W, FU X Y, LIU J, NIE B H. Rapid and sensitive detection of potato virus Y by isothermal reverse transcription-recombinase polymerase amplification assay in potato. Molecular and Cellular Probes, 2020, 50: 101505.
[23] KIM N Y, LEE H J, JEONG R D. A portable detection assay for apple stem pitting virus using reverse transcription-recombinase polymerase amplification. Journal of Virological Methods, 2019, 274: 113747.
[24] Silva G, Bömer M, Nkere C, KUMAR P L, Seal S E. Rapid and specific detection of yam mosaic virus by reverse-transcription recombinase polymerase amplification.Journal of Virological Methods, 2015, 222: 138-144.
[25] BABU B, WASHBURN B K, MILLER S H, PODUCH K, SARIGUL T, KNOX G W, OCHOA-CORONA F M, PARET M L. A rapid assay for detection of rose rosette virus using reverse transcription- recombinase polymerase amplification using multiple gene targets.Journal of Virological Methods, 2017, 240: 78-84.
[26] JIAO Y B, XU C T, LI J L, GU Y, XIA C, XIE Q, XIE Y B, AN M N, XIA Z, WU Y H. Characterization and a RT-RPA assay for rapid detection of chilli veinal mottle virus (ChiVMV) in tobacco. Virology Journal, 2020, 17: 33.
[27] IVANOV A V, SAFENKOVA I V, ZHERDEV A V, DZANTIEV B B. Nucleic acid lateral flow assay with recombinase polymerase amplification: Solutions for highly sensitive detection of RNA virus. Talanta, 2020, 210: 120616.
[28] HARPER S J, CHOOI K M, PEARSON M N. First report of citrus leaf blotch virus in New Zealand. Plant Disease, 2008, 92(10): 1470.
[29] JIAO Y B, JIANG J Y, AN M N, XIA Z H, WU Y H. Recombinase polymerase amplification assay for rapid detection of maize chlorotic mottle virus in maize. Archives of Virology, 2019, 164(10): 2581-2584.
[30] ZENG R, LUO J Y, GAO S G, XU L H, SONG Z W, DAI F M. Rapid detection of cucumber green mottle mosaic virus by reverse transcription recombinase polymerase amplification.Molecular and Cellular Probes, 2019, 43: 84-85.
Detection of citrus leaf blotch virus by reverse transcription-recombinase polymerase amplification (RT-RPA)
DUAN Yu, XU JianJian, MA ZhiMin, BIN Yu, ZHOU ChangYong, SONG Zhen
Citrus Research Institute of Southwest University/Chinese Academy of Agricultural Sciences, Chongqing 400712
【】The objective of this study is toestablish a rapid reversetranscription-recombinase polymerase amplification (RT-RPA) detection system for citrus leaf blotch virus (CLBV), and to provide an efficient and simple detection method for CLBV.【】RT-RPA primers for CLBV detection were designed on conserved sequence of the ORF1, and their specificity was verified. The reaction temperature and reaction time were then optimized to establish RT-RPA system for CLBV detection. Nucleic acids from samples infected with citrus tristeza virus (CTV), citrus yellow vein clearing virus (CYVCV), citrus exocortis viroid (CEVd), citrus tatter leaf virus (CTLV), citrus psorosis virus (CPsV), satsuma dwarf virus (SDV),subsp.() andLiberibacter asiaticus (Las), respectively, were used to evaluate the specificity of the established RT-RPA method. The 10-fold dilution of CLBV positive samples was used to compare the detection sensitivity of RT-RPA, RT-PCR and RT-qPCR. In order to evaluate the applicability of the RT-RPA, 72 citrus samples were parallelly detected by the above three methods.【】In order to establish a RT-RPA method for CLBV detection, a series of experiments have been approached. Firstly, RCLBV-F/RCLBV-R2 that selected out of three designed primer pairs could specifically amplify the ORF1 fragments of CLBV with the aim size of 144 bp by RT-RPA, whereas no specific band could be obtained from samples infected with CTV, CYVCV, CEVd, CTLV, CPsV, SDV,orLas, showing the strong specificity of the RT-RPA method. Secondly, through gradient test of 6 temperatures and 5 reaction times, the optimal reaction temperature and time of the RT-RPA system were determined to be 40℃ and 30 min, respectively. Thirdly, the sensitivity of the RT-RPA was compared to that of RT-PCR and RT-qPCR, which is similar to the former but less than the latter. The RT-RPA established in this study was addressed to detect 72 citrus samples, 11 samples were detected to be CLBV positive with positive rate of 15.28%, which was consistent with that of RT-PCR and RT-qPCR.【】A RT-RPA system for CLBV detection was established and optimized, which has advantages of short time consuming and easy operation with potential usage for a relatively large scale of virus monitoring in field.
citrus leaf blotch virus (CLBV); RT-RPA; rapid detection

10.3864/j.issn.0578-1752.2021.09.008
2020-07-08;
2020-08-04
国家重点研发计划(2018YFD0201500,2017YFD0202002)、国家现代农业柑橘产业技术体系(CARS-26-05B)
段玉,E-mail:982432080@qq.com。通信作者宋震,E-mail:songzhen@cric.cn。通信作者周常勇,E-mail:zhoucy@cric.cn
(责任编辑 岳梅)
