文蛤CDK7基因克隆及其在不同品系生长发育中的表达分析
2021-04-15李秋洁陈爱华陈素华吴杨平张雨曹奕张志东田镇
李秋洁 陈爱华 陈素华 吴杨平 张雨 曹奕 张志东 田镇


摘要:【目的】掌握文蛤(Meretrix meretrix)細胞周期蛋白依赖性激酶7(CDK7)基因(MmCDK7)的时空表达及在不同品系生长发育中的表达规律,从分子水平探究红壳色文蛤新品系的生长优势,为筛选文蛤生长相关基因及揭示其生长发育机制提供理论依据。【方法】利用RACE克隆MmCDK7基因cDNA序列,通过BLAST、ScanProsite、NetPhos 3.0 server及ExPASy等在线软件进行生物信息学分析,使用实时荧光定量PCR检测MmCDK7基因在文蛤不同组织和不同发育时期的表达情况,并比较同一养殖条件下红壳色文蛤(简称红文蛤)和黄壳色文蛤(简称黄文蛤)的壳长、壳长相对增长率及MmCDK7基因表达差异。【结果】MmCDK7基因cDNA序列全长1296 bp,其中,5'端非编码区(5'-UTR)为83 bp,3'端非编码区(3'-UTR)为196 bp,开放阅读框(ORF)为1017 bp,共编码338个氨基酸残基。MmCDK7蛋白分子量约38.32 kD,理论等电点(pI)为8.78,包含丝氨酸/苏氨酸蛋白激酶催化结构域(S_TKc)、酪氨酸激酶催化结构域(TyrKc)及与细胞周期蛋白结合有关的激酶结构域NRTALRE;而S_TKc结构域中有包含蛋白激酶ATP结合位点区域、丝氨酸/苏氨酸蛋白激酶活化位点区域及T-loop环。MmCDK7氨基酸序列与虾夷扇贝CDK7氨基酸序列高度同源,其相似性为75.00%;基于CDK7氨基酸序列相似性构建的系统发育进化树显示,文蛤与中国真蛸、长牡蛎、厚壳贻贝及虾夷扇贝等软体动物先聚为一支。MmCDK7基因在性腺、水管、外套膜和肝胰腺等组织中均有表达,以性腺中的相对表达量显著高于其他组织(P<0.05,下同);MmCDK7基因在2种文蛤的8个发育时期均有表达,均以多细胞时期的相对表达量最高。在同一养殖条件下,除9月18日外,其他采样时间点均表现为红文蛤的壳长显著大于黄文蛤,红文蛤相对于黄文蛤的壳长增长率在2.79%~32.37%;除11月15日和11月30日外,其他采样时间点均表现为红文蛤的MmCDK7基因相对表达量显著高于黄文蛤的相对表达量。【结论】MmCDK7基因属于CDK家族成员,在细胞分裂旺盛的性腺及多细胞时期的相对表达量最高,且在生长速度较快红文蛤中的相对表达量多数情况下显著高于黄文蛤,故推测MmCDK7基因参与调控文蛤的早期生长发育过程。
关键词: 文蛤;CDK7基因;基因克隆;时空表达;生长发育
中图分类号: S968.317 文献标志码: A 文章编号:2095-1191(2021)12-3244-10
Cloning of CDK7 gene in Meretrix meretrix and its expression analysis on growth and development of different strains
LI Qiu-jie1,2, CHEN Ai-hua2*, CHEN Su-hua2, WU Yang-ping2, ZHANG Yu2,
CAO Yi2, ZHANG Zhi-dong2, TIAN Zhen1,2
(1Shanghai Ocean University/National Demonstration Center for Experimental Fisheries Science Education, Shanghai 201306, China; 2Jiangsu Institute of Marine Fisheries Research, Nantong, Jiangsu 226007, China)
Abstract:【Objective】To master the spatiotemporal expression of cyclin dependent kinase 7 (CDK7) gene (MmCDK7) in Meretrix meretrix and the expression law in the growth and development process of different strains , so as to explore the growth advantages of new strains of red clam at the molecular level. It provided a theoretical basis for screening growth related genes of M. meretrix and revealing its growth and development mechanism. 【Method】The cDNA sequence of MmCDK7 gene was cloned by RACE. Bioinformatics analysis was carried out by BLAST,ScanProsite,NetPhos 3.0 server and ExPASy. The expression of MmCDK7 gene in different tissues and different developmental stages of M. meretrix was detected by real-time fluorescence quantitative PCR. The differences of shell length, relative growth rate of shell length and MmCDK7 gene expression between red clam and yellow clam were compared under the same culture conditions. 【Result】The total length of the cDNA sequence of MmCDK7 gene was 1296 bp, of which the 5 ' untranslated region (5'-UTR) was 83 bp, the 3 ' untranslated region (3'-UTR) was 196 bp, and the open reading frame (ORF) was 1017 bp, encoding 338 amino acid. The molecular weight of MmCDK7 protein was about 38.32 kD and the theoretical isoelectric point (pI) was 8.78. It included serine / threonine protein kinase catalytic domain (S_TKc), tyrosine kinase catalytic domain (TyrKc) and kinase domain NRTALRE related to cyclin binding. And S_ TKc domain contained protein kinase ATP binding site, serine / threonine protein kinase activation site and T-loop loop. The amino acid sequence of MmCDK7 was highly homologous with that of Mizuhopecten yessosensis, and its similarity was 75.00%. The phylogene-tic tree constructed based on the amino acid sequence similarity of CDK7showed that M. meretrix and molluscs such as Octopus sinensis, Crassostrea gigas,Mytilus coruscus and M. yessosensis first gathered into one branch. MmCDK7 gene was expressed in gonad, siphon, mantle and hepatopancreas, and the relative expression in gonad was significantly higher than that in other tissues (P<0.05, the same below). MmCDK7 gene was expressed in 8 developmental stages of two species, and the relative expression in multicellular stage was significantly higher than that in other stages. Under the same culture conditions, except September 18, the shell length of red clam was significantly longer than that of yellow clam, and the growth rate of red clam relative to yellow clam was 2.79%-32.37%. Except November 15 and November 30, the relative expression of MmCDK7 gene in red clam was significantly higher than that in yellow clam at other sampling time points. 【Conclusion】MmCDK7 gene belongs to CDK family. The relative expression of CDK7 gene in gonad and multicellular stage with vigorous cell division is the highest, and the relative expression of CDK7 gene in red clam with fast growth rate is significantly higher than that in yellow clam in most cases. Therefore, it is speculated that MmCDK7 gene is involved in regulating the early growth and development of M. meretrix.
Key words: Meretrix meretrix; CDK7 gene; gene cloning; spatiotemporal expression; growth and development
Foundation item: Jiangsu Fishery Science and Technology Major Project(D2018-1); Jiangsu Agricultural Independent Innovation Project [CX (20) 3194];Aquatic Varieties Preservation and Renewal Project of Jiangsu (2020-SJ-006-05); Basic Science Research Project of Nantong(JC2020118)
0 引言
【研究意义】文蛤(Meretrix meretrix)隶属于软体动物门(Mollusca)瓣鳃纲(Lamellibranchia)帘蛤目(Veneroida)帘蛤科(Veneridae),是我国传统滩涂养殖的主要贝类之一。近年来,文蛤野生种质资源量逐年锐减,已出现近交衰退及种质下降等现象,导致文蛤苗种短缺且抗逆性差等问题频出(宋文涛,2013)。细胞周期蛋白依赖性激酶7(Cyclin-dependent kinase,CDK7)是CDK家族中功能较特殊的成员(Malumbres,2014),主要参与生物体细胞的生长发育过程。因此,分析CDK7基因在文蛤生长过程中发挥的功能作用,对推进文蛤良种选育具有重要意义。【前人研究进展】CDK7属于丝氨酸/苏氨酸蛋白激酶家族,具有双重功能:一方面,无活性的CDK7与相应周期蛋白Cyclin H结合,发生双磷酸化后与MAT1结合,构成细胞周期蛋白依赖性激酶活化激酶,即CDK活化激酶(CAK),在哺乳動物体内参与CDK1、CDK2、CDK4及CDK6的激活磷酸化,进而调节细胞周期(Lolli and Johnson,2005;江艳,2010);另一方面,CDK7可作为转录因子TFⅡH的亚基组成成分,负责磷酸化RNA聚合酶Ⅱ大亚基的羧基末端结构域(CTD)及CDK9,在mRNA转录起始、延伸及共转录RNA成熟过程中发挥重要作用(Fisher,2005;Larochelle et al.,2012;张洁妤等,2013)。已有研究证实,CDK7基因在生物体细胞的生长发育过程中发挥重要作用,如线虫(Caenorhabditis elegans)CDK7基因突变株不能激活mRNA转录,且通过RNAi可完全或接近完全抑制整个细胞周期进程(Matthew and Geraldine,2002);有选择性破坏小鼠胚胎成纤维细胞中的CDK7基因,能有效阻碍细胞周期进程,造成早期胚胎死亡(Schachter and Fisher,2013)。此外,有研究表明CDK7基因可促进癌细胞增殖,通过抑制CDK7基因表达可造成mRNA的表达及转录因子活性降低,阻止多种肿瘤细胞的分裂与增殖,因此临床医学上将CDK7基因作为癌症治疗的一个潜在靶点(Wang et al.,2018;Sava et al.,2020)。在水生生物中,已有学者对斑马鱼(Danio rerio)(Liu et al.,2007)、斑节对虾(Penaeus monodon)(Phinyo et al.,2014)及三疣梭子蟹(Portunus trituberculatus)(贾复龙等,2016)等物种的CDK7基因进行初步研究,但针对贝类的研究较少,仅Adzigbli等(2020)对马氏珠母贝(Pinctada martensi)CDK7基因的免疫作用机制进行研究,并未涉及贝类的生长发育。【本研究切入点】江苏省文蛤良种场经过多年的优良品种选育,已培育出红壳色文蛤新品系,与自然群体黄壳色文蛤相比,红壳色文蛤具有生长速度更快、成活率和抗逆性更高的优势(王超等,2016;张雨等,2018),但有关CDK7基因在文蛤生长发育中的作用机制尚未明确。【拟解决的关键问题】利用RACE克隆文蛤CDK7基因(MmCDK7)全长,并通过实时荧光定量PCR检测MmCDK7基因在2种文蛤群体不同组织和不同发育时期的表达情况,分析该基因表达规律与文蛤壳长变化的关系,以期从分子水平探究红壳色文蛤新品系的生长优势,为筛选文蛤生长相关基因及揭示其生长发育机制提供理论依据。
1 材料与方法
1. 1 试验材料
供试红壳色文蛤(简称红文蛤)取自于江苏省文蛤良种场选育的新品系,黄壳色文蛤(简称黄文蛤)为江苏省如东海区的野生群体。红文蛤是以江苏南部沿海自然文蛤的5000粒红壳色文蛤为育种基础群体,以“红壳色+生长”为目标性状,通过闭锁群体选育方式获得的子代选育系,每代留种率大于0.01%。
1. 2 样品采集
挑选4粒黄文蛤成贝,分别采集其肝胰腺、闭壳肌、外套膜、水管、鳃及性腺等组织进行RNA提取;2020年7月选取贝壳完整、健康且性腺成熟的红文蛤和黄文蛤为亲贝,在水温27~29 ℃、盐度29‰左右时进行催产。利用显微镜观察跟踪发育过程,分别选取红文蛤和黄文蛤受精卵、多细胞、囊胚期、原肠胚、担轮幼虫、D形幼虫、壳顶幼虫和稚贝等8个胚胎发育时期的样品用于总RNA提取。
将2020年7月繁育的红文蛤和黄文蛤稚贝置于1.5 t的缸中进行养殖,每缸苗种密度相同,持续充氧,每天换水,投喂相同饵料。红文蛤和黄文蛤各设3组平行。养殖试验于2020年9月开始,9月每周取样1次,10和11月每2周取样1次。每次每组随机取样40粒,测量其壳长,并计算对应壳长相对增长率(R)。测量结束后,取文蛤全组织,经PBS(0.01 mol/L)洗涤,液氮速冻,-80 ℃保存备用。
R(%)=(Lt-Ly)/Ly×100
式中,Lt为红文蛤壳长,Ly为黄文蛤壳长。
1. 3 MmCDK7基因cDNA序列克隆及测序
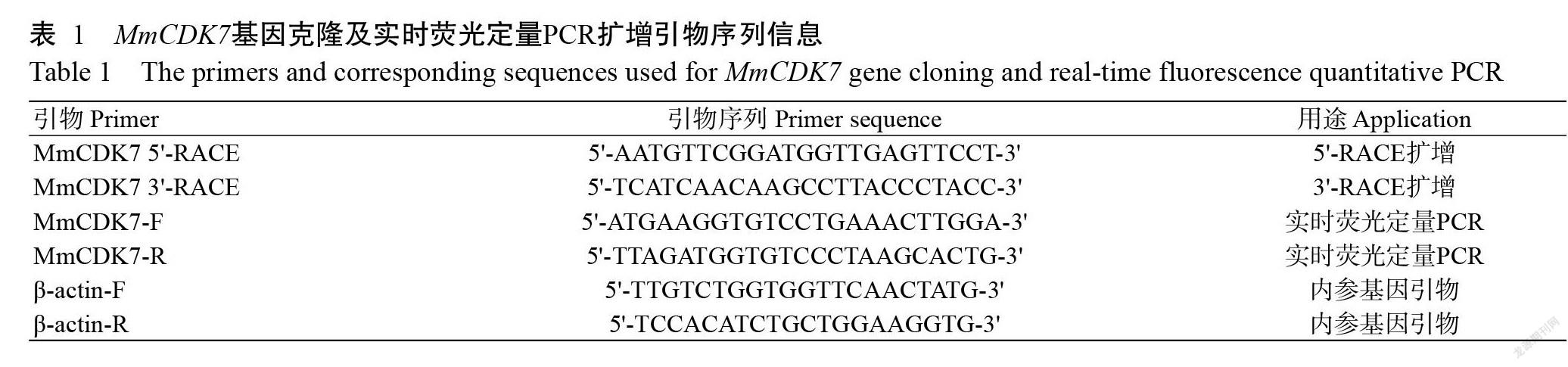
从构建的转录组文库中检索MmCDK7基因EST序列,设计并合成5'-RACE和3'-RACE特异性扩增引物(表1)。使用RNA提取纯化试剂盒[天根生化科技(北京)有限公司]提取文蛤性腺总RNA,然后以RACE试剂盒(Clontech公司)反转录合成cDNA第一链。参照高晓艳(2015)的方法进行RACE扩增,扩增产物经1.0%琼脂糖凝胶电泳检测后割胶回收纯化,纯化产物连接至pMD18-T载体,37 ℃过夜培养后转化大肠杆菌DH5α感受态细胞,筛选阳性克隆送至生工生物工程(上海)股份有限公司测序。
1. 4 生物信息学分析
利用NCBI中的ORF Finder(https://www.ncbi.nlm.nih.gov/orffinder/)进行MmCDK7基因开放阅读框(ORF)寻找,并翻译成氨基酸序列;使用BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行氨基酸序列同源比对分析;采用ScanProsite(https://prosite.expasy.org/scanprosite/)预测MmCDK7蛋白结构域;运用NetPhos 3.0 server(http://www.cbs.dtu.dk/servi-ces/NetPhos-3.0/)预测MmCDK7蛋白磷酸化位点;采用ExPASy(https://web.expasy.org/compute_pi/)预测MmCDK7蛋白分子量及其理论等电点(pI);利用DNAMAN进行多序列比对分析,并以MEGA 5.0中的邻接法(Neighbor-joining method,NJ)构建系统发育进化树。
1. 5 MmCDK7基因表达分析
使用RNA提取纯化试剂盒分别提取文蛤成体不同组织及不同胚胎发育时期的总RNA,通过NanoDrop 2000微量紫外分光光度计和1.0%琼脂糖凝胶电泳检测其浓度和质量;利用FastKing cDNA第一链合成试剂盒将总RNA反转录合成cDNA,-20 ℃保存备用。根据MmCDK7基因序列设计实时荧光定量PCR扩增引物(MmCDK7-F/MmCDK7-R)(表1),以β-actin基因为内参基因,使用Applied Biosystems荧光定量PCR仪测定MmCDK7基因的表达情况。依据SuperReal荧光定量预混试剂盒[天根生化科技(北京)有限公司]说明,配制实时荧光定量PCR反应体系20.0 μL:2×SuperReal Premix Plus 10.0 μL,正、反向引物各0.6 μL, 50×ROX Reference Dye 2.0 μL,cDNA模板1.0 μL,ddH2O 5.8 μL。扩增程序:95 ℃预变性15 min;95 ℃ 10 s,58 ℃ 31 s,72 ℃ 31 s,進行40个循环,收集荧光信号。95 ℃ 15 s,60 ℃ 1 min;95 ℃ 30 s,60 ℃ 15 s,进行融解曲线分析。采用2–ΔΔCT法换算目的基因相对表达量,并以SPSS 20.0进行单因素方差分析(One-way ANOVA)。
2 结果与分析
2. 1 MmCDK7基因克隆及测序分析
通过RACE克隆得到MmCDK7基因cDNA序列全长1296 bp,其中,5'端非编码区(5'-UTR)为83 bp,3'端非编码区(3'-UTR)为196 bp,ORF为1017 bp,共编码338个氨基酸残基(图1)。MmCDK7蛋白分子量约38.32 kD,pI为8.78,包含丝氨酸/苏氨酸蛋白激酶催化结构域(S_TKc)(8~291 aa)、酪氨酸激酶催化结构域(TyrKc)(8~291 aa)及与细胞周期蛋白结合有关的激酶结构域NRTALRE。S_TKc结构域中包含蛋白激酶ATP结合位点区域(14~38 aa)、丝氨酸/苏氨酸蛋白激酶活化位点区域(129~141 aa)及T-loop环(DFGLAKFFGSPNRIYTHQVVTRWYRCPE,151~178 aa)。此外,NetPhos 3.0预测结果显示,MmCDK7蛋白存在12个磷酸化位点,包括6个丝氨酸磷酸化位点、4个苏氨酸磷酸化位点及2个酪氨酸磷酸化位点。
从GenBank搜索参考物种的CDK7氨基酸序列(表2),利用DNAMAN进行多序列比对分析,结果(图2)显示,MmCDK7氨基酸序列与虾夷扇贝(Mizuhopecten yessosensis)的CDK7氨基酸序列高度同源,其相似性为75.00%;与中国真蛸(Octopus sinensis)、厚壳贻贝(Mytilus coruscus)、斑马鱼、黑腹果蝇(Drosophila melanogaster)及木蚁(Camponotus floridanus)的CDK7氨基酸序列相似性分别为71.09%、72.27%、66.09%、61.19%和71.89%,且具有相同活化/结合位点的保守结构域,包括核苷酸结合位点(14~22 aa)、ATP结合位点(37 aa)及质子受体活化位点(133 aa)。基于CDK7氨基酸序列相似性构建的系统发育进化树(图3)显示,文蛤首先与中国真蛸、长牡蛎(Crassostrea gigas)、厚壳贻贝及虾夷扇贝等软体动物聚为一支,然后与斑马鱼、大山雀(Parus major)及猕猴(Macaca mulatta)等脊索动物聚为一大支,而斑节对虾、三疣梭子蟹等节肢动物聚为另一分支。
2. 2 MmCDK7基因在文蛤不同组织中的表达情况
通过实时荧光定量PCR检测MmCDK7基因在文蛤成体不同组织中的表达情况,结果(图4)表明,MmCDK7基因在性腺、水管、外套膜和肝胰腺等组织中均有表达,但各组织中的相对表达量存在差异,以性腺中的相对表达量最高,显著高于其他组织(P<0.05,下同),其次是斧足、外套膜和鳃等组织。
2. 3 MmCDK7基因在文蛤不同发育时期的表达情况
由图5可看出,MmCDK7基因在红文蛤和黄文蛤不同发育时期的表达情况呈现出相同的变化趋势。2种文蛤在多细胞时期的MmCDK7基因相对表达量均达最高值,且显著高于担轮幼虫、D形幼虫、壳顶幼虫及推贝等4个发育时期的相对表达量;发育至担轮幼虫时期,其相对表达量显著下降,稚贝时期的相对表达量最低。
2. 4 MmCDK7基因在不同文蛤品种间的表达差异
在同一养殖条件下,红文蛤和黄文蛤的壳长除了在9月18日的采样中无显著差异(P>0.05,下同)外,其他采样时间点均表现为红文蛤的壳长显著大于黄文蛤,且在养殖后期(11月中下旬)这种差异达极显著水平(P<0.01)(图6)。在壳长相对增长率方面,红文蛤相对于黄文蛤的壳长增长率在2.79%~32.37%,且部分養殖时期存在显著差异(图7)。同步检测MmCDK7基因在文蛤生长过程中的表达变化规律,结果(图8)发现除了在11月15日和11月30日的采样中2种文蛤的MmCDK7基因相对表达量无显著差异外,其他采样时间点均表现为红文蛤的MmCDK7基因相对表达量显著高于黄文蛤的相对表达量。
3 讨论
CDK属于丝氨酸/苏氨酸蛋白激酶家族,在真核生物细胞周期进程中发挥着重要的调控作用,其活性的获得主要通过以下2种途径实现:一方面,CDK的T-loop磷酸化有助于底物进入活性位点,使激酶构象发生变化,进而调节激酶活性(Wu et al.,2003);另一方面,与对应的周期蛋白调节亚基相结合,继而表现出不同的激酶活性(Wood and Endicott,2018;Peissert et al.,2020)。CDK7是CDK家族中的特殊成员,其激活需T-loop的双重磷酸化,以及与Cyclin H和MAT1组成复合体,参与细胞周期调控;也可作为转录因子TFⅡH的亚基,调节CTD激酶活性,发挥转录调控作用(Martinez et al.,1997;Ebmeier et al.,2017)。本研究通过克隆MmCDK7基因cDNA序列并进行测序分析,结果表明,MmCDK7基因具有CDK基因家族典型的结构特征,包括S_TKc结构域、TyrKc结构域、T-loop环、ATP结合位点及调节亚基Cyclin的结合部位NRTALRE等。通过与其他物种的CDK氨基酸序列进行多序列比对分析,发现MmCDK7氨基酸序列与虾夷扇贝的CDK7氨基酸序列高度同源(相似性为75.00%),与斑马鱼和黑腹果蝇的相似性最低,说明CDK7基因在文蛤及其他物种间具有一定的保守性,均含有CDK家族的典型结构域。基于CDK7氨基酸序列相似性构建的系统发育进化树显示,文蛤与中国真蛸、长牡蛎、厚壳贻贝及虾夷扇贝等软体动物聚为一支,亲缘关系较近。可见,克隆获得的MmCDK7基因属于CDK家族成员,可能具有类似于其他物种CDK7基因的功能。
细胞分裂活动的有序进行离不开CDK调控,其中,CDK7通过发挥CAK活性在细胞周期调控过程中发挥重要作用(Ganuza et al.,2012)。CDK7基因可特异性调控细胞分裂,本研究通过实时荧光定量PCR检测MmCDK7基因在文蛤成体各组织中的表达情况,结果发现MmCDK7基因表达具有明显的组织表达特异性,以在性腺中的相对表达量显著高于在其他组织中的相对表达量,可能是由于性腺部位的细胞分裂较旺盛,因此MmCDK7基因在性腺部位高表达,与贾复龙等(2016)研究发现CDK7基因在三疣梭子蟹卵巢/精巢中表达量最高的结论一致。此外,MmCDK7基因在文蛤斧足和外套膜中的相对表达量仅次于性腺,可能是由于文蛤属于埋栖型贝类,需依靠斧足的伸缩活动进行潜钻穴居和运动,而外套膜与幼虫贝壳生长关系密切(王如才,2008;王晓梅,2008),也说明MmCDK7基因在这2个组织的生长发育过程中发挥重要作用。
CDK7在生物体正常生长发育过程中也发挥着至关重要的作用。在斑马鱼的早期胚胎发育过程中,CDK7基因在胚胎动物极的各细胞中均普遍存在,且其过表达可补救因Cyclin H失活造成的细胞凋亡或胚胎畸形(储琳等,2006;刘清云,2006);通过基因敲除技术分析CDK7基因在小鼠体内的生理作用,结果发现CDK7基因缺失能导致体外细胞分裂停止及体内早期胚胎死亡(Ganuza et al.,2012);在适宜温度条件下,热带爪蟾发育速度始终快于非洲爪蟾,与CDK7基因表达量高于非洲爪蟾密切相关(Tanaka et al.,2017)。此外,有研究证实抑制CDK7基因表达能阻止多种肿瘤细胞的分裂与增殖(Wang et al.,2016;Sava et al.,2020),因此可将CDK7基因作为癌症治疗的潜在靶点。本研究对MmCDK7基因在红文蛤和黄文蛤不同发育时期的表达情况进行分析,结果发现MmCDK7基因在2种文蛤的多个发育阶段均有表达,以多细胞时期的相对表达量最高,可能是由于这一时期的细胞分裂活动较旺盛,细胞数目增加,需更多的MmCDK7基因来调控细胞有丝分裂。D形幼虫时期是文蛤幼虫消化道形成,开始摄取外源食物,形成斧足和平衡囊等组织器官的关键时期(李太武等,2006;王如才,2008),因此也需要大量MmCDK7基因以满足机体生长所需;之后MmCDK7基因表达下调,至稚贝时期达最低值,究其原因可能是此时文蛤幼虫生长发育趋于稳定,对MmCDK7基因表达需求量相对较低。
目前,针对CDK7基因功能已有相关研究报道。在斑马鱼(刘清云,2006)、斑节对虾(Phinyo et al.,2014)和马氏珠母贝(Adzigbli et al.,2020)等物种中,发现CDK7基因参与其卵巢/精巢发育、免疫调节及胚胎发育过程,但未见CDK7基因与机体生长发育间的关联性研究。本研究在同一养殖条件下的生长试验中发现,除了个别时间点外,红文蛤壳长显著大于黄文蛤,同时结合红文蛤较黄文蛤的壳长相对增长率,可确定红文蛤的生长速度快于黄文蛤,且其生长优势在此次养殖后期更明显。与此同时,红文蛤MmCDK7基因的相对表达量在多数情况下显著高于黄文蛤的相对表达量,与2种文蛤的壳长对比结果相吻合,故推测红文蛤较黄文蛤生长快速可能与其MmCDK7基因表达量水平较高存在一定联系。
4 结论
MmCDK7基因属于CDK家族成员,在细胞分裂旺盛的性腺及多细胞时期的相对表达量最高,且在生长速度较快红文蛤中的相对表达量多数情况下显著高于黄文蛤,故推测MmCDK7基因参与调控文蛤的早期生长发育过程。
参考文献:
储琳,刘清云,钱旻,严缘昌,李逸平. 2006. 斑马鱼cdk7和cyclin H基因的原核表达及多克隆抗体的制备与鉴定[J]. 细胞生物学杂志,28(6):849-854. [Chu L,Liu Q Y,Qian M,Yan Y C,Li Y P. 2006. Prokaryotic expression and polyclonal antibodies preparation of cdk7 and cyclin H in zebrafish[J]. Chinese Journal of Cell Biology,28(6):849-854.] doi:10.3969/j.issn.1674-7666.2006.06.015.
高曉艳. 2015. 文蛤4个生长相关基因克隆、时空表达及与生长的相关性分析[D]. 宁波:浙江万里学院. [Gao X Y. 2015. Cloning,spatiotemporal expression and association analysis of 4 genes with growth traits in hard clam Meretrix meretrix[D]. Ningbo:Zhejiang Wanli University.]
贾复龙,孟宪亮,刘萍,李健,高保全. 2016. 三疣梭子蟹细胞Cdk7基因克隆及其在卵巢发育中的表达[J]. 中国水产科学,23(5):1032-1040. [Jia F L,Meng X L,Liu P,Li J,Gao B Q. 2016. Cloning and expression analysis of Cdk7,a gene involved in ovarian development,from swimming crab (Portunus trituberculatus)[J]. Journal of Fishery Sciences of China,23(5):1032-1040.] doi:10.3724/ SP.J.1118.2016.15401.
江艳. 2010. BmCycH基因及其在家蚕中表达特性的研究[D]. 杭州:浙江理工大学. [Jiang Y. 2010. Study on expression characteristics of BmCycH gene in silkworm,Bombyx mori[D]. Hangzhou:Zhejiang Sci-Tech University.] doi:10.7666/d.y1747463.
李太武,苏秀榕,季延滨,刘保忠. 2006. 不同发育阶段文蛤同工酶基因的表达研究[J]. 海洋学报(中文版),28(5):162-166. [Li T W,Su X R,Ji Y B,Liu B Z. 2006. Stu-dies on the expressions of isozyme during ontogeny of Meretrix meretrix[J]. Acta Oceanologica Sinica,28(5):162-166.] doi:10.3321/j.issn:0253-4193.2006.05.022.
刘清云. 2006. 斑马鱼中囊胚发育相关基因的克隆及功能研究[D]. 上海:中国科学院研究生院. [Liu Q Y. 2006. Cloning and function of genes related to blastocyst deve-lopment in zebrafish[D]. Shanghai:Graduate School of Chinese Academy of Sciences.]
宋文涛. 2013. 文蛤的养殖和研究现状[J]. 山东工业技术,(11):246. [Song W T. 2013. Cultivation and research status of the clam Meretrix meretrix[J]. Shandong Industrial Technology,(11):246.] doi:10.16640/j.cnki.37-1222/ t.2013.11.107.
王超,陈爱华,曹奕,吴杨平,张雨,姚国兴,蔡永祥. 2016. 6个不同海域文蛤地理群体的亲缘关系分析[J]. 海洋渔业,38(3):262-272. [Wang C,Chen A H,Cao Y,Wu Y P,Zhang Y,Yao G X,Cai Y X. 2016. Genetic relationship analysis of six Meretrix meretrix populations from diffe-rent sea areas[J]. Marine Fisheries,38(3):262-272.] doi:10.13233/j.cnki.mar.fish.2016.03.005.
王如才. 2008. 海水贝类养殖学[M]. 青岛:中国海洋大学出版社. [Wang R C. 2008. Science of marine shellfish culture[M]. Qingdao:China Ocean University Press.]
王晓梅. 2008. 文蛤(Meretrix meretrix)幼虫生长发育相关基因的克隆和功能分析[D]. 青岛:中国科学院研究生院. [Wang X M. 2008. Molecular cloning and functional analysis of genes involved in larval development in clam Meretrix meretrix[D]. Qingdao:Graduate School of Chinese Academy of Sciences.]
張洁妤,张静怡,黄孝天. 2013. TFⅡH参与调控RNA聚合酶Ⅱ介导的mRNA转录的研究进展[J]. 南昌大学学报(医学版),53(7):72-75. [Zhang J Y,Zhang J Y,Huang X T. 2013. Advances in studies of TFⅡH in the regulation of RNA polymeraseⅡmediated mRNA transcription[J]. Journal of Nanchang University (Medical Sciences),53(7):72-75.] doi:10.3969/j.issn.1000-2294.2013.07.025.
张雨,陈爱华,吴杨平,曹奕,姚国兴. 2018. 红壳色文蛤选育子代养殖效果分析[J]. 江苏农业科学,46(6):145-147. [Zhang Y,Chen A H,Wu Y P,Cao Y,Yao G X. 2018. Analysis of breeding offspring culture effect of red-shelled Meretrix meretrix[J]. Jiangsu Agricultural Sciences,46(6):145-147.] doi:10.15889/j.issn.1002-1302.2018.06. 037.
Adzigbli L,Zhao Z H,Wang Z M,Yang C Y,Hao R J,Deng Y W. 2020. Characterization of cyclin dependent kinase-7 and its differential response to grafting challenge in the black shell colored selected line of pearl oyster Pinctada fucata martensii[J]. Fish and Shellfish Immunology,101:277-283. doi:10.1016/j.fsi.2020.04.008.
Ebmeier C C,Erickson B,Allen B L,Allen M A,Kim H,Nova F,Jacobsen J R,Liang K,Shilatifard A,Dowell R D,Old W M,Bentley D L,Taatjes D J. 2017. Human TFIIH kinase CDK7 regulates transcription-associated chromatin modifications[J]. Cell Reports,20(5):1173-1186. doi:10.1016/j.celrep.2017.07.021.
Fisher R P. 2005. Secrets of a double agent:CDK7 in cell-cycle control and transcription[J]. Journal of Cell Science,118(22):5171-5180.
Ganuza M,Sáiz-Ladera C,Ca?amero M,Gómez G,Schneider R,Blasco M A,Pisano D,Paramio J M,Santamaría D,Barbacid M. 2012. Genetic inactivation of Cdk7 leads to cell cycle arrest and induces premature aging due to adult stem cell exhaustion[J]. The EMBO Journal,31(11):2498-2510. doi:10.1038/emboj.2012.94.
Larochelle S,Amat R,Glover-Cutter K,Sansó M,Zhang C,Allen J J,Shokat K M,Bentley D L,Fisher R P. 2012. Cyclin-dependent kinase control of the initiation-to-elongation switch of RNA polymerase II[J]. Nature Structural and Molecular Biology,19(11):1108-1115. doi:10.1038/nsmb.2399.
Liu Q Y,Wu Z L,Lv W J,Yan Y C,Li Y P. 2007. Developmental expression of Cyclin H and Cdk7 in zebrafish:The essential role of Cyclin H during early embryo deve-lopment[J]. Cell Research,17(2):163-173. doi:10.1038/sj.cr.7310144.
Lolli G,Johnson L N. 2005. CAK-Cyclin-dependent activa-ting kinase:A key kinase in cell cycle control and a target for drugs?[J]. Cell Cycle,4(4):565-570. doi:10.4161/cc.4.4.1607.
Malumbres M. 2014. Cyclin-dependent kinases[J]. Genome Biology,15:122. doi:10.1186/gb4184.
Martinez A M,Afshar M,Martin F,Cavadore J C,Labbé J C,Dorée M. 1997. Dual phosphorylation of the T-loop in Cdk7:Its role in controlling cyclin H binding and CAK activity[J]. The EMBO Journal,16(2):343-354. doi:10.1093/emboj/16.2.343.
Matthew R W,Geraldine S. 2002. cdk-7 is required for mRNA transcription and cell cycle progression in Caenorhabditis elegans embryos[J]. Proceedings of the National Academy of Sciences of the United States of America,99(8):5527-5532. doi:10.1073/pnas.082618399.
Peissert S,Schlosser A,Kendel R,Kuper J,Kisker C. 2020. Structural basis for CDK7 activation by MAT1 and Cyclin H[J]. Proceedings of the National Academy of Scien-ces of the United States of America,117(43):26739-26748. doi:10.1073/pnas.2010885117.
Phinyo M,Nounurai P,Hiransuchalert R,Padermsak J,Si-rawut K. 2014. Characterization and expression analysis of Cyclin-dependent kinase 7 gene and protein in ovaries of the giant tiger shrimp Penaeus monodon[J]. Aquaculture,432:286-294. doi:10.1016/j.aquaculture.2014.05. 022.
Sava G P,Fan H L,Coombes R C,Buluwela L,Ali S. 2020. CDK7 inhibitors as anticancer drugs[J]. Cancer Metastasis Reviews,39(3):805-823. doi:10.1007/s10555-020-09885-8.
Schachter M M,Fisher R P. 2013. The CDK-activating kinase Cdk7[J]. Cell Cycle,12(20):3239-3240. doi:10.4161/cc.26355.
Tanaka T,Ochi H,Takahashi S,Ueno N,Taira M. 2017. Genes coding for cyclin-dependent kinase inhibitors are fragile in Xenopus[J]. Developmental Biology,426(2):291-300. doi:10.1016/j.ydbio.2016.06.019.
Wang C,Jin H J,Gao D M,Wang L Q,Evers B,Xue Z,Jin G Z,Lieftink C,Beijersbergen R L,Qin W X,Bernards R. 2018. A CRISPR screen identifies CDK7 as a therapeutic target in hepatocellular carcinoma[J]. Cell Research,28(6):690-692. doi:10.1038/s41422-018-0020-z.
Wang Q H,Li M H,Zhang X L,Huang H,Huang J F,Ke J,Ding H F,Xiao J Z,Shan X H,Liu Q Q,Bao B J,Yang L. 2016. Upregulation of CDK7 in gastric cancer cell promotes tumor cell proliferation and predicts poor pro-gnosis[J]. Experimental & Molecular Pathology,100(3):514-521. doi:10.1016/j.yexmp.2016.05.001.
Wood D J,Endicott J A. 2018. Structural insights into the functional diversity of the CDK-cyclin family[J]. Royal Society Open Biology,8(9):108112. doi:10.1098/rsob. 180112.
Wu S Y,McNae I,Kontopidis G,McClue S J,McInnes C,Stewart K J,Wang S D,Zheleva D,Marriage H,Lane D P,Taylor P,Fischer P M,Walkinshaw M D. 2003. Discovery of a novel family of CDK inhibitors with the program LIDAEUS:Structural basis for ligand-induced disordering of the activation loop[J]. Structure,11(4):399-410. doi:10.1016/S0969-2126(03)00060-1.
(責任编辑 陈德元)
