鲥淀不同基质附着细菌群落结构特征及PICRUSt功能预测分析
2021-04-06裘丽萍孟顺龙陈家长
邓 茹,陈 曦,裘丽萍,孟顺龙,陈家长
(1水产科学国家级实验教学示范中心(上海海洋大学)/农业农村部淡水水产种质资源重点实验室(上海海洋大学)/农业农村部鱼类营养与环境生态研究中心(上海海洋大学),上海 201306;2中国水产科学研究院淡水渔业研究中心/农业农村部水产品质量安全环境因子风险评估实验室(无锡)/中国水产科学研究院内陆渔业生态环境和资源重点开放实验室,江苏无锡 214081)
附着细菌是指水体中一类依附在植物、有机碎屑以及其他物体表面上营专性或兼性附着生活的细菌群体[1]。湖泊生态系统中附着细菌的数量可以达到105~107个/cm2,是湖泊水体中重要的初级生产者之一[2]。附着细菌在富营养化水体中具有显著的脱氮除磷作用,而且能促进大分子有机化合物的矿化[3,4],在重金属吸附过程中附着细菌也发挥着重要作用[5,6]。大量附着微生物群体易在载体表面形成生物膜,而在生物膜中载体是核心部分,在环境中发挥着重要的作用,载体的材质、表面粗糙度、强度及密度等因素对水处理效果具有直接的影响[7-10]。
蒋建文等[11]采用PCR-DGGE技术研究了无纺布载体在池塘和水中表面微生物多样性的差异。辛建美等[12]在研究生物膜对罗氏沼虾育苗体系中水体的净化效果中表明生物膜对水体中污染物的去除率较高,其研究仅介绍了生物膜固着生物量的变化并未涉及对生物膜上微生物群落的具体分析。充分了解湖泊生态系统中微生物群落的组成、分布特征和功能,对湖泊生态环境的管理和维护具有重要意义[13,14]。高通量测序技术可以高通量获得特定的DNA片段,全面展示生物群落的组成和结构[15]。目前,国内外研究人员采用该方法研究了微生物群落的组成、分布特征及其影响因素等[16-18]。PICRUSt(phylogenetic investigation of communities by recon- struction of unobserved states) 是一个预测菌群功能和代谢的软件,通过16S rRNA基因序列预测相应细菌和古菌的代谢功能谱[19]。目前PICRUSt功能预测分析很少应用于微生物研究[20,21],与高通量测序技术相结合将为探明微生物群落组成和功能提供重要帮助。
1 材料与方法
1.1 样品采集与附着基情况

1.2 DNA提取与PCR扩增
DNA提取是根据E.Z.N.A.® soil试剂盒(Omega Bio-tek,Norcross,GA,U.S.)的说明书进行操作,用NanoDrop2000检测DNA浓度和纯度,1%琼脂凝胶电泳检测DNA提取质量。 PCR扩增:
(1) 引物序列为338 F(5′-ACTCCTACGGGAGGCAGCAG-3′)和806 R(5′-GGACTACHVGGGTWTCTAAT-3′),对V3-V4可变区进行扩增。
(2) 扩增程序为:95 ℃ 预变性3 min;27个循环(95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s);最后72 ℃延伸10 min(PCR仪:ABI GeneAmp® 9700型)。
(3) 扩增体系20 μL:4 μL 5 FastPfu缓冲液,2 μL 2.5 mmol/L dNTPs,0.8 μL引物(5 μmol/L),0.4 μL FastPfu聚合酶;10 ng DNA模板。
1.3 高通量测序及PICRUSt功能预测
PCR产物用2%琼脂糖凝胶回收,AxyPrep DNA Gel Extraction Kit(Axygen Biosciences,Union City,CA,USA)纯化,Tris-HCl洗脱,2%琼脂糖电泳检测,利用QuantiFluorTM-ST(Promega,USA)进行检测定量。按照Illumina MiSeq(Illumina,San Diego,USA)标准操作规程,将纯化的扩增片段构建PE 2*300文库。委托上海美吉生物医药科技有限公司进行测序。
PICRUSt功能预测是对OTU丰度表进行标准化;然后通过每个OTU对应的greengene id,获得OTU对应的COG(Clusters of Orthologs Groups)家族信息和KEGG Orthology(KO)信息;并计算各COG的丰度和KO丰度。根据COG数据库的信息,可以从eggNOG数据库中解析到各个COG的描述信息,以及其功能信息,从而得到功能丰度谱;根据KEGG数据库的信息,可以获得KO、Pathway、EC信息,并能根据OTU丰度计算各功能类别的丰度。此外,针对Pathway,运用PICRUSt可获得代谢通路的3个水平信息,并分别得到各个水平的丰度表。
1.4 数据分析与处理
测序序列用Trimmomatic软件控制,用FLASH软件拼接。使用UPARSE软件(version 7.1 http://drive5.com/uparse/),按照97%的相似度对OTU序列进行聚类,并在聚类过程中剔除单序列和嵌合体。采用RDP classifier(http://rdp.cme.msu.edu/)对每个序列的物种分类进行注释,并比较Silva数据库(SSU123),比较阈值为70%。Mothur软件对序列进行抽平计算多样性指数Sobs、Shannon、Simpson、Ace、Chao1指数和Coverage。利用QIIME软件及R语言制成稀释曲线、Venn图、主坐标分析(principal co ordinates analysis,PCoA)。运用Microsoft office和SP SS25软件进行数据处理,进行显著性标记。
2 结果与分析
2.1 稀释曲线

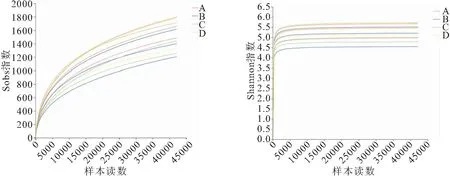
图1 稀释曲线Fig.1 Rarefaction curves of OTU numbers
2.2.1 不同基质细菌群落组成在门水平的分析
通过Silva数据库对所得数据进行比对和物种组成分类,从样品中提取了42 207个微生物基因序列,共检出38门。可发现在门分类水平上(如图2),合并丰度>1%的数值共12门菌群,分别为变形菌门(Proteobacteria)、绿弯菌门(Chloroflexi)、蓝细菌门(Cyanobacteria)、拟杆菌门(Bacteroidetes)、疣微菌门(Verrucomicrobia)、放线菌门(Actinobacteria)、酸杆菌门(Acidobacteria)、浮霉菌(planctomycetes)、WS6、Saccharibacteria、厚壁菌门(Firmicutes)、Deinococcus-Thermns,以上菌门之和在各个样品中占98.39%~98.74%。其中变形菌门是样品中含量最高的门,所占比例分别为棕榈基质45.49%、网片基质36.46%、人工水草基质47.88%、芦苇基质50.80%。绿弯菌门是样品中含量第二高的门,占棕榈、网片、人工水草、芦苇基质的比例依次为20.02%、18.62%、13.04%、16.82%。而蓝细菌为第三大优势菌门,其含量占棕榈、网片、人工水草、芦苇基质的比例依次为14.33%、17.88%、11.32%、5.78%。可发现在芦苇基质上蓝藻细菌含量显著低于其它基质,拟杆菌、WS6、厚壁菌门、Deinococcus-Thermns在芦苇基质上则显著高于其它基质。

图2 门水平上物种相对丰度分布Fig.2 Distribution of relative abundance of species at the phylum level
2.2.2 不同基质细菌群落组成在纲水平的分析
从纲水平上分析,共从样品中提取了42 207个微生物基因序列,可分为91纲。其中相对丰度<1%的有20纲,分别是Alphaproteobacteria(α-变形杆菌纲)、Anaerolineae(厌氧绳菌纲)、Cyanobacteria(蓝细菌纲)、Gammaproteobacteria(丙型变形菌纲)、Spartobacteria、Sphingobacteria(鞘脂杆菌门)、Betaproteobacteria(β-变形菌纲)、Deltaproteobacteria(δ-变形菌纲)、Actnobacteria(放线菌纲)、Acidobacteria(酸杆菌纲)、Caidlineae(暖蝇菌纲)、Chloroflexia(绿屈挠菌属)、Flavobateriia、Norank_p_WS6、Phycisphaerae、Cytophagia(纤维粘网菌)、Norank_p_Saccharibacteria、SM1A07、Clostridia(梭状芽孢杆菌纲)、Deinococci(异常球菌属)。从纲水平上物种相对丰度分布图中可发现不同附着基质上不同属的菌群占比存在差异,且样品中最高不超过3.68%为其它未确定分类的微生物群落(图3),这证明在纲分类水平上Illumina 测序是足够准确的。
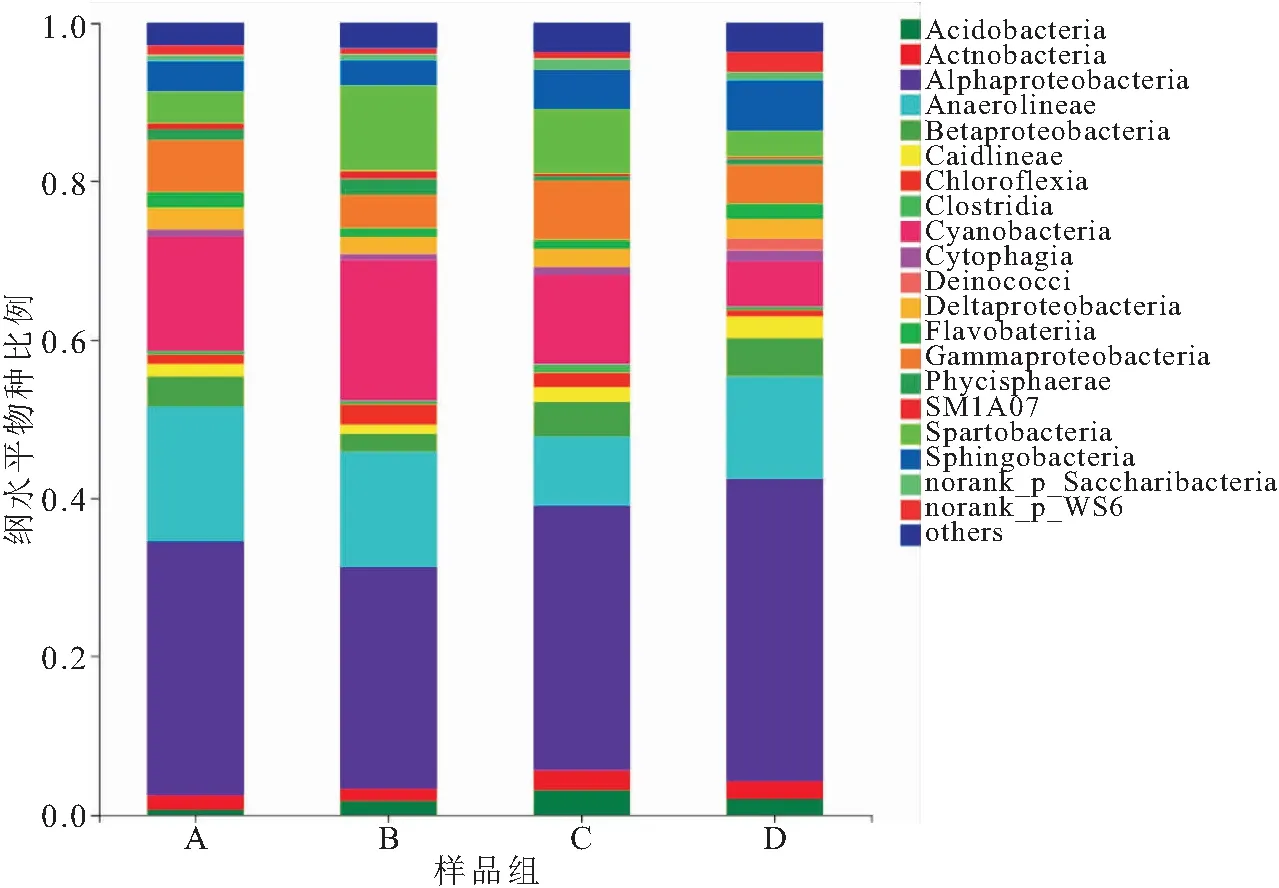
图3 纲水平上物种相对丰度分布Fig.3 Relative abundance distribution of species on the class level
2.2.3 不同基质细菌群落组成在目水平的分析
从样品中提取的42 207个微生物基因序列中可分为183目。如图4所示在目分类水平上,其中相对丰度大于1%的优势目有28个,主要包括Anaerolineales(厌氧绳菌目)、Rhodobacterales(红杆菌目)、Subsectionlll、Rhizobiales(根瘤菌目)、Chthoniobacterales、Sphinomonadales、Sphingobacteiales、Xanthomonadales(黄单胞菌目)、Rhodospirillales(红螺菌目)、Burkholderiales(伯克氏菌目)、Subsectionl、Caulobacterales(柄杆菌目)等。从物种相对分布图中可发现,厌氧蝇菌目在人工水草基质上比其它基质低,红杆菌目在人工水草基质和芦苇基质上的占比相似且均高于棕榈片和网片基质;芦苇基质上Subsectionlll和Chthoniobacterales的占比分别为3.64%和3.24%均显著低于其它基质占比。
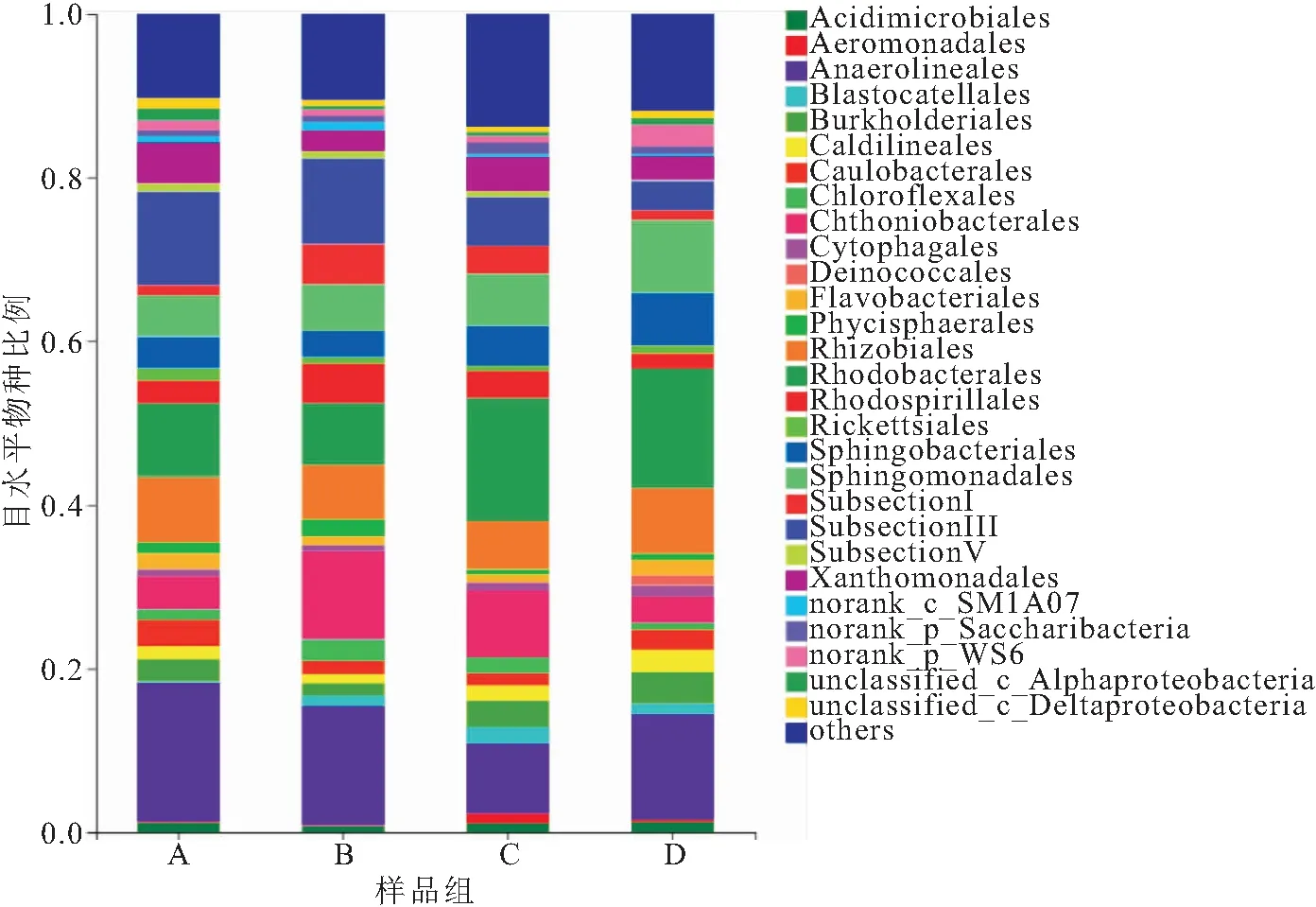
图4 目水平上物种相对丰度分布Fig.4 Relative abundance distribution of species on order level
2.2.4 不同基质细菌群落组成在科水平的分析
通过高通量测序不同基质附着样品一共分为338科,从科水平上观察发现厌氧蝇菌科在棕榈片基质和网片基质上的平均相对丰度最高,在人工水草基质和芦苇基质上的平均相对丰度次之;而红杆菌科在人工水草基质和芦苇基质上的平均相对丰度最高,在棕榈片基质和网片基质上的平均相对丰度次之。除此之外,主要包括Familyl_O_Subsectionlll、LD29、Sphingomonadaceae(鞘脂单胞菌科)、Xanthomonadaceae(黄单胞菌科)、Erythrobacteraceae(赤杆菌科)、Saprospiraceae(腐螺旋菌科)、Hyphomonadacea(生丝单胞菌科)、Hyphomicrobiaceae(生丝微菌科)、Caldilineaceae(暖蝇菌科)、Comamonadaceae(丛毛单胞菌科)、Methylobacteriaceae(甲基杆菌科)、Chitinophagaceae等,通过物种相对丰度分布图(图5)可发现不同基质附着细菌相对丰度在科水平上表现出显著差异。
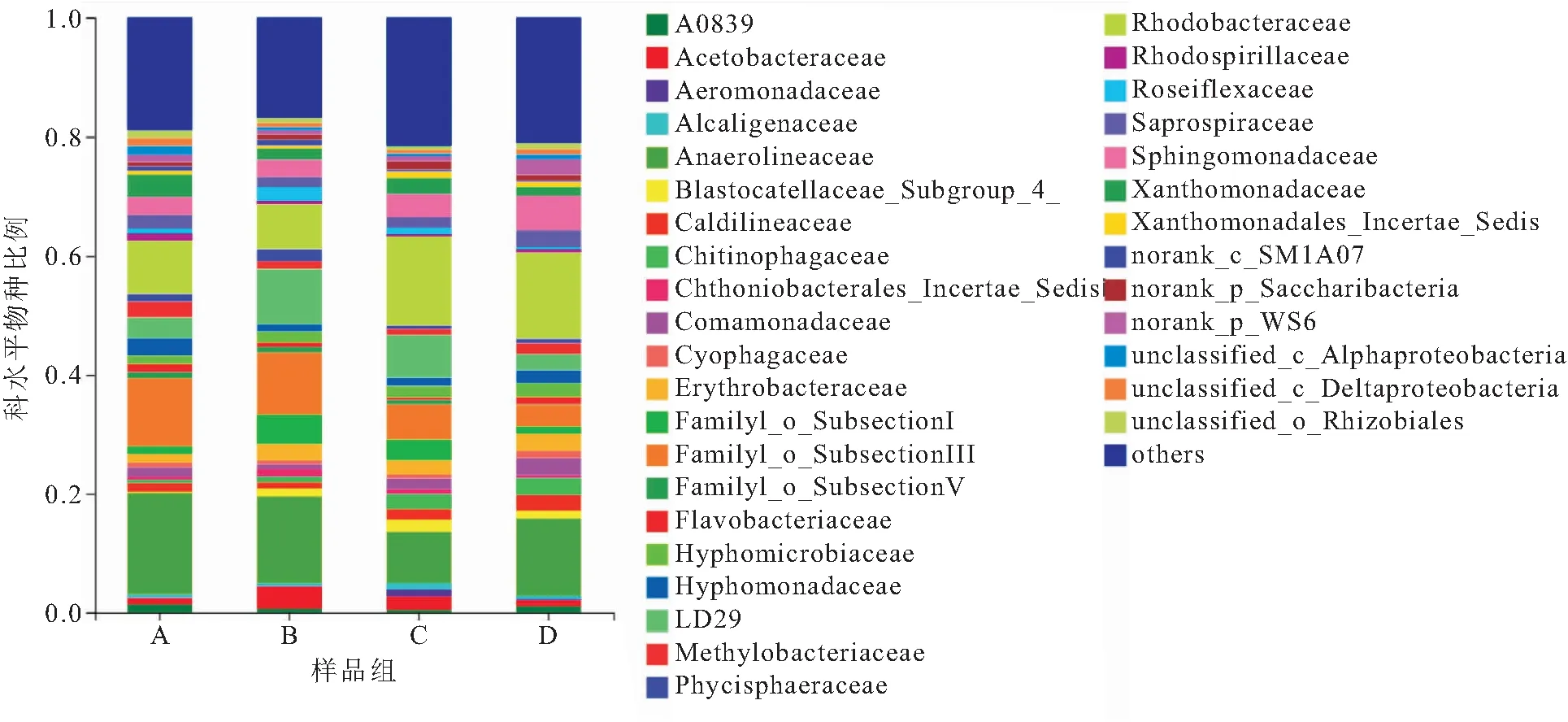
图5 科水平上物种相对丰度分布Fig.5 Relative abundance distribution of species at the family level
2.2.5 不同基质细菌群落组成在属水平分析
样品测序共检测到646属,通过Venn图可以比较直观地展现不同基质附着样本中物种组成相似性及重叠情况(如图6)。从图6中可发现四种不同基质附着细菌大部分存在重叠菌属,其中有435属是属于四种基质共有的,而单独存在于棕榈片基质、网片基质、人工水草基质、芦苇基质上的菌属分别有10个、9个、14个、29个。从总的物种数目柱形图可发现四种不同基质附着细菌属从高到低依次为:芦苇基质>人工水草基质>网片基质>棕榈片基质。
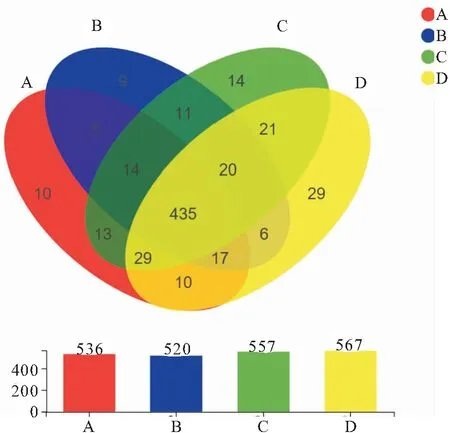
图6 属水平细菌Venn图Fig.6 Venn diagram of a genus of horizontal bacteria
2.3 鲥淀不同基质附着细菌多样性分析
2.3.1 不同基质附着细菌α-多样性分析
通过测序数据对结果进行比对,并对样品进行α-多样性分析,得出多样性指数统计表(表1)。根据表1可以看出所有样品的覆盖度均在98.7%以上,说明测序数据是有效可靠的,可以作为分析的依据。Sobs指数指的是实际观测到的OTU数目,Shannon指数和Simpson指数主要是用来衡量物种的多样性,Ace指数和Chao1指数主要是用来衡量
物种丰度,Coverage指数主要用于检测物种覆盖度。由表1可以看出,Sobs指数OTU数目平均大小为棕榈片基质>芦苇基质>人工水草基质>网片基质;从Shannon指数和Simpson指数反映出不同基质多样性大小依次为芦苇基质>棕榈片基质>人工水草基质>网片基质;从Ace指数和Chao1指数可看出不同基质附着细菌丰富度依次为棕榈片基质>芦苇基质>网片基质>人工水草基质。
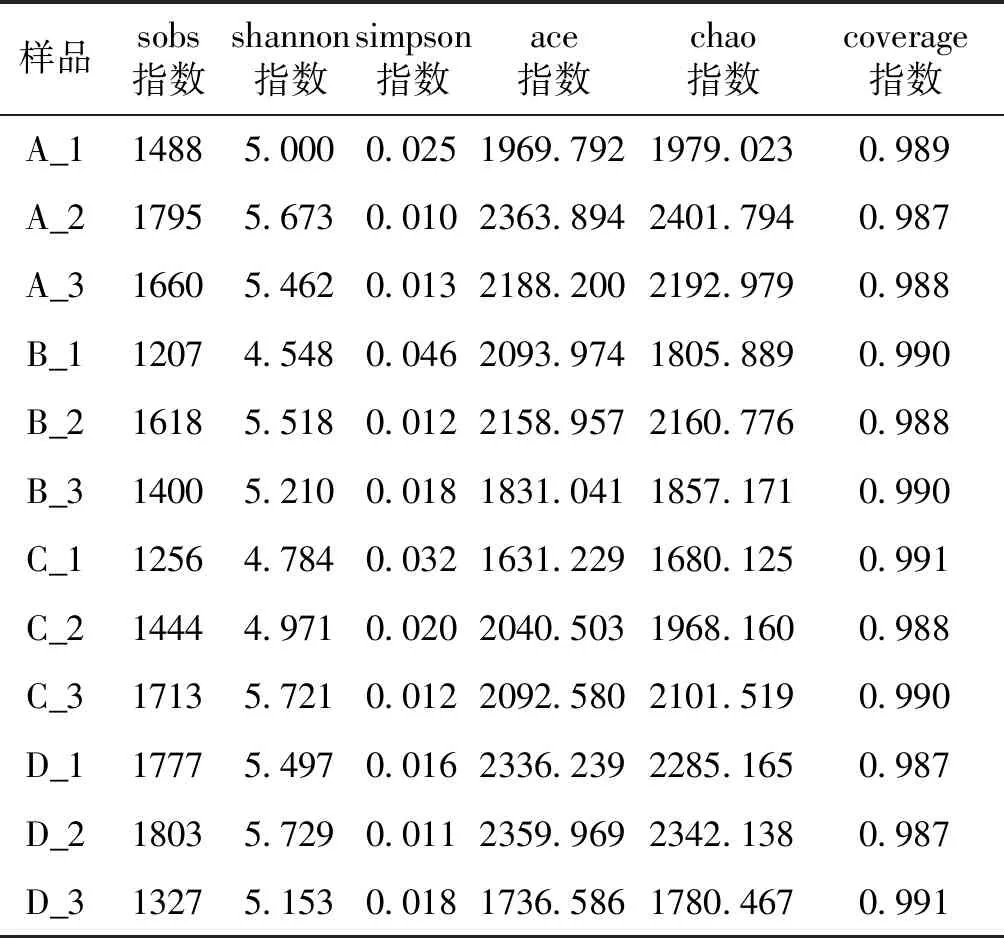
表1 附着细菌的多样性指数分析Tab.1 Analysis of diversity index of attached bacteria
2.3.2 不同基质附着细菌β-多样性分析
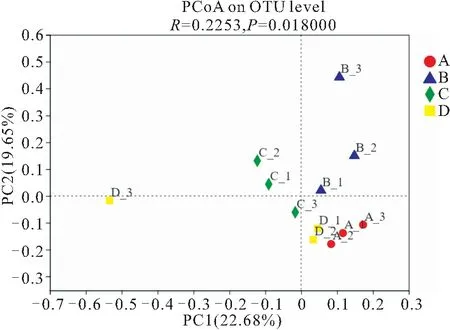
图7 附着细菌多样性的主坐标分析Fig.7 Principal coordinate analysis of the diversity of attached bacteria
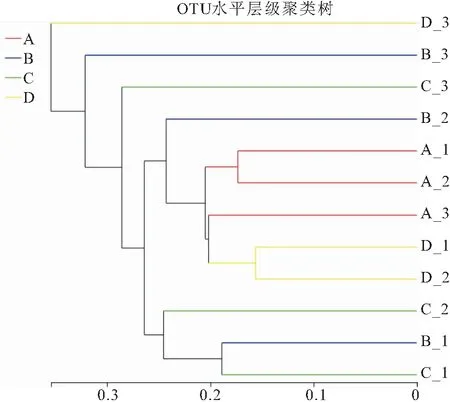
图8 不同样品 UPGMA 聚类图Fig.8 UPGMA cluster diagram of different samples
2.4 附着细菌群落功能预测分析
为了获得附着细菌在不同基质上的功能,本研究使用PICRUSt软件对细菌种群进行预测。基于COG数据库的预测结果显示(如图9所示),共涉及22个基因功能家族,显示出功能上的丰富性。包括氨基酸运输和代谢;细胞膜/细胞壁/膜结构的生物合成;能量转换和产生;信号转导机制;无机离子转运与代谢;碳水化合物的运输和代谢;重建、复制和修复;翻译、生物合成和核糖体结构;转录;蛋白质转换、翻译后修饰、伴侣蛋白;脂质运输和代谢以及辅酶的运输和代谢共十二大类功能基因家族为主要功能基因家族,占比之和达70%以上,未能很好注释的基本预测功能家族占比为8.39%~8.73%和未知功能基因家族占比为10.13%~10.61%。

图9 不同基质附着细菌PICRUSt功能预测Fig.9 PICRUSt function prediction of bacteria attached to different substrates
3 讨论
3.1 不同基质附着细菌群落组成及多样性


3.2 PICRUSt功能预测分析

4 结论

(2)对四种基质α-多样性分析发现附着细菌多样性大小依次为芦苇基质>棕榈片基质>人工水草基质>网片基质,丰富度依次为棕榈片基质>芦苇基质>网片基质>人工水草基质。
(3)PCoA分析和基于Bray_curtis的UPGMA聚类分析显示,棕榈片基质与芦苇基质的附着细菌群落结构相似,而网片基质与人工水草基质相似。
(4)采用PICRUSt软件进行的菌群预测分析表明,主要的COG功能包括氨基酸运输和代谢、细胞壁/细胞膜/膜结构的生物合成、能量产生和转换、信号转导机制、无机离子转运与代谢、碳水化合物的运输和代谢等共涉及22个功能基因家族,表现出功能上的丰富性。
