结合多维度特征的病理图像病灶识别方法
2021-03-23胡伟岸邹俊忠郭玉成
胡伟岸,邹俊忠,郭玉成,张 见,王 蓓
1.华东理工大学 信息科学与工程学院,上海 200237
2.清影医疗科技(深圳)有限公司,广东 深圳 518083
目前,通过病理检查对癌症进行诊断是一种常用的方法,它能够提供明确的疾病诊断,指导病人的治疗。对病理图像进行人工分析本身是一件非常有挑战性的工作,一张病理切片通常包含数百万个细胞[1],一名病理科医生一天需要分析许多病理图像,这给他们带来很大的工作负担,疲劳阅片现象时有发生[2-3]。同时,该领域内专家的培养速度赶不上病例的增加速度,将有限的、珍贵的人力资源大量投入到重复的病理图像的识别诊断中是非常可惜的。通过卷积神经网络[4](Convolutional Neural Network,CNN)快速识别病理图像中的病变区域是本文的主要研究内容。
CNN 是一种高效的学习方法,局部连接和权值共享的特点降低了网络模型的复杂程度,减少了需要学习的参数。将计算机辅助诊断应用于数字病理图像已经得到了研究:在Cossatto等人[5]的研究中,采用人工提取特征,多实例学习的半监督胃癌检测与诊断方法;Spanhol等人[6]的研究中,在BreaKHis乳腺病理数据集上对图像提取了6种特征描述,之后使用机器学习方法对图像进行分类,最终整体准确率达到85%;Ciresan 等人[7]使用有监督的深度神经网络作为分类器,以88%的查准率及70%的查全率赢得了ICPR 2012有丝分裂检测竞赛;王莹等人[8]使用Alexnet及GoogLeNet对临床结直肠病理图像进行图块分类,获得91%的整体准确率。在Sharma等人[9]的研究中,使用了卷积神经网络,对小样本胃癌数据集的三个恶性级别进行了分类,并取得了良好的效果;Li 等人[10]设计了一种基于CNN 的胃癌自动识别框架,获得了97%的图块级别准确率;基于多种结构的融合模型在胃癌病理图像上取得了较好的分类结果[11];Coudray等人[12]使用基于Inception网络的架构,在对肺病理图像中肺组织及肺癌进行区分任务中获得良好的效果;迁移学习的应用在乳腺病理图像的自动分类中获得91%的图块识别准确率[13];以上的研究均证明了卷积神经网络在数字病理图像研究中的实用性。然而,在上述研究中仍存在一些问题,影响了计算机辅助治疗在实际中的应用,包括:手动提取图像特征的过程复杂;网络模型参数多,训练过程耗时;准确率有待提高。
另外,出于多种原因,如染色原料差异、制备方法差异、图像保存时间长短等,病理图像所呈现的颜色都会有所不同。为使得网络在学习时更加关注细胞的形态、结构和排列等特征,期望图像在输入网络前表现出较一致的颜色。使用标准化染色方案和自动染色机可以通过产生更准确和一致的染色来改善染色质量。但是,很难避免所有的外在因素,使最终的染色完全一致[14]。
针对上述问题,本文在预处理阶段采用了染色校正的策略,在不改变原有形态特征的情况下,使图像表现出更加一致的颜色;受文献[15]启发,本文提出一种结合多维度特征的卷积神经网络模型以自动提取病理图像特征,将CNN 中包含纹理等信息的低维特征与包含丰富抽象语义信息的高维特征相结合,使用深度可分离卷积(Depth Wise Separable Convolution)降低模型参数量,减小了计算复杂度,并使用批量规范化(Batch Normalization,BN)及残差连接(Residual Connection)避免梯度消失,改善输出结果。实验结果表明:采用染色校正的处理方法能够提高预测准确率;本文所提出的CNN模型能够自动提取图像特征,且参数少训练快,最终能够达到较好的识别性能。在胃病理病灶识别任务中准确率达到98.67%,TCGA肝癌病理病灶识别任务中准确率达到97.4%,且假阳性及假阴性均较低,在病灶识别任务中能够媲美主流CNN模型所达到的效果。
1 方法
传统的机器学习方法需要以人工提取特征为前提,特征的提取需要设计者具有深厚的专业背景及实际操作经验。然而,使用深度学习的方法能够直接从原始数据中提取特征,LeCun等人[4]提出了卷积神经网络,使用卷积核对输入图像进行多层卷积操作,逐层学习并在更高层直接对抽象的特征进行整合,最终通过图像的高层表征对其做出分类。目前优秀的深度学习分类模型 有 Vgg16[16]、Inception V3[17]、InceptionResnet V2[18]、Resnet50[19]、Densenet169[20]等。以上模型大多使用最后一个卷积层的feature map 来进行分类,它们的缺点是往往在关注网络最后一层特征时忽略了其他层的特征。同时,上述模型在分类任务中能够取得较好的结果,通常是以消耗计算量和时间为代价的,这样以计算资源换取效果的策略在实际应用时常会受到局限。
有研究证明,深度学习模型的各层特征图侧重于不同类型的信息:由于所经过的卷积网络层较少,模型的低层特征图包含较多边缘、纹理等信息;在经过更多的卷积操作后,高层特征包含更多抽象的语义信息;结合浅层特征可以在一定程度上提升模型精度,将不同特征高效融合,是改善模型性能的关键[21]。因此,本文选择将多个维度的特征从top-down方向上采样为同一尺寸后进行融合。
1.1 模型架构
本文所提出的结合多维特征的病灶识别模型结构如图1所示。

图1 结合多维特征的病理图像病灶识别模型
图1 中包含三个模块:Bottom-top 特征学习模块、Top-down特征叠加模块及Modeloutput模型输出模块。
Bottom-top特征学习模块主要由普通卷积、深度可分离卷积及残差学习组成,通过多层卷积自动提取多维度特征。在普通卷积后依次添加BN层及线性整流函数(Rectified Linear Unit,ReLU)。
在对输入图像进行两次普通卷积后,改用深度可分离卷积继续提取特征。在一般卷积中,卷积核对特征图的三个维度进行学习。其中两个维度处于特征图的平面(高和宽),另一个维度处于特征图的通道方向(深度),卷积核能同时学习到特征图的平面相关性和通道相关性。将平面相关性和通道相关性分离开来,是Inception模块的重要思想[13]。在深度可分离卷积中,将卷积操作分为两个部分:首先将所有2维的卷积作用在特征图的每个单层通道上,平面相关性和通道相关性即达到了完全分离的效果,此步骤称为depth-wise卷积;然后使用1×1的卷积核对新的特征空间进行映射,此步骤被称为point-wise 卷积,如图2(a)所示。使用深度可分离卷积能够降低模型参数。假设输入通道数为N,长宽均为Di,且输出通道数为M,长宽均为Do,卷积核的尺寸设置为k,使用普通卷积时参数量为:

在使用深度可分离卷积过程中,depth-wise 卷积参数量与point-wise卷积参数量分别为式(2)、(3)所示:

深度可分离卷积与普通卷积的参数量之比为:

由式(4)可以看出,两者的比值与输出通道及卷积核大小相关,在本文所使用模型中,M的取值包括128、256、512及1 024,卷积核尺寸k均设置为3。因此,所使用的的深度可分离卷积参数量是普通卷积参数量的0.118、0.115、0.113及0.112倍。较少的参数量节省了模型训练及测试时间。
对于卷积神经网络,如果只是简单地堆叠网络层以增加网络深度,容易导致梯度弥散和梯度爆炸。为避免该问题的发生,在本文网络模型中增加了BN层及Residual connection。文中所使用的Residual connection 结构如图2(b)所示。为保证F′(x)与F(x)在相加时尺寸相同,使用简单的1×1 大小卷积层(步长为2)及全零填充(padding=same)对F′(x)的通道数进行匹配。在Residual connection中,F(x)是求和前网络映射,F′(x)是输入x的简单映射,保留了x的大部分原有信息,三者关系如下:

Top-down 特征叠加模块中,在自顶向下的方向对特征进行上采样,并与左侧相邻的残差输出进行连接,如图2(c)中的1×1 卷积被使用以匹配特征图的通道数量,本文中设置卷积核个数d=512。之所以选择将每个残差块的输出作为特征结合的因子是因为这些输出具有每个阶段最具代表性的特征,同时,邻近的特征较为相似并且重复使用这些特征会使得计算量成倍增长。由于网络的前两层卷积所包含浅层特征与原始图像较为相似,且尺寸过大,故不将其作为特征结合的因子。

图2 部分模型结构
特征图上采样能够使卷积核细致学习病理图像中的小特征。特征图上采样的处理可以简单理解为增加了特征映射的分辨率,使用相同卷积核在放大后的特征图进行操作时可以获取更多关于小目标的信息。
特征相加的方式采用主元素相加(add)而非特征图堆叠(concat)。add 方式更加节省参数和计算量,如果使用concat,特征图通道数会是add方法的两倍,如此计算量会是一笔较大的开销。
在Modeloutput 模块中,添加了全局平均池化层(Global Average Pooling,GAP),使用GAP 可以对前一层的每一个特征图求平均值,保留全局信息,同时避免了全连接层(Fully Connected layer,FC layer)训练参数过多,耗时较长的缺点。在GAP 层后添加2 维的输出层,激活函数使用softmax,softmax的计算公式如下:

其中,Vi表示前级第i个单元的输出,i表示类别索引,总的类别个数为k。Si表示的是当前元素的指数与所有元素指数和的比值。通过这种方法,可以将神经元的输出映射到(0,1)区间内,当成概率来理解。对应于softmax,采用交叉熵(CrossEntropy)作为损失函数,当输出为两类时,交叉熵公式如下:

其中,y代表真实样本标签,代表样本标签为1的概率。
表1 给出了本文深度神经网络主要层的参数设定以及输出。
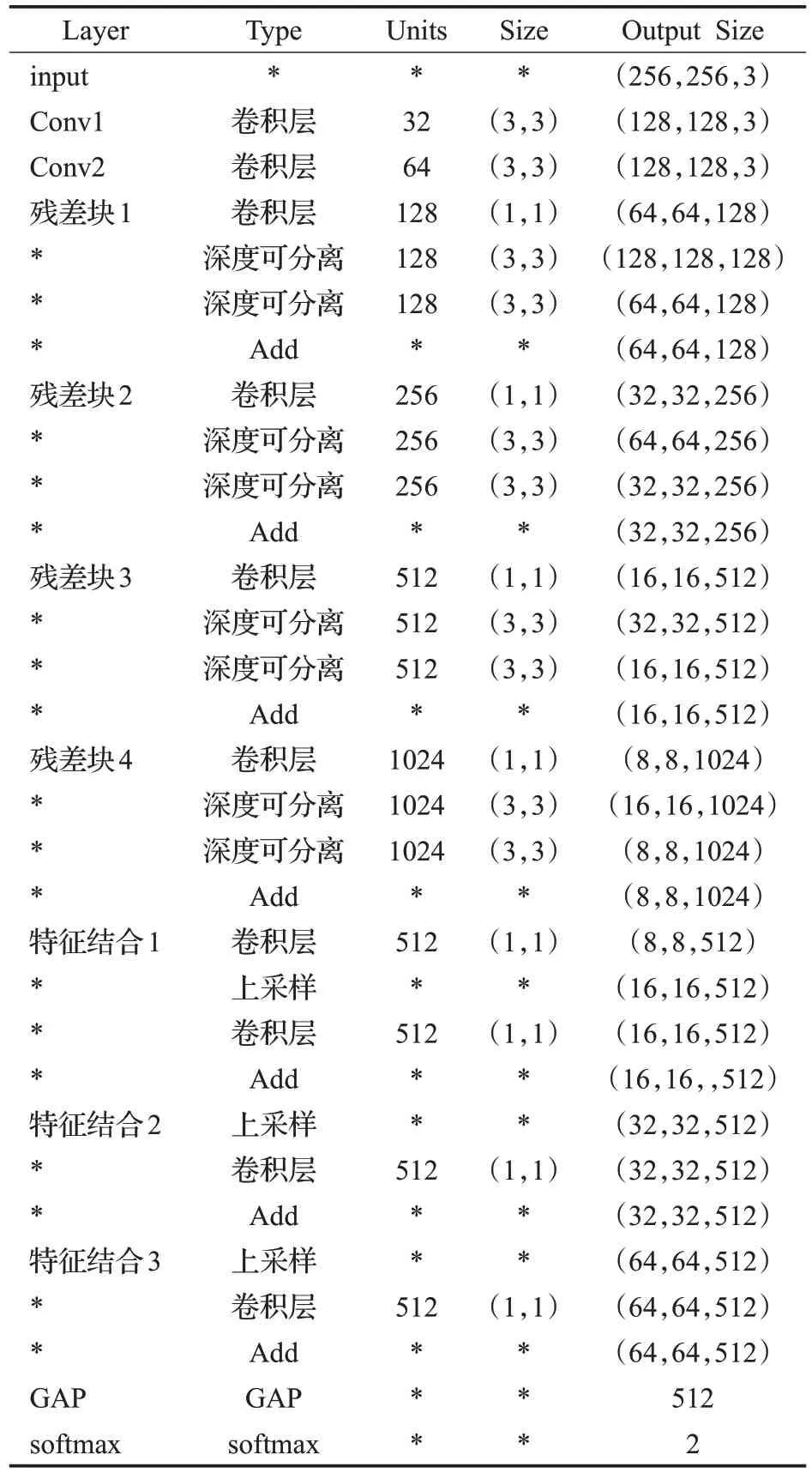
表1 深度神经网络每层参数以及输出
在每层卷积层后分别包含BN 层,ReLU 及最大池化层。
1.2 整体流程
本文所提出的病理图像病变区域检测的主要流程如图3所示。
模型训练阶段,计算机接收数字病理图像,在专家标注内进行图像切割,随后对切割出的图块进行染色校正的操作,再对整个图块数据集进行数据增强,以提高模型的泛化能力,最后将图块数据输入所建CNN 模型进行训练。
在对数字病理图像进行预测的过程中,首先自适应提取感兴趣区(Region of Interests,ROI),在ROI 内进行图像切割;再进行染色校正操作;然后使用CNN对各图块进行预测,计算图块属于病变区域的概率;最后根据图块坐标和预测概率在原始数字病理图像中进行标记,辅助医生进行最终诊断。

图3 检测方法流程图
自适应ROI提取:使用掩膜进行分离是提取ROI的一种可靠方法。在本研究中,观察到不同病理图像呈现的主体颜色存在差异,因此无法直接使用固定阈值对所有图像进行前、背景分离。本研究中采用大津阈值法[22](Otsu)自适应计算各图的阈值T,Otsu也称最大类间方差法,方差是灰度分布均匀性的一种度量,背景和前景之间的类间方差越大,说明构成图像的两部分的差别越大,当部分前景错分为背景或部分背景错分为前景都会导致两部分差别变小。因此,使类间方差最大的分割意味着错分概率最小。以阈值T作为界限将灰度图像分为前景和背景。自适应计算方法公式如下:
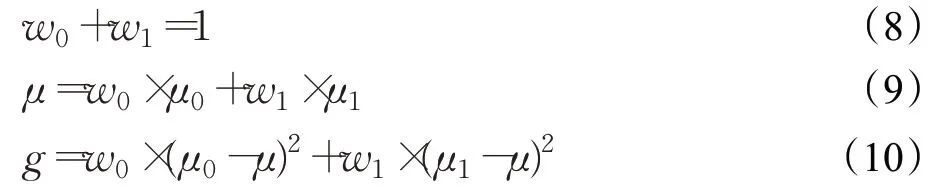
其中,w0、w1分别代表前景和背景像素占整幅图像的比例;μ0、μ1、μ分别代表前景、背景和整幅图像的平均灰度;g表示类间方差。将公式(9)代入公式(10),得到以下公式:

通过不断调整阈值T,使类间方差g获得最大值,对图像进行前、背景分离。
自适应提取ROI 过程:计算原始图像的灰度图;使用大津阈值法获得掩膜;使用形态学闭运算(先膨胀再腐蚀),祛除掩膜中小黑点;剔除面积占整图比例较小区域;获得ROI。
滑动窗图像切割:在标注及ROI 内,从上向下依次滑动,保存滑动窗口内的图块作为后续CNN 的输入数据,滑动窗口的边长与步长相同,为1 024 像素。如果图块处于标注的病变区域内,则将其标记为阳性,标签为1;如果图块处于标注的正常区域内,则将其标注为阴性,标签为0。
数据增强:由于期待卷积神经网络在图块中提取的特征应该是方位不变的,数据增强包括对图块进行旋转、水平或垂直翻转、边缘裁剪和等比例缩放。染色校正会对图像进行色彩上的调整,为保留图像原本的纹理特征,数据增强过程未对图像的亮度和对比度做任何额外的调整。
染色校正:染色校正的目的是降低各图像之间的颜色差异,使得卷积神经网络着重学习病理图像中细胞的排列和结构,组织的纹理等特征。本研究中采取Reinhard颜色迁移方法[23]将模板图像的内部颜色特征应用于目标图像。RGB 各通道之间存在着一定的相关性,这意味着,如果要对图像的色彩进行处理,常常需要对像素的三个通道同时进行修改才不会影响图像的真实感,这将大大增加颜色调整过程的复杂性。
因此,在RGB 色彩空间下进行色彩变化会比较复杂,得到的视觉效果也不自然。本研究中,首先将各图像转移到Lab 色彩空间中,Lab 色彩空间不仅基本消除了颜色分量之间的强相关性,而且有效地将图像的灰度信息和颜色信息分离开来。所以可以分别对三个通道图像进行独立的运算,而不需要修改另外两个通道的信息,从而不会影响原图像的自然效果。色彩空间转换过程如下:

其中,R、G、B代表像素点RGB 通道分量;L、M、S代表像素点LMS 通道分量;l、α、β代表像素点Lab 通道分量。
此方法的主要思想是根据图像的统计分析确定一个线性变换,使得目标图像和模板图像在Lab色彩空间中有相同的均值和方差,计算公式如下:
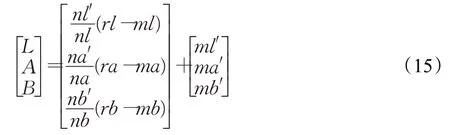
其中,rl、rα、rβ分别是源图像Lab通道原有的数据,L、A、B分别是变换后得到新的源图像Lab通道的值,ml、mα、mβ和ml′、mα′、mβ′分别是源图像和着色图像的三个颜色通道的均值,nl、nα、nβ和nl′、nα′、nβ′表示它们的标准方差。在获得L、A、B后,通过公式(12)~(14)获得染色校正后新图像的RGB通道数值。
经过染色后的图像在不改变原有纹理特征的情况下,使得RGB各通道有着更为相似的分布,有着类数据归一化的作用。图4所示为染色前后的图像对比图。
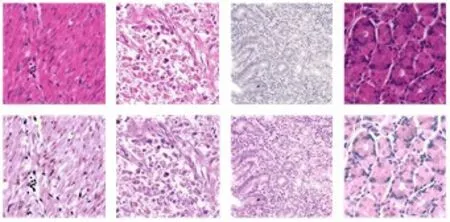
图4 染色效果图
2 实验结果与分析
实验时,所使用的 CPU 型号为 3.4 GHz、16 核AMD1950x,内存容量为 64 GB,GPU 为 12 GB 显存NVIDIA TITAN-V。软件方面,使用Python作为设计语言,配合openslide、opencv 等视觉库进行代码编写。深度学习模型框架使用Keras 以完成对模型的构建、训练与预测。
实验过程中使用了不同数据集对本研究中的网络模型进行了验证,包括胃病理数据集和TCGA肝癌公开数据集。为了定量地评估染色效果及不同算法的性能,采用了以下评价指标:分类准确率Accuracy,敏感性Sensitivity,特异性Specificity。
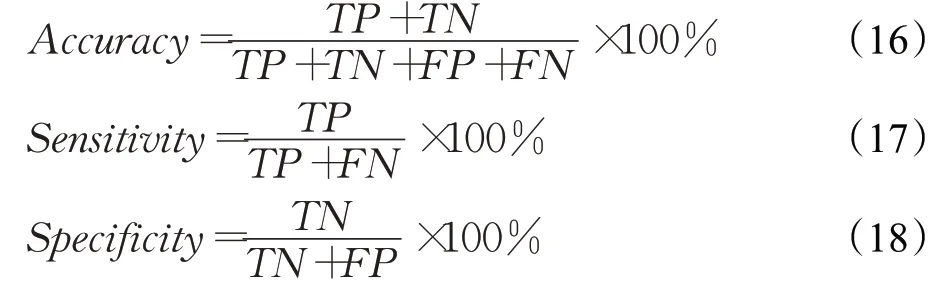
其中,TP为真阳性图块数量,TN为真阴性图块数量,FP为假阳性图块数量,FN为假阴性图块数量。
2.1 特征可视化
本次研究中,为探究不同维度特征在病灶识别中的意义,将Bottom-top模块中常规卷积层及各残差块中最后深度可分离卷积层的输出特征进行可视化,如图5所示。
图5 中,左侧为进行染色校正后的病理图块;右侧为对当前图块进行不同卷积后的可视化特征图,每次卷积取两层特征图作为示例。为突出特征图的细节,将其颜色风格设置为紫红色以凸显对比度。可以观察到,在Con1、Conv2中,由于卷积处于浅层位置,特征图中仍能保留原始图像中的较多信息,大量纹理特征被学习;随着网络加深,Block1中特征图经过下采样,分辨率降低,但与上层特征仍然较为相似,这也是本文网络选择将此处特征图作为特征结合最后一项因子的原因;Block2中,纹理信息淡化,边缘、轮廓等特征逐渐增强;Block3中基本无法观察到纹理信息,细胞及组织的边缘、轮廓信息愈加明显;网络不断加深使得特征更加抽象化,Block5中生成对分类有较强决策意义的语义特征。

图5 特征图可视化
病理医生在对图像进行诊断时需要不断地调整视野及关注区域,需对细胞的轮廓及大小,组织边缘的光滑程度,纹理的排列特征等进行观察,最后整合信息形成判断。从特征可视化图像可以观察到,CNN 对图像进行学习的方式与人工判断的方式有着相似之处,不同维度特征在病灶识别任务中具有实际意义。
2.2 胃病理数据集
本次研究中的胃病理数据集来自于国内某三甲医院,共包含355 张tif 格式的全视野数字病理切片(Whole Slide Images,WSIs),所有切片均使用 HE(Hematoxylin-Eosin)染色法进行着色,且以40X的倍率进行数字化。专业病理科医生在ASAP 标注软件中进行标注,所有标注经过另一名医生复核。
以8∶2 的比率将WSI 分为两部分,其中284 张作为训练集的原始数据,另外独立的71 张作为验证集原始数据。经过切割后的图块数据分布如表2所示。

表2 数据分布表 张
训练过程中,初始学习率设置为0.000 1,使用Adam优化器优化训练,与其他自适应学习率算法相比,Adam的收敛速度更快,且能够避免损失函数波动大的问题。Batchsize 设置为64,即每轮随机输入图64 张。设置迭代次数epoch 值为20。刚开始训练的时候使用较大的学习率,能够避免模型陷入到局部最优;在模型的效果趋于平缓后,使用衰减后较小的学习率,使模型能够收敛到最优值。
将图1本文模型的Top-down叠加模块去除(即Bottomtop 输出直接连接到 Modeloutput),记为 Modelb。在染色校正后的数据集上与本文模型进行对比,对比效果如表3所示。
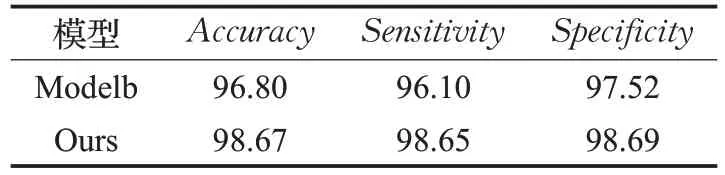
表3 特征叠加效果对比 %
由表3 可知,在添加特征叠加模块后,预测准确率提高了1.87%,这得益于多维度特征结合时对各层次特征的联合学习以及上采样后对细小特征的精细学习。
另选取多种主流CNN模型及其他方法进行实验对比,各模型对独立的验证集图像进行预测的信息如表4所示。

表4 染色校正前后各模型效果对比
可以发现,在对病理图像的图块进行预测时,各深度学习方法都能够获得较高的准确率,均高于95%。在同等条件下,本文所建立的模型能够获得更好的预测效果,InceptionResnet V2 所达到的准确率与本文模型相似,但是训练参数量却高达14 倍之多。表4 中,使用相同的网络模型且加入染色校正的预处理方法后,各性能指标均有提升,各模型预测准确率最多提高了3.42%。染色校正能够降低各图像之间的颜色差异,使模型能够着重于学习细胞的形态、结构和排列等特征。在对图块进行预测前仍进行了染色校正的预处理操作,所以网络查找相关特征不会受到颜色差异的影响。使用染色校正方法后,本文模型在验证集上可以获得最高98.65%的灵敏度,同时特异性达到98.69%。虽然特异性没有达到所有模型中的最高,但是灵敏度作为一个非常重要的指标,能够反映模型检测出病变区域的能力。越高的灵敏度值说明漏诊率的下降,这对于病人提早发现病情,接收早期治疗有着重要意义。特异性高说明模型未将正常区域错分为病变区域。本文模型对染色校正后的图块预测准确率最终为98.67%,这已经达到了相当高的准确率。结合各模型中最高的敏感性98.65%,本次研究中所应用的方法能够为病理科医生提供可靠的诊断基础。
本次研究中,在完成每轮(epoch)训练后使用当次训练所得参数对验证集图像进行测试。图6 所示即为染色后的图块验证集在各模型上的准确率-时间曲线。为直观体现各模型在耗时上的对比,横轴设置为时间轴,代表在整个过程中获得对应坐标点所经历的训练与验证时间总和。对图6观察可知,对于相同规模的图像数据集,本文模型在训练及验证时长上远低于其他模型。本文模型每训练1 个epoch 约需40 min,其余模型每训练1 个epoch 的时间在45~73 min 内。由于每个模型都需要训练多次,因此本文模型能够减少大量训练时间。由图6 可以发现,本文模型在第11 个epoch,约440 min时达到最佳效果,其余模型中最快在600 min时才能达到最高的准确率。

图6 染色后图像验证集准确率曲线
综上所述,本文模型在病灶识别任务中准确率高,并且较其他方法有更强的病灶检出能力,同时,模型参数少极大节省了训练时间。在图像不断累积,模型参数需要不断更新的现实情况下具有实际应用意义。
2.3 TCGA肝癌公开数据集
为进一步验证本文模型在不同种类病理上的适用性,对肝癌数据集进行了测试。癌症基因组图谱(The Cancer Genome Atlas,TCGA)收录了各种人类癌症的临床数据,选择其中的肝癌病理图像作为实验对象。TCGA肝癌数据集共包含379张svs格式的WSI,所有图像标注由专家提供并复核。选择302 张WSI 作为训练集,另外77 张WSI 作为验证集。在标注内对图像进行切割后,数据分布如表5所示。

表5 数据分布表 张
经图像切割后,使用染色校正方法对数据进行处理,将染色后的图块输入网络进行训练和预测。模型参数设置与上文相同。对验证集进行预测后的混淆矩阵如表6所示。
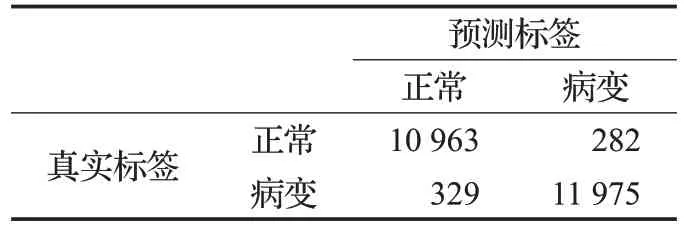
表6 验证集混淆矩阵
由混淆矩阵可以得出,本文模型在TCGA肝癌数据集上的准确率可以达到97.4%,敏感性达到97.3%。数据证明,本文方法可以在不同的病理图像上获得良好效果。
3 结论
为了提高病理图像病灶识别的准确率及效率,提出了一种结合多维度特征的病理图像病灶识别方法。本文中的模型以深度可分离卷积为基础,降低了模型的训练参数量;在Bottom-top 提取特征后,将多维度特征在Top-down 方向进行上采样并进行叠加,使低维特征与高维语义特征进行关联,特征上采样能够使卷积学习到图像中更精细的特征,叠加特征可以提高特征利用率;最终获得的模型在训练参数量上较主流模型得到大幅降低。同时,本研究在预处理过程中采用染色校正的方法,降低了不同图像的颜色差异。本文在胃病理数据集及TCGA 肝癌公开数据集上验证了所提模型的性能。实验结果表明,所提出的模型与其他方法相比,具有参数少,训练快,准确率高的优点,能够更准更快地实现病理图像病灶标注。
