凡纳滨对虾SNP 标记开发与家系亲缘关系验证分析*
2021-03-19刘峻宇刘均辉孟宪红罗曹宝祥陈宝龙
刘峻宇 刘均辉 孔 杰 代 平 于 洋 孟宪红罗 坤 曹宝祥 陈宝龙 高 焕 栾 生①
(1. 江苏海洋大学 海洋生命与水产学院 江苏省海洋生物资源与生态环境重点实验室 连云港 222005;2. 中国水产科学研究院黄海水产研究所 农业农村部海洋渔业可持续发展重点实验室青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 青岛 266071;3. 中国科学院海洋研究所 中国科学院实验海洋生物学重点实验室 青岛 266071)
凡纳滨对虾(Litopenaeus vannamei),亦称南美白对虾、太平洋白对虾,是一种热带虾,具有生长快、抗病力和适应性强、味道鲜美等特点(唐扬等, 2018;王兴强等, 2004)。凡纳滨对虾20 世纪80 年代引入我国,90 年代解决了规模化苗种繁育关键技术后,养殖规模迅速扩大,2018 年产量超过170 万t (农业部渔业渔政管理局, 2018),已发展为我国单一产值最高的水产养殖种类之一。凡纳滨对虾种业体系较为健全,我国每年的种虾需求量超过100 万对,苗种需求超过6000 亿尾。以规模化家系为基础的选择育种,是当前国内外10 多家对虾育种单位主要采用的技术体系,在该体系中,遗传进展主要取决于目标性状的遗传力和选择强度。研究表明,在奠基者群体已经构建遗传力等参数固定的情况下,育种过程中增加家系数量、扩大选育群体规模和提高选择强度可有效增加遗传进展(栾生等, 2018)。
在传统的选择育种中,主要是应用“可视嵌入性橡胶标志”(Visible implant elastomer, VIE)标识不同家系。但在高强度的选育计划中,由于养殖家系数量太多,受限于可使用的颜色数量,难以同时区分超过200 个以上家系。而且对大量的个体进行物理标记的成本太高且耗时耗力,在养殖过程中容易发生物理标记漂移脱落、人工读取标记错误等现象,也会导致个体所属家系识别错误,产生系谱不准确的问题,从而影响遗传评估和选择的准确性。
随着分子标记的发展和分型成本的降低,微卫星(Simple sequence repeat, SSR)、SNP(Single nucleotide polymorphism)等分子标记已成为解决上述问题的有效途径(桂建芳等, 2012)。其中,SNP 标记具有数量多、分布广泛,便于高通量、高度自动化检测分析等优点,通过研究其在生物基因组中的分布即能够全面反映群体的遗传及变异水平,在凡纳滨对虾(于洋等,2014; 王冉等, 2018)、日本沼虾(Macrobrachium nipponense)(张洪伟等, 2010)、栉孔扇贝(Chlamys farreri)(Jiang et al, 2011)等物种中已开展应用。利用分子标记特别是SNP 标记进行亲缘关系鉴定,在畜牧育种中也已得到广泛应用(李东等, 2011; 余国春等,2014),但在水产动物中相关研究还较少。根据其他物种的研究结果推测,在虾类高强度选育中,对人工选择出来的优质候选个体进行SNP 标记分型,通过个体聚类分析及亲缘关系鉴定,可有效区分候选个体的亲本信息,构建更为准确的系谱。
当前,通过DNA 和RNA 高通量测序可以获得大量的多态性SNP 位点。然而,受基因组组装质量、样本测序和分型算法等多种因素影响,很多位点可能是假阳性位点。因此,利用较大样本量,对已有位点进行分型验证,开发高质量的多态性SNP 位点,是开展分子标记辅助选择的重要基础工作。本研究利用飞行时间质谱分析技术,开发了37 个源自转录组序列的SNP 标记,并对22 个凡纳滨对虾家系进行遗传多样性、家系聚类和亲子鉴定分析,旨在探讨SNP标记在凡纳滨对虾选育中应用的可行性,以期为分子标记辅助育种提供参考。
1 材料与方法
1.1 实验材料
在农业农村部海水养殖遗传育种中心(中国水产科学研究院黄海水产研究所鳌山基地)利用G1 代核心群留种个体,通过巢式交配设计和定向交尾的方法生产51 个家系,构建凡纳滨对虾G2 代核心育种群体,并在–20℃温度下保留亲本肌肉组织。从开展生长和存活性状测试的G2 代家系中选择22 个全同胞家系(含4 个半同胞家系),每个家系随机取9~10 尾个体,共计218 尾个体,加上22 尾父本和20 尾母本,作为本实验的样本材料。子代个体取腹部肌肉组织后,放入冻存管,–80℃保存。本研究中的转录组数据由中国科学院海洋研究所实验海洋生物学重点实验室提供,共134 个SNP 位点及其序列信息。
1.2 实验方法
DNA 提取参照王伟继(2008)的方法。DNA 的质量和浓度使用超微量紫外分光光度计测量,保证DNA 的浓度>50 ng/μl,吸光度比值(OD260nm/OD280nm)在 1.7~2.0 之间。采用 1%的琼脂糖凝胶电泳检测DNA 的完整性,检测完毕的样品置于–20℃冰箱中冷冻保存。
1.3 SNP 分型
随机取24 尾家系亲本个体,平均分为2 组,每12 尾个体的DNA 混在一起。根据样品DNA 定量结果,将混合模板的DNA 浓度稀释到50~100 ng/μl。根据序列信息,通过Primer Premier 5.0 软件设计上下游引物,扩增含有该位点的DNA 片段,PCR 反应体积为50 μl:0.25 μl Taq 酶(5 U/μl),引物各2 μl (10 pmol/μl),2 μl DNA(50 ng/μl),4 μl Dntp(2.5 mmol/L),5 μl 10×PCR Buffer (Mg2+plus)和34.75 μl ddH2O。程序:94℃预变性5 min,94℃ 30 s,合适退火温度下1 min,72℃ 1 min,35 个循环;最后72℃延伸10 min。引物合成和测序由上海生工生物工程股份有限公司完成。
利用直接测序法验证SNP 位点是否存在双峰,若扩增产物同时出现双峰,即认为该位点在群体内存在多态性。针对存在双峰的位点,通过Genotyping Tools 及MassARRAY Assay Design 软件设计PCR 扩增引物及单碱基延伸引物,利用多重PCR 设计2 个多重组合,对所有个体上机测试。最后采用基质辅助激光解吸附电离飞行时间质谱分析技术(Matrixassisted laser desorption/ionization-time of flight,MALDITOF)分型检测,此过程由北京博奥晶典生物技术有限公司支持完成。
1.4 数据处理与分析
SNP 位点的期望杂合度(He)、观测杂合度(Ho)和多态信息含量(PIC)分析通过Cervus 3.0 (Kalinowski et al, 2010)软件进行。家系间的遗传距离通过POPGENE 1.32(Yeh et al, 1999)软件计算。基于非加权配对算数平均法,使用MEGA 7(Tamura et al, 2011)绘制22 个家系聚类图。
目前,最简单常用的亲子鉴定方法是排除法(Jones et al, 2010),其中,用累积排除概率(Cumulative probability of exclusion, CPE)表示排除不是真实亲本的候选亲本的概率(Dodds et al, 1996; Heaton et al,2002)。亲缘关系鉴定运用Cervus 3.0 (Kalinowski et al,2010)软件通过似然对数比(log likelihood ratio, LOD)值判断分析。
2 结果
2.1 SNP 分型
测序结果显示,2 组混合DNA 样品中有83 个位点出现明显双峰,剩余位点显示单一的峰图或测序失败。本实验设计2 个多重组合(包含的位点数分别为27 个和28 个),最终2 个组合中分别有18 和19 个SNP 位点在所有样本中均能扩增成功。最后利用飞行时间质谱分析技术对每个样品的37 个SNP 位点进行SNP 分型检测,多态性位点的引物信息见表1。

表1 凡纳滨对虾37 个SNP 位点的引物信息Tab.1 Information of 37 SNP loci primers in L. vannamei
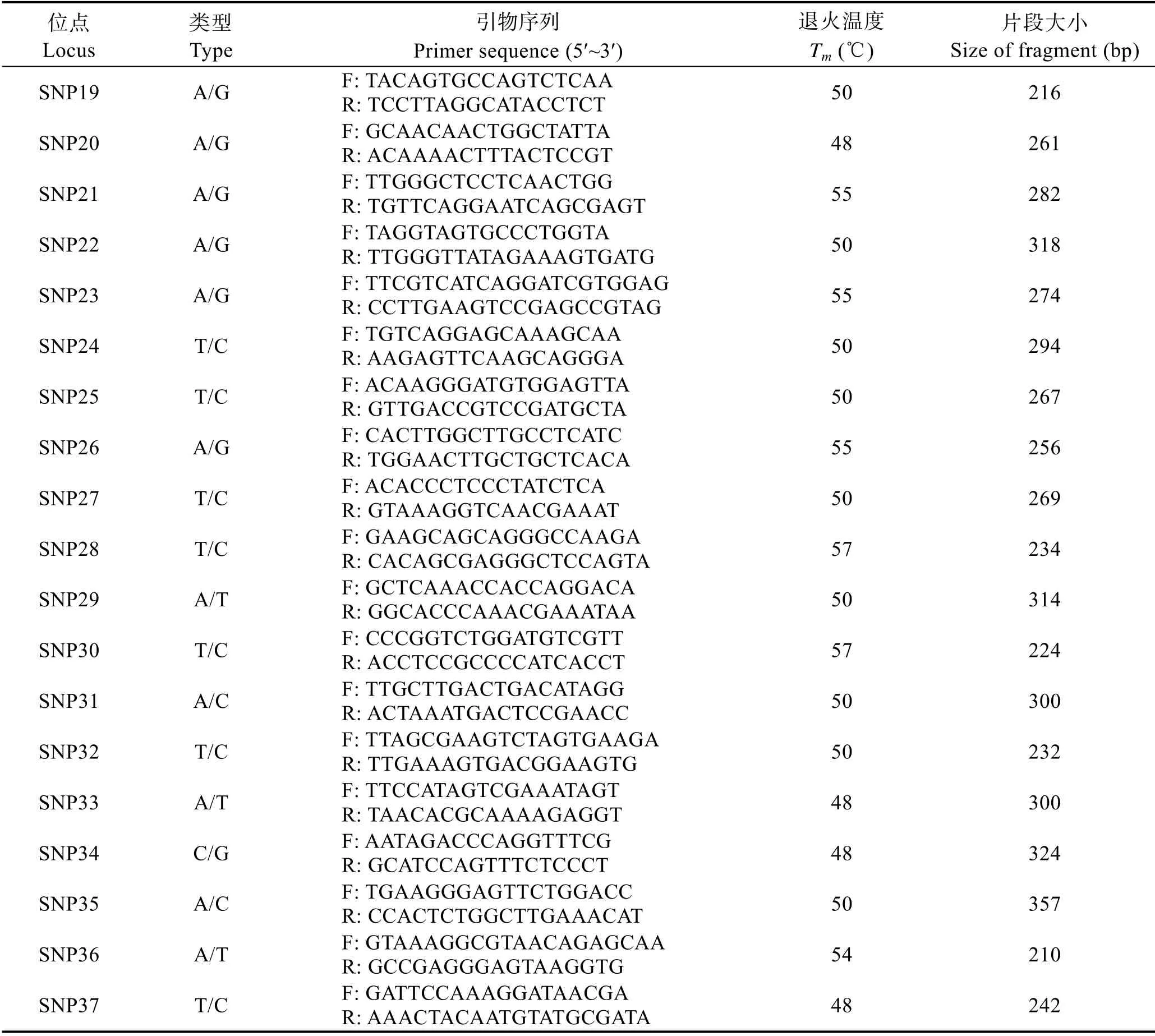
续表1
2.2 家系遗传多样性分析
利用37 个SNP 标记的分型信息对凡纳滨对虾22 个家系进行遗传多样性分析。结果显示,平均期望杂合度(He)为0.38;平均观测杂合度(Ho)为0.34。平均多态信息含量(Polymorphism information content,PIC)为0.30,属于中度多态性(0.25<PIC<0.5)(表2)。
2.3 亲子关系鉴定
在95%的置信水平下,凡纳滨对虾37 个SNP 位点的遗传多样性见表2,218 尾子代亲子关系鉴定准确率见表 3。单亲性别已知的情况下,CPE 值在95.04%~99.75%之间,观测鉴定率在40%左右;双亲性别已知情况下,CPE 值超过99.99%,观测鉴定率为100%,实际鉴定率为77.27%。
2.4 家系遗传距离和聚类分析
基于凡纳滨对虾22 个家系间的遗传距离,绘制家系间的聚类分析图,见图1。家系F11 和F17、F8和F10 分别为两对半同胞家系,其中,F11 和F17 家系间的遗传距离最小(0.04),最先聚在一起;F8 和F10家系间遗传距离为0.10,未能最先聚在一起。
3 讨论
3.1 遗传多样性分析
本研究基于凡纳滨对虾转录组数据开发了37 个多态性SNP 标记。多态信息含量(PIC)是检测群体DNA 变异程度的指标之一,根据Botstein 等(1980)首先提出来的PIC 划分规则,本研究群体中平均PIC含量为0.30,属于中度多态性(0.25<PIC<0.5)。与利用 SNP 标记获得的凡纳滨对虾、中国对虾(Fenneropenaeus chinensis)群体遗传多样性分析结果类似(张建勇等, 2011; 吴莹莹等, 2013)。但研究表明,利用微卫星分析凡纳滨对虾群体,获得了较高的PIC值,平均值为0.51~0.58(陈锦豪等, 2019; 黄小帅等,2019; 孔张伟, 2018; 李东宇等, 2015)。究其原因,主要是因为SNP 标记仅有2 个等位基因,与SSR 标记相比,其携带的PIC 较低(Kong et al, 2014)。
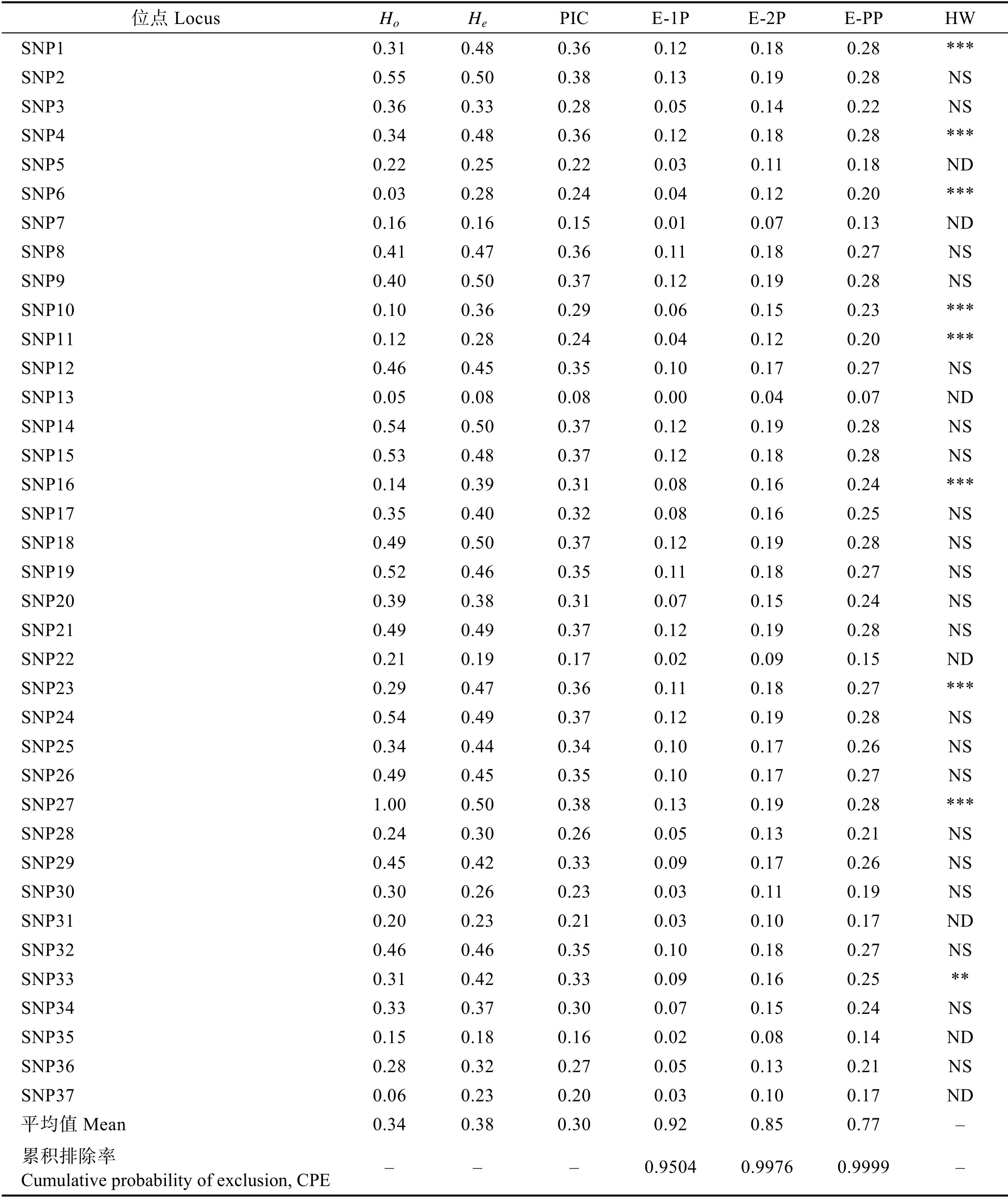
表2 凡纳滨对虾22 个家系在37 个SNP 位点上的遗传多样性Tab.2 Genetic diversity estimated using 37 SNP loci for 22 families in L. vannamei
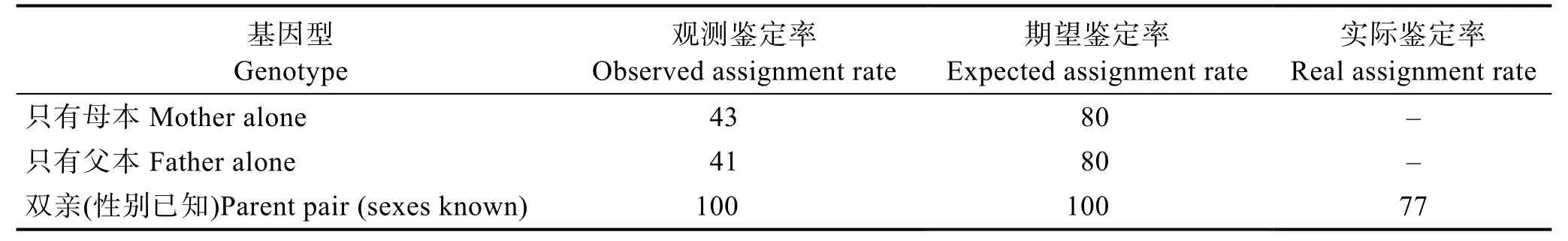
表3 凡纳滨对虾218 尾子代亲子关系鉴定准确率Tab.3 Accuracy of parentage assignment for 218 offspring in L. vannamei (%)
凡纳滨对虾22 个家系Ho和He平均值分别为0.34和0.38,且HW 平衡检验结果表明,有30%的位点极显著偏离平衡,说明该群体经过了多代选育,存在着杂合子缺失的现象。与已报道的凡纳滨对虾群体遗传特性一致(Jimenez et al, 2004),在中国对虾(张天时等, 2005)、斑节对虾(Penaeus monodon)(Xu et al, 2001;Premruthai et al, 2002)育种群体的研究中也发现杂合子缺失。育种群体杂合子缺失将会导致纯合子增加,提高有害等位基因纯合的几率,从而导致近交衰退几率升高,降低群体对环境变化的适应性(杨锐等,2000)。
3.2 家系聚类和亲子鉴定分析
从家系聚类中可以观察到(图1),两组半同胞家系中只识别出其中1 组(F11 和F17 最先聚在一起)。另外一组半同胞家系F8 和F10 中,F8 与F12、F6 和F10 最先聚在一起。所以,对4 个家系(F6、F8、F10和F12)中的共39 尾个体进行聚类(图2)。从图2 可以观察到,F10 家系中的第4、第5 个个体聚类在F12家系中,F12 家系中的第10 个个体聚类在F10 家系中。F8 和F10 未能最先聚在一起,原因可能是F10家系中混入F12 家系的个体,导致F10 家系的平均遗传距离增大,在聚类时未能优先与F8 家系聚类在一起;且结合系谱观察到,F8 和F12 家系的母本来源于同一祖先,2 个家系间亲缘关系较近,造成优先聚类的现象。
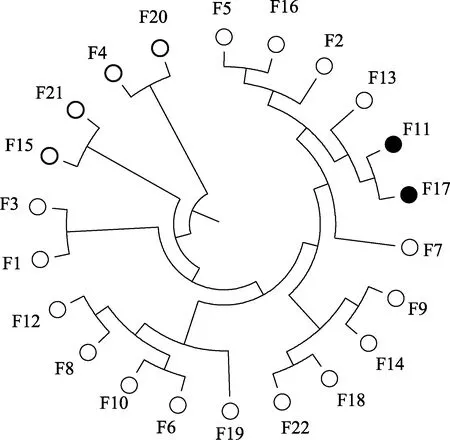
图1 凡纳滨对虾22 个家系聚类分析Fig.1 Cluster analysis between 22 families in L. vannamei
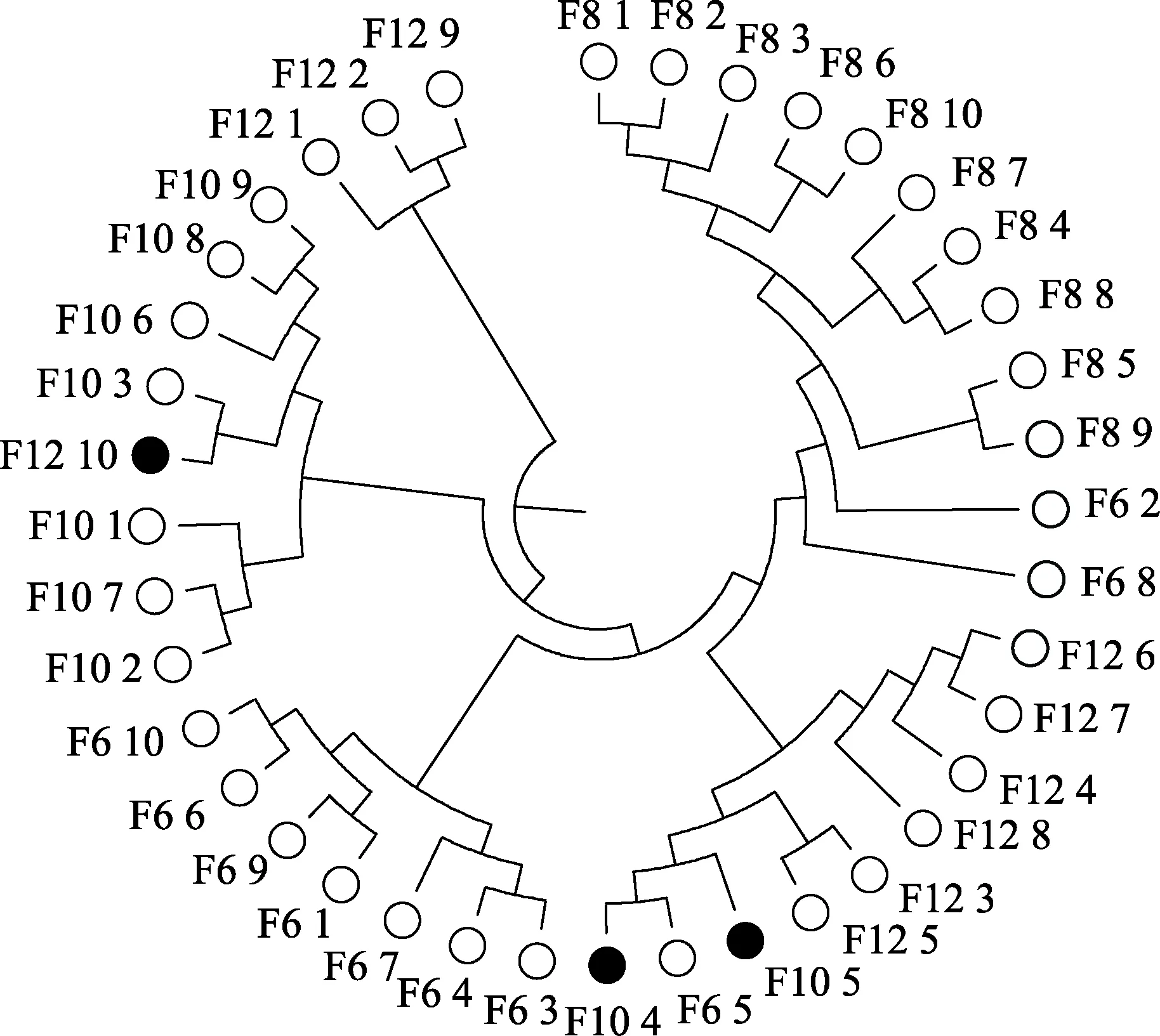
图2 凡纳滨对虾F6、F8、F10 和F12 家系个体聚类分析Fig.2 Cluster analysis among individuals in family F6, F8, F10 and F12 of L. vannamei
在父母本双亲性别已知的情况下,亲子间的CPE为99.99%,观测鉴定率达到100%,实际鉴定率达到77.27%。Kazuhiro 等(2010)利用29 个SNP 对日本黑牛(Bovine)群体进行亲子鉴定分析,单亲已知和双亲已知的情况下,CPE 分别为96.93%和99.69%。相关研究表明,在20~32 个SNP 范围内,亲子间累积排除率在95.6%~99.9%之间(Werner et al, 2004; Anderson et al, 2006; Heaton et al, 2002),与本研究结果一致。Zhu 等(2017)利用6 对微卫星标记对斑节对虾进行亲缘关系鉴定,实际鉴定率达到89%。郭立平等(2013)利用50 个高多态性的SNP 位点对938 头西门塔尔牛(Bos taurus)进行亲子鉴定,最终实际鉴定率为41.04%,Sellars 等(2014)利用SSR 体系和SNP 体系对斑节对虾家系鉴定能力进行比较,各组鉴定结果表明,60 个SNP 标记鉴定率在99%左右。本研究实际鉴定率相对较低,原因在于SNP 位点的数量少和存在杂合子缺失,无法提供充足的信息来鉴定出所有个体的真实父母本,且候选亲本中存在一定程度的亲缘关系,遗传相似度较高,导致亲缘关系的误判;Sellars等(2014)研究表明,随着SNP 位点数的提高,亲缘关系鉴定的准确性也会增加。刘均辉(2016)通过拟合指数曲线推测,在凡纳滨对虾群体中,至少需要110~130个随机选取的SNP 位点,亲子关系鉴定效力方可达到100%。
本研究结果表明,在性状测试和系谱追踪过程中,利用高质量的SNP 标记代替物理标记识别家系是一种较为准确可行的方法。同时,应增加SNP 位点的数量,提高家系系谱鉴定的准确性。
