利用基因组特异性引物和Cas12a介导技术快速检测稻瘟菌小麦致病型
2021-03-17康厚祥彭烨KngyuHu邓雨飞MriBllizziDipliRniGuptNurUinMhmuAlfroUrshimSnjoyKumrPulGryPtrson周益林周雪平TofzzlIslm王国梁
康厚祥*, 彭烨#, Kngyu Hu , 邓雨飞 Mri Bllizzi , Dipli Rni Gupt , Nur Uin Mhmu ,Alfro S.Urshim , Snjoy Kumr Pul , Gry Ptrson , 周益林 周雪平 M Tofzzl Islm ,*,王国梁,*
a S tate Key Laboratory for Biology of Plant Diseases and Insect Pests, Institute of Plant Protection, Chinese Academy of Agricultural S ciences, Beijing 100193, China
b Department of Plant Pathology, The Ohio S tate University, Columbus, OH 43210, US A
c Institute of Biotechnology and Genetic Engineering (IBGE), Bangabandhu S heikh Mujibur Rahman Agricultural University, Dhaka 1706, Bangladesh
dCentro de Ciências Agrárias, Universidade Federal de S ão Carlos,Araras, S P CEP 13600-000, Brazil
e Foreign Disease-Weed S cience Research Unit(FDWS RU), United S tates Department of Agriculture-Agricultural Research S ervice (US DA-ARS), Ft.Detrick, MD 21702, US A
1.引言
半活体营养型真菌稻瘟菌侵害许多谷物,包括两种最重要的主要粮食作物——水稻和小麦。稻瘟病是由稻瘟菌(M.oryzae Oryza,MoO)致病型引起的,近三个世纪前在中国和日本有报道,目前已在超过85 个国家发现[1]。1985 年,由稻瘟菌小麦致病型(M.oryzae Triticum,MoT)引起的小麦麦瘟病在巴西巴拉那州的6 个市被首次发现[2],并且20世纪90年代初其在南美洲的分布逐渐扩大到约3×106hm2[3]。2016年,这种病害首次在南美洲以外被发现,麦瘟病暴发于孟加拉国的8个地区,并迅速成为对小麦生产的严重威胁[4]。2017 年年初,多家报纸报道,在印度-孟加拉国边境附近的印度麦田首次观察到类似麦瘟病的症状[5]。2016—2019 年间,该病害蔓延至孟加拉国的20 个区,并继续对孟加拉国的粮食营养安全构成严重威胁[6]。根据2016 年在孟加拉国首次发现麦瘟病的地区的农业气候条件,最近的一项研究报告称,印度、巴基斯坦和孟加拉国多达7×106hm2的麦田容易受到MoT感染[7]。保守估计产量损失5%~10%,每年潜在经济损失可能为1.32 亿~2.64 亿美元。因此,迫切需要一种综合的方法来控制孟加拉国麦瘟病发生,并防止这种病害传播到世界其他地区。
MoT 如何感染小麦植株,其宿主的替换,以及那些有利于麦瘟病流行的气候因素目前还不清楚。虽然MoT既可以侵染小麦植株的叶片,也可以侵染小麦植株的穗部,但穗部感染会造成更大的经济影响。叶轴或花梗的感染会阻碍光合作用产物的运输,杀死穗状花序的上部。在对病原菌有利的条件下,该病可造成高达100%的产量损失[8]。然而,由于穗部麦瘟病是在没有叶瘟病视觉症状的田野中发生的,两者之间的相关性尚不明确。因此,通过使用杀菌剂来控制麦瘟病穗部发生可能是困难的,因为农民不能在抽穗期之前根据观察叶片病斑来使用杀菌剂。此外,麦瘟病也是一种通过种子传播的病害[9]。对种子进行筛选并对其进行MOT 鉴定将降低病害进一步传播到其他国家的风险。因此,开发一种快速、灵敏、低成本的方法检测田间小麦幼苗、邻近麦田的其他寄主、商业运输中的小麦种子中的MoT,对控制南美洲和孟加拉国的小麦麦瘟病至关重要。
Pieck等[10]鉴定了MoT菌株独有的DNA标记。其中一个标记MoT3对来自多个国家的11个寄主物种的284株稻瘟菌株的DNA进行检测时显示出特异性。其在MoT株系中扩增出一条360 个碱基对(bp)的MoT3标记片段,而在MoO 株系中未扩增出。然而,该片段位于编码视黄醇脱氢酶的单拷贝基因内,该基因存在于MoT、MoO 和MoL(稻瘟菌黑麦草致病型)菌株中。最近的一项研究分析了81个先前组装的Magnaporthe基因组的多态性,发现MOT3序列并不存在于所有的MoT 菌株[11]中。另一种DNA 标记C17,是新的具有高灵敏度的小麦系特异性的标记[11]。在实时荧光定量聚合酶链反应(qRT-PCR)中,该方法成功扩增了30 个小麦携带菌株100%的DNA。然而,目前还不清楚在受感染的小麦植株中,MOT3和C17是否都适用于快速检测MoT。
近年来,已经开发了几种新技术以在实验室和田间条件下快速检测植物病害。例如,环介导等温扩增(LAMP)[12‒ 13]和重组酶聚合酶扩增(RPA)[14]正被广泛用于植物病害检测。值得一提的是,最近开发了一种革命性的人类疾病检测方法,它将Cas蛋白(如Cas12a和Cas13a)可识别的编码核苷酸靶标与核酸快速侧向流动免疫分析(NALFIA)方法相结合[15‒ 17]。这种方法是一种准确、快速、特异和经济有效的替代方法,可以很简易地用于植物病害检测。
本研究旨在利用RPA、CAS12a 和NALFIA 建立一种新的田间快速准确检测MoT 的方法。首先,我们利用下一代测序(NGS)策略对巴西两个小麦麦瘟菌株系的基因组进行了重测序;其次,我们使用BLAT软件[18]对MoO和MoT 基因组进行了成对的基因序列比对分析,从而鉴定出两个MoT 特异片段:MoT-6098和MoT-6099;然后,根据MoT-6098和MoT-6099序列设计了几组引物,并通过PCR 和LAMP 验证了引物的敏感性;最后,我们将基于Cas12a 的检测方法与RPA 和NALFIA 技术相结合,开发了一种灵敏、低成本的田间受侵染小麦植株中MoT 的检测方法。本研究为小麦毁灭性病害麦瘟病提供了一种新的检测方法。
2.材料和方法
2.1.真菌材料
本研究所用的MoO 菌株(RB22、R01-1、P131、Guy11、N60、N63 和N71)在中国农业科学院植物保护研究所培养。所有MoT 菌株的DNA 样本均由邦格班杜(Bangabandhu)·谢赫·穆吉布·拉赫曼农业大学、位于美国德特里克堡(Ft.Detrick)的美国农业部外来病害和杂草研究所、位于巴西圣卡洛斯的联邦大学农业研究中心准备。
2.2.本研究使用的引物
表1列出了本研究中使用的所有引物。

表1 本研究中使用的引物和sgRNA序列列表
2.3.基因组测序与比较基因组分析
本研究利用Illumina 测序平台(Hisep X_ten, Bio Marker Co., China)对MoT 菌株PR01-37.V.1 和PR01-37.V.3 的基因组DNA 进行了测序。简言之,PR01-37.V.1 和PR01-37.V.3的DNA被随机剪切成约500 bp的片段,并克隆到测序载体上构建文库,然后用Illumina系统对文库进行测序。对序列质量控制(QC)后获取准确无误的序列读取数据。使用SPADS-3.13.0 软件(俄罗斯圣彼得堡州立大学)对两个基因组进行从头组装。使用Augus‐tus.2.5.5 软件(德国哥廷根大学)对MoO 和MoT 基因组进行基因预测。然后开发了一种以BLAT[18]为核心比对工具的成对基因比对通道。通过对两株MoT菌株(PR01-37.V.1 和PR01-37.V.3)和三株MoO 菌株进行基因序列比对分析,确定了潜在的MoT 特异性基因。然后将潜在的特定基因与额外的MoO和MoT基因组进行比对[4,19]。具体地说,采用两步法来鉴定MoT 特异性基因。首先,我们将378个候选基因与NCBI-nr数据库进行比对,以移除MoO 基因。其次,我们将100 个候选MoT 基因与序列更高的MoT和MoO基因组进行比对,以测试它们是否存在于所有MoT 基因组中,而不存在于所有MoO 基因组中。最后,在分析中鉴定出两个MoT 特异性基因。详细的生物信息学分析过程如图1所示。
2.4.环介导等温扩增法
环介导等温扩增法(LAMP)反应混合物的最终体积为20 μL,包含1 μL模板DNA(20 μmol∙L−1)、2.0 μmol∙L−1的正向和反向内引物(FIP和BIP)、0.5 μmol∙L−1的F3和B3 引物(表1)、1 mmol∙L−1的dNTP、8 U 的Bst DNA 聚合酶(美国新英格兰生物实验室)和1×ThermoPol反应缓冲液[20 mmol∙L−1Tris-HCl、10 mmol∙L−1KCl、2 mmol∙L−1MgSO4、10 mmol∙L−1(NH4)2SO4、0.1% Triton X-100, pH 8.8]。将混合物在65 ℃保持1 h,然后将混合物移至85 ℃下保持5 min后终止反应。
2.5.LAMP反应产物的检测
在LAMP 扩增产物中加入2 μL 体积的1∶10 稀释的SYBR Green I(10 000×,美国Invitrogen)。变成绿色的反应被记录为阳性,而那些保持棕色的反应被记录为阴性。扩增的LAMP反应也在由溴化乙锭染色2%琼脂糖凝胶上进行分析。该凝胶使用Gel Doc XR 成像仪系统(美国Bio-Rad)在紫外线下拍摄。
2.6.重组酶聚合酶扩增
按Piepenburg 等[14]先前所述,RPA 使用了中国安普未来生物科技有限公司(AMP-Future Biothech Co.Ltd.)的试剂盒(#WLN8203KIT)进行。对于第一个RPA,设计了MoT-6098/MoT-6099特异性引物对,并添加了四氢呋喃(THF)探针作为Nfo酶识别位点。扩增子应至少包含一个用于识别Cas12a 保护间隔相邻基序(PAM)位点(四个碱基“TTTA”)。对于第二个RPA,设计了ssDNA特异性引物对,并在5'端用FAM标记,在3'端用生物素标记。RPA 扩增按照RPA 试剂盒中的说明进行操作。简而言之,将引物与含有重组酶、聚合酶和缓冲液的RPA 溶液混合,然后加入2 μL的样品DNA(或Cas12a消化的溶液),然后将制剂(总体积为30 μL)在37~39 ℃保持8~12 min。
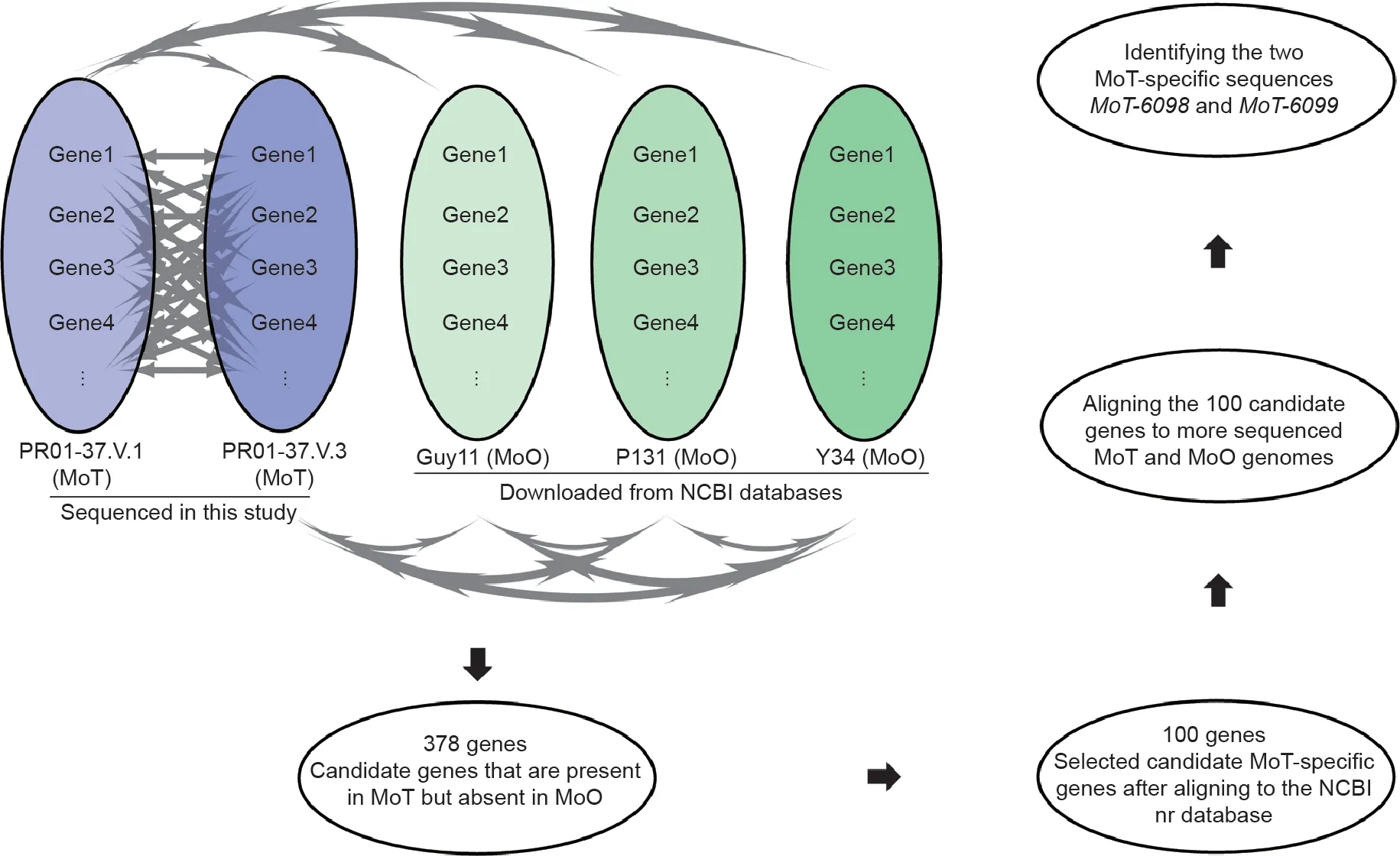
图1.识别两个MoT特定序列的途径。图左上角的省略号表示不同的麦瘟病菌基因组和基因1,2,3,4,…。N代表测序基因组中的所有预测基因。两个基因组中两个基因之间的箭头表示双向成对的基因序列比对,菌株之间的拱形箭头表示两个基因组中单个基因的序列比对。nr:非冗余蛋白序列。
2.7.ssDNA设计
ssDNA 设计有两个要求:①ssDNA 应较短(便于快速PCR检测);②ssDNA与目标基因组序列之间不存在完全相同的序列。为了满足这些要求,我们使用计算机程序随机设计了10 000 个120 bp 的DNA 序列。然后使用BLAST软件将这些120 bp的序列与小麦、MoT和MoO基因组进行比对[20],并丢弃E值≤e−10或与靶序列相同≥10 bp的序列。以下是从与小麦、MoT和MoO序列无明显相似性的序列中随机选择:“ACGTCGTCACATAGGGTATATCTTTTCACAACAGTAACCTACACTCGACTCACC GCAGTTCCCCAGACGCAGTATCGTAGCCGCGGATAGT TAGTGCTTCTAAGACCGCGAATGACAACA”。
2.8.基于CAS12a的检测
对于带有Cas12a 的RPA,该研究使用了之前描述的方法[16]。Cas12a 蛋白购自新英格兰生物实验室(NEB#M0653S,美国)。简而言之,将Cas12a 和sgRNA(等摩尔体积)与反应缓冲液混合,并在室温下孵育约10 min。然后将RPA扩增的靶DNA溶液在37 ℃的Cas12a混合物中孵育约10 min,以激活Cas12a ssDNA 的酶切活性。最后,将设计的ssDNA 与活化的Cas12a 蛋白结合,进行ssDNA酶切。
2.9.用PCRD试纸进行NALFIA检测
使用Abingdon Health PCRD 测试盒(#FD51673,美国)用于DNA检测可视化。简而言之,将5 μL的RPA反应扩增产物与70 μL的PCRD提取缓冲液混合,并将总体积(75 μL)加入PCRD试纸条的样品孔中。3~5 min后评定结果。
3.结论
3.1.用比较基因组学方法鉴定MoT特异性序列
为了研究MoT和MoO的基因组差异,我们从巴西小麦病株上提取了两个MoT 菌株:PR01-37.V.1 和PR01-37.V.3 的DNA。然后,我们使用Illumina 测序平台(美国)对这两个菌株进行了重测序,基因组覆盖率约为150。基因组拼接后,获得了PR01-37.V.1 约38.5 Mb 的基因组序列和PR01-37.V.3 约40.0 Mb 的基因组序列。我们还从GenBank[21]下载了三株MoO菌株:Guy11、P131和Y34的基因组序列。通过成对的基因序列比对,我们鉴定了378个在三株MoO菌株中缺失的MoT基因。将这378个基因与GenBank 蛋白质数据库(nr)进行比对,鉴定出289个基因与数据库中的基因高度同源。其中189 个为含有MoO 基因,其余100 个为MoT 特异的候选基因(见附录A 中的表S1)。除了少数与细菌基因高度同源的基因外,大多数基因都是真菌基因,其中一半以上(100个基因中的57个)属于6个真菌物种(见附录A中的表S2)。将这57个基因与其他20个测序的MoT和MoO基因组进行比对时[4,19],我们发现了两个MoT特异的基因:MoT-6098和MoT-6099。MOT-6098编码一个假定的酸性海藻糖酶蛋白,而MoT-6099编码一个DUF341 家族蛋白。因为MoT-6098和MoT-6099存在于所有测序的MoT基因组中,而不存在于任何测序的MoO 基因组中,所以我们选择这两个MoT特异性基因来开发MoT特异性标记。
3.2.区分MoT 和MoO 的MoT-6098 和MoT-6099 引物对的灵敏度
为了确定MoT 和MoO 菌株中MoT-6098和MoT-6099序列的特异性,我们设计了一组引物(MoT-6098F 和MoT-6098R,MoT-6099F和MoT-6099R,见表1),并用它们对53个不同的稻瘟菌菌株的DNA进行了PCR反应。这53 个菌株来自9 个国家的14 种禾本科植物(表2、表3),包括15个来自水稻的菌株,19个来自小麦的菌株,19个来自可能的其他寄主菌株,如大麦、燕麦、狗尾草、臂尾草、黑麦草、雀麦、穇、画眉草、羊茅、狗尾草和狭叶狗尾草(表2、表3)。我们在Urashima 教授和Islam 教授实验室分别对从巴西和孟加拉国收集的25 个菌株进行了PCR 检测(表2)。其中包含两个在Urashima 教授实验室分离出的引起小麦赤霉病的禾谷镰刀菌的DNA,以检测真菌种间的引物特异性。此外,我们还收集了Wang实验室提供的4 个侵染水稻病原菌和侵染黑麦草的病原菌的DNA 样本作为对照。PCR 分析表明,这两个基因存在于所有8 个MoT 菌株、1 个大麦菌株和1 个美国黑麦草菌株中,但不存在于水稻、狗尾草、臂形草、燕麦和两个禾谷镰刀菌中(图2和表2)。随后,我们测试了美国农业部外来病害和杂草研究所保存的24 株稻瘟菌菌株(表3)。除两株来自臂形草的菌株外,其余菌株均含有这两个基因。在22株MoT阳性菌株中,11株从小麦中分离而得,其余的分离于从巴西、玻利维亚、巴拉圭、日本和美国等地采集的MoT 其他宿主,包括黑麦草、穇、雀麦、画眉草、偏序钝叶草和羊茅(见附录A中的S1和表3)。
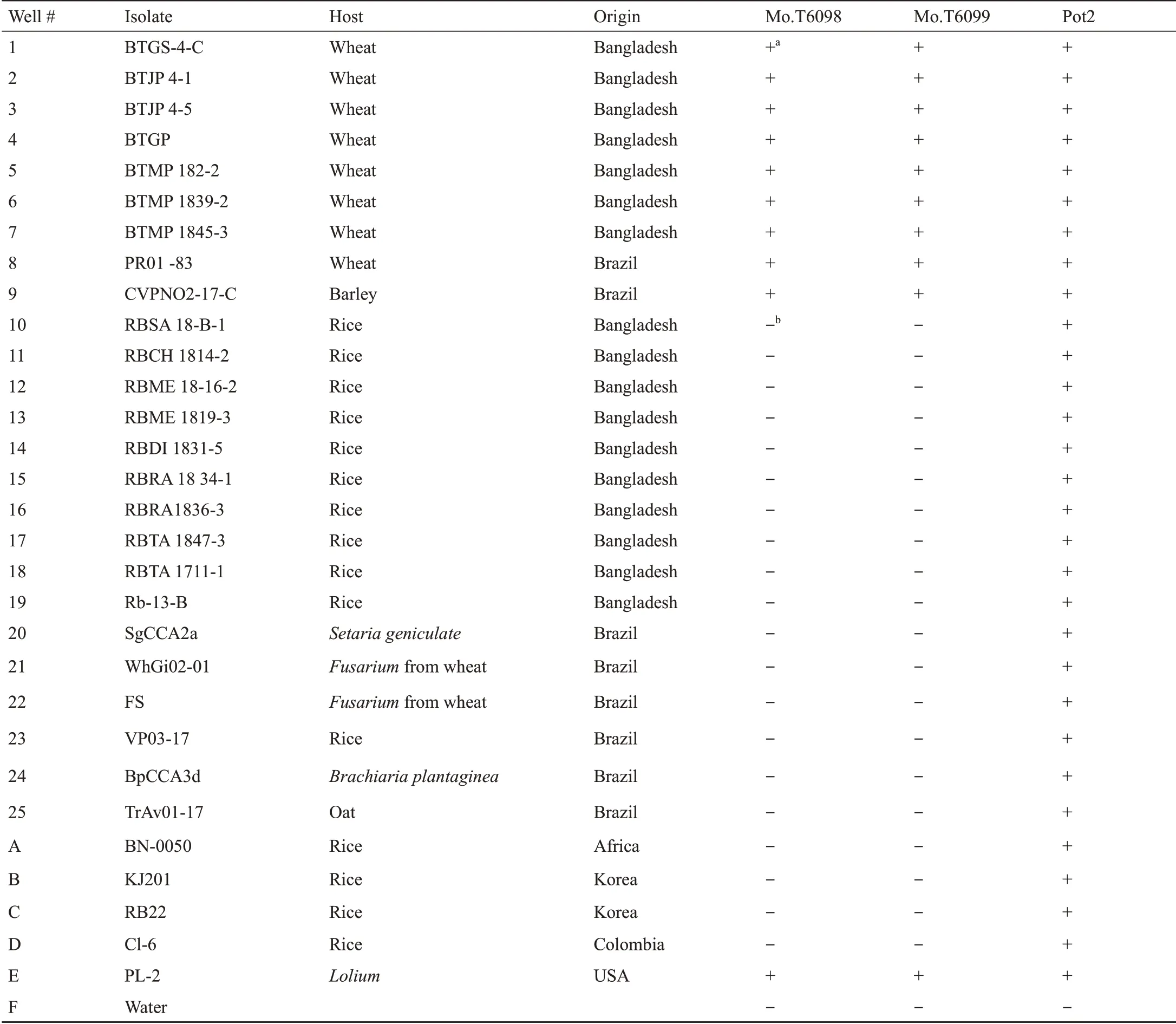
表2 用于PCR检测MoT-6098和MoT-6099片段的孟加拉国和巴西稻瘟菌分离菌株

表3 用于PCR检测MoT-6098和MoT-6099片段的来自USDA-ARS的稻瘟菌分离菌株
然后我们进行了灵敏度试验来检测这两对引物的敏感性。将MoT菌株BO12TA301培养物中5个浓度(0.01 ng·μL−1、0.1 ng·μL−1、1 ng·μL−1、10 ng·μL−1和50 ng·μL−1)的DNA 与MoT-6098F/R 和MoT-6099F/R 引物对进行PCR 反应。以Pot2R/F引物对为对照。PCR结果显示,0.1 ng·μL−1的DNA 能用MoT-6099F/R 扩增出条带,10 ng·μL−1的DNA能用MoT-6098f/R扩增出条带[图3(a)]。我们还用与上述常规PCR相同的DNA稀释度,使用实时荧光定量PCR检测引物的敏感性。两对引物在仅含0.01 ng·μL−1模板DNA的反应中产生了正常的循环阈值(Ct),表明这些引物对目标序列的扩增非常敏感[图3(b)~(d)]。
3.3.感染植物中MoT的检测
为了确定MoT-6098F/R和MoT-6099F/R引物对是否可以用于检测感染小麦中的MoT,我们在邦格班杜·谢赫·穆吉布尔·拉赫曼农业大学的生长室中,从感染MoT菌株BTJP4-5的小麦植株叶片和穗中提取了DNA。为了控制变量,我们还从自然侵染的水稻穗、MoT分离物BTJP4-5和MoO 分离物RB-13b 的菌丝中提取了DNA。PCR 结果表明,MoT-6098 和MoT-6099 扩增带在被MoT 感染的小麦叶片、穗部和MoT菌丝的DNA中均有扩增,而被MoO侵染的水稻叶片或MoO 菌丝中分离的DNA 没有扩增(图4)。这些结果表明,MoT-6098和MoT-6099引物能有效检测受感染小麦植株中的MoT。
3.4.使用LAMP检测MoT
由于LAMP是一种在等温条件下特异、快速、高效的DNA 检测方法,我们利用LAMP 对MoT 中的MoT-6098和MoT-6099片段进行了检测。扩增后的DNA在琼脂糖凝胶和1000×SYBR Green 染料中均能显示。在琼脂糖凝胶电泳实验中,从3 株MoT 菌株提取的DNA 均出现扩增条带,而5 株从MoO 菌株中提取的未出现扩增条带[图5(a)]。与凝胶法结果一致,含3 个MoT 菌株DNA 的SYBR Green 染料溶液中显示出绿色而含5 个MoO 菌株DNA 的溶液均未变色[图5(b)]。结果表明,在等温条件下,LAMP可以快速、高效地检测MoT。
3.5.将Cas12a 蛋白与RPA 和NALFIA 技术相结合,用于田间MoT的快速检测
LAMP 技术由于需要高温条件(60~65 ℃),限制了其在田间的应用。Cas12a 是最新发现的一种核酸酶,能在sgRNA介导的DNA结合靶向序列后非特异性地消化单链DNA 或单链RNA(ssRNA)[12]。基于Cas12a 的序列识别特异性和ssDNA 核酸酶活性,我们将该蛋白质与RPA 和NALFIA 技术相结合用于MoT 的快速检测[图6(a)、(b)]。该过程首先利用RPA 技术扩增MoT 的特异性序列,然后将扩增产物与靶向MoT-6098和MoT-6099序列的sgRNA以及Cas12a蛋白共同孵育。如果MoT的DNA样品包含靶序列,Cas12a的核酸酶活性将被激活,Cas12a蛋白将非特异性地消化反应中随机生成的ssDNA 探针。第二个RPA 将生物素标记的引物(左引物)和FAM 标记的引物(右引物)用于ssDNA 片段的检测,扩增产物装在试纸条(测流免疫分析;英国Abingdon Health 公司)上用于ssDNA 片段的检测。如果MoT 样品中含有sgRNA靶序列,激活的Cas12a 将消化ssDNA,因此,生物素和FAM 标记的ssDNA 片段不会出现在PCRD 侧流条上[图6(a)]。反之,ssDNA 将在第二个RPA 中扩增,PCRD 条上将出现生物素和FAM标记的ssDNA的扩增[图6(b)]。我们选取4 株MoT 和4 株MoO 菌株进行RPA 和NALFIA检测,其中还包含一个随机生成120 个碱基对的DNA 片段(见附录A 中的实验步骤)用于Cas12a 的消化。琼脂糖凝胶分析证实MoT-6098和MoT-6099片段在4个MoT样品的第一次RPA 反应中扩增,而在4 个MoO 样品中未扩增[图6(c)]。第二次RPA后,将反应液放入到PCRD条上进行NALFIA检测。结果显示,ssDNA条带(自上而下的第二个条带)在4个MoO样品和阳性对照中有明显带,而在4个MoT样品或阴性对照中则没有[图6(d)]。
此外,当对MoT感染的麦叶中分离的DNA进行检测时,NALFIA测定在人工接种的叶子和自然感染的麦穗中都检测到了MoT [图6(e)]。为了进一步检验基于Cas12a的NALFIA方法的灵敏度,我们将其与使用不同浓度真菌菌丝DNA的常规PCR方法进行比较(图7)。如图7(a)和(c)所示。当真菌DNA 浓度为0.01 μg∙μL−1时出现了一条弱带。相比之下,基于Cas12a 的NALFIA 方法能在真菌DNA浓度仅0.001 μg∙μL−1内清晰检测到靶序列。叶片接种后4 天,PCR 就能从其分离的DNA 中检测到靶序列[图7(b)]。然而,基于Cas12a的NALFIA方法早在接种后两天就检测到了靶序列[图7(d)]。这些结果表明,新的检测方法能够快速、准确、高效地识别麦田中的MoT,从而为南美洲和南亚国家的破坏性小麦瘟病检测提供了一个简单、经济的平台。
4.讨论
随着NGS 技术的发展,研究人员现在可以快速、经济地对单个基因组进行测序。尽管NGS 技术使研究人员能够识别紧密相关生物之间的基因组差异,但识别特定物种或致病类型特有的序列仍是个挑战。为了鉴定MoT 中存在而MoO 中不存在的序列,我们对来自巴西的两个MoT 菌株进行了重测序。采用双序列、两步全局比对的方法,将两个MoT 菌株中潜在差异的基因与测序的MoO和MoT菌株进行比对,以鉴定MoT的特异性基因。在57个MoT 特异候选基因中,我们发现了两个序列,它们存在于MoT 和从其他寄主中采集的稻瘟菌菌株中,但不存在于任何MoO菌株中。我们对53株稻瘟病菌的PCR分析证实了这一结果,表明在比较两个高度相似的基因组时,将NGS 测序与两步全局比对法相结合,能够快速准确地鉴定出唯一序列。
为了控制农作物的病害,人们需要快速、准确地诊断出突发和再发病害。在某些情况下,能否控制病害取决于能否立即查明田间的致病病原体。20 世纪70 年代以来,以免疫学为基础的检测技术,如酶联免疫吸附试验(en‐zyme-linked immunosorbent assay, ELISA)被用于田间病原检测。由于传统IBD方法中使用的抗体不稳定,该方法现在使用的是经过活化的修饰胶体金或其他替代颗粒,这些粒子被激活并因此可以通过特异性抗体达到可视化。IBD方法的局限性在于无法获得大多数植物病原物的特异性抗体。在过去的40年里,PCR和RT-PCR被广泛用于检测感染植物的病原DNA 或RNA 序列[22]。PCR 方法的缺点在于其需要在实验室的仪器中进行而且获得诊断结果所需的时间相对较长(4~8 h)。相比之下,NALFIA是一个基于试纸条的方法,可以检测和定量复合物中的待分析物并在5~30 min内提供结果[23]。该方法的成本低而且更易操作,所以使得NALFIA的应用范围在不断扩大,包括植物病原检测在内的其他多个领域。近年来,将NALFIA与LAMP、RPA等核酸检测方法相结合,已成为检测植物病原菌的一种流行方法[24]。另一种诊断方法是利用Cas12a蛋白,该蛋白可以不加选择地切割ssDNA,并在sgRNA与靶序列结合后完全降解ssDNA分子[16]。通过将Cas12a单链脱氧核糖核酸酶激活与等温扩增相结合,发展了一种新的方法——DNA内切酶靶向CRISPR反式报告基因的方法,能够快速、特异地检测人体样本中的病毒[16]。
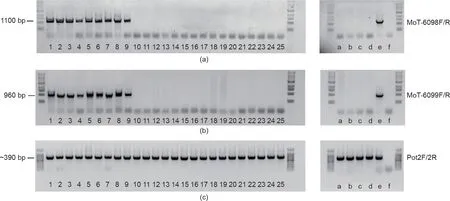
图2.MoT-6098和MoT-6099在所有被检测的MoT菌株中均存在,而在所有MoO株中均不存在。MoT-6098F/R引物对(a)、MoT-6099F/R引物对(b)、对照Pot2F/2R引物对(c)扩增得到的MoT和MoO样品凝胶照片(1%)。DNA样本信息(凝胶中的1~25泳道和a~f泳道)列于表2。
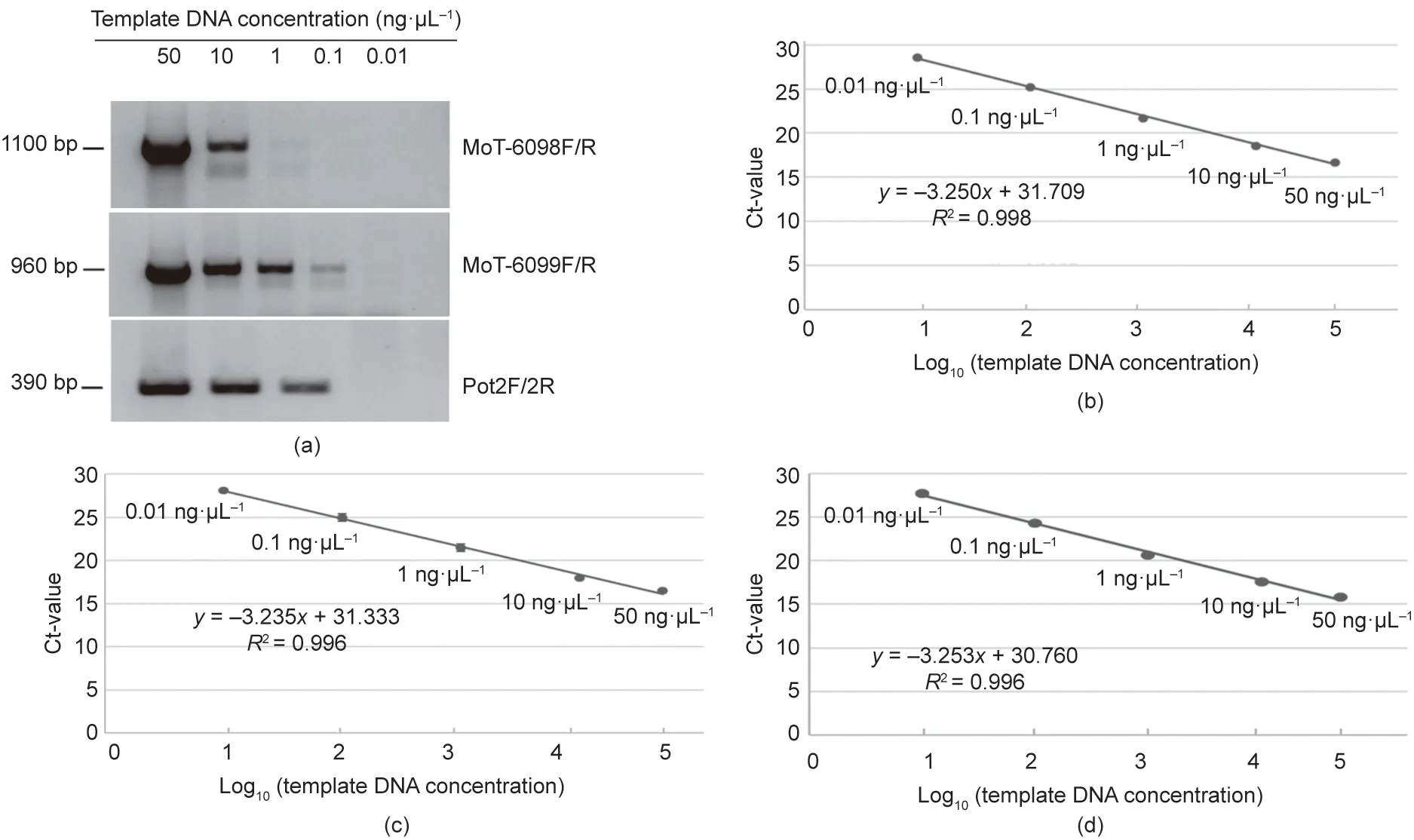
图3.用MoT菌株BO12TA301的DNA进行常规和实时荧光定量PCR,对6098和6099引物对进行敏感性分析。(a)采用常规PCR进行敏感性分析。用引物组合MoT-6098F/R、MoT-6099F/R和Pot2F/2R对1%琼脂糖凝胶进行成像,并对每个DNA样品的特定稀释物和扩增产物的大小进行了说明。(b)~(d)采用实时荧光定量PCR进行敏感性分析。分别采用MoT-6098F/R(b)、MoT-6099F/R(c)、Pot2F/2R(d)引物组合,通过实时荧光定量PCR技术,描绘出Ct值(样品的荧光超过背景荧光)与DNA模板浓度对数的关系。引物列于表1。R2:决定系数;y:PCR数据的线性模型。
在本研究中,我们首先用PCR 和LAMP 方法在等温条件下测定了引物的灵敏度。随后,我们在第一次RPA反应中加入Cas12a 蛋白和一个随机DNA 片段,用MoT-6098 或MoT-6099 引物对MoT 中的靶序列在37 ℃下进行扩增,在第二次RPA 反应中,使用生物素和FAM 标记引物扩增了随机DNA 片段并进行了PCRD 检测。用从MoT菌丝组织和感染MoT小麦叶片中分离的DNA为模板,我们获得了阳性结果。整个过程在25~40 ℃下进行,只需约半个小时,无需任何实验室设备。使用商品化PCRD条带时,每份样品的检测总费用约为6美元。如果采用非专利的方法进行大规模生产,每份样品的成本可以降低到4美元以下。植物病理学家、检疫专家和农业推广工作者很容易采用本研究开发的简单方法,在受感染的植物、种子批和其他寄主中快速检测MoT。
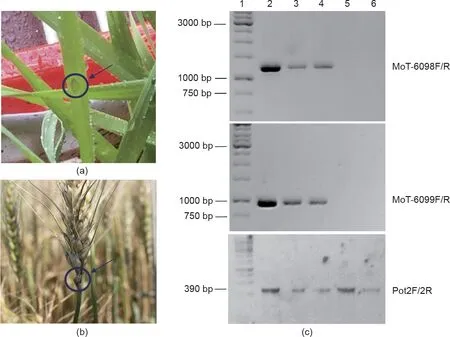
图4.检测受感染小麦植株中的MoT。(a)叶片感染的早期阶段(圆圈中表示播种后14天后叶片出现渗水病斑);(b)穗部感染(圆圈中表示在播种105天后穗颈上出现灰斑);(c)利用琼脂糖凝胶电泳对侵染后的幼株和成株的MoT序列进行PCR检测。第1泳道是DNA标记道。泳道2~6代表分离自下列样本的DNA:MoT菌株BTJP4-5的菌丝体(泳道2)、人工接种MoT菌株BTJP4-5的小麦叶片(泳道3)、MoT侵染麦田而自然感染的小麦植株的穗颈(泳道4)、MoO菌株RB-13b的菌丝体(泳道5)和MoO侵染稻田而自然感染的水稻植株的穗颈(泳道6)。
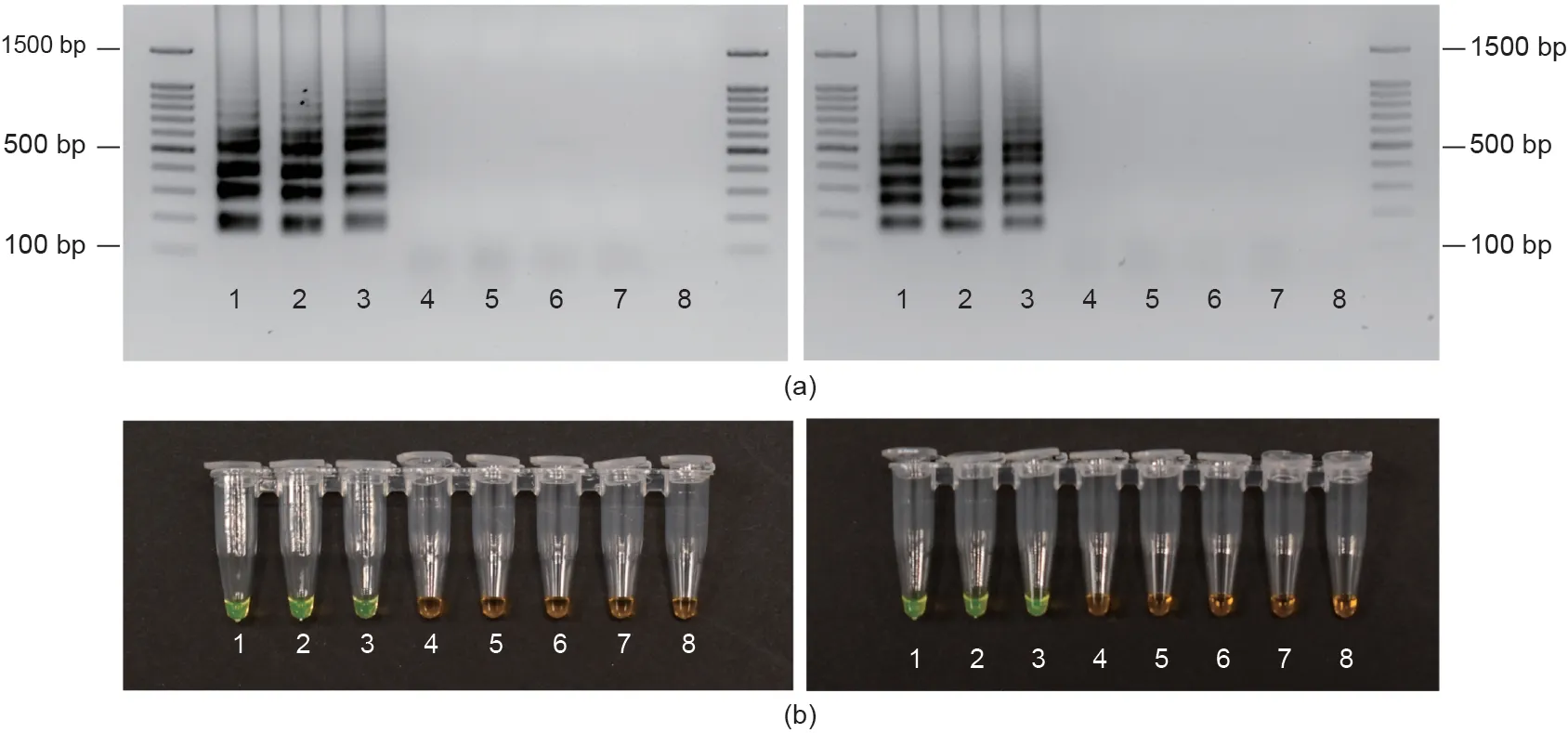
图5.用LAMP 检测MoT 序列。对8 份样本(标记为1~8)样品进行LAMP 检测,其中3 份含有MoT 基因组DNA(样本1~3),5 份含有MoO 基因组DNA(样品4~8)。(a)LAMP扩增的基因组DNA显示在2%的琼脂糖凝胶上;凝胶的左侧和右侧分别显示了针对MoT-6098和MoT-6099的LAMP引物的结果。(b)用SYBR Green染料直观展示了LAMP扩增的基因组DNA。左侧和右侧分别表示用针对MoT-6098和MoT-6099的LAMP引物得到的结果。
Acknowledgements
This research was supported by grants from the SEEDS program of the Ohio Agricultural Research and Development Center (OARDC) and the Coordinated Research Program of the International Atomic Energy Agency (IAEA) to GLW.The financial support from the Krishi Gobeshona Foundation(KGF) of Bangladesh under project KGF TF 50-C/17 to To‐fazzal Islam of the Institute of Biotechnology and Genetic En‐gineering of BSMRAU,Bangladesh,is highly acknowledged.Assistance from Dr.Francesca Hand for the LAMP assay is appreciated.Mention of trade names or commercial productsin this publication is solely for the purpose of providing spe‐cific information and does not imply recommendation or en‐dorsement by the U.S.Department of Agriculture.USDA is an equal opportunity provider and employer.
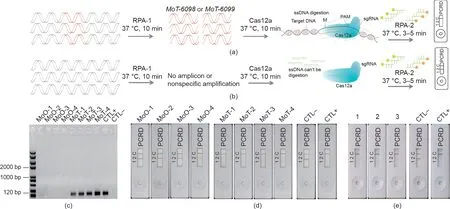
图6.通过将Cas12a蛋白与RPA和NALFIA技术相结合快速检测MoT。(a)当DNA样品中含有MoT-6098或MoT-6099序列时的Cas12a介导的检测方法;(b)DNA样品不含有MoT-6098或MoT-6099序列时的Cas12a介导的检测方法;(c)用MoT-6098引物对琼脂糖凝胶上第一个RPA扩增的DNA进行检测;(d)第二次RPA扩增MoO和MoT菌株条上用NALFIA技术检测MoT-6098的序列;(e)利用从MoT菌株BTJP4-5(泳道1)菌丝体、人工接种MoT菌株BTJP4-5(泳道2)的小麦叶片和田间自然感染MoT的小麦植株穗颈分离的DNA进行MoT-6098序列的NALFIA检测(泳道3)。
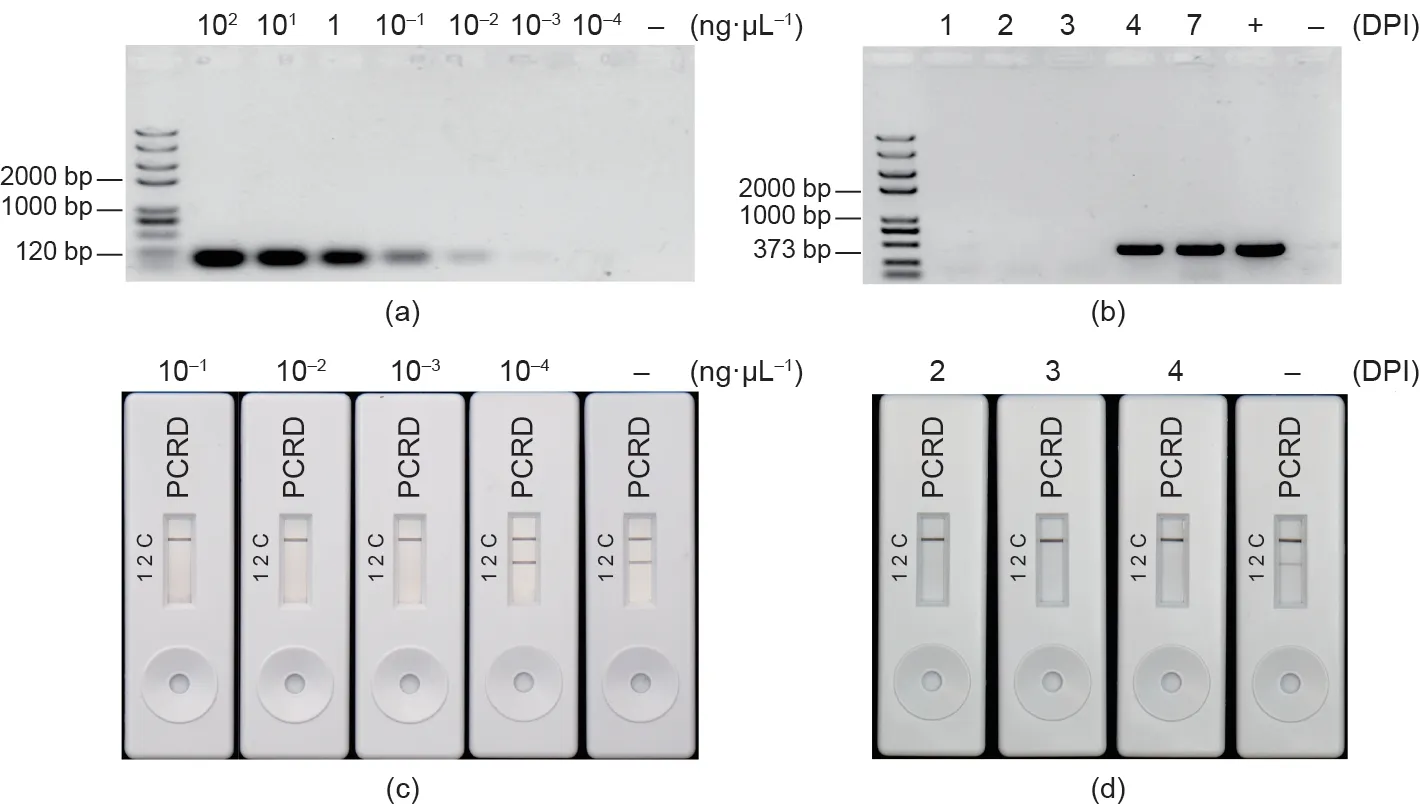
图7.Cas12a-RPA-NALFIA介导法的灵敏度测试及其与常规PCR的比较。(a)利用不同浓度的模板DNA在琼脂糖凝胶上显示PCR扩增MoT-6098序列的灵敏度;(b)在琼脂糖凝胶上显示接种后叶片模板PCR 扩增Mo-6098 序列的灵敏度;(c)PCRD 条带显示Cas12a-RPA-NALFIA 介导法检测菌丝MoT-6098序列的灵敏度;(d)PCRD条带显示Cas12a-RPA-NALFIA介导法检测接种叶片模板扩增MoT-6098序列的灵敏度。
Compliance with ethics guidelines
Houxiang Kang, Peng Ye, Kangyu Hua, Yufei Deng,Maria Bellizzi, Dipali Rani Gupta, Nur Uddin Mahmud,Alfredo S.Urashima, Sanjoy Kumar Paul, Gary Peterson,Yilin Zhou, Md Tofazzal Islam, Xueping Zhou and Guo-Liang Wang declare that they have no conflict of interest or financial conflicts to disclose
Appendix A.Supplementary data
Supplementary data to this article can be found on‐line at https://doi.org/10.1016/j.eng.2020.07.016.
