茶特征成分合成相关新转录因子鉴定
2021-01-29邰玉玲,杨林,王欢欢,刘春
邰玉玲,杨林,王欢欢,刘春



摘要:茶是世界流行的三大无酒精饮料之一,由茶树叶片加工而成,因其中含有三大茶树特征代谢产物(儿茶素、茶氨酸和咖啡碱)而具有独特风味。转录因子不仅在植物生长发育中起重要调控作用,也影响植物的次级代谢产物合成,而茶树中关于转录因子调控儿茶素,茶氨酸和咖啡碱的报道较少。本研究结合茶树全器官转录组数据与茶树基因组数据重构转录本,获得大量新基因和新转录本,进而结合茶树不同器官中儿茶素、茶氨酸和咖啡碱的含量差异,分别建立茶树中儿茶素、茶氨酸和咖啡碱与差异表达基因之间的相关性,筛选鉴定与茶树中儿茶素、茶氨酸和咖啡碱相关的基因及转录因子,并分别对与茶树中儿茶素、茶氨酸和咖啡碱合成相关的3条转录因子WAKY、bZIP、BES基因进行克隆,结果表明,其ORF框大小分别为1 011 bp,1 692 bp和867 bp,与转录组分析结果一致。
关键词:茶树;转录因子;茶氨酸;儿茶素;咖啡碱
中图分类号:S571.1文献标识码:A文章编号:1000-4440(2021)06-1534-11
Identification of new transcription factors related to the synthesis of characteristic components in tea
TAI Yu-ling1,2,YANG Lin1,WANG Huan-huan1,LIU Chun1
(1.School of Life Science, Anhui Agricultural University, Hefei 230036, China;2.State Key Laboratory of Tea Plant Biology and Utilization, Anhui Agricultural University, Hefei 230036, China)
Abstract:Tea is one of the three major non-alcoholic beverages in the world. It is processed from tea leaves and has unique flavor due to the three characteristic metabolites (catechin, theanine and caffeine) of tea. Transcription factors not only play an important role in the regulation of plant growth and development, but also affect the synthesis of secondary metabolites. There are few reports on the regulation of catechin, theanine and caffeine by transcription factors in tea plants. In this study, a large number of new genes and transcripts were reconstructed by combining whole-organ transcriptome data with genomic data of tea tree. Furthermore, the correlation between catechin, theanine and caffeine and differentially expressed genes was established based on the content differences of catechin, theanine and caffeine in different organs of tea tree. The genes transcripts and transcription factors related to the three characteristic metabolic components of tea tree were screened and identified. In addition, three transcription factors (WAKY, bZIP and BES) genes associated with the synthesis of catechin, theanine and caffeine in tea tree were cloned, and the sizes of open reading frame (ORF) were 1 011 bp, 1 692 bp and 867 bp, respectively. The results were consistent with those of transcriptome analysis.
Key words:tea;transcription factor;theanine;catechin;caffeine
茶樹(Camellia sinensis L.)作为中国重要的木本经济作物之一,在中国拥有悠久的栽培历史。而由茶树叶片加工而成的茶是世界三大无酒精饮料之一,富含大量生物活性成分。茶作为一种重要的保健饮品在全世界风靡,其保健功能与儿茶素、茶氨酸、咖啡碱等茶树特征性代谢产物有关[1-2],这些次生代谢成分在茶树生长发育过程中产生,不仅影响到茶树自身的生长发育和新陈代谢,也影响到成茶品质和保健功能[3]。
儿茶素具有苦涩味,是茶树特征次生代谢物质之一,对茶的色、香、味等品质具有重要作用。同时儿茶素也是茶保健功能的重要组成成分,具有降血脂[4]、降血压[3]、增强免疫力、抗菌、抗氧化[5]、预防心血管疾病[6]等多种药理作用。茶树中主要有2种类型的儿茶素(非酯型儿茶素和酯型儿茶素),其中非酯型儿茶素包括儿茶素(Catechin,C)、表儿茶素(Epicatechin,EC)、没食子儿茶素(Gallocatechin,GC)、表没食子儿茶素(Epigallocatechin,EGC)。酯型儿茶素有2种,分别为表儿茶素没食子酸酯(Epicatechin-3-gallate,ECG)和表没食子儿茶素没食子酸酯(Epigallocatechin-3-gallate,EGCG)[7]。酯型儿茶素是茶汤苦涩味的主要来源,其中EGCG含量最高,抗氧化能力也最强 [8],为茶中特色成分。
茶氨酸是植物中罕有的非蛋白氨基酸,也是茶树特有的次级代谢产物,占茶树游离氨基酸含量的40%~70%,占茶叶干质量的1%~2%[9]。迄今茶氨酸只在山茶科植物和蕈中[10-11]被检测出,且含量少[12]。茶氨酸作为茶树特征性成分之一,具有独特的鲜爽味,可以缓解茶汤的苦涩味,增加其甜味,极大地影响茶汤品质,也是鉴定绿茶品质的一项重要标准。此外,茶氨酸具有抗肿瘤、抗疲劳、降血压、镇静安神[13-15]和提高记忆力[6]等多种保健功能,在食品和医药领域被广泛应用[16-17]。研究结果表明,茶氨酸主要在茶树根部合成,再从根部运输至芽、叶并积累[18]。
茶树中主要含有3种嘌呤碱,分别为咖啡碱、可可碱和茶叶碱,其中咖啡碱含量最高,约占茶叶干质量的2%~5%[19],在茶树芽中的含量较高。咖啡碱是具有苦味的植物嘌呤碱,是茶树重要的次生代谢产物之一,也是影响茶汤滋味品质的主要成分之一,具有兴奋、利尿、解乏、增加心血管系统活动、抗菌和提高思维效率等保健作用。
植物生长的自然环境复杂多变,因此植物会根据自然环境变化调控体内特定基因的表达以提高对环境的适应性。植物中各种诱导型基因的表达主要受特定转录因子在转录水平上的调控,转录因子在植物生长发育及其对外界环境的反应中起重要作用。转录因子(反式作用因子)能与启动子中的顺式作用元件发生特异性结合,从而激活或抑制转录[20]。茶树三大代谢物相关的研究主要集中在活性成分提取鉴定以及合成途径、基因克隆验证等,关于转录因子调控其合成研究较少,近期有研究者在茶树中鉴定并研究了 bHLH 的功能,发现 bHLH 转录因子主要和茶树抗性相关[21-22]。 而关于转录因子调控茶树次生代谢成分合成相关基因的报道较少,李明卓等[23]发现MYB 转录因子与茶树黄酮类及木质素合成相关,Wen等[24]对参与茶氨酸合成调控的 MYB 进行了研究,结果表明,MYB 与茶氨酸的积累呈负调控关系。本研究结合已发表的茶树基因组数据[25],利用茶树全器官转录组数据[26]重构转录本,并对这些重构的转录本进行鉴定及分析。同时鉴定与茶树三大特征代谢物(儿茶素、茶氨酸、咖啡碱)合成相关的转录因子,并对这些转录因子基因进行克隆验证。本研究利用重构转录本的新方法对茶树基因组及转录组数据进行分析,为利用茶树基因组及转录组数据提供重要的参考方法,同时鉴定出的转录因子为进一步研究茶树三大特征代谢物合成调控奠定基础。
1材料与方法
1.1参考基因组比对
对测序获得的茶树全器官原始测序数据(Raw reads)进行过滤,去除低质量,包含接头污染以及未知碱基氮含量高于5%的reads,获得高质量的数据(Clean reads),并保存为FASTQ格式[27],以保证后续数据分析结果的可靠性。用HISAT软件[28]将过滤后的Clean reads比对到茶树参考基因组(http://tpia.teaplant.org/download.html)。
1.2新转录本及其编码能力预测
利用StringTie软件[29]对方法1.1里获得的数据进行转录本重构,之后用Cuffcompare 将重构后的转录本与参考注释信息进行比较分析,挑选 Class code 类型为u、i、o、j的转录本,定义为新转录本和新基因(即Novel gene,指原基因组未注释到的基因)。将得到的新转录本和新基因用CPC[30]进行蛋白质编码能力预测,进一步鉴定可能具有蛋白质编码能力的新转录本和新基因,将预测具有蛋白质编码能力的新转录本和新基因加入到参考基因序列中进而补充茶树基因组的基因注释信息,获得完整的参考序列信息。
1.3基因表达量分析
将具有蛋白质编码能力的新转录本和新基因與参考基因序列构建成为完整的参考基因数据库。使用Bowtie 2[31]将茶树全器官Clean reads比对到参考序列,基于比对后的bam文件,使用RSEM软件[32]计算基因和转录本的表达水平。根据表达信息,使用R语言绘制箱线图,展示茶树全器官样品基因表达水平的分布情况。
1.4差异表达基因检测与样品间差异表达基因分析
根据各个样品基因表达水平检测不同样品间的差异表达基因。使用DEseq2算法对样品进行差异表达基因分析(差异倍数≥2.00,Q≤0.05)。由于儿茶素、茶氨酸和咖啡碱主要集中在茶树芽和幼嫩的叶片中,因此本研究以茶树芽(CS-B)作为对照,分别与茶树茎(CS-S)、茶树根(CS-R)、茶树花(CS-FL)、茶树果实(CS-FR)进行比较,鉴定共有差异表达基因。同时,以茶树第一叶(CS-L1)作为对照,分别与茶树茎、茶树花、茶树根和茶树果实进行比较,鉴定共有差异表达基因。最后,以茶树根作为对照,分别与茶树茎、茶树花、茶树芽、茶树第一叶和茶树果实进行比较,鉴定共有差异表达基因。采用RSEM软件中的hclust 函数对样品进行层次聚类分析,并绘制韦恩图用来展示样品间基因表达分布。
1.5儿茶素、茶氨酸和咖啡碱相关基因分析
儿茶素、茶氨酸和咖啡碱是茶中三大特征性代谢成分,因而本研究主要鉴定和筛选与三大特征代谢成分相关的基因。首先,根据鉴定到的差异表达基因,使用R语言的cor.test软件包计算差异表达基因与儿茶素、茶氨酸和咖啡碱的皮尔逊相关系数,筛选相关系数绝对值>7且P<0.05的基因作为候选基因。由于儿茶素在幼嫩芽、叶中含量较高,本研究进一步筛选在茶树第一叶和茶树芽中显著差异表达的基因。同时,由于茶氨酸在茶树根中含量较高,本研究进一步筛选在茶树根中显著差异表达的基因,绘制韦恩图展示交集和并集的信息。
1.6儿茶素、茶氨酸和咖啡碱相关转录因子基因克隆分析
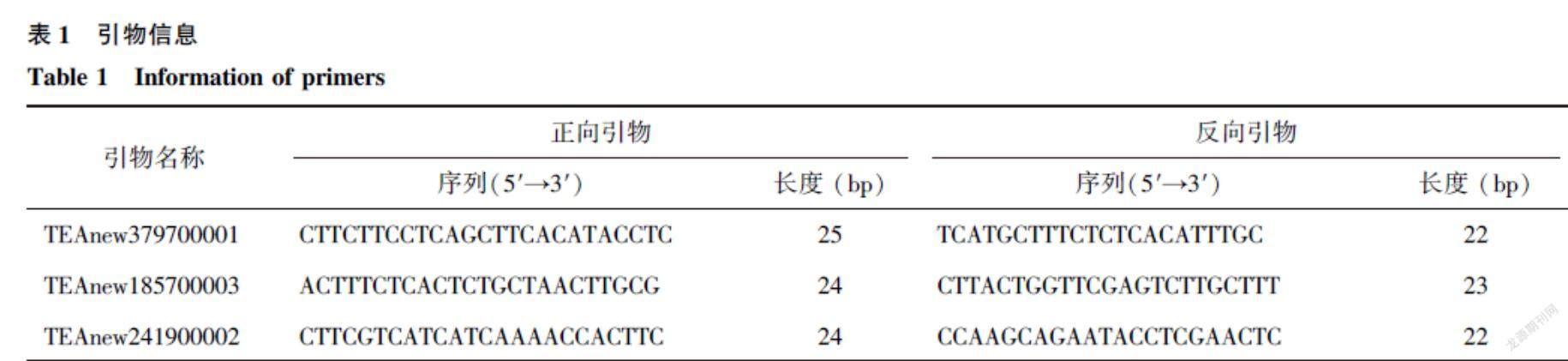
为了鉴定得到与茶树三大特征代谢成分相关的转录因子,在方法1.5的基础上,结合TFDB[33]数据库和iTAK [34]软件,从三大特征代谢成分相关基因中鉴定得到转录因子。为了验证本研究获得的新基因的准确性,我们筛选了3个与儿茶素、茶氨酸和咖啡碱具有较高相关性的转录因子进行验证,分别为TEAnew241900002、TEAnew379700001、TEAnew185700003,并分别对3个候选转录因子基因进行克隆验证和分析。
1.6.1总RNA提取取0.2 g茶树样品,按照Tai等[12]的方法提取茶树总RNA。提前将交联聚乙烯吡咯烷酮(PVPP)加入预冷的研钵中,加入组织样品后液氮磨成粉末,取适量粉末加入已加入600 μl Buffer RLB和30 μl β-巯基乙醇的离心管中。加入50 μl Plantaid混匀,然后参考天根生化科技(北京)有限公司的RNAprep pure Plant Kit试剂盒说明书操作,用琼脂糖凝胶电泳对总 RNA 完整性进行检测。
1.6.2目的基因克隆及分析用TransScript反转录试剂盒对获得的RNA进行反转录。根据筛选的候选基因,结合NCBI确定候选基因的ORF框,采用Primer 5软件设计引物(表1),采用PCR方法进行扩增。PCR反应体系为20.0 μl,分别为10×PCR Buffer 2.0 μl,dNTP Mixture 2.5 μl,上下游引物各0.8 μl,25 mmol/L MgSO4 1.5 μl,KOD酶 0.5 μl,添加dd H2O至20.0 μl。PCR程序:94 ℃ 2 min;98 ℃ 10 s,55 ℃ 30 s,68 ℃ 1 min,68 ℃ 10 min,16 ℃保存,设置30个循环,PCR后,用琼脂糖凝胶(1.5%)电泳检测目的片段。利用胶回收试剂盒对获得的PCR产物进行纯化,用克隆载体pEASY-Blunt Zero进行连接,转至DH5α大肠杆菌感受态细胞培养并筛选阳性克隆送至北京六合华大基因科技有限公司进行基因测序,用 DNAMAN、Bioedit、seqMan 等软件对测序结果进行分析,并与 NCBI 进行 Blast 分析,确认是否获得完整的 ORF框。
2结果与分析
2.1参考基因组比对
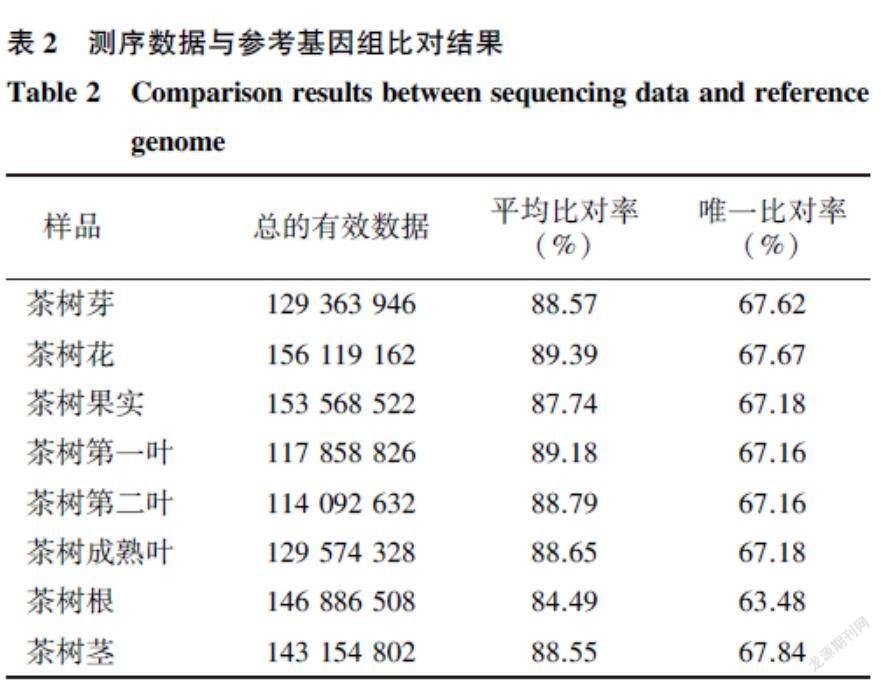
利用HISAT软件将Clean reads 比对到参考基因组序列(表2)。每个样品的平均比对率均大于84%,唯一比对率均大于63%,说明测序结果较好,符合进一步分析的要求。
2.2新转录本重构及编码能力预测
将与参考基因组比对后的样品进行转录本重构和注释[35],共获得81 045个新转录本。其中具有蛋白质编码能力的新转录本有50 111个,不具有蛋白质编码能力的新转录本有30 934个,在茶树基因组中,已有基因的新转录本有34 155个,新基因转录本有15 956个。
2.3基因表达量分析
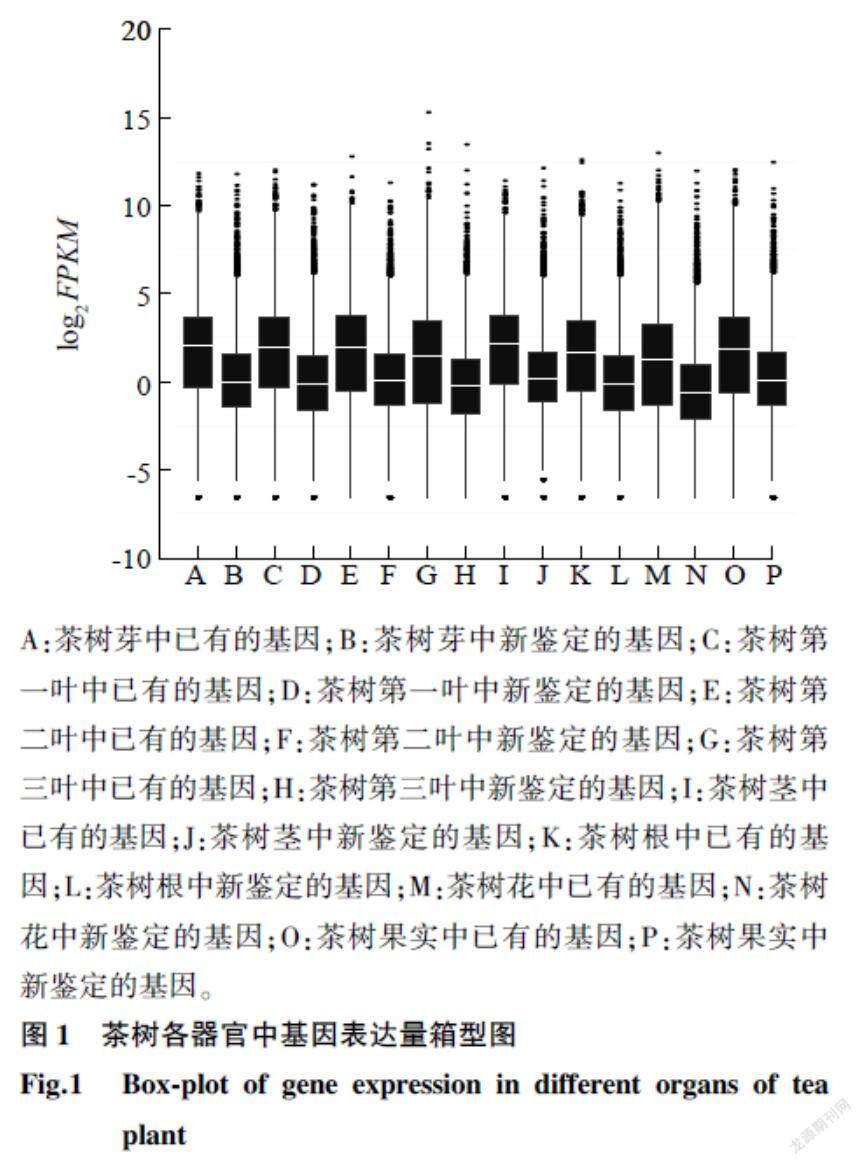
将茶树各器官样品的Clean reads 通过Bowtie2比对到参考序列。采用箱线图(图1)展示各样品基因表达水平的分布情况,log2(FPKM)值大小可表示表达量的高低。图1表明,茶树各器官新鉴定的转录本的表达量都低于已知转录本,表明基因组装的时候对于表达量较高的转录本的组装效果较好,而对于一些表达量低或者组织特异表达的基因鉴定效果較差,利用转录本重构的方法可以鉴定出表达量较低的转录本。
A:茶树芽中已有的基因;B:茶树芽中新鉴定的基因;C:茶树第一叶中已有的基因;D:茶树第一叶中新鉴定的基因;E:茶树第二叶中已有的基因;F:茶树第二叶中新鉴定的基因;G:茶树第三叶中已有的基因;H:茶树第三叶中新鉴定的基因;I:茶树茎中已有的基因;J:茶树茎中新鉴定的基因;K:茶树根中已有的基因;L:茶树根中新鉴定的基因;M:茶树花中已有的基因;N:茶树花中新鉴定的基因;O:茶树果实中已有的基因;P:茶树果实中新鉴定的基因。
2.4差异表达基因检测
根据茶树各器官样品基因表达水平,可以检测样品之间的差异表达基因,检测结果见图2。以茶树花作为对照,分别将茶树芽、茶树第一叶、茶树根与茶树花做差异表达基因分析。其中茶树花和茶树芽比较组上调表达的差异基因个数最多(13 042个),其次为茶树花和茶树第一叶比较组(12 140个)和茶树第三叶和茶树根比较组(11 836个)。下调表达的差异基因个数最多的为茶树茎和茶树根比较组(10 589个),其次为茶树第二叶和茶树根比较组(9 418个)、茶树第一叶和茶树根比较组(9 064个)。
由于儿茶素和咖啡碱在茶树芽中含量最高,在茶树的其他器官中含量较低。因此将茶树芽中与茶树根、茶树花、茶树茎、茶树果实中的差异表达基因分别进行比较,鉴定出的共有差异表达基因命名为DEG-CS-B。采用R语言软件包进行统计分析,对样品组的差异表达基因进行层次聚类分布,用韦恩图来表示样品间差异表达基因的分布(图3A),获得3 859个共同差异表达基因,茶树第一叶中儿茶素和咖啡碱的含量仅次于芽,以茶树第一叶与其他器官中差异表达基因进行比较, 鉴定共有差异表达基因命名为DEG-CS-L1,以韦恩图展示样品间差异表达基因的分布(图3B)。茶氨酸在茶树根中的含量最高,以根作为对照,分别用茶树根与其他器官进行比较,采用R语言软件分析统计,绘制韦恩图来表示样品间的差异表达基因分布(图3C), 鉴定出的共有差异表达基因命名为DEG-CS-R。获得5 540个共同差异表达基因。
a:茶树花和茶树芽比较组;b:茶树花和茶树第一叶比较组;c:茶树花和茶树根比较组;d:茶树果实和茶树芽比较组;e:茶树果实和茶树第一叶比较组;f:茶树果实和茶树根比较组;g:茶树第一叶和茶树根比较组;h:茶树第二叶和茶树芽比较组;i:茶树第二叶和茶树第一叶比较组;j:茶树第二叶和茶树根比较组;k:茶树第三叶和茶树芽比较组;l:茶树第三叶和茶树第一叶比较组;m:茶树第三叶和茶树根比较组;n:茶树根和茶树芽比较组;o:茶树根和茶树第一叶比较组;p:茶树茎和茶树芽比较组;q:茶树茎和茶树第一叶比较组;r:茶树茎和茶树根比较组。
A:茶树芽与茶树根、茶树花、茶树茎、茶树果实差异表达基因比较分析;B:茶树第一叶与茶树根、茶树花、茶树果实、茶树茎差异表达基因比较分析;C:茶树根与茶树第一叶、茶树花、茶树果实、茶树茎差异表达基因比较分析。CS-FL-VS-CS-B:茶树花和茶树芽比较组;CS-S-VS-CS-B:茶树茎和茶树芽比较组;CS-R-VS-CS-B:茶树根和茶树芽比较组;CS-FR-VS-CS-B:茶树果实和茶树芽比较组;CS-FL-VS-CS-L1:茶树花和茶树第一叶比较组;CS-S-VS-CS-L1:茶树茎和茶树第一叶比较组;CS-R-VS-CS-L1:茶树根和茶树第一叶比较组;CS-FR-VS-CS-L1:茶树果实和茶树第一叶比较组;CS-FL-VS-CS-R:茶树花和茶树根比较组;CS-FR-VS-CS-R:茶树果实和茶树根比较组;CS-S-CS-R:茶树茎和茶树根比较组。
2.5与儿茶素、茶氨酸和咖啡碱合成相关的差异表达基因分析
首先,通过相关性分析,获得与儿茶素、茶氨酸、咖啡碱具有相关性的差异表达基因。
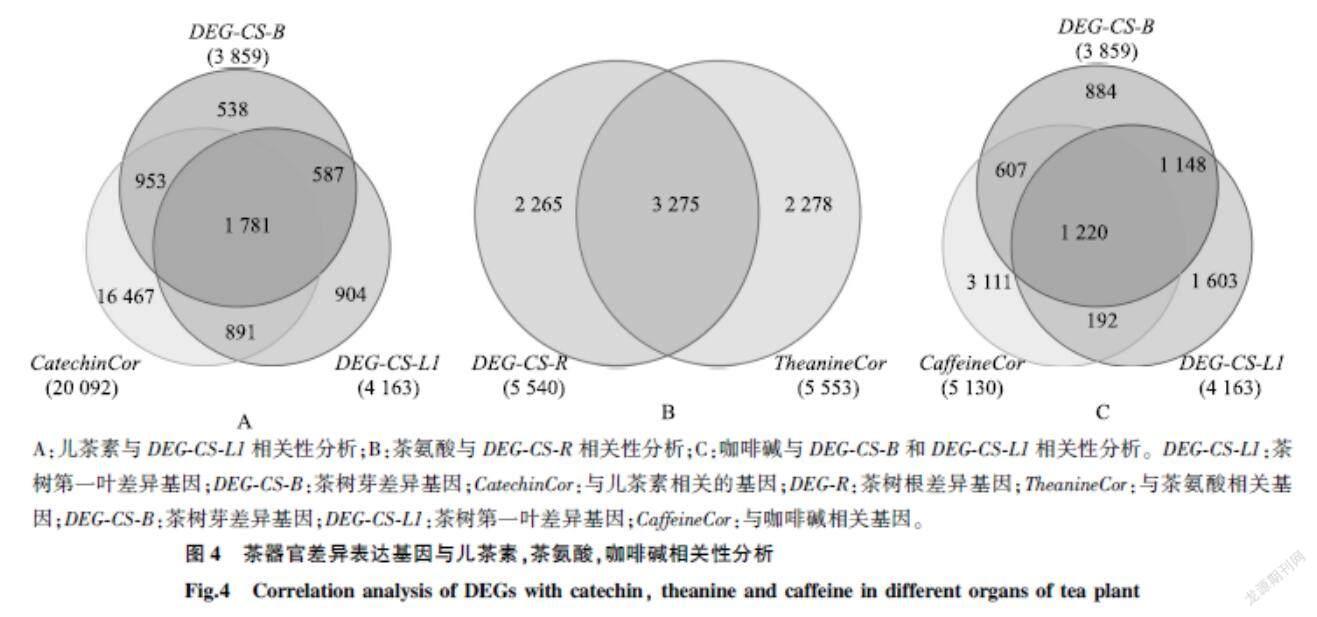
由于茶树芽与第一叶中儿茶素含量较高,本研究进一步筛选CatechinCor(与儿茶素合成相关的基因)且属于DEG-CS-L1和DEG-CS-B的基因,获得与儿茶素具有相关性且在芽和第一叶中显著差异表达的基因,这些基因可能在茶树的儿茶素积累中起到作用(图4A)。与儿茶素合成相关的共有差异表达基因1 781个,芽中与儿茶素合成相关基因2 734个,第一叶中与儿茶素合成相关基因2 672个。芽中与儿茶素合成相关基因略高于第一叶,而芽中的儿茶素含量也略高于第一叶,推测这些差异表达基因可能与儿茶素合成代谢相关,且与儿茶素合成代谢相关的差异表达基因越多,儿茶素的含量也越高。
茶树中,根中的茶氨酸含量最高,筛选TheanineCor(与茶氨酸合成相关的基因)且属于DEG-CS-R的基因,获得跟茶氨酸合成相关的基因且在根中显著差异表达的基因(图4B)。与茶氨酸合成相关的共有的差异表达基因有3 275个。
A:儿茶素与DEG-CS-L1相关性分析;B:茶氨酸与DEG-CS-R相关性分析;C:咖啡碱与DEG-CS-B和DEG-CS-L1相关性分析。DEG-CS-L1:茶树第一叶差异基因;DEG-CS-B:茶树芽差异基因;CatechinCor:与儿茶素相关的基因;DEG-R:茶树根差异基因;TheanineCor:与茶氨酸相关基因;DEG-CS-B:茶树芽差异基因;DEG-CS-L1:茶树第一叶差异基因;CaffeineCor:与咖啡碱相关基因。
茶树芽中咖啡碱含量最高,其次为第一叶,因此,筛选CaffeineCor(与咖啡碱合成相关的基因)且属于的DEG-CS-L1和DEG-CS-B的基因,获得与咖啡碱合成相关的基因且在芽和第一叶中显著差异表达的基因(图4C)。分析获得与咖啡碱合成相关的共有差异表达基因1 220个。芽中与咖啡碱合成相关基因有1 827个,第一叶中与咖啡碱合成相关基因有1 412个。芽中与咖啡碱代谢有关的基因高于第一叶,而芽中咖啡碱含量高于第一叶,推测这些差异表达基因可能与咖啡碱合成代谢相关,且与咖啡碱合成代谢相关的差异表达基因越多,咖啡碱含量也越高。
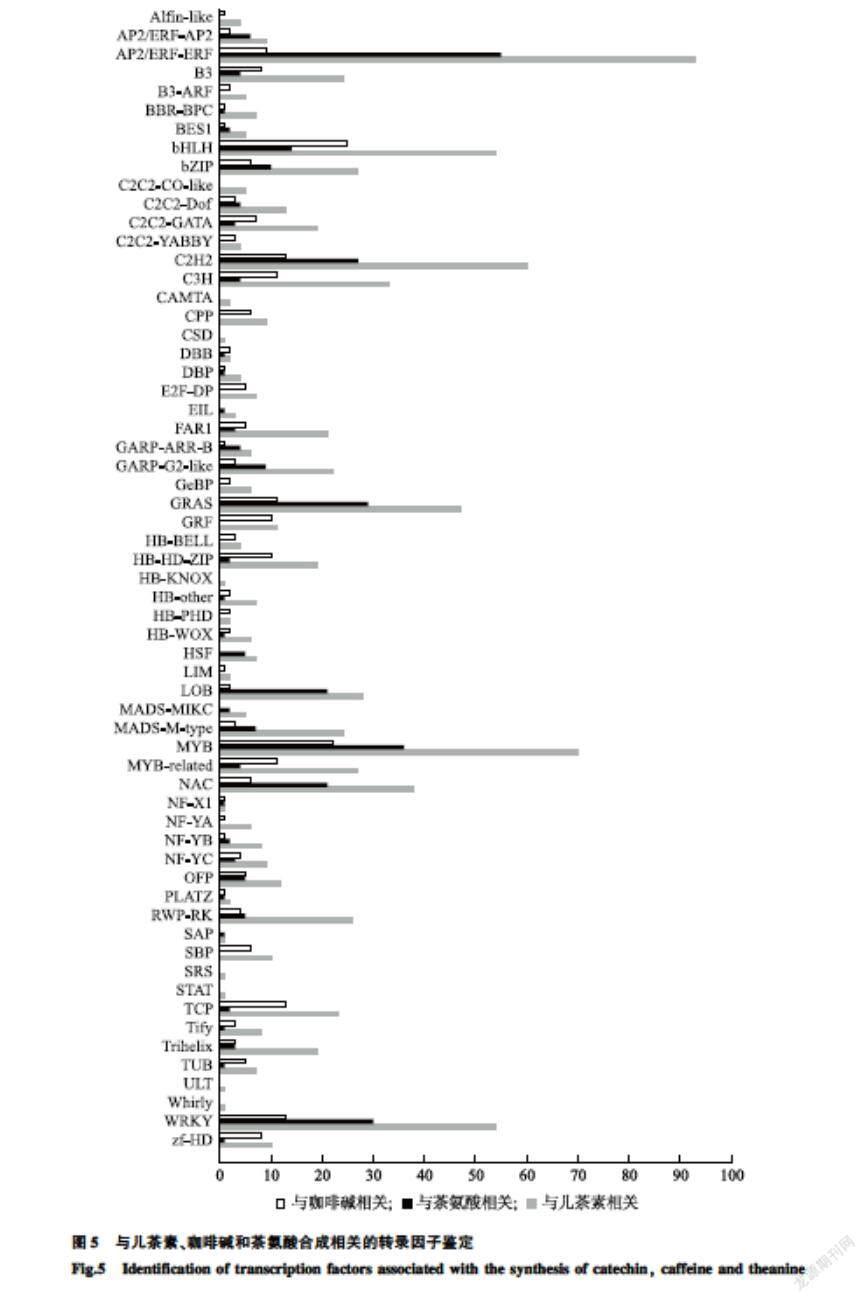
2.6与茶树中儿茶素、茶氨酸和咖啡碱合成相关的转录因子的鉴定
对筛选得到的与儿茶素、茶氨酸和咖啡碱合成相关的转录因子进行分析和鉴定,结果(图5)表明,与儿茶素合成相关的转录因子数量最多,其次为茶氨酸,与咖啡碱合成相关的转录因子数量最少。其中AP2/ERF-ERF、MYB、C2H2、WRKY和bHLH等转录因子在三大代谢成分相关的转录因子中占的比例较高。
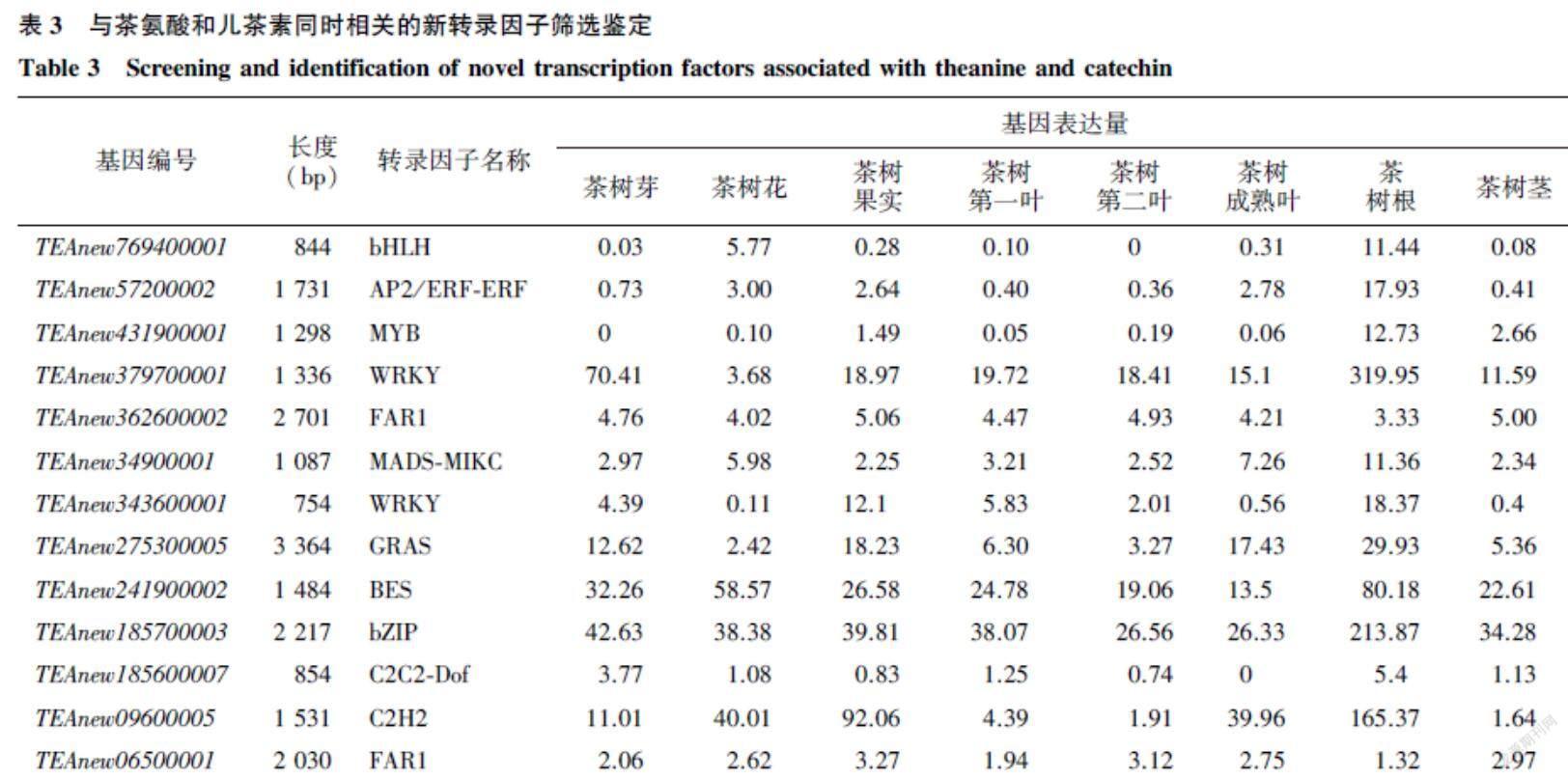
与儿茶素合成相关的转录因子主要有AP2/ERF-ERF、MYB、C2H2、WRKY和bHLH,与茶氨酸合成相关的转录因子有AP2/ERF-ERF、MYB、WRKY、GRAS和C2H2,与咖啡碱合成相关的转录因子有bHLH、MYB、WRKY、TCP和C2H2。在与儿茶素合成相关的转录因子中,鉴定了注释为WRKY、MYB和bHLH的新转录因子,在与茶氨酸合成相关的转录因子中,鉴定了注释为WRKY、NAC、MYB、MADS-M-type、MADS-MIKC、LOB、GRAS、FAR1、C2H2、C2C2-Dof、bZIP、bHLH和AP2/ERF-ERF的新转录因子,这些转录因子与茶树三大代谢物密切相关。本研究进一步分析了这些新转录因子并注释其功能,并筛选新转录因子进行功能验证(表3)。
2.7与儿茶素,茶氨酸和咖啡碱合成相关的转录因子基因的克隆验证
通过PCR方法克隆获得与茶树中儿茶素,茶氨酸和咖啡碱合成相关的3条转录因子[TEAnew379700001(WAKY)、TEAnew185700003(bZIP)、TEAnew241900002(BES)]基因,其ORF框大小分别为1 011 bp,1 692 bp和867 bp,分别编码336个,563个和288个氨基酸(图6)。分别将克隆获得的3个转录因子基因编码的氨基酸序列与通过转录本重构方法获得的序列进行比较,发现其相似度均大于99% (图 7),其中BES的相似度为100%,表明,转录本重构获得新转录本的方法准确度较高。
3讨论
茶具有特殊的滋味和香气,同时具有多种保健功能,其保健功能主要和茶树活性次级代谢物相关,如儿茶素、茶氨酸和咖啡碱等[36-37],这3种成分作为茶叶的特征性代谢产物而受到广泛关注。本研究主要利用已有的茶树转录组和基因组数据,对茶树转录本进行重构,鉴定新转录本,同时结合儿茶素,茶氨酸和咖啡碱在茶树根、茎、叶等器官中的含量差异,分别建立儿茶素、茶氨酸和咖啡碱与茶树转录本的相关性,鉴定与茶树三大特征代谢成分相关的关键酶基因及转录因子。
对于茶树中儿茶素、茶氨酸和咖啡堿的研究主要集中在成分含量检测,关键酶基因的克隆及不同器官、不同品种的茶树转录组测序。儿茶素合成途径中一些关键酶已基本被克隆[38],与儿茶素合成途径有关的其他基因也相继被报道[39]。茶氨酸合成途径中相关酶的研究不多,且参与茶氨酸合成的酶大多较不稳定,分离纯化和酶活性检测技术的欠缺,使得与茶氨酸合成途径相关的酶的研究仍在初级阶段。现今对茶树中儿茶素相关的转录因子MYB和bHLH已有研究,但茶氨酸和咖啡碱相关的转录因子研究较少。本研究结果表明,与儿茶素和咖啡碱合成相关的基因在茶树芽和第一叶中数量较高,而与茶氨酸合成相关的基因在茶树根中数量最高,这些结果与以往的研究结果[12,39]一致。
利用茶树全器官转录组数据重构转录本,将得到的新转录本通过已发表的茶树基因组数据进行鉴定及分析,鉴定出与茶树儿茶素、茶氨酸和咖啡碱合成相关的新转录本及转录因子,同时对鉴定重构转录因子基因进行克隆验证。转录本重构的方法,能够鉴定出基因组组装过程中没有鉴定到的基因,或者组织器官特异表达的转录本,对与茶树中儿茶素、茶氨酸和咖啡碱合成相关的基因进行了补充,也为进一步研究茶树次生代谢及基因功能提供了大量的数据支撑。通过对筛选的转录因子基因进行克隆,再次对重构转录本方法进行验证。
参考文献:
[1]LIANG Y R, MA W Y, LU J L, et al. Comparison of chemical compositions of Ilex latifolia Thumb and Camellia sinensis L.[J]. Food Chemistry, 2001, 75(3): 339-343.
[2]MAMATI G E, LIANG Y R, LU J L. Expression of basic genes involved in tea polyphenol synthesis in relation to accumulation of catechins and total tea polyphenols [J]. Journal of the Science of Food & Agriculture, 2010, 86(3): 459-464.
[3]SUEOKA N, SUGANUMA M, SUEOKA E, et al. A new function of green tea: prevention of lifestyle-related diseases [J]. Annals of the New York Academy of Sciences, 2010, 928(1): 274-280.
[4]THIELECKE F, BOSCHMANN M. The potential role of green tea catechins in the prevention of the metabolic syndrome-a review [J]. Phytochemistry, 2009, 70(1): 11-24.
[5]YANG L, COLDITZ G A. Prevalence of overweight and obesity in the United States, 2007-2012 [J]. Jama Internal Medicine, 2015, 175(8): 1412.
[6]林智,吕海鹏,张盛. 茶叶活性成分的化学和药理作用 [J]. 中国茶叶, 2018, 40(11): 1-6.
[7]YANG C S, CHEN G, WU Q. Recent scientific studies of a traditional chinese medicine, tea, on prevention of chronic diseases [J]. Journal of Traditional & Complementary Medicine, 2014, 4(1): 17-23.
[8]方芳,崔志清,韩永晶. 茶儿茶素的药效研究概况 [J]. 中草药, 2000, 31(5): 396-398.
[9]杨洲. 茶氨酸保健作用研究进展 [J]. 茶叶通讯, 2018, 45(2):3-7.
[10]CHUNG F L, SCHWARTZ J, HERZOG C R, et al. Tea and cancer prevention: studies in animals and humans [J]. Journal of Nutrition, 2003, 133(10): 3268.
[11]LI J, LI P, LIU F. Production of theanine by Xerocomus badius (mushroom) using submerged fermentation [J]. LWT - Food Science and Technology, 2008, 41(5): 883-889.
[12]TAI Y L, WEI C L, YANG H, et al. Transcriptomic and phytochemical analysis of the biosynthesis of characteristic constituents in tea ( Camellia sinensis ) compared with oil tea ( Camellia oleifera ) [J]. Bmc Plant Biology, 2015, 15(1): 190.
[13]ZHANG G Y, MIURA Y, YAGASAKI K. Effects of dietary powdered green tea and theanine on tumor growth and endogenous hyperlipidemia in hepatoma-bearing rats [J]. Journal of the Agricultural Chemical Society of Japan, 2002, 66(4): 711-716.
[14]譚俊峰,林智,彭群华,等. 茶氨酸复合制剂缓解体力疲劳功能评价 [J]. 茶叶科学, 2012, 32(6): 530-534.
[15]林伟东,孙威江,郭义红,等. 茶叶中茶氨酸的研究与利用 [J]. 食品研究与开发, 2016, 37(20): 201-206.
[16]张莹,施兆鹏,施玲. 茶氨酸的研究进展 [J]. 天然产物研究与开发, 2003, 15(4): 369-372.
[17]林雪玲,程朝辉,黄才欢,等. 茶氨酸对小鼠学习记忆能力的影响 [J]. 食品科学, 2004(5): 171-173.
[18]HO K, KATO T, XU H L. Transport of nitrogen assimilation in xylem vessels of green tea plants fed with NH+4-N and NO-3-N[J]. Pedosphere, 2008, 18(2): 222-226.
[19]LIU Y J, GAO L P, TAO X, et al. Investigation of the site-specific accumulation of catechins in the tea plant (Camellia sinensis (L.) O. Kuntze) via vanillin-HCl staining [J]. Journal of agricultural and food chemistry, 2009, 57(21): 10371-10376.
[20]刘强,张贵友. 植物转录因子的结构与调控作用 [J]. 科学通报, 2000, 45(14): 1465-1474.
[21]CUI X, WANG Y X, LIU Z W, et al. Transcriptome-wide identification and expression profile analysis of the bHLH family genes in Camellia sinensis [J]. Functional & Integrative Genomics, 2018, 18(5): 489-503.
[22]韓永涛. 茶树CsbHLH2基因克隆及功能分析 [D].杨凌:西北农林科技大学,2015.
[23]李明卓. 茶树CsMYB4a转录因子以及靶基因Cs4CLs的功能验证 [D]. 合肥:安徽农业大学, 2016.
[24]WEN B B, LUO Y, LIU D M, et al. The R2R3-MYB transcription factor CsMYB73 negatively regulates l -Theanine biosynthesis in tea plants ( Camellia sinensis L.) [J]. Plant Science, 2020, 298: 110546.
[25]WEI C L, YANG H, WANG S B, et al. Draft genome sequence of Camellia sinensis var. sinensis provides insights into the evolution of the tea genome and tea quality [J]. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(18): 4151-4158.
[26]TAI Y L, LIU C, YU S W, et al. Gene co-expression network analysis reveals coordinated regulation of three characteristic secondary biosynthetic pathways in tea plant (Camellia sinensis) [J]. Bmc Genomics, 2018, 19(1): 616.
[27]COCK P J A, FIELDS C J, NAOHISA G, et al. The sanger FASTQ file format for sequences with quality scores, and the Solexa/Illumina FASTQ variants [J]. Nucleic Acids Research, 2009, 38(6): 1767-1771.
[28]DAEHWAN K, BEN L, SALZBERG S L. HISAT: a fast spliced aligner with low memory requirements [J]. Nature Methods, 2015, 12(4): 357-360.
[29]PERTEA M, KIM D, PERTEA G M, et al. Transcript-level expression analysis of RNA-seq experiments with HISAT, StringTie and Ballgown [J]. Nature Protocols, 2016, 11(9): 1650.
[30]LEI K, YONG Z, ZHI Q Y, et al. CPC: assess the protein-coding potential of transcripts using sequence features and support vector machine [J]. Nucleic Acids Research, 2007, 35: 345.
[31]LANGDON B W. Performance of genetic programming optimised Bowtie2 on genome comparison and analytic testing (GCAT) benchmarks [J]. Biodata Mining, 2015, 8(1): 1.
[32]MIHAELA P, PERTEA G M, ANTONESCU C M, et al. StringTie enables improved reconstruction of a transcriptome from RNA-seq reads [J]. Nature Biotechnology, 2015, 33(3): 290-295.
[33]JIN J P, TIAN F, YANG D C, et al. PlantTFDB 4.0: toward a central hub for transcription factors and regulatory interactions in plants [J]. Nucleic Acids Research, 2017, 45(1) :1040-1045.
[34]ZHENG Y, JIAO C, SUN H H,et al. iTAK: a program for genome-wide prediction and classification of plant transcription factors, transcriptional regulators, and protein kinases. [J]. Molecular Plant, 2016, 9(12):1667-1670.
[35]SHIHAO S, JUW WON P, ZHI-XIANG L, et al. rMATS: robust and flexible detection of differential alternative splicing from replicate RNA-Seq data [J]. Proc Natl Acad Sci USA, 2014, 111(51): 5593-5601.
[36]劉文静,傅建炜,何明燕. 福建3类陈年老茶有机酸组分及含量比较分析[J].江苏农业科学,2020,48(20):224-229.
[37]杨转,刘玉飞,郭桂义,等. 黄金叶与信阳群体种信阳毛尖茶的感官品质与化学成分比较[J]. 江苏农业科学,2019,47(20):206-208,217.
[38]马春雷,陈亮. 茶树功能基因分离克隆研究进展 [J]. 分子植物育种, 2006, 4(1): 16-22.
[39]XIONG L G, LI J, LI Y H, et al. Dynamic changes in catechin levels and catechin biosynthesis-related gene expression in albino tea plants ( Camellia sinensis L.) [J]. Plant Physiol Biochem, 2013, 71(71): 132-143.
(责任编辑:陈海霞)
收稿日期:2021-08-19
基金项目:安徽农业大学青年基金项目(2016ZR012)
作者简介:邰玉玲(1987-),女,山东临沂人,博士,副教授,主要从事植物次生代谢及分子生物学研究。(Tel) 13335655226;(E-mail)taiyuling1102@126.com。杨林为共同第一作者。
