黄秋葵实时荧光定量PCR内参基因的克隆与筛选评价
2021-01-28李永平叶新如陈敏氡刘建汀朱海生温庆放
李永平 叶新如 王 彬 陈敏氡 刘建汀 朱海生 温庆放
(福建省蔬菜遗传育种重点实验室/福建省农业科学院作物研究所/福建省农业科学院蔬菜研究中心/福建省蔬菜工程技术研究中心,福建 福州 350013)
黄秋葵(HibiscusesculentusL.)属于秋葵属,其嫩果可食用,含有丰富的蛋白质、维生素及矿物盐、多糖聚合体等营养物质,是一种具有较高营养价值、保健功能的新型蔬菜[1-2],已成为许多国家运动员的首选蔬菜[3]。
实时荧光定量PCR(quantitative real-time PCR,RT-qPCR)能够精确定量复杂混合物中特定核酸,是定量分析各种目的基因和基因表达量的重要技术手段[4]。影响RT-qPCR分析结果准确性的因素包括RNA质量、内参基因及引物特异性等[5],其中内参基因的稳定性对其结果影响最大。因此,有必要选择表达稳定的内参基因对目标基因表达量进行校正和标准化[6]。然而,内参基因在不同试验要求下表达可能出现差异[7-8],因此根据不同物种和不同试验要求筛选能稳定表达的内参基因对基因定量分析具有重要的意义。
肌动蛋白基因(actin,ACT)、18S核糖体RNA基因(18S ribosomal RNA,18SrRNA)、真核生物延伸因子(elongation factor 1 alpha,EF-1α)、甘油醛-3-磷酸-脱氢酶基因(glyceraldehyde-3-phosphate dehydrogenase,GAPDH)、α-微管蛋白基因(α-tubulin,TUA)和β-微管蛋白基因(β-tubulin,TUB)等看家基因常被作为内参基因[9-11]。目前,国内外关于黄秋葵内参基因的筛选及表达分析鲜有报道,仅王旭等[12]发现,研究黄秋葵查尔酮合成酶基因(chalcone synthase,AeCHS)表达时应选用陆地棉β-actin(FJ560483.1)为内参基因。因此,本研究根据黄秋葵RNA-seq数据库,筛选并验证获得18SrRNA、ACT、EF-1α、TUA、TUB、GAPDH等基因开放阅读框(open reading frame,ORF)序列;以绿白1号的不同组织、不同发育时期果实、叶片及不同胁迫条件下的叶片为材料,利用RT-qPCR测定基因表达量,结合GeNorm、NormFinder和BestKeeper软件评价6个内参基因表达的稳定性,旨在筛选适合的黄秋葵内参基因,为黄秋葵相关基因表达研究奠定基础。
1 材料与方法
1.1 试验材料
1.1.1 植物材料 黄秋葵,为福建省农业科学院作物研究所选育的绿白1号,种植于福建省农业科学院作物研究所蔬菜科研基地。待幼苗长至三叶一心时移入培养箱进行胁迫处理。
胁迫处理试验分为3组,分别为低温(A组)、高温(B组)和干旱处理组(C组);正常栽培条件下,分别采集盛果期不同组织(D组)、叶不同发育时期(E组)和果实不同发育时期(F组)的样品,分析胁迫处理组和正常栽培条件下黄秋葵18SrRNA、TUB、ACT、GAPDH、EF-1α和TUA基因的表达情况。采样后液氮冷冻,-80℃保存。试验处理及样品见表1。
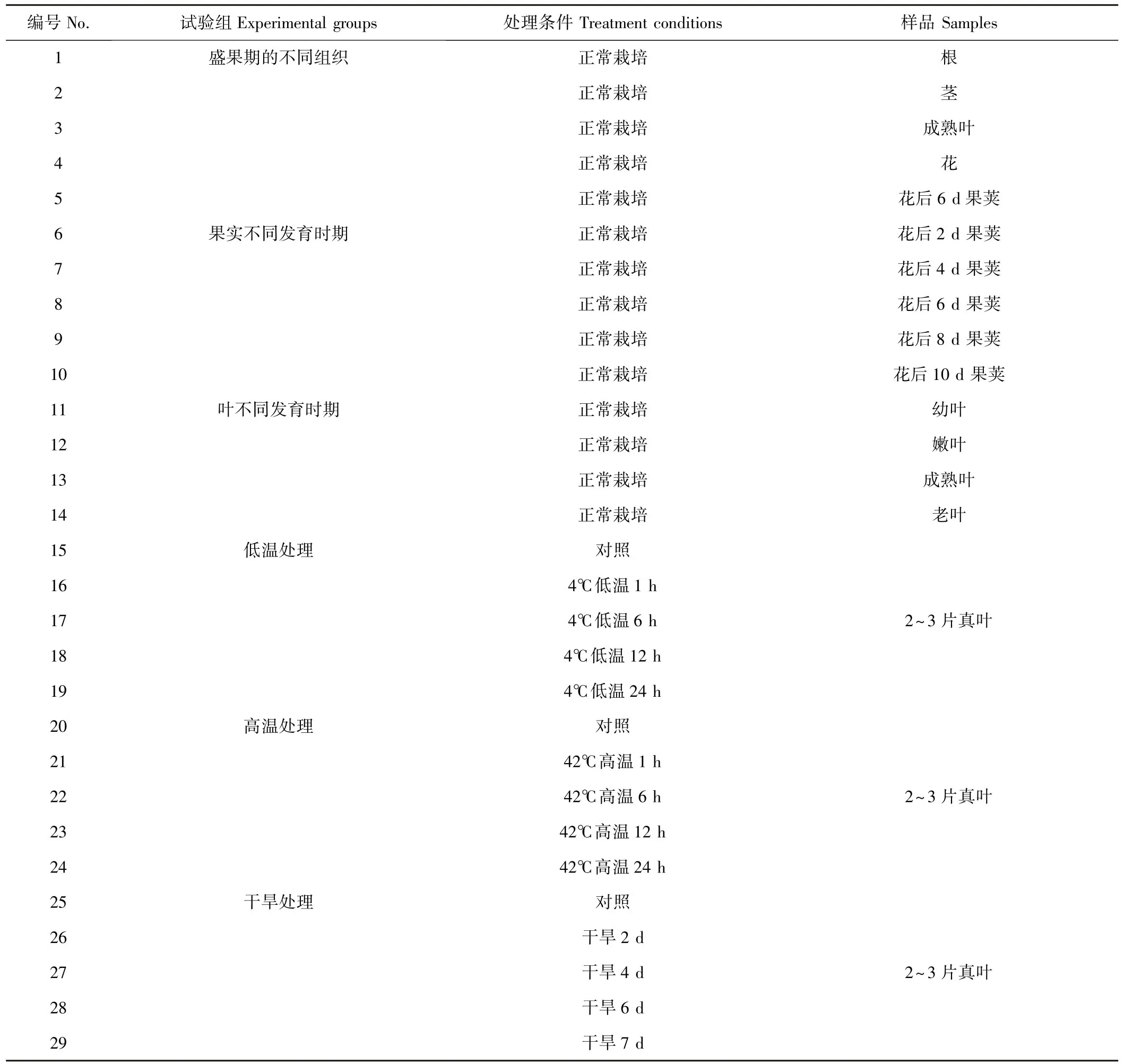
表1 黄秋葵试验处理样品Table1 Test sample and treatments of Hibiscus esculentus L.
1.1.2 主要试剂与数据分析来源 通用植物总RNA提取试剂盒购自北京百泰克生物技术有限公司;RNA反转录试剂盒(PrimeScriptTM 1st Strand cDNA)、Taq DNA Polymerase、dNTPs、pMD18-T simple,购自宝生物工程(大连)有限公司;胶回收试剂盒、质粒提取试剂盒购自瑞士Omega公司;引物合成和测序由尚亚生物技术(福州)有限公司完成。
黄秋葵RNA-seq数据库由广州基迪奥生物科技有限公司采用Illumina HiSeq TM 2500 PE125系统对福建省农业科学院作物研究所黄秋葵种质资源库中的绿色黄秋葵资源(全株及嫩果均为绿色,果实六棱至八棱,以六棱为主)进行转录组测序获得。
1.2 方法
1.2.1 引物设计 对黄秋葵RNA-seq所得数据进行过滤、拼接和组装,并在NCBI中进行功能注释(E值<10-5)[13-14]。根据黄秋葵RNA-seq数据库的功能注释,在NCBI上比对含有完整ORF,且与其他作物的同源性高,筛选得到18SrRNA、ACT、EF-1α、TUA、TUB和GAPDH基因全长序列,应用Primer Express 3.0软件分别设计其基因ORF扩增引物、荧光定量特异引物(表2)。

表2 6个内参基因ORF扩增及定量表达引物序列Table2 ORF amplification and quantitative expression primer sequence of six internal reference genes
1.2.2 黄秋葵基因的ORF扩增 应用反转录试剂盒将黄秋葵各处理材料的总RNA反转录形成cDNA第一链。PCR反应体系(25μL):50 ng·μL-1cDNA 2μL、正反引物(10μmol·L-1)各1μL、10 mmol·L-1dNTP 0.5μL、5 U Taq酶0.3μL、1.5mmol·L-1MgCl2的10×PCR缓冲液2.5μL,超纯水补足至25μL。PCR反应程序见表3。PCR扩增产物采用0.8%琼脂糖电泳检测,回收目的片段,转化并测序。
1.2.3 常规PCR检测定量引物 以反转录的黄秋葵cDNA为模板,用1.2.1中设计的荧光定量PCR引物进行常规PCR检测。PCR反应体系同1.2.2,PCR反应程序见表3第2个程序。PCR扩增产物采用1.8%琼脂糖电泳检测。
1.2.4 实时荧光定量PCR检测定量引物 定量检测使用SYBR®Premix Ex TaqTM荧光定量PCR试剂盒(TakaRa公司,大连)。反应体系(20μL):10μL Power SYBR®Green PCR Master Mix、2μL cDNA、正、反向引物各0.5μL(浓度为10μmol·L-1),用蒸馏水补足至20 μL。3个重复,反应程序见表3第3个程序。
1.2.5 基因表达稳定性分析 利用荧光定量PCR分析基因表达稳定性。以陆地棉β-actin(FJ560483.1)为内参基因,将试验的29个黄秋葵样品cDNA采用SYBR Green染料法进行实时荧光定量PCR分析,反应体系同1.2.4,反应程序见表3第4个程序。每个反应设3个重复,采用2-ΔΔCt法计算目的基因的相对表达情况。
1.3 数据分析
采用GeNorm、NormFinder和BestKeeper软件分析6个内参基因表达的稳定性。GeNorm软件中,内参基因平均表达稳定值(M)越小稳定性越高;ⅤN/ⅤN+1值确定最适内参基因数量,若ⅤN/ⅤN+1值<0.15,则最适内参基因的数量是N个,当ⅤN/ⅤN+1值>0.15时,最适内参基因的数量为N+1个[15]。NormFinder软件对基因稳定性评定是根据基因表达稳定值(S),S值越小基因稳定性越好[16]。BestKeeper软件中,标准偏差(standard deviation,SD)>1时,该内参基因表达不稳定;SD<1时,变异系数(coefficient of variation,CV)越小,内参基因稳定性越高[17]。
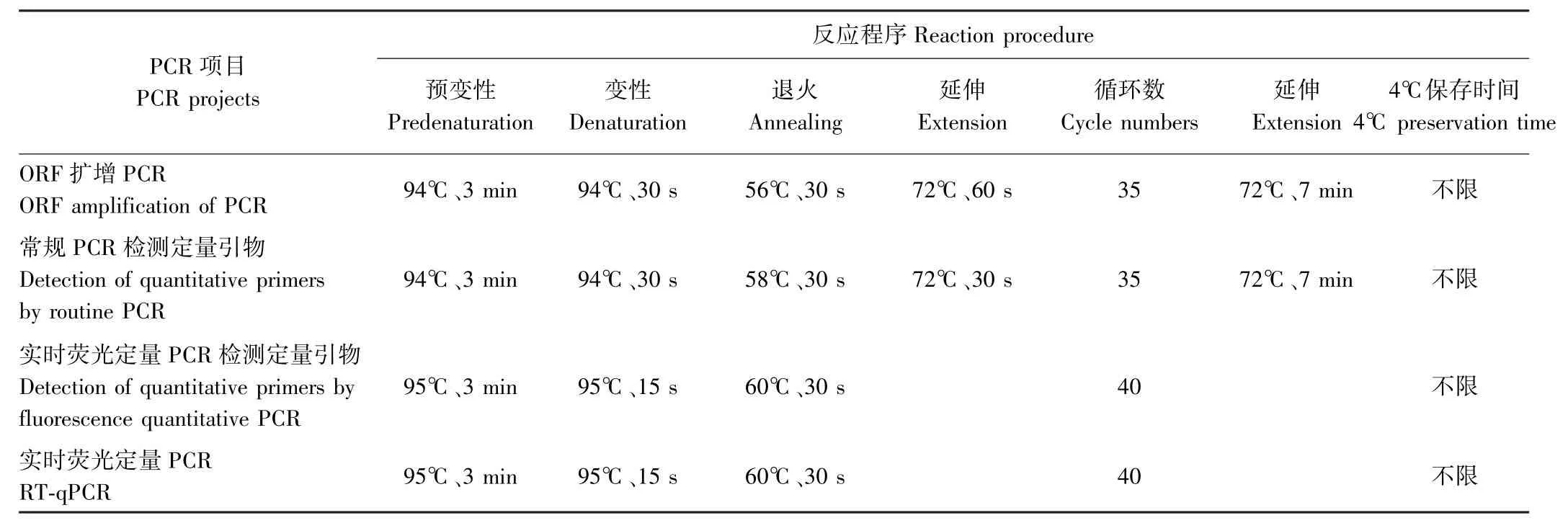
表3 PCR反应程序Table3 PCR reaction procedure
2 结果与分析
2.1 黄秋葵内参基因扩增结果
从黄秋葵果实转录组测序的RNA-seq数据库中筛选得到ACT、TUB、18SrRNA、TUA、EF-1α、GAPDH基因全长序列,设计各基因ORF引物进行验证(图1)。经回收、连结、转化、测序,结果表明,ACT、TUB、18SrRNA、TUA、EF-1α、GAPDH基因的ORF大小分别为1 134、1 338、1 464、1 353、1 407、1 455 bp。
2.2 基因引物设计及常规PCR检测
依据ACT、TUB、18SrRNA、TUA、EF-1α和GAPDH基因ORF序列,各基因设计出一对荧光定量特异引物,用PCR检测引物特异性,结果表明(图2),6个内参基因扩增产出大小与预期相符的单一条带,表明所设计引物无二聚体、特异性强,可用于RT-qPCR研究。
2.3 实时荧光定量PCR检测引物
以黄秋葵嫩叶cDNA为模板,6对引物RT-qPCR扩增结果显示,3次重复的RT-qPCR扩增曲线较一致(图3)。各基因的融解曲线(图4)显示3次重复的平均溶解温度(melting temperature,Tm)基本一致,都只有一个特异峰,无引物二聚体,无非特异性扩增,表明试验设计的引物特异性优、重复性好,可作为黄秋葵RT-qPCR的引物。
2.4 基因表达稳定性分析
由图5可知,各处理组黄秋葵样品中18SrRNA、ACT、EF-1α、TUA基因的相对表达量无显著差异,而在高温处理下GAPDH基因的相对表达量有差异,在黄秋葵各组织、果荚发育不同时期、低温处理、干旱处理下TUB基因的相对表达量均差异较大,表明18SrRNA、ACT、EF-1α、TUA基因在黄秋葵不同组织、果荚发育、叶发育,以及各种胁迫下均能较稳定地表达。
2.5 内参基因CT值分析
6个基因CT(cycle threshold)值在14.02~32.12之间,表达丰度分布范围广,其中18SrRNA的低于其他内参基因CT值,且数值集中;EF-1α、ACT、GAPDH、TUA的CT值在16.20~23.83之间,表达丰度居中,除GAPDH外数值较集中;TUB的CT值最大且变化较大,表达量最低(图6)。从各基因在不同处理下CT值分布来看,在黄秋葵不同组织中EF-1α、18SrRNA、ACT的CT值比较稳定,TUA、GAPDH的CT值略有变化,TUB的CT值变化较大(图7-A);果荚发育过程中除TUB的CT值有较大变化外,其他5个基因的CT值都相对稳定(图7-B);叶发育过程中,6个基因的CT值都相对稳定(图7-C);低温处理和干旱处理时GAPDH、TUB的CT值变化较大(图7-D、F);干旱处理时GAPDH的CT值变化较大(图7-E)。由此可初步判断,18SrRNA、EF-1α、ACT和TUA表达较为稳定,GAPDH和TUB表达差异较大。
2.6 黄秋葵内参基因的筛选
GeNorm软件的配对差异值(ⅤN/ⅤN+1)用于确认所需内参基因的最适数目,以降低单一内参基因引起的偏差和波动。由图8可知,在盛果期不同组织、果实不同发育时期、叶不同发育时期、低温胁迫和高温胁迫、干旱胁迫下,均有Ⅴ2/Ⅴ3值小于0.15,因此,各试验组的最适内参基因数均为2个。
2.6.1 盛果期不同组织、果荚发育、叶发育时期黄秋葵内参基因表达的筛选 由表4可知,经GeNorm程序分析,在黄秋葵盛果期各组织中6个内参基因表达稳定值(M)排序为EF-1α=18SrRNA<TUA<ACT<GAPDH<TUB;果荚不同发育时期M值排序为EF-1α=ACT<TUA<GAPDH<TUB;叶不同发育时期M值排序为18SrRNA=ACT<EF-1α<GAPDH<TUA<TUB。经NormFinder软件分析,黄秋葵盛果期不同组织中6个内参基因S值排序为18SrRNA<EF-1α<ACT<TUA<GAPDH<TUB;果荚不同发育时期6个内参基因S值排序为EF-1α<ACT<18SrRNA<TUA<GAPDH<TUB;叶不同发育时期6个内参基因稳定值(S)排序为EF-1α<18SrRNA=ACT<GAPDH<TUA<TUB。经BestKeeper软件分析,黄秋葵盛果期不同组织中表达稳定性18SrRNA>EF-1α>ACT>TUA>GAPDH>TUB,其中GAPDH和TUB稳定性差,不可用;果荚不同发育时期EF-1α>ACT>18SrRNA>TUA>GAPDH>TUB,其中TUB稳定性差,不可用;叶不同发育时期18SrRNA>ACT>EF-1α>GAPDH>TUA>TUB。
2.6.2 不同胁迫处理下黄秋葵内参基因的筛选 由表5可知,经GeNorm程序分析,黄秋葵低温胁迫不同时段的M值排序为EF-1α=18SrRNA<ACT<TUA<GAPDH<TUB;高温胁迫不同时段M值排序为EF-1α=TUA<18SrRNA<ACT<TUB<GAPDH;干旱胁迫不同时段M值排序为EF-1α=18SrRNA<TUA<ACT<GAPDH<TUB。根据NormFinder软件分析结果可知,在4℃低温胁迫条件下6个内参基因S值排序为ACT<18SrRNA<EF-1α<TUA<GAPDH<TUB;42℃高温胁迫条件下6个内参基因S值排序为EF-1α<TUA=18SrRNA<ACT<TUB<GAPDH;干旱胁迫条件下6个内参基因S值排序为18SrRNA<EF-1α<TUA<ACT<GAPDH<TUB。BestKeeper软件直接利用CT值进行数据分析,结果表明,在低温胁迫条件下稳定性ACT>18SrRNA>EF-1α>TUA>GAPDH>TUB,TUB稳定性差,不可用;高温胁迫条件下稳定性EF-1α>TUA>18SrRNA>ACT>TUB>GAPDH,GAPDH稳定性差;干旱胁迫条件下稳定性18SrRNA>EF-1α>TUA>ACT>GAPDH>TUB,TUB稳定性较差。
采用GeNorm、NormFinder和BestKeeper软件分析全部29个黄秋葵样品,结果显示,18SrRNA、EF-1α、ACT均排名前三,且数值较低,适合作为黄秋葵荧光定量分析的内参基因;TUA居第四名,稳定性尚好,亦可选用;GAPDH和TUB居末位,稳定性较差,不宜选用。
3 讨论
目前,RT-qPCR是基因表达分析主要工具,是明确植物基因功能和调控机制的重要途径。然而,样品自身差异、RNA质量等会导致分析结果出现误差。为提高结果可靠性,引入内参基因校正表达数据。内参基因稳定性直接严重影响RT-qPCR分析结果准确度[18]。研究表明,内参基因在不同胁迫和不同组织表达会出现变化[19-20]。因此,应根据试验要求选择最适宜的内参基因或组合。本研究利用GeNorm、NormFinder和BestKeeper软件分析黄秋葵6个看家内参基因在低温、高温、干旱胁迫处理不同时间和正常处理盛果期不同组织、果实不同发育时期、叶不同发育时期表达的稳定性结果不同处理和发育时期表达最稳定的基因不尽相同。
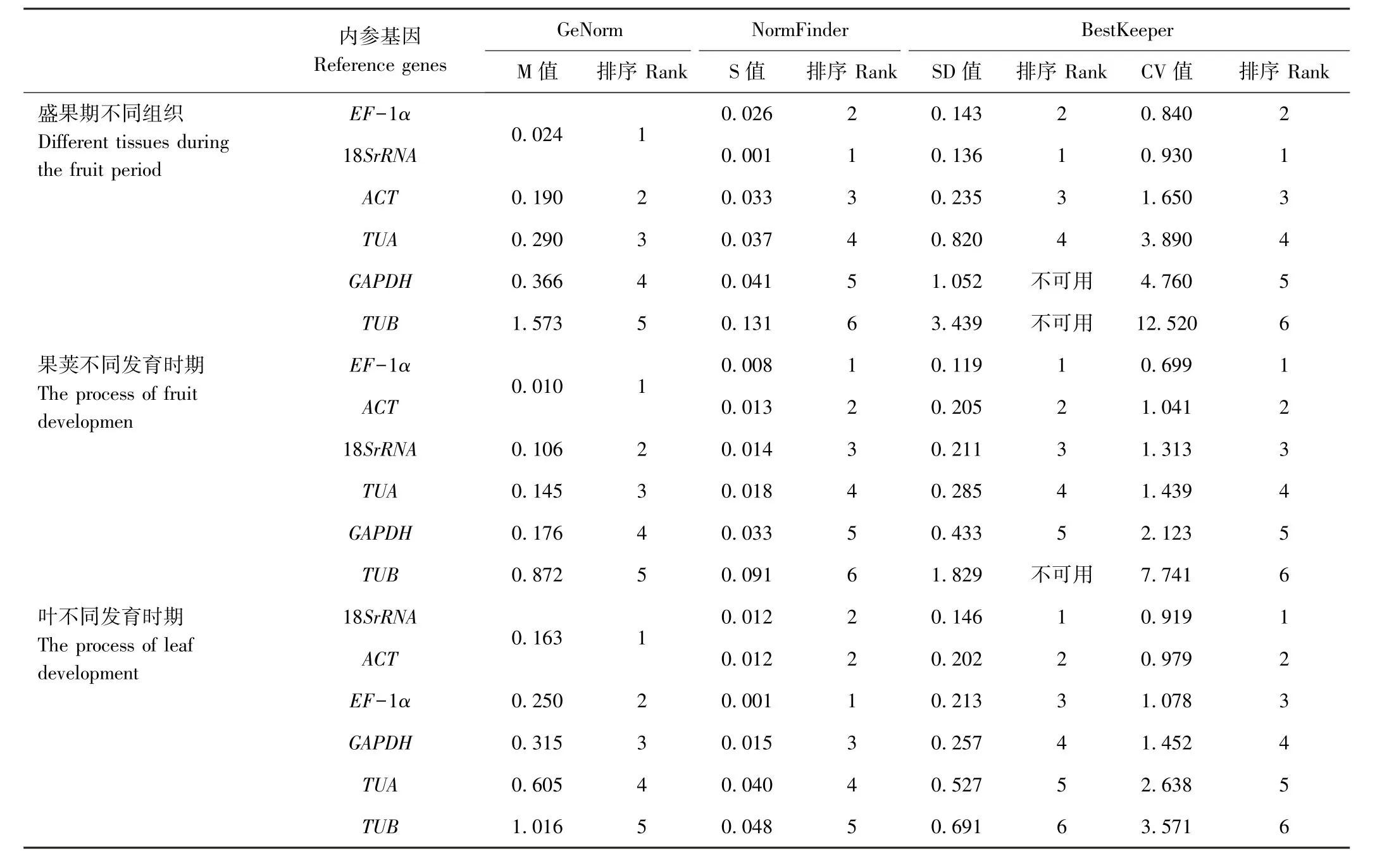
表4 不同软件分析6个内参基因在黄秋葵盛果期不同组织、果荚不同发育时期、叶不同发育时期表达稳定性Table4 Expression stability of the 6 reference genes under different different tissues during the fruit period,fruit developmen and leaf development conditions by software analysis
前人对非生物胁迫条件下植物中内参基因已有研究报道。叶新如等[21]研究表明,冬瓜(Benincasa hispidaCogn.)高温、干旱胁迫的内参基因是TUA,而EF-1α适合于低温胁迫;肖翠等[22]研究发现柑橘(CitrusL.)在干旱胁迫下GAPDH、18SrRNA表达最稳定,而在低温胁迫下ACT、18SrRNA表达稳定;Lovdal等[23]研究表明,番茄(LycopersiconesculentumMill.)在低温胁迫下EF-1α和RPLA表达稳定,GAPDH稳定性最差;庞强强等[24]发现茄子(SolanummelongenaL.)在高温胁迫下表达稳定性最好的是SmEF1a;杜辉辉[25]发现Ha18SrRNA、HaTUA5及HaUBQ10是梭梭(Haloxylonammodendron)干旱胁迫下最适内参基因;EF-1α是茅苍术(RhizomaAreactylodisLanceae)在干旱胁迫下最佳内参基因[26]。本研究结果表明,黄秋葵在高温胁迫下EF-1α表达稳定性最好,这与庞强强等[24]在茄子上的研究结果一致;干旱胁迫下黄秋葵18SrRNA表现最为稳定,杜辉辉[25]在梭梭上也得到此结果;低温胁迫下黄秋葵ACT表达稳定与柑橘[22]、花椰菜(BrassicaoleraceaL.)[27]的研究结果一致。

表5 不同软件分析不同胁迫条件下黄秋葵6个内参基因的表达稳定性Table5 Expression stability of the 6 reference genes under different stress conditions by software analysis
本研究中,黄秋葵根、茎、叶等不同组织最适内参基因为EF-1α和18SrRNA,与铁皮石斛(Dendrobiumofficinale)的相关研究一致[28],冬瓜不同组织中EF-1α表达稳定性最好[21],核桃(Juglans regia)EF-1α和GAPDH在不同组织和发育时期中表达最稳定[29];在印度南瓜不同组织中EF-1α表达稳定[30]。EF-1α在黄秋葵果实不同发育时期的表达最稳定,而猕猴桃果实不同发育时期18SrRNA表达最稳定[31],这可能与试验材料的物种、内参基因筛选范围不同有关。
内参基因稳定性分析与筛选,是得到准确基因表达分析结果的必要前提。基因稳定性分析常用GeNorm、NormFinder和BestKeeper软件。GeNorm原则是2个稳定内参基因的表达水平在试验样品中一致,根据ⅤN/ⅤN+1值确定最适内参基因个数,并选出最优组合,以校正系统偏差,从而获得更可靠的表达分析结果,对于研究基因表达的细微变化具有极其重要的意义[32]。NormFinder可分析内参基因表达的稳定性,还能比较目标基因的表达水平。本试验利用GeNorm、NormFinder和BestKeeper软件分析黄秋葵6个内参基因的稳定性及表达水平,分析结果略有不同,这种不一致性可能是由于计算机软件的统计学算法不同及试验数据的误差引起的。
4 结论
本研究克隆获得黄秋葵18SrRNA、ACT、EF-1α、TUA、TUB、GAPDH基因全长序列,分析了这6个基因在黄秋葵盛果期不同组织、果实不同发育时期、叶不同发育时期,以及不同胁迫下的表达水平,结合GeNorm、NormFinder和BestKeeper软件评价其表达稳定性,结果表明,各处理中18SrRNA、EF-1α、ACT表达均较为稳定,GAPDH表达稳定性较差,TUB的表达稳定性最低。本研究结果对于黄秋葵的基因表达等相关分子生物学研究具有借鉴意义。
